Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de la Facultad de Ciencias Veterinarias
versión impresa ISSN 0258-6576
Rev. Fac. Cienc. Vet. vol.56 no.2 Maracay jul. 2015
Estandarización de la técnica de PCR para la detección de ADN de Leishmanía sp. en muestras de sangre de caninos
Jesús Reyes*, Mercedes Viettri*, Angeline Rivas*, María Lares*, Leidi Herrera**, Cruz M. Aguilar*** y Elizabeth Ferrer*,****, 1
*Instituto de Investigaciones Biomédicas (BIOMED) Dr. Francisco J. Triana Alonso, Facultad de Ciencias de la Salud, Universidad de Carabobo Sede Aragua, Maracay, Venezuela.
** Instituto de Zoología y Ecología Tropical (IZET), Facultad de Ciencias, Universidad Central de Venezuela (UCV), Caracas, Venezuela.
***Centro de Investigaciones en Enfermedades Tropicales (CIET-UC), Facultad de Ciencias de la Salud, Universidad de Carabobo, san Carlos, Cojedes, Venezuela.
****Departamento de Parasitología, Facultad de Ciencias de la Salud, Universidad de Carabobo, Sede Aragua, Maracay, Venezuela. Correo-E: elizabeth.ferrer@gmail.com
RESUMEN
(Palabras clave: Leishmaniasis; perro; diagnóstico; PCR)
Leishmaniasis is a disease with clinical and epidemiological diversity, caused by several species of protozoan parasites of the genus Leishmania. These parasites infect a wide variety of mammalian hosts, and are transmitted by insects of the genus Lutzomyia in our country. Canines have been implicated as potential reservoirs of the parasite. For the diagnosis of leishmaniasis, parasitological techniques generally with low sensitivity and immunological techniques, with poor specificity, are used. Because of limitations in the diagnosis, and the difficulty to obtain, transport, and store samples, the aim of this research was to standardize a Leishmania nested PCR test (Ln-PCR), for the detection of Leishmania sp. DNA in blood samples from dogs collected in filter paper. For that purpose, concentrations of the PCR reagent for DNA parasite amplification were titrated and the analytical sensitivity and specificity of the technique were determined. A total of 36 canine blood samples (6 infected and 30 uninfected) were evaluated. The optimal reaction conditions were: 0.2 mM dNTPs; 0.4 µM of each primer; and 1 U of Taq polymerase. The analytical sensitivity of the Ln-PCR was 10 fg and the specificity was 100% in detecting Leishmania sp. DNA, since no amplification was observed with DNA from other parasites, human DNA or canine DNA. The six samples from infected canines evaluated amplified Leishmania sp. DNA by PCR, whereas in none of the 30 samples from uninfected canines the amplification was observed. The DNA extraction from blood samples collected in filter paper was efficient for the PCR amplification, a technique that can be very useful for the diagnosis of leishmaniasis in animals and their involvement as potential reservoirs.
Recibido: 04/03/15 - Aprobado: 11/12/15
La leishmaniasis es una enfermedad que presenta diversas formas clínicas y características epidemiológicas. Es producida por parásitos del género Leishmania y transmitidos por insectos dípteros hematófagos del género Lutzomyia en el nuevo mundo [1]. La infección en el vector ocurre cuando se alimenta de sangre de hospedadores mamíferos parasitados, cuyos macrófagos contienen las formas amastigotas de Leishmania sp. Clínicamente, se han descrito las formas cutánea, mucocutánea y visceral de la enfermedad. La forma cutánea se caracteriza por lesiones dérmicas, en la forma mucocutánea los parásitos se diseminan hacia las mucosas, mientras que en la forma visceral se distribuyen en órganos internos como bazo, hígado y médula ósea. Los caninos han sido implicados como reservorios del parásito, pudiendo ser asintomáticos o sufrir la enfermedad, dependiendo de la respuesta inmunológica del animal [2, 3].
A nivel mundial se estima que existen 12 millones de infectados. Anualmente ocurren 1,3 millones de casos y 30.000 muertes [3, 4]. En Venezuela, las formas cutáneas de leishmaniasis tienen una tasa anual promedio de 10/100.000 habitantes y la de leishmaniasis visceral era de 0,22/100.000 habitantes durante el 2005 [5]. Es importante resaltar que no se cuenta con cifras de prevalencia actualizadas en el país. Sin embargo, por algunos boletines epidemiológicos se conoce que para 2013 se reportaron 112 nuevos casos por el Instituto de Biomedicina y en 2014 se reportaron en el país 808 casos totales [6, 7]. en caninos, la prevalencia no se conoce con certeza, en algunos estudios se han reportado seroprevalencias alrededor del 20% en ciertas zonas endémicas [8, 9].
El diagnóstico de la enfermedad se basa en la aplicación de métodos parasitológicos e inmunológicos, variando en función del cuadro clínico [2]. Los métodos parasitológicos permiten la observación del parásito en el tejido infectado, mediante biopsias del borde de las lesiones en los casos de leishmaniasis cutánea y mucosa, o punciones de bazo o médula ósea, en el caso de leishmaniasis visceral. Son métodos invasivos y generalmente presentan baja sensibilidad en cargas parasitarias escasas. Otros, como cultivos o inoculación en animales de laboratorio, son laboriosos y la sensibilidad dependerá de la carga parasitaria y de la cepa del parásito [2,10]. El diagnóstico inmunológico permite detectar anticuerpos anti-Leishmania sp. mediante inmunoensayo enzimático (ELISA), hemaglutinación indirecta (HAI) o inmunofluorescencia indirecta (IFI). Son técnicas sencillas, pero de sensibilidad y especificidad limitadas por reacciones cruzadas con otros parásitos como Trypanosoma cruzi y T. rangeli, dificultando el diagnóstico en las zonas coendémicas [10,11].
Se han desarrollado varios protocolos de reacción en cadena de la polimerasa (PCR, Polymerase Chain Reaction, por sus siglas en inglés), amplificando diferentes secuencias de ADN del parásito para el diagnóstico de leishmaniasis, de forma sensible, específica, sencilla y rápida [12-18]. En Venezuela, algunos estudios han permitido la detección molecular mediante PCR para diagnóstico de leishmaniasis en humanos y algunos reservorios animales [19-21]. En el 2002, Cruz et al. [16] diseñaron una PCR anidada (Ln-PCR, Leishmania nested Polymerase Chain Reaction) para la amplificación del ADN ribosomal del parásito, la cual permitió la detección de ADN de Leishmania sp. en muestras de sangre y médula ósea.
Este trabajo tuvo como objetivo, estandarizar la técnica de Ln-PCR, así como el análisis de su sensibilidad analítica y la especificidad de la misma, para la amplificación de ADN de Leishmania sp. y su posterior uso en muestras de sangre de caninos.
MATERIALES Y MÉTODOS
Muestras Biológicas
Para la estandarización y evaluación de la sensibilidad de la PCR, y como controles, se emplearon aislados de parásitos del género Leishmania en cultivo, de las siguientes especies; Leishmania amazonensis PH8 (IFLA/BR/1967/PH8), L. braziliensis M2903, (MHOM/BR/1975/M2903), L. mexicana M7326, (MHOM/BZ/1982/BEL21), y L. chagasi (IOC/L579). Para la determinación de la especificidad de la PCR se utilizaron aislados de parásitos relacionados, tales como: Trypanosoma cruzi aislado Dm28c [22], y otros como Trypanosoma rangeli aislado DOG-82 [23] y Toxoplasma gondii, así como también ADN canino y ADN humano.
Se utilizaron además, 36 muestras de sangre de caninos, 6 de perros infectados, positivos serológicamente (presencia de anticuerpos anti-Leishmania sp.) y asintomáticos y 30 de perros no infectados (ausencia de anticuerpos anti-Leishmania sp.) y asintomáticos, todos procedentes de zonas endémicas de leishmaniasis del estado Cojedes. Las muestras fueron colectadas en papel de filtro y tomadas previo consentimiento informado de sus dueños.
Los aislados parasitarios de Trypanosoma y Leishmania mencionados anteriormente, se cultivaron in vitro y se expandieron para su masificación en medio LIT (Liver-Infusion-Tryptose) con suero fetal bovino al 10%. En la fase de crecimiento logarítmico, se recolectaron los parásitos para la posterior extracción de ADN, utilizando la resina Chelex®-100 (BioRad).
Para la extracción de ADN a partir de los aislados parasitarios en cultivo se tomó 200 µL de los cultivos y se les añadió 800 µL de agua destilada estéril, incubándose 30 min a temperatura ambiente. Posteriormente, se centrifugó durante 3 min a 16.000 g y se retiró el sobrenadante obtenido dejándose un volumen de 50 mL, al cual se le agregó 200 µL de la resina Chelex®-100 al 5%, incubándose durante 30 min a 56 ºC. Luego, se agitó en vortex por 10 seg, y se incubó a 100ºC durante 10 min para más tarde agitarse durante10 seg nuevamente. La resina y las proteínas desnaturalizadas fueron separadas mediante centrifugación por 3 min a 16.000 g y se recogió el sobrenadante que contenía el ADN. Para la extracción de ADN, a partir de las muestras de sangre colectadas en papel de filtro, se tomó con una perforadora los círculos impregnados, de 6 mm de diámetro, que corresponde aproximadamente a 50 µL de sangre. Se colocaron los círculos de papel de filtro en un microtubo y se les añadió 1 mL de agua destilada estéril, incubándose por 30 min a temperatura ambiente y se continuó con la técnica como fue descrita anteriormente.
Determinación de la Concentración y Pureza del ADN
La concentración y pureza del ADN extraído se determinó por los métodos descritos por Sambrook y Russell [24]. La concentración de ADN fue estimada en un espectrofotómetro UV/Visible BioPhotometer Plus de Eppendorf, midiendo la absorbancia a 260 nm (A260) y 280 nm (A280), utilizando la fórmula siguiente: [ADN]= A260 nm x factor de dilución x 50 mg/ml. El grado de pureza se calculó dividiendo la absorbancia a 260 nm entre la absorbancia a 280 nm. Valores óptimos de pureza se encuentran entre 1,7- 1,9.
Electroforesis de ADN en Geles de Agarosa
La calidad e integridad del ADN extraído se analizó por electroforesis en gel de agarosa (D-1 LE GQT, Scientific Trade Corp, Miami, USA) al 1%, utilizando TAE (tampón tris ácido acético 40 mM, EDTA 0,5 M pH 8,0). La separación del ADN se realizó en una cámara de electroforesis horizontal (Minicell® EC 370M), se empleó un voltaje constante de 60-100 V, dependiendo del tamaño del gel. Las bandas de ADN se visualizaron mediante el sistema Gel Doc 1000 de BioRad®. El tamaño de las bandas de ADN se estimó comparándolas con el marcador de tamaño molecular l-BstEII (Promega) con un rango de tamaño de 117 pb a 8.454 pb.
Estandarización de la Ln-PCR para la Amplificación del ADN de Leishmania sp.
Con el fin de obtener fragmentos correspondientes al ADN ribosomal de Leishmania sp., se optimizaron las condiciones de la reacción en un volumen final de 50 µL mediante la variación de la concentración de determinados componentes. Específicamente, los cebadores se evaluaron a concentraciones de 0,2 mM y 0,4 mM, los desoxinucleótidos (dNTPs, 2-desoxynucleoside 5-triphosphate) a 0,1 mM y 0,2 mM y la enzima Taq ADN polimerasa desde 0,25 U hasta 1 U (0,25 U, 0,5 U y 1 U).
La PCR se realizó siguiendo la metodología descrita por Cruz et al. [16]. En la primera PCR para la detección de una banda de 603 pb correspondiente al ADN codificante del ARN de la subunidad pequeña ribosomal, se utilizaron los cebadores [R221 (cebador directo): 5- GGT TCC TTT CCT GAT TTA CG - 3/R332 (cebador reverso): 5- GGC CGG TAA AGG CCG AAT AG - 3] y las siguients condiciones de tiempo y temperatura: desnaturalización inicial a 95ºC por 60 seg, y 35 ciclos de los procesos de desnaturalización (94ºC, 30), hibridación (60ºC, 30) y extensión (72ºC, 30). Adicionalmente, se realizó una extensión final a 72ºC por 10 min. La segunda PCR se realizó para amplificar un producto entre 200 y 600 pb (dependiendo de la especie involucrada), utilizando cebadores internos [R223 (cebador directo): 5- TCC CAT CGC AAC CTC GGT T - 3´/ R333 (cebador reverso): 5- AAA GCG GGC GCG GTG CTG – 3] [12,16]. Las condiciones de tiempo y temperatura fueron: desnaturalización inicial a 95ºC por 60 seg y 35 ciclos de los procesos de desnaturalización (94ºC, 30), hibridación (65ºC, 30) y extensión (72ºC, 30). Adicionalmente, se realizó una extensión final a 72ºC por 10 min. El resultado de la migración electroforética en los geles de agarosa al 2% se observó en un equipo de fotodocumentación de geles Gel Doc 1000 de BioRad®.
Sensibilidad y Especificidad de la Técnica de PCR
La sensibilidad de la técnica se determinó a partir de ADN extraído de parásitos en cultivo, utilizando las siguientes cantidades de ADN molde: 1 mg, 100 ng, 10 ng, 1 ng, 100 pg, 10 pg, 1 pg, 100 fg, 10 fg y 1 fg. Esto permitió determinar la mínima cantidad de ADN capaz de producir amplificación. La especificidad se determinó utilizando ADN molde de otros parásitos relacionados; Trypanosoma cruzi y T. rangeli, el parásito apicomplexa Toxoplasma gondii, así como ADN humano y ADN canino.
En la técnica Ln-PCR se probaron condiciones cercanas a las concentraciones descritas [16], (Cuadro 1).
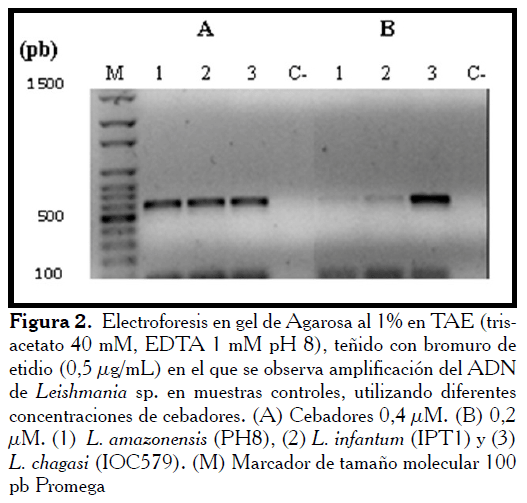
En el caso de los dNTPs, se observó a una concentración de 0,2 mM la presencia de bandas nítidas e íntegras para todas las cepas [L. amazonensis (PH8), L. infantum (IPT1) y L. chagasi (IOC579)] (Figura 1 panel A); mientras que cuando se utilizó una concentración menor (0,1 mM) solamente amplificaron L. infantum (IPT1) y L. chagasi (IOC579), destacando la banda de esta última (Figura 1 panel B). Ha sido reportado por algunos autores que existen diferencias en la cantidad de ADN de las diferentes especies de Leishmania, afectan la sensibilidad de la técnica de PCR, por lo que pudiese tener menor número de copias de alguna diana para determinada especie [17-18] y en este caso la concentración óptima de algunos reactivos podría ser determinante para algunas especies en particular. Estos resultados permitieron observar cómo la concentración de 0,2 mM era óptima para amplificar la región de ADN, previamente mencionada, de las tres cepas analizadas, tal como lo describiera previamente Cruz et al. [16] y otros autores [14,17]. Con relación a los cebadores, resultó de especial interés observar como la concentración de 0,4 µM permitió la amplificación en todos los casos (Figura 2 panel A), mientras que a 0,2 µM, a pesar de que se observaron bandas correspondientes al tamaño esperado (Figura 2, panel B), no resulta ser una concentración ideal ya que ante cargas parasitarias bajas, se corre el riesgo de generar un resultado falso negativo. Esta concentración fue menor que la empleada en el protocolo original de Cruz et al. [16] en el que se empleó 0,6 µM y en otros protocolos de otros autores, en los cuales se llega a emplear hasta 1 µM [17]. En cuanto a la enzima Taq polimerasa, se observa que todas las especies utilizadas en el ensayo mostraron amplificación con 1 U de la enzima (Figura 3 panel A), utilizando 0,5 U las bandas correspondientes a L. amazonensis (PH8) y L. infantum (IPT1) aparecen débiles (figura 3 panel B), y con 0,25 U (figura 3 panel C), no se observa amplificación en ninguno de los casos. En este caso podemos utilizar una concentración menor a la reportada por Cruz et al. [16], quienes emplearon 1,4 U y que la empleada por la mayoría de los autores, los cuales llegan a utilizar hasta 4 U por reacción [12]. Este proceso de estandarización permitió la disminución de las concentraciones de los cebadores y Taq polimerasa, obteniendo bandas diagnósticas definidas y disminuyendo los costos de la técnica.
Se obtuvo que la mínima cantidad de ADN de Leishmania, que amplifica mediante la técnica de Ln-PCR fue de 10 fg, una sensibilidad un poco menor a la reportada por Cruz et al. [16], la cual fue de 1 fg, pero igual o superior a la reportada en otros protocolos de la PCR [12, 14]. La sensibilidad varía según los distintos protocolos de la PCR empleados, tal como demuestra Lachaud et al. [17], en un estudio en el cual se compararon seis métodos de PCR usando sangre periférica para la detección de leishmaniasis visceral en caninos, observándose que todos los métodos detectaron ADN equivalente a menos de un parásito por tubo de reacción, sin embargo, la sensibilidad varió entre ellos. Sólo dos técnicas de PCR de ADN Kinetoplasto (K13AK13B y RV1-RV2) pudieron detectar el ADN del parásito en el 100% de los perros infectados sintomáticos, a diferencia de los métodos basados en ADN genómico, los cuales detectaron un 62%. Es importante destacar que la sensibilidad puede depender del tipo y cantidad de ADN de cepas de Leishmania que pudiesen estar infectando un determinado canino y de las técnicas de extracción de ADN.
Después de determinar la cantidad mínima de ADN que puede ser amplificado, se evalúo la especificidad de la técnica de Ln-PCR, la cual fue 100% específica con las muestras utilizadas, ya que los cebadores empleados para Leishmnania sp. no reconocen secuencias de ADN de otros parásitos como T. cruzi, T. rangeli, los cuales comparten secuencias de ADN muy parecidas a la de Leishmania por pertenecer al orden Kinetoplastida. Tampoco amplificó el ADN de T. gondii, que aunque no es kinetoplastida, es un protozoario que también infecta caninos. No se observó amplificación ni con ADN de canino, ni con ADN humano, confirmando que los cebadores utilizados son específicos para la amplificación solo de ADN del parásito Leishmania, obteniéndose de esta forma una especificidad igual a la reportada por la mayoría de los autores [12-18]. La especificidad es un aspecto muy importante en las técnicas de diagnóstico ya que muchas de las zonas endémicas de leishmaniasis en nuestro país, lo son también para la enfermedad de Chagas, y en las técnicas inmunológicas se pueden observar reacciones cruzadas, por lo que se requiere de técnicas de diagnóstico que puedan diferenciar estas infecciones [25-26].
Con la PCR estandarizada se probaron las muestras de caninos positivas (asintomáticos) y negativas a Leishmania sp. del estado Cojedes, observándose que las seis muestras positivas, previamente, todas presentaron amplificación, mientras que en las negativas en ningún caso se obtuvo amplificación. Por lo que, tanto la extracción de ADN de muestras colectadas en papel de filtro, como la técnica de PCR estandarizada fueron adecuadas para el correcto diagnóstico de las muestras. El uso del papel de filtro presenta ventajas para la recolección, transporte y almacenamiento de las muestras, como ha sido descrito también por otros autores [27].
La técnica de PCR puede ser una herramienta muy útil para el diagnóstico de leishmaniasis canina y la implicación de los caninos como reservorios en las distintas zonas endémicas, ya que ha mostrado en muchos casos mejor sensibilidad y especificidad que las técnicas convencionales, siendo de gran importancia para la detección de los casos oligosintomáticos y los asintomáticos y permite detectar infección activa y reciente [28-30]. Sin embargo, es importante enfatizar que no siempre la técnica de PCR muestra mejores índices diagnósticos que las técnicas convencionales [31], por lo que es recomendable su uso en conjunto con las técnicas parasitológicas e inmunológicas para el diagnóstico certero de leishmaniasis y para el fortalecimiento de los programas de control.
En la técnica de Ln-PCR estandarizada se empleó menos reactivos (cebadores y Taq polimerasa), que la utilizada por los autores originales, lo cual representa ahorro de reactivos, a pesar que la sensibilidad analítica fue un poco menor (10 fg vs. 1 fg). La sensibilidad y la especificidad de la técnica permiten su uso para diagnóstico, ya que fue capaz de identificar correctamente las muestras de caninos, positivas y negativas, empleadas en el estudio. La recolección de las muestras de sangre en papel de filtro, facilitó la obtención, transporte y almacenamiento de las muestras y fue adecuada para el aislamiento de ADN empleado en la técnica de Ln-PCR.
Los autores declaran no tener ningún tipo de conflicto de intereses
NOTA:
1 A quien debe dirigirse la correspondencia (To whom correspondence should be addressed)
1. Botero D, Restrepo M. Parasitosis Humana. (5a ed), Corporación para investigaciones biológicas (CIB) Medellín, Colombia. 2012; p. 315-49. [ Links ]
2. Alvar J. Las Leishmaniasis: de la biología al control. 2a ed. Laboratorios Intervet S.A, Salamanca España. 2001; p. 15-45.
3. WHO. World Health Organization. Leishmaniasis. 2015; Fact sheet N° 375. [ Links ]
4. Desjeux P. Leishmaniasis: current situation and new perspetives. Comp Immunol Microbiol Infect Dis. 2004; 27(5): 305-18. [ Links ]
5. Rosilved R, Silva B. Factores de riesgo involucrados en la infección por Leishmania infantum/Leishmania chagasi. Rev Inst Nac Hig Rafael Rangel, 2008; 39(2): 35-41. [ Links ]
6. MPPPS. Memoria 2013. Servicio Autónomo Instituto de Biomedicina. Unidad de Informática. Venezuela. Caracas. [Internet]; 2014; p. 322. [acceso 26 de julio de 2015]. Disponible en: http://transparencia.org.ve/wp-content/uploads/2012/10/Memoria-MPPS-2013.pdf. [ Links ]
7. MPPPS. Boletin epidemiológico N° 44. 2014; p. 5. [acceso 26 de julio de 2015]. Disponible en: http://www.mpps.gob.ve/index.php?option=com_phocadownload&view=category&id=43:ano2014&Itemid=915. [ Links ]
8. Zerpa O, Ulrich M, Negrón E, Rodríguez N, Centeno M, Rodríguez V, et al. Canine visceral Leishmaniasis on Margarita Island (Nueva Esparta, Venezuela). Trans R Soc Trop Med Hyg. 2000; 94(5): 484-7. [ Links ]
9. Albarracin K, Molina N, Romero J. Seroprevalencia de leishmaniasis canina en las localidades Topo I, Topo II y las Cumbres de Zamora de la parroquia capital de San Mateo municipio Bolívar, estado Aragua. Rev Fac Cienc Vet, 2011; 52:2. [ Links ]
10. Bonfante R, Barroeta S. Leishmanias y Leishmaniasis en América con especial referencia a Venezuela. 1a ed. Tipografía y Litografía Horizonte C.A. Barquisimeto. Estado Lara, Venezuela. 2002; p. 130-45.
11. Becerril F, Romero C. Parasitología Médica. 73-83. Editorial Mc. Graw Hill. México. 2001; p. 73-80. [ Links ]
12. Van Eys GJ, Schoone GL, Kroon NC, Ebeling SB. Sequence analysis of small subunit ribosomal RNA genes and its for detection and identification of Leishmania parasites. Mol Bichem Parasitol. 1992; 51(1):133-42. [ Links ]
13. Cupolillo E, Grimaldi JG, Momen H, Beverley SM. Intergenic region typing (IRT): a rapid molecular approach to the characterization and evolution of Leishmania. Mol Bichem Parasitol. 1995; 73(1-2): 145-55. [ Links ]
14. Harris E, Kropp G, Belli A, Rodriguez B, Agabian N. Single-step multiplex PCR assay for characterization of New World Leishmania complexes. J Clin Microbiol. 1998; 36(7):1989-95. [ Links ]
15. Chiurillo MA, Sachdeva M, Dole VS, Yepes Y, Miliani E, Vazquez L, et al. Detection of Leishmania causing visceral leishmaniasis by a polymerasa chain reaction assay based on telomeric sequences. Am J Trop Med Hyg. 2001; 65(5):573-82. [ Links ]
16. Cruz I, Cañavate C, Rubio JM, Morales MA, Chicharro C, Laguna F, et al. Spanish HIV-Leishmania Study Group. A nested polymerase chain reaction (Ln-PCR) for diagnosing and monitoring Leishmania infantum infection in patients co-infected with human immunodeficiency virus. Trans R Soc Trop Med Hyg. 2002; 96 S1:S185-9. [ Links ]
17. Lachaud L, Marchergui-Hammami S, Chabbert E, Dereure J, Dedet JP, Bastien P. Comparison of six PCR methods using peripheral blood for detection of canine visceral leishmaniasis. J Clin Microbiol. 2002; 40(1): 210-5. [ Links ]
18. Schönian G, Nasereddin A, Dinse N, Schweynoch C, Schallig H, Presber W, et al. PCR diagnosis and characterization of Leishmania in local and imported clinical samples. Diagn Microbiol Infect Dis. 2003; 47(1): 349-58. [ Links ]
19. Zulueta AM, Villarroel E, Rodríguez N, Feliciangeli MD, Mazzarri M, Reyes O, et al. Epidemiological aspects of american visceral leishmaniasis in an endemic focus in eastern Venezuela. Am J Trop Med Hyg. 1999; 61(6):945-50. [ Links ]
20. Rodriguez N, De Lima H, Aguilar CM, Rodríguez A, Barker DC, Convit J. Molecular epidemiology of cutaneous leishmaniasis in Venezuela. Trans R Soc Trop Med Hyg. 2002; 96 (S1):105-9. [ Links ]
21. De Lima H, Carrero J, Rodríguez A, de Guglielmo Z, Rodríguez N. Trypanosomatidae of public health importance occurring in wild and synanthropic animals of rural Venezuela. Biomédica, 2006; 26(1): 42-50. [ Links ]
22. Contreras VT, Araujo-Jorge TC, Bonaldo MC, Thomaz N, Barbosa HS, Meirelles M de N, et al. Biological aspects of the Dm 28c clone of Trypanosoma cruzi after metacyclogenesis in chemically defined media. Mem Inst Oswaldo Cruz. 1988; 83(1):123-33. [ Links ]
23. Añez N, Nieves E., Cazorla D. Estudies on Trypanosoma rangeli Tejera, 1920, IX. Course of infecction on different stage of Rhodnius prolixus. Mem Inst Oswaldo Cruz. 1987; 82(1):1-6. [ Links ]
24. Sambrook J, Russell D. Molecular Cloning. A Laboratory Manual, 3a ed. Cold Spring Harbor Laboratory Press. New Cork. 2001; p. 6.3-6.30. [ Links ]
25. de Oliveira I, Silva R, Sucupira M, da Silva E, Reis A, Grimaldi G, et al. Multi-antigen print immunoassay (MAPIA)-based evaluation of novel recombinant Leishmania infantum antigens for the serodiagnosis of canine visceral leishmaniasis. Parasit Vectors. 2015; 8:45. doi: 10.1186/s13071-015-0651-6. [ Links ]
26. Faria AR, de Castro Veloso L, Coura-Vital W, Reis AB, Damasceno LM, Gazzinelli RT, et al. Novel Recombinant Multiepitope Proteins for the Diagnosis of Asymptomatic Leishmania infantum-Infected Dogs. PLoS Negl Trop Dis. 2015 Jan 8;9(1):e3429. doi: 10.1371/journal.pntd.0003429. eCollection 2015. [ Links ]
27. Da Silva ES, Gontijo CM, Pacheco RS, Brazil RP. Diagnosis of human visceral leishmaniasis by PCR using blood samples spotted on filter paper. Genet Mol Res. 2004; 3(2):251-7. [ Links ]
28. Regina-Silva S, Fortes-Dias CL, Michalsky ÉM, França-Silva JC, Quaresma PF, da Rocha Lima AC, et al. Evaluation of parasitological examination, kDNA polymerase chain reaction and rK39-based immunochromatography for the diagnosis of visceral leishmaniasis in seropositive dogs from the screening-culling program in Brazil. Rev Soc Bras Med Trop. 2014; 47(4):462-8. [ Links ]
29. Santos TR, Carreira VS, Ferrari HF, Moreira MA, Luvizotto MC. Comparison of PCR with stained slides of bone marrow and lymph nodes aspirates with suspect diagnosis for leishmaniasis. Acta Trop. 2014; 140:137-40. [ Links ]
30. Costa LN, Borba AS, Castagna CL, Carvalho Filho EB, Marson FA, Sá Junior FF, et al. The Leishmaniasis Study Group. Evaluation of PCR in the diagnosis of canine leishmaniasis in two different epidemiological regions: Campinas (SP) and Teresina (PI), Brazil. Epidemiol Infect. 2014; 143(5): 1088-95. [ Links ]
31. Ferrer E, Lares M, Viettri M, Medina M. Comparación entre técnicas inmunológicas y moleculares para el diagnóstico de la Enfermedad de Chagas. Enferm Infecc Microbiol Clin. 2013; 31(5): 277-82. [ Links ]