Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de la Sociedad Venezolana de Microbiología
Print version ISSN 1315-2556
Rev. Soc. Ven. Microbiol. vol.36 no.1 Caracas June 2016
Comparación del sistema BacT/ALERT® 3D con los métodos de cultivo Lowenstein-Jensen y Ogawa-Kudoh para el aislamiento de micobacterias
Mirexia Eva Marcano-Vásqueza,*, Ilsy Andreína Ramíreza, Omaira Josefina Da Mata-Jardínb,c, Sandra Fernández-Figueirasb
a Escuela de Bioanálisis, Universidad Central de Venezuela. b Laboratorio de Diagnósticos Especiales, Instituto Nacional de Higiene Rafael Rangel. c Postgrado en Ciencias Biológicas, Universidad Simón Bolívar. Caracas, Venezuela.
* Correspondencia: E-mail: eva_marcano20@hotmail.com
Resumen: La tuberculosis es una enfermedad causada por bacterias pertenecientes al Complejo Mycobacterium tuberculosis. El diagnóstico se dificulta por su sintomatología inespecífica y los métodos bacteriológicos que ofrecen resultados tardíos. El objetivo del presente estudio fue comparar el desempeño del sistema automatizado BacT/ALERT® 3D con los métodos convencionales de cultivo en medio Lowenstein-Jensen (L-J) y Ogawa-Kudoh (O-K) para el aislamiento de micobacterias. Se procesaron 266 muestras provenientes de pacientes con sospecha de tuberculosis entre mayo y octubre de 2013. Se aislaron 63 bacilos acido-resistentes: 46 M. tuberculosis y 17 micobacterias no tuberculosas (MNT). Al comparar los tres métodos, se observó que todos presentaron desempeño similar en el aislamiento de M. tuberculosis. Las tasas de recuperación obtenidas no mostraron diferencia estadísticamente significativa (p>0,05) sin embargo, con BacT/ALERT® 3D se aislaron mayor número de MNT, con diferencia significativa respecto al L-J (p<0,05). El tiempo de detección promedio de M. tuberculosis fue de 11 días por BacT/ALERT® 3D, 20 días en L-J y 23 días en O-K. La contaminación cruzada, fue de 0,38%. Se concluyó que BacT/ALERT® 3D es una excelente herramienta para el aislamiento de M. tuberculosis, mejora la recuperación de las MNT y reduce significativamente el tiempo de diagnóstico, lo que permitiría un tratamiento oportuno, con mayor probabilidad de sobrevida del paciente.
Palabras clave: micobacterias, Complejo Mycobacterium tuberculosis, BacT/ALERT® 3D, medios sólidos, medios líquidos, cultivos.
Comparison of BacT/ALERT 3D® system with the culture methods of Lowenstein-Jensen and Ogawa-Kudoh, for the isolation of mycobacteria
Abstract: Tuberculosis is a disease caused by species of the Mycobacterium tuberculosis complex. The diagnosis is difficult due to nonspecific symptoms and late results of bacteriological culture methods. The aim of this study was to compare the efficiency of the BacT/ALERT® 3D automated system with conventional methods of culture on Lowenstein-Jensen (L-J) and Ogawa-Kudoh (O-K). A total of 266 specimens from patients with clinical suspected tuberculosis were studied from May to October 2013. We recovered, 63 acid fast bacilli isolates: 46 identified as M. tuberculosis and 17 as nontuberculous mycobacteria (NTM). The three methods had a similar performance for isolation of M. tuberculosis; recovery rates obtained showed no statistically significant difference (p> 0.05). However, with the BacT/ALERT® 3D system, a larger number of MNT were isolated, with significant statistic difference for L-J (p <0.05). The average detection time for M. tuberculosis was 11 days with the BacT/ALERT® 3D system, with significant statistic difference for LJ (20.4 days) and O-K (23.2 days). Additionally, cross-contamination was determined as 0.38%. The study results showed that the BacT/ALERT® 3D system is an excellent tool for isolation of M. tuberculosis and it improves the recovery of NTM. Also, the time of diagnosis is significantly reduced, leading to earlier treatment that could improve patient recovery.
Keywords: mycobacterias, Mycobacterium tuberculosis complex, BACT / ALERT 3D, solid medium, liquid medium, cultures.
Recibido 13 de julio de 2015; aceptado 21 de febrero de 2016
Introducción
La tuberculosis (TB) es una enfermedad infecciosa, causada por micobacterias pertenecientes al Complejo Mycobacterium tuberculosis (CMT), siendo M. tuberculosis el principal agente causal, constituyendo uno de los principales problemas de salud pública en la actualidad. Esta patología afecta a la población a nivel mundial, pero especialmente la localizada en los países tropicales y en vías de desarrollo, entre los cuales se encuentra Venezuela, donde la TB se ubica entre las 20 causas principales de muerte [1,2]. Para el año 2013, la prevalencia de TB fue de 49 por cada 100.000 habitantes y la incidencia 33 por cada 100.000 habitantes [3].
Uno de los pilares en el control de esta enfermedad es mejorar el diagnóstico precoz y la rapidez de identificación del agente causal. Cualquier retardo en la detección de M. tuberculosis conducirá, inevitablemente, a una demora en el inicio de una terapia efectiva e incrementará la probabilidad de diseminación de la enfermedad. El diagnóstico de TB se realiza mediante la orientación clínica y epidemiológica, y a nivel microbiológico se basa en la observación directa de M. tuberculosis a través de la baciloscopia y su aislamiento en medios de cultivo. La baciloscopia es un método rápido y sencillo, pero de baja sensibilidad, ya que requiere aproximadamente 104 bacilos por mL de muestra para la obtención de un resultado positivo; para mejorar su valor predictivo positivo (VPP) se debe efectuar la toma de dos muestras, con la primera se logra detectar aproximadamente el 80% de los casos y con la segunda un 15% adicional, logrando elevar hasta un 95% la detección del bacilo tuberculoso. El cultivo en cambio, si bien ofrece una mayor sensibilidad (detecta de 10 a 100 bacilos/mL de muestra), requiere de un prolongado tiempo de incubación para evidenciar el crecimiento del agente etiológico, aproximadamente de 2 a 6 semanas, dependiendo de la carga bacilar [4].
Debido a esto, existe la necesidad de evaluar e implementar nuevos métodos de diagnóstico confiables y con tiempos de respuesta cortos, por lo que se planteó como objetivo, en el presente estudio, comparar el desempeño del BacT/ALERT® 3D con los métodos convencionales de cultivo Lowenstein-Jensen (L-J) y Ogawa-Kudoh (O-K) en cuanto a tasa de recuperación, tiempo de detección, sensibilidad, especificidad, VPP, valor predictivo negativo (VPN) y tasa de contaminación con flora asociada. Además se propuso descartar y/o confirmar la presencia de posibles casos de contaminación cruzada, obtenida con el sistema automatizado, mediante la técnica de tipificación molecular VNTR-MIRU (del inglés: Variable Number Tandem Repeat-Mycobacterial interspersed repetitive units), ya que los medios líquidos tienen mayor tendencia a la contaminación debido a la formación de aerosoles durante el procesamiento de las muestras.
Materiales y métodos
Se seleccionaron muestras de pacientes con sospecha clínica de TB, remitidas al Laboratorio de Diagnósticos Especiales del Instituto Nacional de Higiene Rafael Rangel, en el periodo comprendido entre mayo y octubre de 2013. Como criterio de inclusión se consideraron muestras clínicas pulmonares y extrapulmonares, excluyendo las provenientes de médula ósea o sangre. Se procesaron 266 muestras: 256 pulmonares (224 esputos, 30 lavados bronquiales, 2 líquidos pleurales) y 10 extrapulmonares (2 biopsias, 7 orinas y 1 líquido ascítico). Todas las muestras fueron procesadas en un tiempo no mayor a 48 horas después de su recepción. Este estudio fue aprobado por el Comité de Bioética de la Escuela de Bioanálisis de la Facultad de Medicina de la Universidad Central de Venezuela.
Procesamiento de las muestras: Todos los especímenes fueron examinados microscópicamente a través de la baciloscopia (a excepción de las muestras de orina) y posteriormente, fueron procesados, mediante las tres metodologías a comparar, por los mismos operadores. La cantidad de la muestra fue tomada de acuerdo a la metodología. Las muestras no estériles fueron sometidas a un proceso de descontaminación-digestión a través del método N-acetil-L-cisteína-hidróxido de sodio al 2% (NALC-NaOH) [4] y posteriormente sembradas en el medio de L-J y en botellas MP (Mycobacteria process) conteniendo suplemento antibiótico (anfotericina B, azlozilina, ácido nalidíxico, polimixina B, trimetoprim y vancomicina) e introducidas en el sistema BacT/ALERT®. En el método de O-K se empleó hidróxido de sodio al 4% (NaOH) para la descontaminación de las muestras y posteriormente fueron inoculadas en medio sólido Ogawa [4]. Los líquidos estériles se inocularon directamente (5 mL), sin centrifugar, en las botellas MB (Mycobacteria blood) [5], suplementadas con el fluido de enriquecimiento MB/BacT, y posterior a la centrifugación se sembraron 0,5 mL del sedimento en los medios L-J y Ogawa. Los medios sólidos se incubaron por 8 semanas a 37 °C.
Se realizó la observación de los cultivos en medios sólidos semanalmente, hasta completar el tiempo de incubación, mientras que los medios líquidos procesados por la metodología del BacT /ALERT® fueron monitoreados por el sistema cada media hora durante 42 días. Tanto los reactivos como los medios solidos fueron preparados en la División de Producción de Reactivos y Medios de Cultivo del Instituto Nacional de Higiene Rafael Rangel. Los medios líquidos y los suplementos del sistema automatizado fueron suministrados por el fabricante (bioMérieux).
Identificación de los aislados: Se evaluó la presencia de bacilos ácido resistentes (BAR) en los cultivos con desarrollo bacteriano mediante la tinción de Ziehl Neelsen (ZN). A partir de los cultivos positivos en medio líquido, se centrifugó durante tres minutos 0,5 mL del medio a 14.000 revoluciones por minuto (rpm), se descartó el sobrenadante y se resuspendió en TE 1X (Buffer Tris 10 mM- EDTA 1 mM, pH 8,0). Este procedimiento se realizó dos veces en cada muestra para eliminar interferentes o inhibidores de la Reacción en Cadena de la Polimerasa (PCR). Luego las muestras fueron sometidas a 100 °C durante 10 min para obtener el ADN. En cuanto a los cultivos positivos en medios sólidos, se prepararon suspensiones bacterianas a partir de 3 a 5 colonias en buffer TE 1X pH 8,0 y se procedió a realizar la extracción de ADN como se describió previamente.
La identificación molecular de los aislados de crecimiento lento se realizó mediante la determinación de las regiones de diferencia RD1, RD4, RD9 y RD12, de acuerdo a Warren et al. [6] con modificaciones. Brevemente, para la reacción de amplificación se utilizó un volumen final de 50 µL, con las siguientes concentraciones finales: buffer 1X (200 mM Tris-HCl pH 8,4 y 500 mM KCl); MgCl 3,5 mM; dNTPs 0,6 mM; betaine 0,5 M; 1,25 U taq ADN polimerasa (PROMEGA), 25 pmol de cada iniciador, y se adicionó 10 µL del ADN extraído del medio de cultivo mediante el método de ebullición [7]. La PCR se llevó a cabo en 35 ciclos a 94 ºC por 1 min, 58 ºC por 1 min y 72 ºC por 1 min, con un paso previo a 95 ºC por 5 min y uno adicional a 72 ºC por 10 min. Como control positivo se utilizó ADN de la cepa M. tuberculosis H37Rv. Los productos amplificados se analizaron mediante electroforesis en gel de agarosa al 3%, a 6 V/cm por 2 h, con búfer TBE 0,5X. Para la visualización, los geles fueron teñidos en una solución de bromuro de etidio (1 µg/mL), posteriormente se lavaron con agua destilada y las imágenes fueron digitalizadas en el equipo Gel DOC XR de Bio-Rad. La determinación de los tamaños en pares de bases (pb) de los amplicones se hizo mediante el programa Quantity One®.
Los cultivos con aislamiento de BAR de crecimiento rápido y los de crecimiento lento con resultados negativos en la PCR múltiple de las RD, se identificaron mediante el análisis del polimorfismo del gen hsp65, descrito por Telenti et al. [8] con modificaciones. Brevemente, se realizó la reacción de amplificación con un volumen final de 50 µL, con las siguientes concentraciones finales: buffer 1X (200 mM Tris-HCl pH 8,4 y 500 mM KCl); MgCl 1,5 mM; dNTPs 0,2 mM; 1,25 U de la Taq ADN polimerasa (PROMEGA) y 25 pmol de cada iniciador. Se adicionó 10 µL de la muestra de ADN extraído como se describió previamente. Como control positivo se utilizó ADN de la cepa ATCC®19977 M. abscessus. La visualización de los amplicones se realizó mediante electroforesis en gel de agarosa al 1%, 6 V/cm por 10 min en búfer TBE 0,5X. Posteriormente se llevó a cabo la restricción enzimática de los productos de amplificación con 5 U de BstE II y Hae III (PROMEGA) por dos horas, bajo las condiciones sugeridas por el fabricante. El análisis de los patrones de restricción se realizó mediante electroforesis en gel de agarosa para pequeños fragmentos al 3% durante 2 horas a 6 V/cm. Se determinó el tamaño en pb de cada fragmento mediante el sistema de Gel DOC de Biorad® y el programa QuantityOne®. Los patrones obtenidos fueron ingresados en la base de datos de PRASITE para su identificación [9].
Evaluación de la contaminación cruzada: La contaminación cruzada es la transferencia accidental de microorganismos, en este caso de bacilos de la TB de una muestra a otra al ser procesadas en serie en el laboratorio. Las fuentes de este tipo de contaminación son generalmente muestras que contienen abundantes bacilos. Este error puede originar falsos resultados positivos del cultivo. Se consideró la contaminación cruzada en aquellas muestras que tenían un ZN y cultivo en medio sólido negativos en conjunto con una botella de BacT/ALERT® 3D positiva con una baja densidad de crecimiento, y que fueron procesadas el mismo día que otra muestra con alta carga bacilar. Adicionalmente, se realizó una revisión de los antecedentes clínicos de los pacientes, es decir, si tenían diagnóstico previo de TB, si recibían tratamiento, ausencia de síntomas y posible relación epidemiológica entre ellos. Todos los aislados que cumplían con estas características se seleccionaron para la tipificación mediante la técnica de VNTR-MIRU, siguiendo el protocolo descrito en la literatura [10].
Contaminación por flora asociada: Se determinó el porcentaje de contaminación considerando el número de muestras con crecimiento de flora asociada (bacterias no BAR) a las 24 a 48 horas con respecto a las siembras totales para cada metodología.
Análisis estadístico: Se utilizó el cultivo en L-J como el estándar de oro, y se comparó el desempeño del BacT/ALERT® 3D y el método O-K, en términos de sensibilidad, especificidad, VPP y VPN. Para determinar si hubo diferencia significativa se utilizó la prueba de chi cuadrado (X2) con un nivel de confianza de 95%. Para determinar si hubo diferencias significativas en el número de días requerido para la recuperación de micobacterias, se realizó la prueba t de Student, utilizando igualmente un nivel de confianza de 95%; valores de p< 0,05 fueron considerados como significativos.
Resultados
Se obtuvieron 63 aislamientos de BAR por los tres métodos empleados, de los cuales 46 fueron identificados como M. tuberculosis y 17 como micobacterias no tuberculosas (MNT). A través del sistema BacT/ALERT® 3D se obtuvieron 54 cultivos positivos (41 M. tuberculosis y 13 MNT), 42 por el medio L-J (38 M. tuberculosis y 4 MNT) y 47 por el método O-K (41 M. tuberculosis y 6 MNT) (Tabla 1).
Tabla 1. Comparación de las tasas de recuperación de micobacterias según el método de cultivo utilizado.
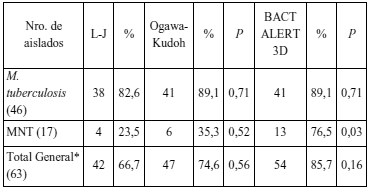
L-J = Lowenstein-Jensen. Los números entre paréntesis indican la cantidad total de aislados. *Total general: número de muestras positivas con las tres metodologías.
Con base en los resultados anteriores se calcularon las tasas de recuperación por cada método. En cuanto a M. tuberculosis, la tasa de recuperación que se obtuvo en el medio L-J fue de 82,6%, mientras que los métodos O-K y BacT/ALERT® 3D fue de 89,1%, observándose que no hubo diferencia significativa para el aislamiento de esta especie entre los tres métodos (p=0,71). En el caso de MNT, el BacT/ALERT® 3D presentó mayor porcentaje de recuperación (76,5%), encontrándose diferencia significativa con respecto al medio L-J (23%) (p=0,03), (Tabla 1). Se presentaron 3 casos de infección por MNT confirmados microbiológicamente de acuerdo a los criterios sugeridos en la literatura [11]. En el primer caso, por BacT/ALERT® 3D se aisló M. avium de las dos muestras de esputo procesadas, mientras que en el L-J se aisló el mismo microorganismo solo en una de las muestras, y en O-K no hubo aislamiento. En el segundo caso, por BacT/ALERT® 3D se aisló M. porcinum en las dos muestras de esputo procesadas, mientras que en los otros medios de cultivo no pudo ser detectado. Por último, en el tercer caso, se aisló del BacT/ALERT® 3D M. abscessus tipo 2 en las dos muestras, el cual solo pudo ser detectado en una sola de las muestras sembradas en L-J y nuevamente en O-K no se obtuvo aislamiento.
El 95,1% de los aislados de M. tuberculosis fueron identificados a partir de ADN extraído directamente del medio líquido de BACT/ALERT® 3D, mientras que para las MNT sólo se logró la identificación del 46,6%. En aquellos aislados donde no se logró la identificación usando ADN extraído del medio líquido se utilizó ADN extraído a partir de repique en medio L-J.
Del total de muestras con baciloscopia positiva, 26 tuvieron cultivo positivo por el método O-K, 25 por L-J y 26 por BacT/ALERT® 3D (Tabla 2).
Tabla 2. Número de aislamientos de micobacterias de acuerdo a la baciloscopia.

L-J: Lowenstein-Jensen. + Positivo. − Negativo. *Nota: Fueron excluidos de esta tabla los cultivos contaminados.
El equipo BacT/ALERT® 3D presentó una sensibilidad y especificidad de 97,6% y 93,9% respectivamente, un VPP de 78,4% y VPN de 99,4%. Para el método O-K, la sensibilidad y especificidad fue de 85,7% y 95,5% respectivamente, VPP de 80% y VPN de 96,9%. El tiempo de detección promedio de M. tuberculosis fue de 11 días por el BacT/ALERT® 3D; 20,4 días por el L-J, y 23,1 días por el método O-K, encontrándose diferencia significativa entre el sistema BacT/ALERT® 3D y el medio de referencia L-J (p=0,03). En 11 casos se presentaron falsas alarmas en el equipo BacT/ALERT® 3D, el cual indicó positividad en algunas botellas en las que no se observó bacterias con la tinción de ZN, ni crecimiento en subcultivos en placas de agar tripticasa de soya a partir del sedimento, por lo tanto se procedió a reintroducir las botellas en el equipo.
La tasa de contaminación por flora asociada para todas las muestras procesadas con el BacT/ALERT® 3D fue de 7,9%, la cual se encuentra dentro de los valores aceptables establecidos, según la literatura internacional; la del medio L-J fue de 8,3% y la del método O-K fue de 1,1%.
En cuanto a la contaminación cruzada, se realizó la tipificación mediante VNTR-MIRU de siete aislados que reunían los criterios descritos en la sección de materiales y métodos. Cuatro de los aislados (A, C, D, F) presentaron patrones diferenciables de VNTR, por lo que la contaminación cruzada fue descartada. Los otros tres aislados B, E y G mostraron patrones genéticos idénticos (Figura 1).
Figura 1. Dendograma obtenido a través de análisis comparativo de los patrones genéticos de VNTR de 7 aislados de muestras de pacientes referidos al INHRR mediante UPGMA. Los aislados B, E y G presentaron patrones genéticos idénticos; B y E pertenecen a un mismo paciente y el G a un paciente diferente.
Discusión
Las tasas de recuperación de M. tuberculosis observadas en el presente estudio, al comparar el sistema BacT/ALERT® 3D con el medio L-J y el método O-K, fueron similares; sin embargo, la siembra en medios líquidos en el sistema automatizado fue más eficiente en cuanto al tiempo de detección, el cual fue significativamente menor que el de los medios sólidos, ofreciendo un diagnóstico rápido de TB, representando una mayor oportunidad de sobrevida para el paciente, a la vez que reduce la posibilidad de diseminación. En cuanto a la sensibilidad, el BacT/ALERT® 3D fue superior fue superior con el sistema BacT/ ALERT® 3D, encontrándose dentro del rango de 78-99% reportado internacionalmente para este sistema automatizado, siendo mayor a la obtenida por Solórzano et al. [12] y similar a la de Martínez y col. [13].
En el año 2012, se realizó un estudio con el objetivo de evaluar la posibilidad de utilizar el sistema BacT/ALERT® 3D y el medio de cultivo 7H10 Middlebrook (MB7H10) como medios de aislamiento primario de micobacterias; ambos fueron comparados con el medio L-J como método de referencia. En los resultados, de las 236 muestras evaluadas, se obtuvieron 116 aislamientos reconocidos en su totalidad por el BacT/ALERT® 3D, 82 por el medio L-J y 62 por el medio MB7H10. Por otra parte, el BacT/ALERT® 3D y el L-J en conjunto lograron 82 aislamientos, 62 se obtuvieron del conjunto MB7H10 y BACT/ALERT 3D, 58 de L-J junto con MB7H10 y 58 de los tres en conjunto. Además, los tiempos de detección para el L-J, MB7H10 y el BacT/ALERT® 3D fueron de 30, 31 y 18 días, respectivamente. Los autores del estudio concluyeron que, ni el medio L-J ni el MB7H10 pueden ser considerados como único medio para el aislamiento de micobacterias, y la combinación de medios no demuestra ser superior al uso de BacT/ALERT® 3D, por lo que éste puede considerarse superior comparado con los otros dos en términos de tasa de aislamiento y rapidez, con un promedio de positividad entre los 7 y 10 días [14].
Adicionalmente en nuestro estudio, la metodología automatizada presentó un mejor desempeño para el aislamiento de MNT, siendo este un resultado importante, ya que podría indicar que el sistema automatizado empleado mejora la oportunidad de recuperación de estas especies. De acuerdo a la Sociedad Americana de Tórax, uno de los criterios bacteriológicos para considerar como patógena una MNT incluye el aislamiento de la misma a partir de 2 muestras de esputo, tomadas en diferentes momentos [11]. Considerando este criterio, con el uso del BacT/ALERT® se detectaron 3 casos, que no pudieron ser evidenciados con los otros medios. Por lo tanto, el BacT/ALERT® 3D demostró ser una herramienta valiosa para el diagnóstico de micobacteriosis pulmonar, afección que pudiera pasar desapercibida con el uso exclusivo de medios sólidos, ya que el aislamiento en una sola muestra pudiera ser considerado contaminación o colonización.
En la actualidad hay diversos estudios que refieren la importancia del método de descontaminación para el diagnóstico de la micobacteriosis pulmonar, siendo uno de los factores que pueden ocasionar un subdiagnóstico [11]. La concentración y tiempo de exposición al agente descontaminante afecta el aislamiento de MNT, siendo éstas más susceptibles a agentes muy agresivos, tales como el NaOH al 4% empleado en el método O-K. Por otra parte, los medios enriquecidos como los del BacT/ALERT® 3D permiten el aislamiento de MNT que requieren ciertos factores de crecimiento que no están disponibles en medios sólidos usados en la rutina de aislamiento de especies del complejo de M. tuberculosis. Otro factor importante a considerar es la temperatura; varias especies requieren temperaturas entre 25 y 30 °C para su crecimiento. Alfa et al. [15] adicionaron al sistema BacT/ALERT® 3D un módulo con temperatura de incubación de 30 °C, facilitando el aislamiento de otras especies de MNT como M. chelonae, M. marinum y M. ulcerans. Esto es un elemento de interés a considerar en futuras evaluaciones.
La Organización Panamericana de la Salud establece un rango de contaminación por flora asociada, entre el 3 y el 4% para el método de Petroff en medios sólidos y entre 8 a 9% para medios enriquecidos (líquidos), o cuando se utiliza una concentración menor de NaOH en los métodos de descontaminación [3]. Los métodos de siembra en medios sólidos evaluados en el presente estudio mostraron tasas de contaminación fuera de este rango, a diferencia del sistema BacT/ALERT® el cual mostró una tasa de contaminación dentro de los rangos aceptados para este tipo de metodología. A pesar de que la literatura refiere que el método de descontaminación que debe utilizarse para el BacT/ALERT® 3D es NALC-NaOH al 2%, diversos estudios han reportado que con otros métodos de descontaminación como el Petroff modificado, se ha obtenido una tasa de contaminación menor (6,7%) para este sistema [12]. Esto demuestra la necesidad de evaluar otros métodos de descontaminación que ofrezcan buena recuperación de micobacterias, así como tasas más bajas de contaminación.
Por otra parte, de las 231 muestras que presentaron baciloscopia negativa, 28 presentaron cultivo positivo en BacT/ALERT® 3D, 17 en L-J y 21 en O-K, lo que demuestra que si bien la baciloscopía es una herramienta rápida no es lo suficientemente sensible ni puede considerarse por sí sola como método diagnóstico; el cultivo es la única prueba confiable para el diagnóstico de TB, por lo que siempre debe ser incluido como método confirmatorio, siendo aún más importante en pacientes paucibacilares.
En cuanto a contaminación cruzada, sólo se encontró 1 caso (0,38%), realizándose la confirmación a través de una segunda muestra solicitada al paciente; este porcentaje es menor que los valores encontrados en la literatura [4].
Si bien el sistema automatizado presenta una gran cantidad de ventajas frente a los medios sólidos empleados, no está exento de desventajas. Entre ellas, las falsas alarmas que presentó el equipo, posiblemente causadas por diversos elementos, como por ejemplo las células. Por otra parte, para la identificación de las micobacterias aisladas mediante las diversas técnicas de biología molecular empleadas, se hizo necesario un doble lavado del sedimento, ya que el medio líquido de las botellas puede contener elementos que inhiben la amplificación mediante PCR; esto sugiere que no es suficiente la extracción de ADN mediante el método de ebullición y son necesarios métodos de purificación para la obtención del ADN directamente de los medios líquidos del BacT/ALERT® 3D.
Conclusiones
De acuerdo a los resultados obtenidos en esta investigación se puede concluir que el sistema BacT/ALERT® 3D es un método eficiente para la recuperación de M. tuberculosis procedente de muestras clínicas al compararlo con los medios sólidos L-J y Ogawa-Kudoh. El sistema BacT/ALERT® 3D también mostró un mejor desempeño para el aislamiento de MNT y podría representar una herramienta valiosa para el diagnóstico de micobacteriosis. Adicionalmente, el sistema automatizado presentó un tiempo de detección significativamente menor que los medios sólidos en estudio, ofreciendo un diagnóstico rápido de TB (11 días).
Agradecimientos.
Estos resultados forman parte del Trabajo Especial de Investigación para optar al título de Licenciado en Bioanálisis de los autores Marcano-Vásquez, Mirexia Eva y Ramirez-Araujo, Ilsy Andreina. El estudio fue financiado por la Gerencia de Docencia e Investigación, y la Gerencia de Diagnóstico y Vigilancia Epidemiológica del Instituto Nacional de Higiene Rafael Rangel.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Referencias
1. De Hoyos V. Métodos para la caracterización fenotípica y genotípica de la resistencia de Mycobacterium tuberculosis a los antimicrobianos [Tesis]. Pontificia Universidad Javeriana Bogotá; 2009. [ Links ]
2. Ministerio del Poder Popular para la Salud (MPPS). Anuario de Mortalidad. 2009. [ Links ]
3. World Health Organization (WHO). Disponible en: https://extranet.who.int/sree/Reports?op=Replet & name=%2FWHO_HQ_Reports%2FG2%2FPROD%2FEXT%2FTBCountryProfile & ISO2=VE & LAN=EN & outtype=html. Acceso: 15 noviembre 2014. [ Links ]
4. Organización Panamericana de la Salud (OPS). Manual para el Diagnóstico bacteriológico de la tuberculosis parte II: Cultivo. 2008. [ Links ]
5. Mattei R, Savarino A, Fabbri M, Moneta S, Tortoli E. Use of the BacT/Alert MB mycobacterial blood culture system for detection of mycobacteria in body fluids other than blood. J Clin Micribiol. 2009; 47:711-4. [ Links ]
6. Warren R, Gey van Pittius N, Barnard M, Hesseling A, Engelke E, de Kock M, et al. Differentiation of Mycobacterium tuberculosis complex by PCR amplification of genomic regions of difference. Int J Tuberc Lung Dis. 2006; 10:818-22. [ Links ]
7. Sambrook J, Russel D. Molecular cloning: A laboratory manual. Volumen 1. 3th ed. New York: Cold Spring Harbor Laboratory Press; 2001. [ Links ]
8. Telenti A, Marchesi F, Balz M, Bally F, Böttger E, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J Clin Microbiol. 1993; 31:175-8. [ Links ]
9. PRASITE. Identification of mycobacteria. [Online] 2007. Disponible en: http://app.chuv.ch/prasite/index.html. Acceso 11 de noviembre 2014. [ Links ]
10. Institut de Biologie/Institut Pasteur de Lille. MIRU-VNTRplus, web application. 2005. Disponible en: http://www.miru-vntrplus.org/MIRU/index.faces. Acceso 23 de mayo 2014. [ Links ]
11. Griffith D, Aksamit T, Brown-Elliot B, Catanzaro A, Daley C, Gordin F, et al. An official ATS/IDSA statement: Diagnosis, treatment and prevention of nontuberculous mycobacterial disease. Am J Res Crit Care Med. 2007; 175:367-416. [ Links ]
12. Solorzano A, Soria I, Roman J, Huertas P, Soto M, Piedrola G, et al. Comparative evaluation of three culture methods for the isolation of mycobacteria from clinical samples. Microbiol Biotechnol. 2009; 19:1259-64. [ Links ]
13. Martínez-Romero M, Sardiña-Aragón M, García-León G, Mederos-Cuervo L, Vega-Riverón B, Díaz-Rodríguez R. Evaluación del sistema automatizado BacT/ALERT 3D para el aislamiento de micobacterias en el LNRTB-IPK. Neumol Cir Torax. 2012; 71:333-8. [ Links ]
14. Naveen G, Basavaraj V. Comparison of the Lowenstein Jensen medium, the Middlebrook 7H10 medium and MB/BACT for the isolation of Mycobacterium tuberculosis from clinical specimens. J Clin Diagn Res. 2012; 6:1704-9. [ Links ]
15. Alfa M, Manickam K, Sepehri S, Sitter D, Lenton P. Evaluation of BacT/Alert 3D automated unit for detection of nontuberculous mycobacteria requiring incubation at 30 °C for optimal growth. J Clin Microbiol. 2011; 49:2691-3. [ Links ]