Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Tropical
versión impresa ISSN 0002-192X
Agronomía Trop. vol.64 no.3-4 Maracay dic. 2014
Asociación entre marcadores microsatélites y el gen de resistencia a la bacteriosis común en familias F2:4 de Phaseolus vulgaris L.
Rossmary Castañeda1*, Catalina Ramis2 y Delis Pérez1
1Instituto Nacional de Investigaciones Agrícolas, Laboratorio de Biotecnología Vegetal (INIA-Lara) y Banco de Germoplasma Leguminosas (INIA-CENIAP).
2Universidad Central de Venezuela, Facultad de Agronomía, Instituto de Genética. *Correo electrónico: rossmarych@gmail.com.
RESUMEN
La incidencia de enfermedades es uno de los factores que limita la producción de la caraota (Phaseolus vulgaris L.). La bacteriosis común de la caraota causada por Xanthomonas phaseoli (Xph) es la enfermedad bacteriana más importante del cultivo, reduce la producción de grano en un 45% y también afecta la calidad de la semilla. Es necesario incorporar genes de resistencia a través de programas de cruzamientos. El objetivo del presente trabajo fue verifi car la asociación entre marcadores moleculares tipo microsatélites (SSR) y el gen de resistencia a la bacteriosis común en familias F2:4 de caraota. La evaluación molecular se realizó mediante la amplifi cación de cebadores PV-ctt001, PV-atgc001, BM-140, BM-156 y BM- 202 que mostraron posible asociación con el carácter resistencia. Para la amplifi cación de SSR se utilizaron las condiciones estandarizadas en el Laboratorio de Genética Molecular, Facultad de Agronomía, Universidad Central de Venezuela. Se contrastó la segregación esperada de la población F2:4 para cada SSR con el programa MapDisto Genetics V 6.3. BM-140, el cual se comportó como marcador monomórfi co y no fue posible diferenciar entre familias resistentes a Xph y PV-atgc001 como un locus independiente. Existió asociación entre los SSR: BM-156, BM-202 y PV-ctt001 y el gen mayor de resistencia en las familias F2:4. La selección asistida hacia el alelo b utilizando esos SSR será de gran utilidad para identifi car plantas portadoras de la resistencia, y darle continuidad al programa de mejoramiento genético.
Palabras clave: Xanthomonas phaseoli, marcadores genéticos, mejoramiento genético, caraota.
Association between microsatellite markers and the resistance gene to common bacterial blight in F2:4 families of Phaseolus vulgaris L.
ABSTRACT
The incidence of disease is one of the factors limiting the production of black bean (Phaseolus vulgaris L.). The common bacterial blight of bean caused by Xanthomonas phaseoli (Xph) is the most important crop bacterial disease because reduces grain yield by 45% and also affects the quality of the seed. It is necessary to incorporate resistance genes through crossing programs. The object of this study was to verify the association between type microsatellite molecular markers (SSR) and the resistance gene to common bacterial blight in families F2:4 of black bean. The molecular evaluation was performed using the amplifi cation primers PV-ctt001, PV-atgc001, BM- 140, BM-156 and BM-202, that has previously showed a possible association with resistance. For amplifi cation of SSR, the standardized conditions in the Molecular Genetics Laboratory Agronomy Faculty, Central University of Venezuela were used. Expected segregation of F2:4 families for each SSR was verifi ed using MapDisto Genetics V 6.3 program. BM-140 behaved as a monomorphic marker and PV-atgc001 as an independent locus, so it was not possible to differentiate between resistant families using these markers. Signifi cant association between SSR: BM-156, BM-202 and PV-ctt001, and resistance controlled by a dominant gene in families F2:4 was found. Assisted selection to the b allele using these SSR will be useful to identify plants carrying the resistance in black bean breeding programs.
Key words: Xanthomonas phaseoli, genetic markers, plant breeding, black bean.
Recibido: 21/03/14 Aprobado: 25/03/15 Publicado: 31/03/17
INTRODUCCIÓN
La caraota (Phaseolus vulgaris L.), constituye la leguminosa de grano de mayor consumo en Venezuela. Representa uno de los alimentos básicos en la dieta del venezolano y es la principal fuente de proteína vegetal (Mekbib, 2003; Mwale et al., 2008).
Sin embargo, la incidencia de enfermedades es uno de los factores que limita la producción de este cultivo; siendo la bacteriosis común de la caraota, causada por Xanthomonas phaseoli (Xph), la enfermedad bacteriana más importante (Maselli et al., 2006) y una de las principales causas de los bajos rendimientos, lo que reduce la producción del grano hasta en un 45% y afectando además, la calidad de la semilla (Cruz et al., 2001; Mutlu et al., 2008; Nunes et al., 2008).
La resistencia genética se presenta como la alternativa de control económicamente efectiva y ecológicamente sustentable (Yu et al., 1998; Cruz et al., 2001; Santos et al., 2003; Cruz et al., 2004), especialmente cuando se combina con prácticas de cultivo que previenen el establecimiento de patógenos en el campo (Santos et al., 2003; Nunes et al., 2008).
Ante tal contexto, los marcadores moleculares asociados a genes de resistencia son una herramienta útil para la evaluación y selección de plantas. El primer paso de un programa de mejoramiento, mediante la selección asistida por marcadores moleculares (SAM), es la identifi cación de los marcadores de las regiones del genoma asociadas a tal resistencia.
Los genes que condicionan la resistencia a determinados aislamientos de Xph han sido reconocidos al emplear marcadores moleculares (Nodari et al.., 1993; Jung et al.., 1994; Miklas et al.., 1996; Park et al., 1996; Jung et al.., 1997; Lagarde et al.., 2010). Estos genes han logrado explicar y son responsables, en forma individual, del 18 al 53% de la variación fenotípica del carácter bajo estudio. Cuando se analizan en grupos de dos, estos valores oscilan entre 46 y 65%, mientras que en conjunto alcanzan hasta aproximadamente el 75% (Nodari et al.., 1993; Milklas et al.., 1996; Jung et al.., 1997; Salomón, 2002; Lagarde et al.., 2010).
Los microsatélites o secuencias simples repetidas (SSRs) son regiones de secuencias pequeñas (2 a 10 pares de bases) repetidas y arregladas en serie, las cuales se asume que están distribuidas al azar por todo el ADN. Se caracterizan por su hipervariabilidad, abundancia, reproducibilidad, herencia mendeliana simple, son multialélicos de naturaleza codominante y genomas eucariotas (Yu et al.., 2000; Gaitán et al.., 2002).
En este proceso, es importante la identificación de marcadores moleculares tipo SSRs asociados a la resistencia a Xph, ya que, permitirán a los programas de mejoramiento iniciados, la obtención de cultivares de caraota mejorados para tal característica, con mayor efi ciencia y eficacia.
Por tanto, en un programa de mejoramiento financiado por el proyecto del Banco Interamericano de Desarrollo y el Fondo Nacional de Ciencia, Tecnología e Innovación (BID-FONACIT) N° 26110 2004-000410, a través del Sub-proyecto Búsqueda de Marcadores Moleculares Asociados a la Resistencia a la Bacteriosis común en caraota, se realizaron cruzamientos usando parentales resistentes a la enfermedad, originando 9 poblaciones F2.
Las plantas F2 fueron caracterizadas y evaluadas con marcadores moleculares SSRs, donde cinco marcadores resultaron polimórfi cos; y se realizó el análisis de asociación entre los marcadores moleculares polimórfi cos y el gen de mayor resistencia identificado.
Se verificó el origen heterocigoto de las plantas F1 por medio de los SSR polimórficos y la segregación obtenida para los marcadores moleculares con la segregación esperada en la población F2 (Ramis et al., 2007). A partir de las familias F2:3 disponibles se obtuvieron las 98 familias F2:4, material utilizado en este estudio. El objetivo del presente trabajo fue verificar la asociación entre marcadores moleculares tipo microsatélites (SSR) y el gen de resistencia a la bacteriosis común en familias F2:4 de caraota.
MATERIALES Y MÉTODOS
La evaluación molecular se realizó en el Laboratorio de Genética Molecular del Centro de Investigaciones en Biotecnología Agrícola (CIBA) de la Facultad de Agronomía de la Universidad Central de Venezuela, Maracay, estado Aragua.
Material vegetal
Para el estudio se utilizaron 102 genotipos que incluían las familias F2:4 y los testigos. En un principio se trabajó con 104 familias, sin embargo no todas sobrevivieron, por lo que solo se extrajo ADN de 98 y se realizó la evaluación molecular de asociación para un máximo de 71.
Las familias F2:4 provenían del cruce entre el progenitor resistente XAN-154 y el susceptible MEM-0301013 a la bacteria Xph. Se utilizaron como testigos resistentes: XAN-154 y XAN-149, líneas avanzadas procedentes del Centro Internacional de Agricultura Tropical (CIAT) y conservados en el Banco de germoplasma de la Unidad de Recursos Fitogenéticos del Instituto de Genética, Facultad de Agronomía de la Universidad Central de Venezuela; y como testigos susceptibles MEM- 0301013 y Tacarigua, procedentes del Banco de germoplasma del INIA-CENIAP.
Extracción de ADN
El ADN fue obtenido a partir de hojas jóvenes no expandidas, de cada una de las familias F2:4 de caraota y testigos. El tejido fue macerado con nitrógeno líquido y se procedió a efectuar la extracción con tampón CTAB (2%), siguiendo la metodología de mini extracción de ADN de Gepts y Clegg (1989), Gepts y Koinange (1992); y Murray y Thompson (1980), empleadas en el Laboratorio de Genética Molecular (CIBA). La calidad y cantidad del ADN se verificó en geles de agarosa teñidos con bromuro de etidio. Las muestras de ADN fueron preparadas a una concentración final de 2,5 ng μL-1.
Evaluación molecular
Con base en los resultados de Ramis et al. (2007) se obtuvo el genotipo para cada planta de las familias F2:4 para los cebadores PV-ctt001, PV-atgc001, BM-140, BM-156 y BM-202 que mostraron posible asociación con el carácter resistencia cuando fue evaluado molecularmente el polimorfismo entre los progenitores del cruce simple, lo que permitió la obtención de las familias F2:4, (Cuadro 1).
Amplifi cación de cada SSRs a través de la técnica de PCR
En general se utilizaron las condiciones estandarizadas por Ramis et al. (2007), tanto para la mezcla de reacción de amplificación (Cuadro 2), como para los programas del termociclador. La temperatura de alineación (TM) varió según el SSRs a amplificar. En los Cuadros 3 y 4 se presentan los programas de PCR utilizados para cada SSRs. Los fragmentos se separaron en geles de poliacrilamida al 6% con revelación en nitrato de plata (Arnao, 2003).
Análisis estadístico
Con los perfiles electroforéticos visualizados para cada familia y microsatélite se preparó una matriz de genotipaje, donde se le asignó alelo(s) a cada familia (Figura 1). La segregación esperada de la población F2:4 para cada marcador molecular, se determinó a través del programa MapDisto Genetics V 6.3.
A fin de realizar el análisis de asociación de cada marcador con la resistencia a la bacteriosis común de la caraota, las plantas F2:4 se clasificaron en resistentes cuando el valor de la escala de evaluación estaba comprendida entre 1 y 4; y susceptibles para más de 5. En vista de que Ramis et al. (2007) demostraron que la resistencia es un carácter dominante, a las plantas se le asignó el genotipo R cuando eran resistentes y rr para las susceptibles; y se realizó un análisis para detectar los grupos de ligamiento empleando el mismo programa.
RESULTADOS Y DISCUSIÓN
Análisis de segregación
En el Cuadro 5 se presentan los genotipos para cada marcador SSRs y progenitor, así como la frecuencia de las 71 familias homocigotas: heterocigotas para cada marcador. Durante el proceso de endocría desde F2 hasta F4 no se realizó ninguna selección dirigida, por tal razón se espera que la frecuencia de genotipos para cada gen SSRs corresponda a la esperada para una población de líneas endocriadas.
El microsatélite BM-140 se comportó como marcador monomórfico, por lo que no fue posible diferenciar entre familias resistentes a Xph, tampoco se pudo realizar el análisis de asociación, ni seleccionar las plantas a partir del registro del marcador.
Las frecuencias de genotipos en las familias F2:4 no correspondió a la esperada 1:1; por el contrario, se obtuvo un exceso de homocigotas, hacia el progenitor XAN-154 (bb), con la excepción del SSR PV-atgc001 donde las homocigotas correspondieron al genotipo del progenitor MEM-0301013 (Cuadro 5). Estos resultados pudieran deberse a la presión de selección presente en las plantas F2.
Las plantas F2 fueron inoculadas con la bacteria Xph, la presión de selección ejercida ocasionó la muerte de numerosas plantas susceptibles, por lo que la descendencia obtenida, semillas F2:3, en su mayoría corresponderían a las de las plantas resistentes. De existir alguna asociación entre los SSRs y la resistencia, es de esperar la orientación hacia el progenitor resistente, para los alelos de los marcadores moleculares.
Es significativo señalar que la evaluación molecular de asociación se realizó para un máximo de 71; no con los 98 materiales, y posiblemente las proporciones esperadas no correspondan con las observadas (se rechaza la hipótesis nula de la segregación), tal vez por el número de materiales evaluados.
Es importante señalar que se identificaron familias con presencia de plantas heterocigotas, lo cual persiste en etapas tempranas de endocría. A nivel del programa de mejoramiento, se señala que se debe seguir avanzando hasta llegar a unmayor nivel de homocigosis, y entonces realizar la selección individual de plantas; finalmente obtener una línea experimental homocigota de alta resistencia y rendimiento, para iniciar el proceso de evaluación en distintas localidades.
con la resistencia
La asociación entre marcadores moleculares y características agronómicas, para poblaciones segregantes con menos de 100 familias, ha sido realizada por varios autores en caraota. Santos et al. (2003) para complementar un mapagenético del frijol común basado en el cruce entre un genotipo susceptible HAB-52 y un genotipo resistente BAC-6 utilizaron 88 plantas F2 para mapear y analizar regiones genómicas relacionadas con la resistencia a Xanthomonas.
El análisis de ligamiento se llevó a cabo por el Software MAPMAKER / EXP usando 143 marcadores RAPD, lo que genera un mapa que se utiliza para detectar QTL asociados con Xanthomonas en caraota.
Igualmente, Lara (2012) realizó la búsqueda de un marcador molecular del gen circumlineatusen semillas de Phaseolus vulgaris para su incorporación en el mapa genético del frijol,empleando 12 plantas de genotipos contrastantesen poblaciones segregantes hasta F5; siendo analizados usando PCR con 57 microsatélites, entre los que se encontraban BM-156 y BM-202
Los Cuadros 6 y 7 presentan los valores de la fracción de recombinación (r) y los de Lod Score obtenidos entre los marcadores SSRs y el gen mayor de resistencia, respectivamente. Los resultados obtenidos confi rman la asociación entre los SSRs BM-156, BM-202 y PV-ctt001. A pesar de haber evaluado 71 familias, se detectó asociación entre esos marcadores y el gen mayor de resistencia a Xph, lo que corrobora la utilidad de los SSR como una herramienta en los programas de mejoramiento para la resistencia.
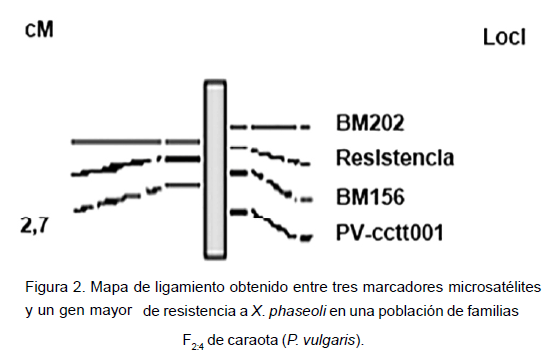
Los SSRs BM-156, BM-202 y PV-ctt001 se encuentran ligados al gen de resistencia.No así con el marcador PV-atgc001 que se comportó como un locus independiente. Resultados similares obtuvieron Ramis et al. (2007), encontrando un valor r de 0,10 entre los marcadores BM-156 y BM-202.
Con respecto a la resistencia, Ramis et al. (2007) lograron detectar la asociación con el marcador Pv-ctt001 con un valor de r de 0,41. En esta oportunidad, y en consecuencia a la selección realizada por la inoculación con la bacteria Xph en las plantas F2, se incrementó la frecuencia de plantas resistentes y por lo tanto la mayor expresión de la asociación.
El valor del Lod Score en el análisis de asociación entre una característica y un marcador molecular, se puede interpretar como la probabilidad que los datos observados se derivan de un QTL presente de uno ausente. Si el valor de Lod Score sobrepasa un umbral, generalmente entre 2 y 3, es un indicio de que un QTL está en el marcador molecular evaluado.
En el Cuadro 7, los valores para la asociación entre el gen de resistencia y los SSR BM-156, BM-202 y PV-ctt001, fueron de 8,49; 7,48 y 5,89; respectivamente. Esto es importante debido a que los valores de Lod Score siempre fueron mayores a 5, teniendo en cuenta que 3 es el valor mínimo de Z que se requiere para poder afi rmar que existe ligamiento signifi cativo entre dos loci. Existe una relación de ligamiento entre la resistencia a Xph y los SSRs BM-156, BM-202 y PV-ctt001; indicando que los loci están suficientemente próximos entre sí, de forma que no se produce recombinación entre ellos en cada división meiótica. Garzón (2012) y Herrera (2011), en sus trabajos utilizando SSR en Phaseolus vulgaris hallaron a BM-156 ubicado en el grupo de ligamiento B02; también reportan que el BM-202 no ha sido mapeado por lo que no presenta anclaje a un grupo de ligamiento.
En la Figura 2 se presenta gráficamente el grupo de ligamiento para los SSRs BM-156, BM-202 y PV-ctt001 asociados al gen de la resistencia Xph, el cual se puede utilizar para identificar familias que porten el alelo b (XAN-154) en estado homocigota para los tres microsatélites. Dichas familias pueden escogerse mediante la selección asistida con marcadores (SAM).
CONCLUSIONES
Se verificó la asociación entre microsatélites (SSRs): BM-156, BM-202 y PV-ctt001 y el gen mayor de resistencia a Xph, en familias avanzadas F2:4 de caraota (Phaseolus vulgaris L.); los SSR BM-156, BM-202 y PV-ctt001 se encuentran ligados al gen de resistencia. La selección asistida utilizando los SSRs BM-156, BM-202 y PV-ctt001 hacia el alelo b, será de gran utilidad para identifi car familias portadoras de la resistencia con las cuales continuar el programa de mejoramiento, escogiendo plantas que porten el alelo b en estado homocigota para los tres microsatélites.
LITERATURA CITADA
1. Arnao, E. 2003. Aplicación de marcadores moleculares en la recuperación del padre recurrente en el mejoramiento por retrocruza para la resistencia a Pyricularia grisea (Cooke) Sacc. Trabajo de grado para optar al título de magister scientiarum. Postgrado en Agronomía. Universidad Central de Venezuela. Maracay, Venezuela. 66 p. [ Links ]
2. Cruz, S., P. Ramírez, R. García, F. Castillo y J. Sandoval. 2004. Selección para la resistencia al tizón común en frijol. Revista Fitotecnia Mexicana. 27(2):141-147. [ Links ]
3. Cruz, S., P. Ramírez, B. Tlapal, I. Ramírez, R. García, J. Sandoval y F. Castillo. 2001. Producción masiva de Xanthomonas axonodopis pv. phaseoli (Smith). Agrociencia. 35:575-581. [ Links ]
4. Gaitán, E., M. Duque, K. Edwards and J. Tohme. 2002. Microsatellite Repeats in Common Bean (Phaseolus vulgaris): Isolation, Characterization, and Cross-Species Amplification in Phaseolus ssp. Crop Science. 42:2128-2136. [ Links ]
5. Garzón, L. 2012. Homólogos de genes de resistencia en fríjol (Phaseolus vulgaris) y su aplicación en resistencia a Colletotrichum lindemuthianum. Trabajo de grado para optar al Título de doctor en Ciencias Agropecuarias, área agraria con énfasis en Fitopatología. Universidad Nacional de Colombia Facultad de Agronomía, Escuela de posgrados Bogotá, Colombia. 217 p. [ Links ]
6. Gepts, P. and E. Koinange. 1992. Hybrid Weakness in Wild Phaseolus vulgaris L. The Journal of Heredity. 83(2):135-139. [ Links ]
7. Gepts, P. and M. Clegg. 1989. Genetic Diversity in Pearl Millet (Pennisetum glaucum) [L.] R. Br.) at the DNA Sequence Level. The Journal of Heredity. 80(3):203-208. [ Links ]
8. Herrera, N. 2011. Saturación del mapa genético de una población intra-acervo de fríjol (Phaseolus vulgaris L.) empleando marcadores moleculares tipo SSR y SSCP. Trabajo de grado para optar al título de bióloga. Universidad del Quindío. Facultad de Ciencias Básicas y Tecnologías. Colombia. 122 p. [ Links ]
9. Jung, G., P. Skroch, D. Coyne, J. Nienhuis, E. Arnaud, H. Ariyarathne, S. Kaeppler and M. Basset. 1997. Molecular-marker based genetic analysis of tepary bean derivedcommon bacterial blight resistance in different developmental stages of common bean. Journal of the American Society for Horticultural Science. 122(3):329-337. [ Links ]
10. Jung, G., D. Coyne, P. Kroch, J. Nienhuis, E. Arnal, J. Bokosi, S. Kaeppler, and J. Steandman. 1994. Construction of a genetic linkage map and location of common blight, rust resistance and pubescence loci in Phaseolus vulgaris L. using amplified polymorphic DNA (RAPD) markers. Bean Improvement Cooperative. 37:37-38. [ Links ]
11. Lagarde, P., A. Medina, C. Ramis y A. Maselli. 2010. Evaluación de la resistencia a la bacteriosis común causada por Xanthomonas phaseoli en plantas F3 de caraota (Phaseolus vulgaris). Fitopatología Venezuela. 23(2):35-39. [ Links ]
12. Lara, L. 2012. Evaluación del gen circumlineatus en la semilla de frijol común (Phaseolus vulgaris) con marcadores moleculares microsatélites. Proyecto especial de graduación del programa de Ingeniería Agronómica. Escuela Agrícola Panamericana. Zamorano, Honduras. 17 p. [ Links ]
13. Maselli, A., L. Rosales y Y. Guevara. 2006. Uso de extractos vegetales sobre Xanthomonas phaseoli, causante de la quemazón en Phaseolus vulgaris L. CENIAP HOY, N° 12. [ Links ]
14. Mekbib, F. 2003. Yield stability in common bean (Phaseolus vulgaris L.) genotypes. Euphytica. 130:147-153. [ Links ]
15. Miklas, P., E. Johnson, V. Stone, J. Beaver, C. Montoya and M. Zapata. 1996. Selective mapping of QTL conditioning disease resistance in common bean. Crop Science. 36:1344-1351. [ Links ]
16. Mutlu, N., K. Vidaver, D. Coyne, J. Steadman, P. Lambrecht and J. Reiser. 2008. DifferentialPathogenicity of Xanthomonas campestris pv. phaseoli and X. fuscans subsp. fuscans Strains on Bean Genotypes with CommonBlight Resistance. Published by The American Phytopathological Society. Plant Disease. 92(4):546-554. [ Links ]
17. Murray, M. and W .Thompsom. 1980. Rapid isolation of high molecular weight DNA. Nucleic Acids Reporter. 8:4321-4325. [ Links ]
18. Mwale, M., J. Bokosi, C. Masangano, M. Kwapata, V. Kabambe and C. Miles. 2008. Yield performance of dwarf bean (Phaseolus vulgaris L.) lines under Researcher Designed Farmer Managed (RDFM) system in three bean agro-ecological zones of Malawi. African Journal of Biotechnology. 7(16):2847-2853. [ Links ]
19. Nodari, R., S. Tsai, P. Guzman, R. Gilbertson and P. Gepts. 1993. Towards an integratedlinkage map of common bean. Mapping genetic factors controlling host-bacteria interactions. Genetics. 134:341-350. [ Links ]
20. Nunes, W., M. Corazza, S. Dias, S. Mui and E. Kuramae. 2008. Characterization of Xanthomonas axonopodis pv. phaseoli isolates. Summa Phytopathologica. Botucatu, Brasil. 34(3):228-231. [ Links ]
21. Park, S., A. Dursun, D. Coyne and G. Jung. 1996. Identification of RAPD markers linked to major genes for common bacteria. Annual Report Bean Improvement Cooperative. 39:128-129. [ Links ]
22. Ramis, C., A. Medina, A. Maselli, M. Pérez, O. Movil, L. Salazar, J. Jiménez, A. Bedoya, M. Gutiérrez, D. Pérez y M. Gutierrez. 2007. Informe final del subprograma: Búsqueda de marcadores moleculares asociadosa la resistencia a la bacteriosis común (Xanthomonas phaseoli) en caraota. BID-FONACIT II; Instituto Nacional de Investigaciones Agrícolas INIA-CENIAP, Laboratorio de Bacteriología; Universidad Central de Venezuela, Facultad de Agronomía, Centro de Investigación en Biotecnología Agrícola (CIBA). Maracay, Venezuela. [ Links ]
23. Santos, A., R. Bressan, M. Pereira, R. Rodrigues and C. Ferreira. 2003. Genetic Linkage Map of Phaseolus vulgaris and Identification of QTLs Responsible for Resistance to Xanthomonas axonopodis pv. phaseoli. Fitopatología Brasileira (jan-fev). 28(1):5-10. [ Links ]
24. Salomón, J. 2002. Evaluación y selección de familias F2 en F3 de caraota (Phaseolus vulgaris L.) por su reacción a Xanthomonas axonopodis pv. phaseoli. Trabajo de grado para optar al título de Magister Scientiarum. Postgrado en Agronomía. Universidad Central de Venezuela. Maracay, Venezuela. 70 p. [ Links ]
25. Yu, K., S. Park, V. Poysa and P. Gepts. 2000. Integration of Simple Secuence Repeat (SSR) Markers Into a Molecular Linkage Map of Common Bean (Phaseolus vulgaris L.) The American Genetic Association. 91:429-434. [ Links ]
26. Yu, Z., R. Stall and C. Vallejos. 1998. Detection of genes for resistance to common bacterial blight of beans. Crop Science. 38:1290-1296. [ Links ]