Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Tropical
versión impresa ISSN 0002-192X
Agronomía Trop. vol.64 no.3-4 Maracay dic. 2014
Marcadores bioquímicos y moleculares asociados a la tolerancia al estrés hídrico en arroz
Gelis T. Torrealba-Núñez1*, Iris Pérez-Almeida2, Catalina Ramis3, Nelly Delgado4, Luis Angulo3, Yreny K. De Faria3, Carlos Marín2, Oralys C. León Brito5 y Getssy C. Martínez Zapata1
1Instituto Nacional de Investigaciones Agrícolas (INIA-Guárico),
2Centro Nacional de Investigaciones Agropecuarias (CENIAP),
3Universidad Central de Venezuela (UCV), Maracay, estado Aragua.
4INIA-Portuguesa,
5INIA-Monagas. *Correos electrónicos: gelistorrealbaarroz@gmail.com, gtorrealba@inia.gob.ve
RESUMEN
En Venezuela, la sequía se ha convertido en un factor limitante para la producción arrocera nacional. Las variedades cultivadas tradicionalmente están adaptadas a una alta suplencia de agua. Esta investigación tuvo como propósito identificar marcadores bioquímicos (isoenzimas) y moleculares (microsatélites SSR) asociados a la tolerancia al estrés hídrico en arroz (Oryza sativa L.). El estudio de cartografía génica fue realizado en una población RC1F2 producto del cruce (LO2 x Fonaiap 2000) x LO2, empleando el programa MapDisto y el método de un marcador a la vez. Se identificaron 89 caracteres cuantitativos (QTL) putativos asociados a las características de la fase vegetativa, reproductivas y de rendimiento; usando cinco isoenzimas (PRX, G6PDH, ACP, 6PGD, SDH) y 40 (SSR) se estimaron los índices de susceptibilidad a la sequía (ISS). Seguidamente, se identifi có la asociación entre las isoenzimas, la tolerancia al estrés hídrico y los ISS en las familias de la población segregante, para variables relacionadas a la fase vegetativa, reproductiva y de maduración del grano de arroz. En los 2 grupos de ligamiento (cromosoma 3 y 9) se detectaron 4 QTL asociados a los ISS, 2 regiones asociadas con la floración al 50%, 1 para el desgrane (cromosoma 3) y 1 para la fertilidad (cromosoma 9) en RM107, RM442 y RM570. Esta información puede ser empleada como criterio en la selección asistida por medio de marcadores moleculares, en los futuros programas de mejoramiento genético del arroz, a fin de identificar familias y genotipos tolerantes a la condición de estrés hídrico.
Palabras clave: Oryza sativa L., sequía, producción, isoenzimas, microsatélites.
Biochemists and molecular markers associated with water stress tolerance in rice
ABSTRACT
Drought in Venezuela has become a limiting factor for national rice production, as the cultivated varieties traditionally are adapted to high water supply. This study aimed to identify biochemical (isozymes) and molecular markers (microsatellites, SSR) associated with water stress tolerance in rice (Oryza sativa L.). Gene mapping study was conducted in a BC1F2 population product of the crossing (LO2 x Fonaiap 2000) x LO2 using the MapDisto program and the method of one marker at a time. Eighty-nine putative quantitative characters (QTLs) associated with vegetative, reproductive and yield traits were identifi ed. Using five isozymes (PRX, G6PDH, ACP, 6PGD and SDH) and 40 SSR drought susceptibility indexes (ISS) were estimated. Next, the association between isoenzymes, water stress tolerance and ISS in the families of the segregating population were identified for variables related to the vegetative, reproductive and maturation of the grain phase in rice. In the 2 linkage groups (chromosome 3 and 9) 4 QTL associated to ISS, 2 regions associated with 50% flowering, 1 for threshability (chromosome 3) and 1 for fertility (chromosome 9) were detected in RM107, RM442 and RM570. This information can be used as a criterion in molecular marker assisted selection in future rice breeding programs in order to identify families and genotypes tolerant to the water stress condition.
Key words: Oryza sativa L., drought, production, Isozymes, microsatellite.
Recibido: 07/05/14 Aprobado: 15/02/16 Publicado: 31/03/17
INTRODUCCIÓN
La escasez de agua es uno de los grandes retos que afronta la humanidad, elemento muy abundante en el planeta Tierra. Sin embargo, el 97% del total está formada por agua salada perteneciente a mares y océanos, el restante 3% formado por agua dulce, está poco disponible, porque se encuentra congelada (Cortés, 2004).
La agricultura es la actividad de mayor consumo de agua dulce del planeta, utilizando aproximadamente el 70% del agua potable. Algunos países en desarrollo destinan cerca de 90% del líquido a esta importante actividad. Se estima que para producir un kilo de arroz se necesitan de 1 a 3 m3 de agua, de allí la importancia de un manejo sostenible de los recursos hídricos, para asegurar el abastecimiento de alimentos y mitigar los efectos del cambio climático (FAO, 2002).
En Venezuela, la sequía se ha convertido en un factor limitante para la producción arrocera nacional, las variedades de arroz cultivadas tradicionalmente están adaptadas a una alta suplencia de agua. Sin embargo, instituciones del país como el Instituto Venezolano de Investigaciones Científicas (IVIC), la Universidad Central de Venezuela (UCV), y el Instituto Nacional de Investigaciones Agrícolas (INIA) a través de investigadores en el área, han realizado estudios para conocer la respuesta de los diferentes genotipos de arroz frente al déficit hídrico (Ghneim et al., 2005; Ghneim-Herrera 2006; Pieters y Núñez 2008; García 2008 y Torrealba et al., 2010).
La tolerancia a la sequía es una característica compleja, expresada por la acción e interacción de diferentes caracteres: morfológicos (sistema radical eficiente, reducción del macollamiento, entre otros); y comportamientos fisiológicos (reducción de la transpiración, alta eficiencia en el uso del agua, cierre estomático, ajuste osmótico), controlados por numerosos genes. La obtención de cultivares de arroz tolerantes a la sequía es un objetivo claro de los programas de mejoramiento genético, como alternativa para el aumento y mantenimiento de la productividad nacional del arroz, como rubro estratégico para la alimentación nacional.
En vista de la complejidad del control genético de las características asociadas a la tolerancia al estrés por déficit hídrico, se ha propuesto la aplicación de la selección asistida por marcadores moleculares (SAM) para una mayor eficacia y eficiencia del proceso de selección.
Los marcadores moleculares son secuencias de ADN que se encuentran en determinados lugares del genoma; y se defi nen como características heredables que permiten clasificar el genotipo de un individuo, identificando regiones del genoma asociadas con un carácter fenotípico cuantitativo específi co (QTL). La identificación del QTL es de gran utilidad en plantas, ya que permite la manipulación de características agronómicas como el rendimiento, la calidad nutricional y la floración, entre otros (Grandillo y Tanksley, 1996). Las bases de detección de los QTL son la identificación de las asociaciones entre fenotipos determinados genéticamente y marcadores genéticos específicos (McCouch y Doerge, 1995).
Teng et al. (2002) empleando una variedad indica (Zhaile Ye Quing8), una japónica (Jing Xi 17) y sus dobles haploides, lograron identificar 2 QTL: qDR-5 y qDR-12 para tolerancia a la sequía en arroz. Más recientemente, Vikram et al. (2011) encontraron un QTL (qDTY 1.1) asociado a la tolerancia a la sequía en cuanto a la producción de grano ubicado en el cromosoma 1, flanqueado por los microsatélites RM11943 y RM431, de efecto mayor y consistente en cuanto a su expresión en diferentes ambientes.
Aunque no se han identificado isoenzimas asociadas a características cuantitativas relacionadas con la tolerancia al défi cit hídrico, se espera obtener cierta relación con distintas isoformas de enzimas vitales para la producción de granos. El grupo de isoenzimas de la glutamina sintetasa (GS) son responsables de la asimilación y movilización dentro de la planta de las distintas fuentes de nitrógeno (N). Existen formas de GS, tanto en cloroplastos como en citosol; en el caso del arroz se conocen tres isoformas del citosol.
Bajo estrés por déficit hídrico, tanto las isoenzimas de GS de cloroplasto como del citosol son afectadas, lo que disminuye la tasa de asimilación de N en forma de amonio, y en consecuencia al llenado de granos (Swarbreck et al., 2011). Se espera que las plantas con alguna isoforma más tolerante al estrés hídrico, permita a la planta un mejor comportamiento bajo esas condiciones, evidenciado en un mayor rendimiento.
Por otra parte, Lee et al. (2007), propusieron que las enzimas relacionadas al proceso de lignificación, responsables de los mecanismos de tolerancia a diferentes estrés, son activadas por el estrés causado por el déficit hídrico. En ese sentido, encontraron en trébol que las enzimas peroxidasas eran impulsadas bajo condiciones de sequía, con reducción de la biomasa foliar. En vista de la importancia del cultivo de arroz para el país y el uso eficiente del recurso hídrico, el objetivo del presente estudio fue identifi car marcadores bioquímicos y moleculares asociados a la tolerancia al estrés hídrico en arroz con fines de selección.
MATERIALES Y MÉTODOS
La identificación de QTL asociados a caracteres fenotípicos relacionados a la tolerancia al déficit hídrico se llevó a cabo en cuatro etapas, que comprenden: la creación de una población segregante a partir del cruce entre progenitores contrastantes en cuanto al comportamiento fenotípico; la evaluación fenotípica de la población segregante obtenida bajo condiciones con o sin estrés por défi cit hídrico (Torrealba, 2010; Torrealba et al., 2010); la selección de marcadores polimórfi cos entre los dos progenitores y; finalmente, el estudio de segregación y asociación de los marcadores con los caracteres fenotípicos.
Estas dos últimas etapas, correspondientes al análisis molecular, se llevaron a cabo en el Laboratorio de genética molecular del Centro de Investigaciones en Biotecnología Agrícola (CIBA) de la Universidad Central de Venezuela, Facultad de Agronomía, núcleo Maracay.
Material vegetal
Se empleó una población RC1F2 del cruce (LO2 x Fonaiap 2000) x LO2, conformada por 149 familias, desarrollada por Torrealba (2010) en el campo experimental de INIA Guárico, Bancos de San Pedro, bajo la metodología de Torrealba et al. (2010), considerando las características asociadas al estrés hídrico, señaladas por Babu et al. (2002), Toorchi et al. (2002), Courtois y Lafitte (2003). Esto permitió escoger dos progenitores contrastantes en su respuesta al estrés hídrico, medida a través del ISS de Fischer y Maurer (1978) para enrollamiento de la hoja, secado de la hoja y fertilidad de las espiguillas.
Se consideraron variables de crecimiento vegetativo y reproductivo para determinar los ISS (Torrealba, 2010): 1. altura de planta; 2. exerción de la panícula; 3. ácame; 4. enrollamiento de la hoja; 5. secado de la hoja; 6. longitud de la panícula; 7. promedio de panículas/planta; 8. número de hijos/planta; 9. contenido relativo de agua en las hojas; 10. temperatura de la hoja; 11. coeficiente de clorofila; 12. índice de área foliar; 13. longitud de las raíces; 14. volumen radical; 15. relación parte aérea y parte radical; 16. biomasa; 17. peso seco parte aérea; 18. peso seco de raíces; 19. floración al 50%; 20. floración como relación granos llenos y granos vanos; 21. senescencia; 22. fertilidad; y 23. aceptación fenotípica.
Con relación al rendimiento, se tomó en cuenta: 1. desgrane de la panícula; 2. número de granos llenos; 3. número de granos vanos; 4. peso de granos llenos; 5. peso de granos vanos; 6. peso de 1000 g; 7. índice de cosecha; y 8. rendimiento (g 5 m-2).
Evaluacion molecular
Se evaluó el polimorfismo entre los progenitores utilizando 15 sistemas de marcadores isoenzimáticos (SMI) y 258 SSR distribuidos en todo el genoma. Para los SMI se utilizaron plúmulas de 4 a 15 días de edad, de acuerdo a la metodología empleada en arroz por Ortiz et al. (2002), abreviatura y código por Manchenko (1994).
Los marcadores fueron los siguientes: (1) Glucosa-6-fosfato isomerasa (GPI: 5.3.1.9); (2) glucosa-6-fosfato deshidrogenasa (G6PDH: 1.1.1.49); (3) 6-fosfogluconato deshidrogenasa (6PGD: 1.1.1.44); (4) fosfatasa ácida (ACP: 3.1.3.2); (5) alfa-esterasa (αEST: 3.1.1); (6) beta-esterasa (βEST: 3.1.1); (7) glutamato deshidrogenasa (GDH: 1.4.1.2); (8) malato deshidrogenasa (MDH:1.1.1.37); (9) enzima málica (ME: 1.1.1.40); (10) fosfoglucosa Isomerasa D-glucosa-6-fosfato (PGI: 5.3.1.9); (11) peroxidasa (PRX: 1.11.1.7); (12) siquimato deshidrogenasa (SDH: 1.1.1.25); (13) alcohol deshidrogenasa (ADH: 1.1.1.1); (14) isocitrato deshidrogenasa (IDH: 1.1.1.42); y (15) fosfoglucomutasa (PGM: 2.7.5.1), en un sistema discontinuo de electroforesis en geles de policrilamida (6/10%) bajo condiciones nativas (PAGE), siguiendo la metodología descrita por Ortiz et al. (2004) y Velásquez (2001).
Los SSR fueron seleccionados de la base de datos de GRAMENE (McCouch et al., 2002) y Plantmarkers (Rudd et al., 2005).
Seguidamente, a fines de realizar la mini preparación de ADN y visualizado en geles de agarosa al 0,8% teñido con bromuro de etidio (10 mg ml-1) el ADN fue obtenido siguiendo el método de Zambrano et al. (2002), cuantificando la cantidad de ADN a partir del Fago Lambda de concentración conocida. La reacción en cadena de polimerasa (PCR), los ciclos en el termociclador MJ Research PTC-200 y la tinción de los geles de poliacrilamida al 6%, fueron realizadas siguiendo la metodología descrita por Pérez-Almeida et al. (2011).
Análisis estadístico
Empleando el programa MapDisto, versión 1.7 (Lorieux, 2008), se procedió a verifi car la segregación mendeliana (1:1), correspondiente a una retrocruza, para cada marcador molecular y la información genotípica de las 149 familias RC1F2. Asimismo, con este programa se realizó el mapeo y la identificación de los grupos de ligamiento a partir de los alelos presentes de cada genotipo para cada marcador molecular polimórfico.
Fue utilizado el método de mapeo para un marcador a la vez, como lo indica Xiao et al. (1998); Thomson et al. (2003); Septiningshigh et al. (2003); Wan et al. (2005); Lee et al. (2005) y Praddep et al. (2005). Este método fue empleado por el bajo número de marcadores polimórficos detectados entre los progenitores. Se consideró la existencia de un QTL y la asociación entre el valor fenotípico de cada ISS de las 31 variables evaluadas por Torrealba (2010) y el locus de cada marcador molecular, para un valor mínimo de LOD Score de 3,0 y fracción de recombinación de 0,35.
Nomenclatura para QTL
Los caracteres cuantitativos (QTL) fueron nombrados de acuerdo a la nomenclatura descrita por McCouch et al. (1997), utilizando dos letras abreviadas de las características de estudio, seguida por el número del cromosoma donde fue detectado el QTL.
RESULTADOS Y DISCUSIÓN
Evaluación del polimorfismo
De los 15 sistemas de marcadores isoenzimáticos sólo 7 resultaron polimórficos: G6PDH: 1.1.1.49; 6PGD: 1.1.1.44; ACP: 3.1.3.2; αEST: 3.1.1; βEST: 3.1.1; PRX: 1.11.1.7 y SDH: 1.1.1.25. De esos marcadores se seleccionaron: ACP, PRX, G6PDH, 6PGD y SDH, por su resolución, calidad de los geles y visualización de las bandas obtenidas. En la evaluación del polimorfismo de los SSR, 66 fueron polimórficos; de los cuales se utilizaron 52 por sus características de discriminación entre los progenitores (Cuadros 1a y 1b).
Verificación de la segregación mendeliana
De los 57 marcadores moleculares evaluados en la prueba de bondad de ajuste de Ji-cuadrado (Cuadro 2), sólo dos no segregaron en las proporciones esperadas para una retrocruza (1:1). La distorsión en la segregación es muy común en los estudios de mapeo, señalada para la mayoría de los marcadores genéticos, como: desviaciones estadísticas; tipo de población de estudio; causas biológicas, como irregularidades durante la meiosis, competencia entre las gametas durante la fertilización o incompatibilidad genética; y por la cercanía de los marcadores al centrómero (Pradeep et al., 2005; Semagn et al., 2006; Zhao et al., 2006).
Mapeo y grupos de ligamiento
o se identificó ningún grupo de ligamiento con los marcadores isoenzimaticos, mientras que seis de los SSR pudieron ser asignados en tres grupos. El primer grupo de ligamiento estuvo conformado por los marcadores RM278 y RM107; el segundo por RM201 y RM309 y el tercero por los marcadores RM570 y RM442.
Asociación entre marcadores moleculares y variables del estrés hídrico
Se identificaron 89 QTL putativos asociados a las características de la fase vegetativa, reproductiva y rendimiento (Cuadro 3). Es importante destacar la presencia de ocho asociaciones entre los marcadores bioquímicos y los índices de susceptibilidad a la sequía (ISS): G6PDH al coeficiente de clorofila y peso seco (radical); ACP y el índice de área foliar; 6PGD para la fertilidad, número de granos vanos y peso seco (radical); y SDH al peso de 1000 g.
Con respecto a la peroxidasa (PRX) se encontró asociación con el contenido relativo de agua en las hojas y floración 50%, coincidiendo con lo planteado por Lee et al. (2007) en vista de su intervención en el proceso de lignifi cación como una respuesta al estrés por déficit hídrico. Esto indica la importancia del uso de este tipo de marcador en la identificación de caracteres cuantitativos. Con 40 SSR se detectaron 65 QTL putativos para las variables evaluadas. El RM231 (cromosoma 3) mostró asociación para la excersión de la panícula y enrollamiento de la hoja; RM290 (cromosoma 2) para las variables enrollamiento de las hojas y el contenido relativo de agua en las hojas. Lafitte et al. (2004) en su evaluación de líneas recombinantes de arroz detectaron QTL asociados al contenido relativo de agua en las hojas y su correlación a las características de floración y radiación solar; así como, la correlación entre las variables de la fase vegetativa, altura de la planta y rendimiento en grano en condiciones de estrés hídrico.
Se asoció la variable volumen radical con 7 microsatélites en distintos cromosomas: RM17 (cromosoma 12); RM220 y RM575 (cromosoma 1); RM324 (cromosoma 2); RM303 (cromosoma 4); RM336 y RM25 de los cromosomas 7 y 8, respectivamente. La característica longitud de las raíces fue asociada a 4 SSR: RM220 (cromosoma 1) altamente signifi cativo, RM324 (cromosoma 2) altamente signifi cativo, RM336 (cromosoma 7) y RM25 (cromosoma 8).
Del mismo modo, fueron detectadas asociaciones entre las variables de la fase reproductiva del arroz; por ejemplo, la variable senescencia fue altamente signifi cativa para los microsatélites RM17 (cromosoma 12) y RM539 (cromosoma 6) mostrando este último, asociación significativa con las características de aceptación fenotípica y fertilidad.
La variable floración 50% fue asociada con los microsatélites: RM259 (cromosomas 1); RM71 (cromosoma 2); RM55, RM442 y RM570 (cromosoma 3) y RM336 del cromosoma 7. Se señala que la floración es un indicador de la respuesta de la planta a la condición de estrés hídrico, así como el contenido relativo de agua en la hoja. Fue detectado también una región en el cromosoma 1 del arroz (gen sd1) asociada al estrés hídrico para el contenido relativo de agua en la hoja y correlacionada a la floración; dos QTL asociados a la floración; 1 QTL a la altura de la planta y otro (RM212) a longitud de la panícula (Lafi tte et al., 2004).
Seguidamente, fue determinada una asociación altamente significativa para el desgrane en los SSR RM20 (cromosoma 12) y RM442 (cromosoma 3); número de granos por planta, RM277 (cromosoma 12) y el peso de granos llenos por planta. Hubo asociación para el número de granos por planta y el peso de granos por planta: RM220 (cromosoma 1), RM277 (cromosoma 12) y RM450 (cromosoma 2).
Los resultados sugieren la importancia de incorporar algunas técnicas de biología molecular como herramienta en los programas de mejoramiento genético, para el estudio de características asociadas a la tolerancia al estrés hídrico, señalado por Mackill (2003).
En el presente estudio se detectaron 89 QTL individuales, 54 relacionados con la fase vegetativa (60,67%), 14 para la fase reproductiva (15,73%) y 21 al rendimiento (23,60%), los cuales son comparables con los resultados obtenidos por Yue et al. (2005). Se detectaron, además un total de 39 QTL individuales para las características relacionadas con el índice de respuesta a la sequía: área foliar, longitud de las raíces (fase vegetativa); floración, fertilidad (fase reproductiva del cultivo) y potencial de rendimiento en arroz; en una población de 180 familias híbridas recombinantes.
El ISS empleado para la identificación de los QTL, mostró ser un indicador preciso para reconocer la habilidad intrínseca de los genotipos de las familias RC1F2, para superar el estrés por el défi cit de agua, bajo las condiciones del cultivo en campo y las bases genéticas del estrés a la sequía. RM220, RM259 y RM575 fueron los marcadores más asociados a los índices de susceptibilidad a la sequía.
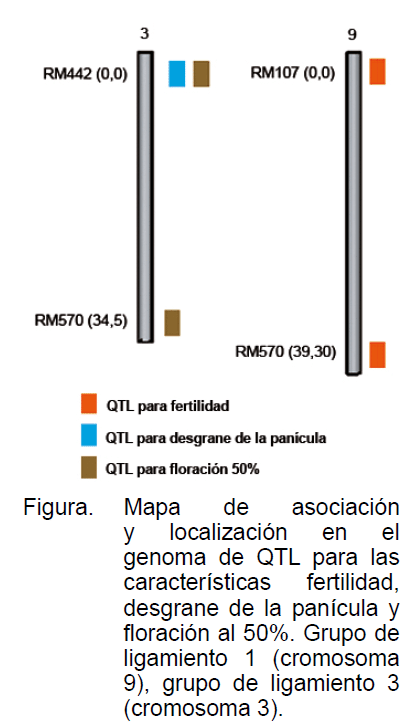
En el análisis de QTL detectados por grupos de ligamientos, se identificaron cuatro asociaciones, una en el cromosoma 9 y tres regiones en el cromosoma 3, para RM107, RM442, RM570 y las variables de fertilidad, desgrane de la panícula y floración al 50% (Figura y Cuadro 4).
Las características de desgrane y floración al 50% fueron altamente significativas en la asociación con los SSR del cromosoma 3. El valor de la fracción de recombinación para los marcadores microsatélites RM107 y RM278 fue 0,328 y 0,299 para RM442 y RM470. Los valores de LOD Score en ambos grupos de ligamiento fueron mayores a 3, lo cual es un indicativo de la significancia del análisis realizado, demostrado por Thomson et al. (2003) y Septiningshigh et al. (2003) para los criterios del análisis de asociación entre marcadores moleculares y caracteres cuantitativos.
Se identificaron 2 regiones en el cromosoma 3 asociadas con floración al 50% para RM442 y RM570, como lo reportado por Lanceras et al. (2004). La distancia entre RM442 y RM570 fue de 34,5 cm; entre RM107 y RM278 fue de 39,3 cm; en ambos casos menores a 50 cm, lo cual es un indicativo de que estos marcadores, se encuentran ligados, confirmando la utilidad de los SSR en estudios de asociación a la tolerancia al estrés hídrico en las etapas vegetativas y reproductivas (Sarkarung, 2002; Toojinda et al., 2003).
Brondani et al. (2002) y Lafitte et al. (2004) asociaron un QTL en el cromosoma 3 para la floración, resultado que confirma el QTL detectado en la presente investigación. La floración ha sido considerada como una variable secundaria asociada a la tolerancia al estrés hídrico. Se han encontraron variaciones entre líneas evaluadas bajo condiciones de estrés hídrico, para la floración al 50%. Esta característica puede ser un criterio de selección e identificación de materiales en sus programas de mejoramiento para la tolerancia a la sequía (Lafitte et al., 2003).
La floración es un carácter importante de adaptación de los cultivos y el crecimiento de las plantas en condiciones de estrés hídrico es muy sensible que afecta un componente signifi cativo del rendimiento, como lo es el porcentaje de fertilidad de las espiguillas (Takahashi et al., 2001).
En el cromosoma 9 se identificó un QTL para la fertilidad de la panícula asociada al marcador RM107. Resultado similar fue encontrado por Temnykh et al. (2001), Mei et al. (2005) y Lang y Buu (2008), quienes detectaron QTL´s altamente significativos para esa característica en el mismo cromosoma. El estrés hídrico reduce del 10 al 16% la fertilidad de las espiguillas y el número de granos por panícula; es decir, la fertilidad y la floración son afectadas por la sequía, su evaluación da información de las respuestas de los genotipos a la condición de estrés. Estas respuestas son el resultado integral de muchos procesos que ocurren durante el establecimiento en campo, desarrollo vegetativo, reproductivo y cosecha de los materiales (Babu et al., 2002).
La característica desgrane fue asociada al marcador RM442 (cromosoma 3). Según el porcentaje de desgrane del material genético, se pueden producir pérdidas de rendimiento de grano en el campo (Quirós, 2003). La disminución del desgrane de la panícula ha sido resultado de la domesticación del arroz, facilitando así la cosecha (Acevedo et al., 2006).
CONCLUSIÓN
Se identificó asociación entre las isoenzimas PRX, G6PDH, ACP, 6PGD, SDH, la tolerancia al estrés hídrico y los índices de susceptibilidad a la sequía en las familias de la población segregante, para variables relacionadas a la fase vegetativa, reproductiva y de maduración del grano del arroz; confi rmando así la utilidad de estos marcadores aplicados a estudios de mapeo y cartografía genética.
Los índices de susceptibilidad a la sequía también fueron identificados en los marcadores tipo microsatélites para características asociadas al estrés hídrico. Su información puede ser empleada como criterio en la selección asistida por medio de marcadores moleculares en los futuros programas de mejoramiento genético del arroz, con la finalidad de identificar familias y genotipos tolerantes a la condición de estrés.
Los resultados constituyen un punto de partida para el posible desarrollo de tecnologías en el cultivo de arroz para afrontar condiciones de bajo suministro de agua, nuevas formas de manejo agronómico del recurso agua, así como la obtención de variedades cuya respuesta al déficit hídrico les permita alcanzar un adecuado desempeño agronómico, en rendimiento y calidad de grano.
LITERATURA CITADA
1. Acevedo, M., W. Castrillo y U. Belmonte. 2006. Origen, evolución y diversidad del arroz. Trabajo Especial. Agronomía Tropical. 56(2):151-170. [ Links ]
2. Babu, R., P. Shanmugasundaram, P. Chezhian, B. Nguyen, P. Jayaprakash, S. Ganesh, V. Chamarerk, A. Palchamy, S. Sadasivam, S. Sarkarung, L. Wade and H. Nguyen. 2002. Molecular Dissection of Drought Tolerance in rice: Association between Physiomorphological Traits and Field Performance. In: Field screening for drought tolerance in Crop Plants with Emphasis on Rice. Proceedings of an International Workshop on Field Screening for Drought Tolerance in Rice, 11-14 December 2000, ICRISAT, Patancheru, India. Patancheru 502 324, Andhra Pradesh, India, and the Rockefeller Foundation, New York. pp. 82- 87. [ Links ]
3. Brondani, C., N. Rangel, V. Brondani and E. Ferreira. 2002. QTL mapping and introgression of yield-related traits from Oryza glumaepatula to cultivated rice (Oryza sativa) using microsatellite markers. Theoretical and Applied Genetics. 104:1192-1203. [ Links ]
4. Courtois, B. and R. Lafitte. 2003. Using marketaided selection for a specific droughttolerance trait. In: Breeding Rice for drought-prone environments. Edited by Fischer, K.S; R., Lafitte; S., Fukai; G., Atlin y B. Gardí. International Rice Research Institute (IRRI), Philippines. pp. 84-89. [ Links ]
5. Cortés, J. 2004. El Agua en el Mundo: Cooperación y Conflicto. In: Observatori Solidaritat UB. Disponible en línea: http://www.solidaritat.ub.edu/observatori/esp/itinerarios/agua/agua.htm. [Abr. 08, 2014]. [ Links ]
6. FAO. (Food and Agriculture Organization) 2002. El agua: un recurso inapreciable y limitado. Enfoques. Disponible en línea: http://www.fao.org/AG/esp/revista/0210sp1.htm. [Abr.08, 2014]. [ Links ]
7. Fischer, R. and R. Maurer. 1978. Drought resistance in spring wheat cultivars. I. Grain yield responses, Aust. J. Agric. Res. 29(5):897-912. [ Links ]
8. Garcia, F. 2008. Caracterización morfoanatómica de genotipos de arroz (Oryza sativa L.) en condiciones de estrés hídrico. Trabajo de grado para optar al título de Ingeniero Agrónomo. Universidad Central de Venezuela. Facultad de Agronomía. 89 p. [ Links ]
9. Ghneim, T., P. Alejandro, I. Pérez, G. Torrealba, C. Martínez, M. Lorieux and J. Tohme. 2005. Plant Breeding News, Edition 160 10. An Electronic Newsletter of Applied Plant Breeding by FAO and Cornell University. Available online: http://www.fao.org/WAICENT/FAOINFO/AGRICULT/AGP/AGPC/doc/services/pbn.html. [Abr. 08, 2014]. [ Links ]
10. Ghneim-Herrera, T., A. Rosales, M. Aguilar, P. Alejandro J., I. Pérez-Almeida y G. Torrealba. 2006. Identificación de cultivares de arroz con alta capacidad de ajuste osmótico para el mejoramiento genético de la tolerancia a la sequía. Agronomía Tropical. 56(4):677-687. [ Links ]
11. Grandillo, S. and S. Tanksley. 1996. QTL analysis of horticultural traits differentiating the cultivated tomato from closely related species Lycopersicum pimpinellifolium. Theoretical and Applied Genetics. 92:935-951. [ Links ]
12. Lanceras, J., G. Pantuwan, B. Jongdee and T. Toojinda. 2004. Quantitative Trait Loci Associated with Drought Tolerance at Reproductive Stage in Rice. Plant Physiology. 135(1):384-399. [ Links ]
13. Lang, N. and B. Buu. 2008. Fine mapping for drought tolerance in rice (Oryza sativa L.) Omonrice. 16:9-15. [ Links ]
14. Lafitte, H., A. Blum and G. Atlin. 2003. Using secondary traits to help identify droughttolerant genotypes. IRRI: Breeding Rice for Drought-Prone Environments. pp. 37-48. [ Links ]
15. Lafitte, H., A. Price and B. Courtois. 2004. Yield response to water deficit in an upland rice mapping population: Associations among traits and genetic markers. Theoretical and Applied Genetics. 109(6):1237-1246. [ Links ]
16. Lee, S., C. Oh, J. Suh, S. McCouch and S. Ahn. 2005. Identification of QTLs for domestication – related and agronomic traits in an Oryza sativa x Oryza rufipogon BC1F7 population. Plant Breeding. 124:209-219. [ Links ]
17. Lee, B. K., K. Y. Kim, W. J. Jung, J. C. Avice, A. Ourry and T. H. Kim 2007. Peroxidases and lignification in relation to the intensity of water-deficit stress in white clover (Trifolium repens L.). Journal of Experimental Botany. 58(6):1271-1279. [ Links ]
18. Lorieux, M. 2008. Map Disto Genetics Software, version 1.7. IRD-CIAT. Available online: http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.464.825&rep=rep1&type=pdf [Abr. 08, 2014]. [ Links ]
19. Mackill, D. 2003. What molecular tools are available for selection for drought tolerance? Section 4.4 Breeding Rice for Drought -Prone Environments. IRRI. pp. 55-61. [ Links ]
20. Manchenko, G. 1994. Handbook of Detection of Enzymes on Electrophoretic gels. Library of Congress Cataloging-in-Publication Data. 341 p. [ Links ]
21. McCouh, S. R., L. Teytelman, Y. Xu, K. B. Lobos, K. Clare, M. Walton, B. Fu, R. Maghirang, Z. Li, Y. Xing, Q. Zhang, L. Kono, M. Yano, R. Fjellstrom, G. DeClerck, D. Schneider, S. Cartinhour, D. Ware and L. Stein. 2002. Development and Mapping of 2240 New SSR Markers for Rice (Oryza sativa L.). DNA Research. 9:199-207. [ Links ]
22. McCouch, S., X. Chen, O. Panaud, S. Temnykh, Y. Xu, Y. Cho, N. Huang, T. Ishii and M. Blair. 1997. Microsatellite marker development, mapping and applications in rice genetics and breeding. Plant Molecular Biology. 35:89-99. [ Links ]
23. McCouch, S. and R. Doerge. 1995. QTL mapping in rice. Trends and Genetics. 11(12):482-487. [ Links ]
24. Mei, H., Q. Shu, Y. Wang, C. Ying, Z. Li, L. Guo, X. Yu and L. Luo. 2005. Gene actions of QTLs affecting several agronomic traits resolved in a recombinant inbred rice population and two backcross populations. Theoretical and Applied Genetics. 110:649-659. [ Links ]
25. Ortiz, A., O. Gómez y C. Ramis, C. 2004. Caracterización bioquímica de poblaciones del complejo de maleza arroz rojo en Venezuela. Agronomía Trop. 54(3):257-273. [ Links ]
26. Ortiz, A., C. Ramis, P. Parra, A. Díaz y L. López. 2002. Patrones isoenzimáticos de variedades de arroz y arroces rojos en Venezuela. Revista Facultad de Agronomía de la Universidad Central de Venezuela (UCV). 28:117-130. [ Links ]
27. Pérez-Almeida, I., E. Torres, L. Angulo y M. Acevedo. 2011. Diversidad genética entre cultivares de arroz de Venezuela con base a la estimación del coeficiente de parentesco y análisis con marcadores moleculares microsatélites (SSR). Interciencia. 36(7):545-551. [ Links ]
28. Pieters, A. J. and M. Núñez. 2008. Photosyntesis, water use efficiency andδ13C in two rice genotypes with contrasting response to water deficit. Photosynthetica. 46(4):574-580. [ Links ]
29. Praddep, M., N. Sarla, L. Reddy and E. Siddiq. 2005. Identification and mapping of yield and yield relative QTLs from an Indian accession of Oryza rufipogon. BMC Genetics. BioMed Central. Research article. Available online: http://www.biomedcentral.com. [Abr. 08,2014]. [ Links ]
30. Quirós, E. 2003. Evaluación morfológica y molecular de líneas avanzadas de mejoramiento genético de arroz (Oryzasativa) del Instituto de Investigación Agropecuaria de Panamá (IDIAP). Tesis para optar al título de maestría. Escuela de Posgrado, Programa de Educación para el Desarrollo y la Conservación del Centro Agronómico Tropical de Investigación y Enseñanza. 136 p. [ Links ]
31. Rudd, S, H. Schoof and K. Mayer. 2005. Plant Markers a database of predicted molecular markers from plants. Nucleic Acids Research. Available online: http://www.ncbi.nlm.nih.gov [Abr. 10, 2014]. [ Links ]
32. Sarkarung, S. 2002. A regional breeding program to develop drought - tolerant rainfed lowland germoplasm: IRRI´s experience in South and Southeast Asia. Breeding Rice for Drought - Prone Environments. IRRI. pp. 70-74. [ Links ]
33. Semagn, K., A. Bjornstad and N. Ndjiondjop. 2006. An overview of molecular markersmethods for plants. African Journal of Biotechnology. 5(25):2540-2568. [ Links ]
34. Septiningshigh, E., J. Prasetiyono, E. Lubis, T. Tai, T. Tjubaryat, S. Moeljopaeiro and S. McCouch. 2003. Identification of quantitative trait loci for yield and yield components in an advanced backcross population derived from the Oryza sativa variety IR64 and the wild relative O. rufipogon. Theoretical and Applied Genetics. 107:1419-1432. [ Links ]
35. Swarbreck, S., M. Defoin-Platel, M. Hindle, M. Saqi and D. Habash. 2011. New perspectives on glutamine synthetase in grasses. Journal of Experimental Botany. 62(4):1511-1522. [ Links ]
36. Takahashi, Y., A. Shomura, T. Sasaki and M. Yano. 2001. Hd6, a rice quantitative trait locus involved in photoperiod sensitivity, encodes the α subunit of protein kinase CK2. Proc Natl Acad USA. Available online: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC35444/ 98(14):7922-7927. [Abr. 08, 2014]. [ Links ]
37. Temnykh, S., W. Ayres, N. Cartinhour, S. Hauck, L. Lipovich, Y. Cho, T. Ishii and S. MCcouch. 2001. Mapping and genome organization of microsatellite sequence in rice (Oryza sativa L.). Theoretical and Applied Genetics. 100(5):697-712. [ Links ]
38. Teng, S., Q. Qian, D. L. Zeng, Y. Kunihiro, K. Fujimoto, D. N Huang and L. H Zhu. 2002. Analysis of gene loci and epistasis for drought tolerance in seedling stage of rice (O. sativa L.) Yi Chuan Xue Bao. Available online: www.ncbi.nlm.nih.gov. 29(3):235-240. [ Links ]
39. Toojinda, T., M. Siangliw, A. Tragoonrung and A. Vanavichit. 2003. Molecular Genetics of Submergence Tolerance in Rice: QTL Analysis of Key Traits. Ann Bot. 91(2):243-253. Available online: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4244984/. [Abr.08, 2014]. [ Links ]
40. Thomson, M., T. Tai, A. McClung, X.-H. Lai, M. Hinga, K. Lobos, Y. Xu, C. Martínez and S. McCouch. 2003. Mapping quantitative trait loci for yield, yield components and morphological traits in an advanced backcross population between Oryza rufipogon and the Oryza sativa cultivar Jefferson. Theoretical and Applied Genetics. 107(3):479-493. [ Links ]
41. Toorchi, M., H. Shashidhar, N. Sharma and S. Hittalmani. 2002. Tagging QTLs for maximum root length in rainfed lowland rice (Oryza sativa L.) using molecular markers. Cell Molecular Biology Lett. 7(2B):771-776. [ Links ]
42. Torrealba, G. 2010. Identificación de marcadores moleculares asociados a la tolerancia al estrés hídrico en arroz (Oryza sativa L.). Trabajo para optar al título de doctora en Ciencias Agrícolas. Universidad Central de Venezuela (UCV), Facultad de Agronomía. Maracay. 199 p. [ Links ]
43. Torrealba, G., I. Pérez, N. Delgado, C. Ramis, L. Angulo, O. León, M. Muñoz y G. Martínez. 2010. Metodología de campo para evaluación de tolerancia al estrés hídrico en el cultivo arroz. INIA Divulga. 16:23-26. [ Links ]
44. Velásquez, R. 2001. Control genético de tres sistemas isoenzimáticos en arroz (Oryza sativa L). Trabajo de ascenso a Asistente. Maracay. Universidad Central de Venezuela (UCV), Facultad de Agronomía. 57 p. [ Links ]
45. Vikram, P., BP. Mallikarjuna, S. Dixit, H. Uddin, MT. Sta Cruz, A. Kumar and Kumar. A. 2011. qDTY1.1, a major QTL for rice grain yield under reproductive-stage drought stress with a consistent effect in multiple elite genetic backgrounds. BMC Genetics. 12:89-104. [ Links ]
46. Wan, X., J. Wan, J. Weng, L. Jiang, J. Bi, C. Wang and H. Zhai. 2005. Stability of QTLs for rice grain dimension and endosperm chalkiness characteristics across eight environments. 2005. Theoretical and Applied Genetics. 110(7):1334-1346. [ Links ]
47. Xiao, J., J. Li, S. Grandillo, S. Ahn, L. Yuan, S. D Tanksley and McCouch S. 1998. Identification of trait improving quantitative trait loci alleles from a wild rice relative, Oryza rufipogon. Genetics. 150(2):899-909. [ Links ]
48. Yue, B., L. Xiong, W. Xue, Y. Xing, L. Luo and C. Xu. 2005. Genetic analysis for drought resistance of rice at reproductive stage infield with different types of soil. Theoretical and Applied Genetics. 111:1127-1136. [ Links ]
49. Zambrano, A., J. Demey, G., Martínez, F. Fuenmayor, Z. Gutiérrez, G. Saldaña y M. Torrealba. 2002. Método rápido, económico y confiable de minipreparación de ADN para amplificaciones por RAPD en bancos de germoplasma. Agronomía Trop. 52(2): 235-243. [ Links ]
50. Zhao, B., M. Deng, Q. Zhang, J. Li, S. Ye, Y. Liang, Y. Peng and P. Li. 2006. Analysis of Segregation Distortion of Molecular Markers in F2 Population of Rice. Acta Genetic Sinica. 33(5):449-457. [ Links ]