Kasmera
versión impresa ISSN 0075-5222
Kasmera v.38 n.1 Maracaibo jun. 2010
Mecanismos de resistencia a antibióticos β-lactámicos en Staphylococcus aureus
Mechanisms of Resistance To β-Lactam Antibiotics in Staphylococcus aureus
Castellano González, Maribel J.1; Perozo-Mena, Armindo J.2
1Cátedra de Bacteriología General. Escuela de Bioanálisis. LUZ 2Cátedra de Práctica Profesional de Bacteriología. Escuela de Bioanálisis. LUZ. Centro de Referencia Bacteriológica –Servicio Autónomo Hospital Universitario de Maracaibo (SAHUM). *E-mail: maribeljo@cantv.net.
Resumen
S. aureus ha demostrado un gran poder de adaptación a los agentes antimicrobianos, adquiriendo paso a paso resistencia a todos los antibióticos disponibles para el tratamiento de las infecciones que ocasiona. Existen tres mecanismos de resistencia a los antibióticos β-lactámicos en S. aureus: resistencia mediada por enzimas (penicilinasas o β-lactamasas) las cuales desactivan al antibiótico; resistencia intrínseca, que no es debida a la inactivación de drogas y es responsable de la resistencia a meticilina; y la modificación de las proteínas de unión a penicilinas (PBPs). Además, S. aureus puede expresar el fenómeno de tolerancia, en el que ocurre una disociación de las acciones inhibitoria y bactericida de los antibióticos β-lactámicos. De éstos, el mecanismo más importante, es la resistencia intrínseca que es probablemente más compleja, debido a que varios factores pueden afectar también su expresión.
Palabras clave: Staphylococcus aureus, antibióticos β-lactámicos, mecanismos de resistencia.
Abstract
S. aureus has shown a great power of adaptation to antimicrobial agents, acquiring, step-by-step, resistance to all available antibiotics for treatment of the infections it causes. S. aureus has three major mechanisms of resistance to β-lactam antibiotics: enzyme mediated (penicillinase or β-lactamase) by which the antibiotic is inactivated; intrinsic resistance, which is not due to drug inactivation and accounts for methicillin resistance; and modifications of penicillin-binding proteins (PBPs). Additionally, S. aureus can express the tolerance phenomenon, in which there is a dissociation of the inhibitory and killing actions of â-lactam antibiotics. Of these, the most important mechanism is intrinsic resistance, which is probably more complex because several factors can affect its expression.
Key words: Staphylococcus aureus, β-lactam antibiotics, resistance mechanisms.
Recibido: 05-10-09 / Aceptado: 25-11-09
Introducción
S. aureus es un microorganismo que posee características particulares de virulencia y resistencia a los antibióticos. Las infecciones que produce, ocurren regularmente en pacientes hospitalizados y tienen severas consecuencias, a pesar de la terapia antimicrobiana. La diseminación de la resistencia antimicrobiana entre cepas de S. aureus es de gran importancia en salud pública, puesto que este microorganismo ha desarrollado resistencia rápidamente a todos los antibióticos introducidos para uso clínico (1).
En la actualidad las cepas de S. aureus tienen un amplio rango de resistencia a los antibióticos y se pueden encontrar cepas resistentes y multirresistentes. El surgimiento de cepas de S. aureus multirresistentes representa una respuesta secuencial a la presión selectiva impuesta por la terapia antimicrobiana. Sin embargo, se ha observado que la acumulación y la diseminación de resistencia en S. aureus es producto del intercambio de determinantes de resistencia pre-existentes portados por elementos genéticos móviles como plásmidos, transposones (Tn) y secuencias de inserción (IS) (2).
La presente revisión tiene por objeto describir, fenotípica y genotípicamente, los mecanismos de resistencia a los antibióticos β-lactámicos en S. aureus. Se han descrito tres mecanismos que explican la resistencia a los β-lactámicos en S. aureus: producción de enzimas inactivadoras (penicilinasas o β-lactamasas), modificación de las Proteínas de unión a penicilinas (PBPs) y la resistencia intrínseca a meticilina, debida a la presencia del gen mecA; siendo este último, el mecanismo más importante desde el punto de vista clínico y el más estudiado. Además, S. aureus puede expresar el fenómeno de tolerancia, en el que ocurre una disociación de las acciones inhibitoria y bactericida de los antibióticos β-lactámicos. Aunque la frecuencia relativa de cada uno de los tipos de resistencia puede variar según el área geográfica y el tipo de paciente, se puede dar la posibilidad de que en una misma cepa coexistan distintos mecanismos (3).
Análisis y Comentarios
Resistencia a penicilina
La introducción de la penicilina a principios de los años 40 como tratamiento en las infecciones causadas por S. aureus abatió de manera importante las enfermedades ocasionadas por este microorganismo. Sin embargo, un año después de su utilización, ya se tenían cepas de S. aureus resistentes a penicilina. Para 1946, en Inglaterra se observó que aproximadamente un 60% de los aislamientos de estafilococos fueron resistentes a penicilina y para mediados de 1950, los aislamientos de S. aureus mostraron niveles más elevados de resistencia. Actualmente se reporta una resistencia a penicilina del 80%-93% o más, en cepas de S. aureus aisladas de hospitales y de la comunidad (2, 4).
La resistencia a penicilina se debe a la producción de penicilinasas (β-lactamasas) y es conferida por una penicilinasa plasmídica, inducible, que inactiva la penicilina G, carboxipenicilinas y ureidopenicilinas. El mecanismo de inducción consiste en que la penicilina y sus análogos favorecen la producción de una proteína antirrepresora que, al inhibir el gen represor de la betalactamasa, aumenta la síntesis de penicilinasa. Esta penicilinasa es inactivada por los inhibidores de β-lactamasas (ácido clavulánico, sulbactam y tazobactam). Las cefalosporinas no son hidrolizadas (Tabla 1) (5).
Tabla 1. Fenotipos de Resistencia a β-lactámicos en S. aureus (30).
| Mecanismo | Penicilina G, Carboxipenicilinas y Ureidopenicilinas | Penicilina + Inhibidor de β-lactamasa | Oxacilina | Cefalosporinas |
| Ninguno | S | S | S | S |
| Producción de β-lactamasa | R | S | S | S |
| Producción de PBP2a (mecA) | R | R | R | R |
| BORSA | R | S/R | R | S |
| MODSA | S | S | R | S |
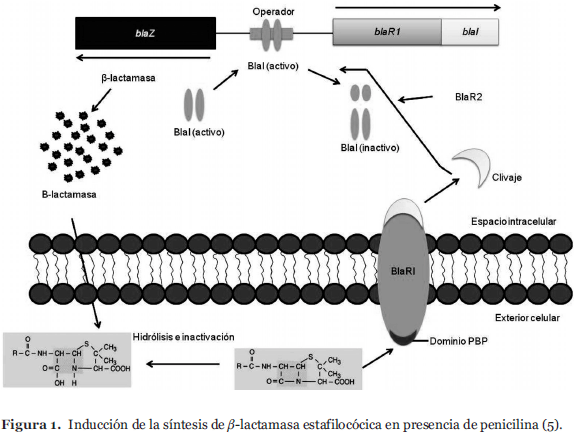
Este mecanismo de resistencia es mediado por blaZ, gen que codifica para la β-lactamasa (Figura 1). La proteína BlaI, proteína de unión al ADN, se une a la región del operón, reprimiendo así la transcripción tanto de blaZ como de blaR1-blaI. En ausencia de penicilina, la β-lactamasa se expresa a bajo nivel. La unión de penicilina al receptor transmembrana (sensor-transductor) BlaR1 estimula la autoactivación catalítica, clivándose a si mismo. BlaR1 activa, directa o indirectamente -mediante la proteína BlaR2- cliva a BlaI produciendo fragmentos inactivos, permitiendo entonces, el inicio de la transcripción de blaZ y blaR1-blaI. La β-lacta- masa, enzima de producción extracelular, inactiva entonces, la penicilina (5).
La presión ejercida por el amplio uso de la penicilina hizo que las cepas productoras de β-lactamasas se hicieran más prevalentes. El incremento de la resistencia a penicilina en S. aureus, producto del uso clínico de esta droga, no se debió sólo a la selección de unos pocos microorganismos mutantes que eran capaces de producir β-lactamasas. Evidencias genéticas indican que los genes que codifican su producción se encuentran en plásmidos y esta información puede ser transferida de un microorganismo a otro por transducción in vivo, este mecanismo desarrollado por S. aureus ha sido demostrado en todo el mundo (6).
En la actualidad más del 90% de las cepas de S. aureus han desarrollado resistencia a penicilina. Sin embargo, cuando la cepa es sensible, este antibiótico constituye una droga de primera línea y de amplio uso; además, presenta una buena disponibilidad con relación a otros antimicrobianos, dado su bajo costo y su toxicidad selectiva; debido a que actúa inhibiendo la síntesis del peptidoglucano, principal constituyente de la pared celular bacteriana, la cual no está presente en las células eucariotas del hospedero (5).
Hiperproducción de β-lactamasas (Resistencia border-linea oxacilina)
En ausencia de mecA, la resistencia a oxacilina en S. aureus puede deberse a la producción excesiva de β-lactamasas, conociéndose a los microorganismos que expresan esta forma de resistencia, como cepas con resistencia borderline (Borderline resistant Staphylocccus aureus-BORSA) (resistencia fronteriza) o resistencia de bajo nivel (Tabla 1) (3). Descubierto inicialmente por McDougal, su mecanismo es una hiperproducción de penicilinasa estafilocócica normal, mediada por plásmidos. Estas cepas producen altas cantidades de enzima, lo que hace que oxacilina y meticilina, que fueron desarrolladas para resistir la acción hidrolítica de la penicilinasa, sean lenta aunque apreciablemente degradadas, presentando una resistencia límite a oxacilina con una Concentración Inhibitora Mínima (CIM) de hasta 8 µg/ml. Esta resistencia se encuentra avalada por la ausencia de PBP2a en su pared celular y por la observación de que la asociación con ácido clavulánico, tazobactam o sulbactam disminuye las CIMs de oxacilina y meticilina en varias diluciones (3).
Las cepas hiperproductoras de β-lac- tamasas poseen un plásmido común de β-lac- tamasa de 17,2 kb que codifica a la β-lac- tamasa estafilocócica del tipo A. Se cree que la acción de resistencia no es sólo debida a una hiperproducción, sino también a una nueva β -lactamasa cuyo gen no se ha identificado aún. Esta hiperproducción de β-lacta- masas requiere altas concentraciones de cloruro de sodio y no induce una CIM mayor en condiciones de estudio normales (3).
Modificación de las PBPs
Descrita por Tomasz y colaboradores (7), corresponde a una modificación mínima (modified S. aureus, MODSA) de las Proteínas de unión a Penicilinas (PBPs) 1, 2 y 4 de peso molecular normal; pero con baja afinidad por los antibióticos β-lactámicos. Al igual que el mecanismo anterior, la respuesta es límite. Estas cepas no producen β-lac- tamasas y presentan bajos niveles de resistencia a meticilina, ello puede ser debido a la hiperexpresión de alguna de estas PBPs o la consecuencia de mutaciones genéticas que alteren la afinidad de la proteína final por el antibiótico (Tabla 1) (3).
Resistencia a meticilina
Debido a la resistencia a penicilina de las cepas de S. aureus, a finales de los años 50, se introdujeron cefalosporinas estables a penicilinasas y penicilinas semisintéticas. Entre éstas estuvo la meticilina, como antibiótico de elección en el tratamiento de S. aureus (2, 4).
La meticilina es un derivado semisintético de la penicilina, introducido en Europa en 1959 (2). Un año después, en 1960, se detectó la primera cepa S. aureus meticilina resistente (methicillin resistant S. aureus, MRSA en inglés, o SAMR en español). La resistencia a meticilina se determina utilizando el antibiótico oxacilina, por lo que se ha sugerido que estas cepas deberían llamarse S. aureus resistentes a oxacilina (oxacillin-resistant S. aureus, ORSA), más que MRSA. Más tarde, en 1963, se reportó el primer brote nosocomial causado por cepas SAMR. Desde entonces se han notificado cepas SAMR en todo el mundo (2,4).
En la actualidad, SAMR es el patógeno gram positivo, resistente a los antibióticos más comúnmente identificado en los hospitales. A pesar que 25% de las cepas de S. aureus aisladas de pacientes externos son resistentes a meticilina, la mayoría de las cepas son recuperadas de individuos que muy probablemente adquieren la infección en el hospital (8, 9); sin embargo, su asociación con los hospitales es indirecta; los contactos de individuos con SAMR adquiridos en el hospital tienen mayor riesgo de colonización por cepas SAMR (10). En un reciente y dramático evento evolutivo, la infección por cepas SAMR adquiridas en la comunidad en individuos previamente sanos sin asociación directa o indirecta con instituciones de salud, ha emergido como un nuevo e importante problema de salud pública (11, 12).
La resistencia a meticilina fue denominada intrínseca debido a que no se produce destrucción del antibiótico por acción de enzimas β-lactamasas (6) y es conferida por una proteína de unión a penicilina (PBP) adicional denominada PB2 o PBP2a, la cual no está presente en las cepas susceptibles a meticilina (Tabla 1) (2).
Las PBPs son enzimas que catalizan las reacciones de transpeptidación del peptidoglucano durante la síntesis de la pared celular. S. aureus produce al menos cuatro PBPS (PBP1, PBP2, PBP3 y PBP4), que son inhibidas por los β-lactámicos, incluyendo la meticilina. Las cepas SAMR además de sintetizar estas PBPs, producen una PBP adicional conocida como PBP2a. Esta proteína permite que, en presencia de meticilina, al ser inhibidas las PBPs normales, continúe la síntesis de pared celular; debido a que bloquea la unión de cualquier antibiótico β-lactámico a su sitio activo; pero permite que continúe el proceso de transpeptidación (2,13). La PBP2a es una transpeptidasa clase B, de alto peso molecular que cataliza la formación de puentes cruzados en el peptidoglucano de la pared celular. Asistida por el dominio transglicosilasa de la PBP2a nativa de S. aureus, toma el control de la función biosintética de la pared celular en presencia de β-lactámicos, cuando normalmente las PBPs son inactivadas al ligarse a estos antibióticos (13). Las cepas de S. aureus que son resistentes a meticilina por este mecanismo, lo son también a todos los β-lactámicos, incluyendo las penicilinas, cefalosporinas (a excepción de las nuevas cefalosporinas anti-SAMR (ceftobiprol y ceftarolina) y carbapenemes (2).
El gen mecA, responsable de la resistencia a meticilina en S. aureus, se encuentra formando parte del llamado SCCmec. y se encuentra localizado en el cromosoma bacteriano de cepas SAMR. El complejo denominado gen mec, una porción de ADN cromosómico adicional, de aproximadamente 30 a 50 Kb, es exclusivo de las cepas meticilino-resistentes. El complejo mec contiene el gen mecA (gen estructural para la PBP2a); mecI (represor) y mecR1 (inactivador de mecI), que actúan como elementos reguladores que controlan la trascripción del mecA y de 20 a 45 Kb del ADN asociado al cromosoma (Figura 2).
Existen cuatro clases genéticas del complejo del gen mec (A-D), los cuales están estructurados de la siguiente manera (Figura 3):
1. Clase A: mecI-mecRl-mecA-IS431;
2. Clase B: IS1272-ΔmecRI-mecA-IS431 (el dominio de unión a penicilina de mecl es reemplazado por IS1272);
3. Clase C1 y C2: IS431- ΔmecRI-mecA-IS431 (la Clase C2 contiene una deleción mayor en el dominio de unión a penicilina en mecRI que la Clase 1);
4. Clase D: ΔmecRI-mecA-IS431 (deleción del dominio de unión a penicilina en mecRI);
5. Clase E: ΔmecRI-mecA-IS431 (existe una deleción de 976 bp en el dominio de membrana de mecRI).
Existen también reportes de complejos mec Clase A1 y B1(14); así como A.3 y A.4(15).
El gen mecA es una región altamente conservada entre las especies de estafilococos. Muestra alto nivel de homología en SAMR y estafilococos coagulasa negativos meticilino resistentes, por lo que se ha descrito como un marcador molecular adecuado en la determinación de resistencia en todos los estafilococos. La región promotora del gen mecA constituida por los primeros 300 nucleótidos, y sus genes regulatorios, son similares en secuencia a las regiones análogas de la -lactamasa estafilocócica (6).
SCCmec es una isla genómica (G island), que comprende un grupo de elementos móviles de ADN, de 21 a 67 kb, que se inserta al final del extremo 3 de un marco de lectura abierta (open reading frame, ORF), denominado orfX, en un sitio único (attBscc), cerca del origen de replicación de S. aureus. SCCmec contiene además del conjunto de genes mec (el gen mecA y sus reguladores), el conjunto de genes ccr, los cuales codifican recombinasas sitio específicas responsables de la movilidad de SCCmec (1;16). Este cassette no es endógeno de esta bacteria y para su transmisión, SCCmec porta uno de tres pares de genes para recombinasas cromosómicas (ccrA, ccrB, ccrC), que codifican para una recombinasa de la familia invertasa-reductasa (13, 16, 17).
SCCmec recuerda una isla de patogenicidad; sin embargo, no contiene genes de virulencia. En este sentido, se le conoce como una isla de resistencia a los antibióticos, ya que aparte de conferir resistencia a todos los antibióticos β-lactámicos, contiene genes de resistencia adicionales como consecuencia de la integración de plásmidos y transposones en el cassette cromosomal. SCCmec también confiere resistencia a eritromicina, espectinomicina, tetraciclina, kanamicina y otros antibióticos (13).
El origen del SCCmec es incierto. No existen reportes de SCCmec en bacterias diferentes a estafilococos (16). El mecanismo responsable de la transferencia de mecA, no se conoce; pero la evidencia sugiere la transferencia horizontal de ADN mecA entre especies de estafilococos y del gen mecA entre diferentes géneros grampositivos. Se ha asumido que los genes ccr y mec fueron traídos conjuntamente a estafilococos coagulasa negativa a partir de una fuente desconocida; donde ocurre la deleción de genes regulatorios, antes que los genes sean transferidos a S. aureus, generando así, las cepas SAMR (3).
Una región homóloga con 88% de similaridad en aminoácidos con el mecA de las cepas meticilino-resistentes de estafilococos ha sido identificada en S. sciuri (18), dicha secuencia es ubicua en esta especie; pero su fenotipo es susceptible, lo que sustenta la hipótesis que el mecA se originó en estafilococos coagulasa negativo, cercanamente relacionadas desde el punto de vista evolutivo con S. sciuri (18).
Todas las cepas SAMR son clones descendientes de las pocas cepas ancestrales que adquirieron el gen mecA (2, 13). Esta afirmación ha conllevado a la creencia que la frecuencia de transmisión intra o interespecie de mecA es más bien un proceso limitado que no es debido a la presencia de maquinaria especializada de transmisión, como los transposones (2, 13).
Hasta la fecha, en S. aureus se han caracterizado y clasificado cinco tipos diferentes con algunas variantes de cassette mec (SCCmec), de acuerdo a los genes de su recombinasa putativa (alotipos ccrAB) y su composición genética en general (19, 20). Se han identificado tres alotipos importantes (ccrAB1 a ccrAB3) y un alotipo esporádico (ccrAB4). Recientemente fue divulgado un nuevo tipo de complejo del gen ccr (ccrC) no estrechamente relacionado con los anteriores (13). SCCmec tipos I al V son definidos en base a la combinación particular del tipo de 1,2 ó 3 pares de genes ccrAB y la clase del complejo mecA, B, C y D (Tabla 2, Figura 4) (1, 13, 21-24).
Tabla 2. Características de los Cassettes Cromosómicos mec de Estafilococos (SCCmec)(23,24).
| Tipo SCC | Tamaño SCCmec | Tipo Gen |
Elementos Complejo mec | Tipo Complejo mec | Otros determi- | Origen | Presencia de PVL | |||
| IS1272 | mecI | mecRI PB/MS | mecA | |||||||
| I | 34 | 1 | + | - | -/+ | + | B | - | Hospital | Infrecuente |
| II | 53 | 2 | - | + | +/+ | + | A | PUB110a Tn554b | Hospital | Infrecuente |
| III | 67 | 3 | + | + | +/+ | + | A | PUB110 PT181c | Hospital | Infrecuente |
| IV | 21-24 | 2 | - | - | -/+ | + | B | - | Comunidad | Frecuente |
| V | 28 | C | - | - | -/- | + | C | - | Comunidad | Desconocido |
ccr: recombinasa del cassette cromosómico
MS: dominio de membrana
PB: dominio de las PBPs
a: codifica resistencia tetraciclina y kamamicina
b: codifica resistencia a macrólidos-lincosaminas y estreptograminas
c: codifica resistencia a tetraciclina
Tipo I SCCmec: Comienza su diseminación entre las cepas SARM en los años 60, en Inglaterra, coincidiendo con el principio de la era antibiótica. Tiene un tamaño de 34Kb. Está formado por el complejo de genes ccr tipo 1 (ccrA1, ccrB1) y el complejo mec clase B (mecA, mecR1 y una copia cortada de la secuencia de inserción IS1272). También contiene la inserción IS431. No posee ningún gen de resistencia excepto el mecA (1, 13, 21).
Tipo II SCCmec (53 Kb): Se aísla en 1982 y es frecuente entre las cepas SARM de Japón, Corea y USA. Comprende el complejo de recombinasas tipo 2 (ccrA2, ccrB2), el complejo mec clase A (mecI, mecR1, mecA), la inserción IS431, el plásmido pUB110 y el transposón Tn554 (1, 13, 21).
Tipo III SCCmec (67 Kb): Se ha descrito en los países europeos, Arabia Saudita, India, Singapur, Hong Kong, Australia y Nueva Zelanda. Lo componen el complejo de recombinasas tipo 3 (ccrA3, ccrB3), el complejo mec clase A, Tn554, pT181, pI256 y las secuencias de inserción IS431 y IS256. También contiene una copia cortada de genes ccr situada entre dos elementos IS431, esto sugiere la posibilidad de que el tipo III SCCmec pueda derivar de la fusión en el pasado de dos elementos SCC. Los tipos II y III SCCmec se encontraban en las cepas dominantes de los años 1980, caracterizadas por contener múltiples genes de resistencia antibiótica (1, 13, 21).
Tipo IV SCCmec (21-24 Kb): contiene el complejo de genes ccr tipo 2 (ccrA2, ccrB2), el complejo mec clase B y la inserción IS1272. Es el elemento más pequeño, lo que le confiere mayor movilidad genética y no lleva factores de virulencia, ni genes adicionales de resistencia para otros antibióticos que no sea meticilina. Esta característica también ocurre en un rango más amplio de grupos genéticos de S. aureus sensibles a meticilina, sugiriendo que ha sido heterólogamente transferido de otras especies estafilocócicas. Posiblemente, estas mismas características hacen que el SCCmec tipo IV se encuentre asociado a cepas SARM adquiridas en la comunidad. Sin embargo, esta asociación no es única ya que aunque el tipo IV es más frecuente entre los SARM adquiridos en la comunidad, también se encuentra entre las cepas de adquisición hospitalaria (1, 13, 21).
Tipo V SCCmec (28 Kb): a diferencia de los otros tipos este posee un nuevo gen ccr denominado ccrC. Se ha encontrado en Australia en un pequeño número de cepas SARM de origen comunitario y aún está en investigación. Dos posibles nuevas variedades de cassette han sido recientemente identificadas en cepas australianas resistentes a meticilina de origen comunitario (1, 13, 21).
Los aislamientos SAMR adquiridos en el hospital (SAMR-AH) difieren sensiblemente de las cepas SAMR adquiridas en la comunidad (SAMR-AC), desde el punto de vista microbiológico y epidemiológico (25, 26). Así, SAMR-AC típicamente, contiene el gen mecA del tipo IV SCCmec; en contraste con el tipo II de las cepas hospitalarias (23, 24). Por otra parte, SAMR-AC frecuentemente porta genes que codifican para la leucocidina Pantone-Valentine (PVL), una citotoxina, factor de virulencia asociado a la infección de tejidos blandos y neumonía necrotizante (Tabla 2) (23, 24).
Desde el punto de vista clínico, las cepas SAMR-AC comparadas a las SAMR-AH, usualmente, afectan individuos más jóvenes, con escasa co-morbilidad y con propensión a infecciones en piel y tejidos blandos (27). Finalmente, SAMR-AC es típicamente, más sensible a los antibióticos que sus homólogos SAMR-AH, probablemente debido a que poseen un SCCmec de menor tamaño, con menos capacidad para portar genes de resistencia antimicrobiana (28-30).
La meticilino-resistencia, dependiendo de la habilidad de S. aureus para organizar el SCCmec y de integrar funcionalmente la PBP2a, es diferente en cada cepa, resultando en una gran variedad de niveles de resistencia (13).
La expresión fenotípica de resistencia a meticilina en estafilococos presenta varias características intrínsecas (7). Las cepas SAMR parecen ser uniformes y poseen el gen mecA y su producto, la PBP2a o PBP2 (78-kDa). Por otra parte, el grado de resistencia (por ejemplo, concentraciones inhibitorias mínimas (CIMs) determinadas por técnicas convencionales de pruebas de susceptibilidad) varía tremendamente entre cepas, oscilando en el rango de pocos microgramos por mililitro (valores no alejados de la CIM para cepas S. aureus meticilino-sensibles, SAMS) a varios miligramos por mililitro (7).
Usando técnicas de mayor resolución (análisis de población), es posible determinar características peculiares de las bacterias: los cultivos en caldo de las cepas SAMR no son uniformes en sus CIMs; están compuestas por variedad de subpoblaciones de bacterias que varían ampliamente en sus grados de resistencia antibiótica. En consecuencia es sorprendente el nivel de heterogeneidad de la expresión fenotípica de resistencia a meticilina entre cepas y aún, entre la progenie de una única cepa SAMR (31). Se conoce que factores ambientales y genéticos influyen en la expresión fenotípica de tal resistencia; sin embargo, las bases de su mecanismo de diversidad no se ha establecido con claridad hasta la fecha (7).
Tomasz y colaboradores (7) definieron cuatro clases de expresión fenotípica de resistencia a meticilina en S. aureus: la clase 4, que es definida por una resistencia homogénea de alto nivel (CIM > 800 µg/ml) y las clases 1, 2 y 3 con resistencia heterogénea a meticilina con CIM de 1,5 a 3; 6 a 12 y 50 a 200 µg/ml, respectivamente.
La resistencia heterogénea es distintiva de la resistencia a meticilina en la cual, el 99,99% o más, es sensible a bajas concentraciones de antibiótico β-lactámico y 1 en 106 crece a concentraciones de meticilina de 50 µg/ml o más. Esta denominación de heterogénea está dada por el siguiente hecho diferencial: en placas con concentraciones elevadas y progresivas de antibiótico, el número de colonias va decreciendo inversamente a la concentración, sugiriendo que sólo una pequeña fracción de la población expresa fenotípicamente la resistencia (3).
Factores que afectan la resistencia (Figura 5)
Los cambios en la expresión de resistencia heterogénea son transitorios y enteramente fenotípicos. El pasaje de estas cepas heterogéneas en medios con antibióticos produce proliferación de cepas altamente resistentes, resultando en una población homogénea. Subcultivos posteriores en medio libre de antimicrobianos vuelven al patrón original heterogéneo (2, 3, 6, 30).
Se desconocen diferencias entre cepas silvestres con expresión fenotípica de resistencia heterogénea u homogénea: no hay deficiencia de factores y la pared de ambas es similar. Una explicación podría ser que cepas heterogéneas tengan alguna deficiencia en una vía bioquímica específica que repercuta en la síntesis de la pared celular, importante para el funcionamiento de la PBP2a y que sea la presión selectiva de β-lactámicos sobre cepas homogéneas, la que provoque mutaciones funcionales sobre la PBP2a (2, 3, 6, 30).
Existe una variedad de factores adicionales a mecA que han sido asociados con la resistencia a meticilina en S. aureus, a saber:
Plásmido de β-lactamasa estafilocócica: podría impedir la deleción espontánea del mec cromosomal o podría inducir un patrón heterogéneo de resistencia, si se inserta en receptores homogéneamente resistentes, entre otros (3).
Factores fem (factores esenciales para resistencia a meticilina): denominado también factor auxiliar (factor aux). Constituido por genes cromosómicos distintos al mec, necesarios para la completa expresión de resistencia, presente en cepas sensibles y resistentes. Destaca fem A, B, C, D, E y F mapeados en diferentes sitios. Mutantes fem alteran la composición del peptidoglucano, excepto fem E cuya función es desconocida (3).
Otros locus cromosomales: llm codifica una proteína hidrófoba de 38 KDa, de función desconocida, cuya inactivación hace que una cepa de resistencia homogénea produzca un patrón heterogéneo de resistencia a meticilina con un aumento en la tasa de autolisis. Los locus agr (gen accesorio regulador) y sar (regulador accesorio estafilocócico) están involucrados en controlar la expresión de numerosos factores de virulencia estafilocócica y exoproteínas. Existen también los genes chr, genes cromosomales cuyas mutaciones conllevan resistencia de alto nivel a meticilina en presencia de PBP2a y que difieren del operón femAB. Hasta el momento se desconoce su posición dentro del cromosoma (3).
Detección de resistencia a meticilina
Dada la alta heterogeneidad (variabilidad en la expresión de la resistencia) en las cepas que llevan el genotipo mecA, la detección de la resistencia en el laboratorio sigue siendo problemática. Las técnicas a usar deben detectar una anormalidad en una población de 100.000 ó 1.000.000 de individuos, por lo que deben manejarse con precaución los factores físicos y químicos que influyen: osmolaridad del medio, luz visible (las cepas pigmentadas incubadas en la oscuridad aumentan la expresión 1,7 veces, y las cepas no pigmentadas son 6.000 veces más resistentes a meticilina), pH del medio (no existiendo resistencia a un pH de 5,2 pero sí a 7,4), agentes quelantes, cationes divalentes, temperatura de incubación, presencia de antibióticos β-lactámicos, entre otros (2, 3, 6, 13, 30).
Se ha sugerido la presencia de un factor adicional, producto de un gen control notando la expresión fenotípica de la resistencia, al que inicialmente denominaron factor X. En aquellas cepas en que la expresión de la resistencia es condicionada por la temperatura alta, el elemento sensible a ésta, no sería en realidad la PBP2a, sino el factor X, cuya acción sería regular su transducción y transcripción o tal vez, interviene en la regulación de la actividad autolítica o en la síntesis de la pared celular de cepas SAMR (3, 13, 30).
Las condiciones ideales que permiten la detección en el laboratorio de cepas resistentes a oxacilina son: pH neutro, medios hipertónicos, temperatura de incubación de 35°C, incubaciones de 24 horas exactas, e inóculos densos. Sin embargo, al comparar distintos métodos de detección de SAMR in vitro, se concluyó que la variante más importante es el suplemento del medio de cultivo con NaCl al 4% (3, 13, 30).
Pese a los numerosos trabajos realizados, no existe en la actualidad uniformidad de criterios sobre que modificación es la determinante principal. El factor de concentración salina pareciera ser decisivo para detectar las cepas heterorresistentes, no ejerciendo gran influencia, la temperatura de incubación, ni el tamaño del inóculo, a pesar que la temperatura permite diferenciar dos tipos de cepas: homogéneas y heterogéneas (32). Las primeras tienen una expresión de resistencia uniforme en el 100% de la población, independientemente de la temperatura de incubación, que puede detectar más de 95% de las cepas resistentes (37 ó 42°C). Son cepas infrecuentes con CIM elevada. En cambio, las heterogéneas son dependientes de la temperatura, con mejor expresión a 37°C y nula a 42°C. En este último grupo coexisten dos subpoblaciones: una más pequeña (1 en 105 a 107) detectable a 37°C, la otra, predominante y mayoritariamente termosensible, que crece a 30°C. Existe aún una mayor eficiencia al adicionar al agar Müeller Hinton, NaCl 4% + 6 µg/ml de oxacilina, en condiciones estandarizadas (oxacillin screen plate). La sensibilidad de este método se aproxima al 100% para la detección de resistencia a meticilina (3, 13, 30).
Otro método utilizado para la detección de resistencia a meticilina en S. aureus, es el de difusión del disco en agar, para el cual se recomienda una temperatura de incubación de 35°C y discos de oxacilina de 1 µg. Es el método menos confiable de detección teniendo baja especificidad. El NaCl adicionado al agar o la incubación mayor a 48 horas mejoran la sensibilidad en desventaja de la especificidad (3, 13, 30). Adicionalmente, se ha demostrado que el cefoxitin (cefamicina) in vitro, induce la producción de PBP2a en cepas de S. aureus meticilino sensibles (SAMS); por lo que el método de difusión del disco usando cefoxitin (FOX 30 µg) ha probado ser un buen ensayo para la detección de resistencia de bajo nivel a oxacilina en cepas de S. aureus (33).
El test de dilución bajo condiciones apropiadas detecta más del 95% de las cepas resistentes. El Instituto para la Estandarización de Laboratorios Clínicos (CLSI) recomienda emplear caldo Müeller Hinton suplementado con NaCl al 2% y un inóculo de 1,5 x 108 UFC/ml y 24 horas de incubación a 35°C (32).
Las pruebas de aglutinación con partículas de látex se basan en una reacción de aglutinación que por medio de anticuerpos monoclonales detectan la presencia de la PBP2a, producto del gen mecA, en aislamientos de S. aureus (34).
Desde el año 2002, el NCCLS (actual CLSI) ha aceptado que la detección de PBP2a es un método alternativo para la evaluación de resistencia a meticilina en S. aureus, el cual correlaciona eficientemente con la presencia del gen mecA, con una sensibilidad y especificidad del 100%. Estas técnicas poseen una ventaja adicional en relación al tiempo de detección, ya que se pueden obtener resultados en sólo 15 minutos después de aislado el microorganismo. Por otra parte, son fáciles de realizar e interpretar y representan, en los casos en que es imposible la detección del mecA por métodos moleculares, una alternativa diagnóstica para laboratorios de rutina (34).
Los sistemas automatizados tienen una excelente especificidad pero baja sensibilidad. Los resultados deberían chequearse con un segundo test como agar screen antes de informar la cepa como susceptible (3, 13, 30).
Como aporte al estudio epidemiológico se han desarrollado técnicas de tipificación que colaboran en la pesquiza de reservorios y vías de diseminación de SAMR. Existen técnicas fenotípicas como: fagotipificación, tipificación por electroforesis de proteínas, antibiotipia, inmunoblot y serotipificación capsular; las técnicas genotípicas se basan en la tipificación del ADN: análisis del ADN plasmídico, análisis del ADN cromosómico por enzimas de restricción, electroforesis de campo pulsado, southerblot y la reacción en cadena de la polimerasa (PCR) (3, 21, 23, 30).
La detección de mecA basada en PCR o hibridación de ADN identificará siempre correctamente la mayoría de las cepas heterogéneas y debería ser considerado como el gold standard para detectar la resistencia a meticilina (3, 23, 30).
Tolerancia a la acción bactericida de los antibióticos β-lactámicos
Afecta a todos los β-lactámicos. Implica que para la lisis y muerte del microorganismo se requieren concentraciones de antibiótico mucho más elevadas que para la inhibición de su crecimiento. Significa una disminución de la actividad autolítica por exceso de inhibidor de autolisinas, lo que conlleva a un efecto bactericida más lento. Se desconoce su base genética (21, 23, 30).
Un cultivo de S. aureus tolerante se define como una cepa que exhibe una disociación muy marcada entre la CIM y la CBM (Concentración Bactericida Mínima) del antibiótico β-lactámico luego de la prueba estándar de dilución. En la práctica, la mayor parte de las cepas de S. aureus es no tolerante; es decir, tiene una relación CIM/CBM mucho mayor de 1:32, pudiendo llegar a 1:2.000. Cuando se observa este fenómeno en una cepa dada para un antibiótico β-lactámico, en general, es reproducible con otros antibióticos del grupo, incluidas las cefalosporinas, y a veces, incluso con vancomicina. Esta característica tiende a desaparecer en el laboratorio cuando se subcultiva el microorganismo (2, 6, 30).
Teniendo como base investigaciones realizadas en otras especies bacterianas, se presume actualmente que, los antibióticos β-lactámicos fallan en la activación de las enzimas autolíticas en cepas tolerantes de S. aureus. En algunos estudios, la tolerancia de S. aureus se ha asociado con una respuesta clínica insuficiente a los β-lactámicos en pacientes con septicemia y endocarditis. Estas observaciones clínicas han sido contradichas por estudios en animales que muestran un comportamiento similar en cepas tolerantes y no tolerantes. La tolerancia puede, sin embargo, tener importancia clínica en infecciones crónicas asociadas con cuerpos extraños (35).
Nuevas cefalosporinas
Ceftobiprol: El ceftobiprol (BAL5788) (Basilea/Johnson & Johnson) es una nueva cefalosporina de amplio espectro, con potente actividad contra las bacterias gramnegativas y grampositivas. Su principal ventaja, en comparación con las otras cefalosporinas, reside en su actividad bactericida contra patógenos grampositivos resistentes a las β-lactamasas, como SAMR y Streptococcus pneumoniae resistente a penicilina y ceftriaxona. Este fármaco también inhibe una amplia gama de patógenos gramnegativos (Figura 6) (36-39).
Al igual que en todos los β-lactámicos, el sitio de unión del ceftobiprol a las proteínas de unión a penicilina (PBP) es el determinante más importante de su actividad antibacteriana. La inhibición potente y prolongada de la PBP2a de S. aureus diferencia el ceftobiprol de otros β-lactámicos y es la base molecular para su actividad contra el estafilococo resistente a meticilina. El ceftobiprol se une en el sitio activo de la PBP2a y rápidamente forma un complejo acil-enzima estable. Esta interacción produce una hidrólisis muy lenta de la molécula que determina la inhibición estable de la enzima (36-39).
Ha sido aprobado por la FDA para el tratamiento de las infecciones complicadas de la piel y tejidos blandos debidas a SAMR y también para el tratamiento de la neumonía nosocomial (36-39). Esta cefalosporina de quinta generación, ha demostrado, además, un bajo potencial de selección de resistencia in vitro. Según hipótesis de Banerjee y cols. (37) la resistencia a este antibiótico podría deberse a mutaciones en el gen mecA. Estos autores seleccionaron mutantes de SARM resistentes a ceftobiprol por pases en caldos de cultivo conteniendo concentraciones subinhibitorias de la droga, utilizando las cepas COLnex (que carece del gen mecA) transformadas con el plásmido pAW8 (plásmido vector), pYK20 (plásmido que contiene el mecA wild type) y el pYK21 (que contiene un mutante de mecA con cinco mutaciones en la PBP2a). Las cepas portadoras del mecA wild type y las mutantes en mecA desarrollaron seis (transformantes pYK20) y cuatro (transformantes pYK21) nuevas mutaciones en mecA. La transformación de COLnex con el plásmido mutante mecA confirió resistencia a ceftobiprol y, la pérdida del gen mecA tornó la cepa nuevamente susceptible a la droga. Se ha propuesto que mutaciones múltiples en el gen mecA alteran la unión del ceftobiprol, provocando resistencia en cepas SAMR. Mutaciones adicionales podrían haber mediado la resistencia al influir sobre otras proteínas. Sin embargo, también se han desarrollado cepas resistentes carentes de mecA, sugiriendo el posible rol de genes cromosomales (37).
Ceftarolina: La ceftarolina es una nueva cefalosporina de amplio espectro con potencial actividad contra cepas SAMR debido a su fuerte afinidad por las PBPs de S. aureus, incluyendo la PBP2a, proteína responsable de la resistencia a meticilina (Figura 7) (40).
Se presenta en forma de acetato de ceftarolina (PPI- 0903), una prodroga soluble en agua que, in vivo, es rápidamente metabolizada en su metabolito activo ceftarolina (PPI-0903 M). En comparación con linezolid y vancomicina, es más efectiva en el tratamiento de infecciones por cepas hGISA (S. aureus con susceptibilidad intermedia heterogénea a glicopéptidos). Esta nueva cefalosporina al parecer, es una opción prometedora y efectiva para el tratamiento de infecciones severas por SAMR (40).
Aún está por determinarse la frecuencia relativa de cada uno de los mecanismos de resistencia a los antibióticos β-lactámicos y existe la posibilidad que en una misma cepa SAMR coexistan distintos mecanismos (30).
Conclusión
La resistencia intrínseca a meticilina es el principal mecanismo de resistencia a β-lactámicos en S. aureus y el más importante desde el punto de vista clínico, puesto que las cepas SAMR se han diseminado de los hospitales a la comunidad y con frecuencia expresan multirresistencia, resultando en ocasiones, sensibles sólo a glicopéptidos. La reciente aparición de cepas con susceptibilidad disminuida e incluso, resistentes a vancomicina, pone en evidencia la potencial infección por un organismo virulento, para el cual las opciones terapéuticas son muy limitadas.
Referencias Bibliográficas
1. Ito T, Ma XX, Takeuchi F, Okuma K, Yuzawa H, Hiramatsu K. Novel Type V Staphylococcal Cassette Chromosome mec Driven by a Novel Cassette Chromosome Recombinase, ccrC. Antimicrob Agents Chemother 2004 Jul 1; 48(7):2637-51. [ Links ]
2. Velázquez-Meza ME. Surgimiento y diseminación de Staphylococcus aureus meticili- no-resistente. Salud Pública Méx 2005; 47:381-7. [ Links ]
3. Gil de M M. Staphylococcus aureus: Microbiologia y aspectos moleculares de la resistencia a meticilina. Revista Chilena de Infectología 2000; 17(2):145-52. [ Links ]
4. Kanafani Z, Fowler V. Staphylococcus aureus Infections: New Challenges from an Old Pathogen. Enferm Infecc Microbiol Clin 2006; 24(3):182-93. [ Links ]
5. Lowy F. Antimicrobial Resistance: the example of Staphylococcus aureus. J Clin Invest 2003; 111(9):1265-73. [ Links ]
6. Chambers HF. Methicillin resistance in staphylococci: molecular and biochemical basis and clinical implications. Clin Microbiol Rev 1997 Oct 1; 10(4):781-91. [ Links ]
7. Tomasz A, Nachman S, Leaf H. Stable classes of phenotypic expression in methicillin-resistant clinical isolates of staphylococci. Antimicrob Agents Chemother 1991 Jan 1; 35(1):124-9. [ Links ]
8. Charlebois E, Bangsberg D, Moss N, Moore M, Moss A, Chambers H, et al. Population-Based Community Prevalence of Methicillin-Resistant Staphylococcus aureus in the Urban Poor of San Francisco. Clin Infect Dis 2002; 34:425-33. [ Links ]
9. Tacconelli E, Venkataraman L, De Girolami PC, DAgata EMC. Methicillin-resistant Staphylococcus aureus bacteraemia diagnosed at hospital admission: distinguishing between community-acquired versus healthcare-associated strains. J Antimicrob Chemother 2004 Mar 1; 53(3):474-9. [ Links ]
10. Calfee DP, Durbin LJ, Germanson TP, Toney DM, Smith EB, Farr BM. Spread of methicillin-resistant Staphylococcus aureus (MRSA) among household contacts of individuals with nosocomially acquired MRSA. Infect Control Hosp Epidemiol 2003 Jun; 24(6):422-6. [ Links ]
11. Deresinski S. Methicillin-resistant Staphylococcus aureus: an evolutionary, epidemiologic, and therapeutic odyssey. Clin Infect Dis 2005; 40(4):562-73. [ Links ]
12. Naimi T, Ledell K, Como-Sabetti K, Borchardt S, Boxrud D, Etienne J, et al. Comparison of community- and health care-associated methicillin-resistant Staphylococcus aureus infection. JAMA 2003; 290 (22):2976-84. [ Links ]
13. Hanssen AM, Ericson Sollid JU. SCCmec in staphylococci: genes on the move. FEMS Immunol Med Microbiol 2006 Feb; 46 (1):8-20. [ Links ]
14. Lim TT, Chong FN, OBrien FG, Grubb WB. Are all community methicillin-resistant Staphylococcus aureus related? A comparison of their mec regions. Pathology 2003 Aug; 35(4):336-43. [ Links ]
15. Shore A, Rossney AS, Keane CT, Enright MC, Coleman DC. Seven novel variants of the staphylococcal chromosomal cassette mec in methicillin-resistant Staphylococcus aureus isolates from Ireland. Antimicrob Agents Chemother 2005 May; 49(5):2070-83. [ Links ]
16. Hiramatsu K, Cui L, Kuroda M, Ito T. The emergence and evolution of methicillin-resistant Staphylococcus aureus. Trends Microbiol 2001 Oct; 9(10):486-93. [ Links ]
17. Ito T, Katayama Y, Asada K, Mori N, Tsutsumimoto K, Tiensasitorn C, et al. Structural Comparison of Three Types of Staphylococcal Cassette Chromosome mec Integrated in the Chromosome in Methicillin-Resistant Staphylococcus aureus. Antimicrob Agents Chemother 2001 Dec 1; 45(12):3677. [ Links ]
18. Wu S, Piscitelli C, de LH, Tomasz A. Tracking the evolutionary origin of the methicillin resistance gene: cloning and sequencing of a homologue of mecA from a methicillin susceptible strain of Staphylococcus sciuri. Microb Drug Resist 1996; 2(4):435-41. [ Links ]
19. Ben NM, Mastouri M, Frih S, Sakly N, Ben SY, Nour M. Molecular characterization of methicillin-resistant Staphylococcus aureus isolated in Tunisia. Diagn Microbiol Infect Dis 2006 May; 55(1):21-6. [ Links ]
20. Katayama Y, Ito T, Hiramatsu K. A New Class of Genetic Element, Staphylococcus Cassette Chromosome mec, Encodes Methicillin Resistance in Staphylococcus aureus. Antimicrob Agents Chemother 2000 Jun 1; 44(6):1549-55. [ Links ]
21. Borras Ordás C. Epidemiología de la resistencia a meticilina en cepas de Staphylococcus aureus aisladas en hospitales españolas. Barcelona, España: Facultad de Medicina. Universidad de Barcelona; 2006. [ Links ]
22. Velazquez-Meza ME, ires De SM, Echaniz-Aviles G, Solorzano-Santos F, Miranda-Novales G, Silva-Sanchez J, et al. Surveillance of methicillin-resistant Staphylococcus aureus in a pediatric hospital in Mexico City during a 7-year period (1997 to 2003): clonal evolution and impact of infection control. J Clin Microbiol 2004 Aug; 42(8): 3877-80. [ Links ]
23. Bustos-Martínez J, Hamdan-Partida A, Gutiérrez-Cárdenas M. Staphylococcus aureus: la reemergencia de un patógeno en la comunidad. Rev Biomed 2006;17:287-305. [ Links ]
24. Diederen BM, Kluytmans JA. The emergence of infections with community-associated methicillin resistant Staphylococcus aureus. J Infect 2006;52(3):157-68. [ Links ]
25. Draghi DC, Sheehan DF, Hogan P, Sahm DF. Current antimicrobial resistance profiles among methicillin-resistant Staphylococcus aureus encountered in the outpatient setting. Diagn Microbiol Infect Dis 2006 Jun; 55(2):129-33. [ Links ]
26. Durand G, Bes M, Meugnier H, Enright MC, Forey F, Liassine N, et al. Detection of New Methicillin-Resistant Staphylococcus aureus Clones Containing the Toxic Shock Syndrome Toxin 1 Gene Responsible for Hospital- and Community-Acquired Infections in France. J Clin Microbiol 2006 Mar 1; 44(3):847-53. [ Links ]
27. Weber J. Community-Associated Methicillin- Resistant Staphylococcus aureus. Clin Infect Dis 2005; 41(Suppl 4): S269-S272. [ Links ]
28. Almer LS, Shortridge VD, Nilius AM, Beyer JM, Soni NB, Bui MH, et al. Antimicrobial susceptibility and molecular characterization of community-acquired methicillin- resistant Staphylococcus aureus. Diagn Microbiol Infect Dis 2002 Jul; 43(3):225-32. [ Links ]
29. Crum NF, Lee RU, Thornton SA, Stine OC, Wallace MR, Barrozo C, et al. Fifteen-year study of the changing epidemiology of methicillin-resistant Staphylococcus aureus. Am J Med 2006 Nov; 119(11):943-51. [ Links ]
30. Chambers HF. Detection of methicillin-resistant staphylococci. Infect Dis Clin North Am 1993 Jun; 7(2):425-33. [ Links ]
31. Hartman BJ, Tomasz A. Expression of methicillin resistance in heterogeneous strains of Staphylococcus aureus. Antimicrob Agents Chemother 1986 Jan 1; 29(1):85-92. [ Links ]
32. CLSI (Clinical Laboratory Standardization Institute). Performance standards for antimicrobial susceptibility testing. Nineteenth Informational Supplement M100-S19. CLSI, editor. 19th[29], 1-156. 2009. USA. [ Links ]
33. Felten A, Grandry B, Lagrange PH, Casin I. Evaluation of Three Techniques for Detection of Low-Level Methicillin-Resistant Staphylococcus aureus (MRSA): a Disk Diffusion Method with Cefoxitin and Moxalactam, the Vitek 2 System, and the MRSA-Screen Latex Agglutination Test. J Clin Microbiol 2002 Aug 1; 40(8):2766-71. [ Links ]
34. Soloaga R, Corso A, Gagetti P, Faccone D, Galas M. Methicillin resistance detection in Staphylococcus aureus: comparison between conventional methods and MRSA-Screen latex agglutination technique]. Rev Argent Microbiol 2004 Jan; 36(1):36-40. [ Links ]
35. Goldman PL, Petersdorf RG. Significance of Methicillin Tolerance in Experimental Staphylococcal Endocarditis. Antimicrob Agents Chemother 1979 Jun 1; 15(6):802-6. [ Links ]
36. Anderson SD, Gums JG. Ceftobiprole: an extended-spectrum anti-methicillin-resistant Staphylococcus aureus cephalosporin. Ann Pharmacother 2008 Jun; 42(6): 806-16. [ Links ]
37. Banerjee R, Gretes M, Basuino L, Strynadka N, Chambers HF. In vitro selection and characterization of ceftobiprole-resistant methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother 2008 Jun; 52(6):2089-96. [ Links ]
38. Pillar CM, Aranza MK, Shah D, Sahm DF. In vitro activity profile of ceftobiprole, an anti-MRSA cephalosporin, against recent Gram-positive and Gram-negative isolates of European origin. J Antimicrob Chemother 2008 Mar 1; 61(3):595-602. [ Links ]
39. Noel GJ. Clinical profile of ceftobiprole, a novel beta-lactam antibiotic. Clin Microbiol Infect 2007 Jun; 13 Suppl 2:25-9. [ Links ]
40. Jacqueline C, Caillon J, Le M V, Miegeville AF, Hamel A, Bugnon D, et al. In vivo efficacy of ceftaroline (PPI-0903), a new broad-spectrum cephalosporin, compared with linezolid and vancomycin against methicillin-resistant and vancomycin-intermediate Staphylococcus aureus in a rabbit endocarditis model. Antimicrob Agents Chemother 2007 Sep; 51(9):3397-400. [ Links ]












 uBio
uBio