Interciencia
versión impresa ISSN 0378-1844
INCI v.33 n.5 Caracas mayo 2008
Marcadores aflp en la evaluación de la diversidad genética de variedades y líneas élites de arroz en venezuela
Erika Arnao, Yorman Jayaro, Patricio Hinrichsen, Catalina Ramis, Carlos Marín R. e Iris Pérez-Almeida
Erika A. Arnao T. Ingeniera Agrónoma y Magíster en Agronomía, Universidad Central de Venezuela (UCV). Investigadora, Fundación para la Investigación Agrícola Danac, Venezuela. Dirección: Fundación Danac, Apartado 182, San Javier, vía Guarataro, Yaracuy, Venezuela. e-mail: erika.arnao@danac.org.ve
Yorman G. Jayaro. Ingeniero Agrónomo, UCV, Venezuela. Investigador, Fundación Danac, Venezuela. e-mail: yorman.jayaro@danac.org.ve
Patricio Hinrichsen. Bioquímico, Universidad de Concepción, Chile. Doctor en Ciencias Biológicas, Pontificia Universidad Católica de Chile. Investigador, Instituto de Investigaciones Agropecuarias, Santiago, Chile. e-mail: phinrichsen@inia.cl
Catalina M. Ramis J. Ingeniera Agrónoma, UCV, Venezuela. Doctora, Université de Rouen, Francia. Profesora, UCV, Venezuela. e-mail: cmcramis@yahoo.es
Carlos Marín R. Ingeniero Agrónomo, UCV, Venezuela. Técnico, Centro Nacional de Investigaciones Agropecuarias (CENIAP), Instituto Nacional de Investigaciones Agrícolas (INIA), Venezuela. e-mail: cmarin@inia.gob.ve.
Iris Pérez-Almeida. IngenieraAgrónoma y Magíster en Ciencias, UCV, Venezuela. Doctora en Biología Molecular de Plantas, Purdue University, EEUU. Investigadora, CENIAP-INIA, Venezuela. e-mail: iperez@inia.gob.ve
RESUMEN
Los marcadores moleculares representan una herramienta de gran precisión para la evaluación de la diversidad genética y la determinación de la identidad de variedades, especialmente en cultivos de estrecha base genética tales como el arroz. El objetivo de este estudio fue evaluar la diversidad genética de variedades de arroz venezolanas y de líneas élites provenientes del Plan Nacional de Mejoramiento Genético de Arroz (INIA y otras instituciones), de la Fundación Danac y de Productores Asociados Chispa, usando cinco combinaciones de iniciadores AFLP. Además se incluyeron dos arroces rojos. Se registraron 220 bandas desde 25 a >300pb, de las que 60 (27,27%) resultaron polimórficas, con promedio de 12 bandas por combinación. El índice de diversidad de Shannon para cada combinación varió de 0,17 a 0,41 con promedio de 0,29 ±0,10. Dicho índice mostró un moderado nivel de polimorfismo genético entre las variedades, líneas élites y arroz rojo. El análisis de agrupamiento permitió identificar cinco grupos, a una distancia de ~0,50 unidades ultramétricas. El primer grupo quedó conformado solo por arroces rojos; los grupos restantes estuvieron constituidos tanto por variedades como por líneas experimentales avanzadas. Las combinaciones AFLP permitieron la diferenciación de todos los materiales, observándose asociación entre los agrupamientos generados y la constitución genética de los genotipos. Este trabajo corrobora la utilidad de los AFLP como herramienta para discriminar entre individuos altamente emparentados.
Aflp markers in the evaluation of genetic diversity of rice varieties and elite lines in venezuela
SUMMARY
Molecular markers represent a high-precision tool for the assessment of genetic diversity and to determine the identity of varieties, especially in crops of limited genetic basis such as rice. The aim of this study was to assess the genetic diversity of Venezuelan rice varieties and élite lines developed by the National Plan of Rice breeding (INIA and other institutions), Danac Foundation and Chispa Associated Producers using five AFLP primer combinations. Two red rice were also included. Two hundred and twenty bands were registered with molecular size from 25 to >300bp, of which 60 (27.27%) resulted polymorphic, the average showing 12 bands per combination. Shannon diversity index for each combination ranged from 0.17 to 0.41 with an average of 0.29 ±0.10. The index showed a moderate level of genetic polymorphism among varieties, élite lines and red rice. Cluster analysis allowed identification of five groups, at a distance of ~0.50 ultrametric units. The first group was formed by red rice only; the remaining groups were formed by both varieties and advanced experimental lines. AFLP combinations allowed differentiation of all the materials, there being association between generated clusters and genotype genetic constitution. This work confirms AFLP usefulness as a tool for discrimination among highly related individuals.
Marcadores aflp na avaliação da diversidade genética de variedades e linhas elites de arroz na venezuela
RESUMO
Os marcadores moleculares representam uma ferramenta de grande precisão para a avaliação da diversidade genética e a determinação da identidade de variedades, especialmente em cultivos de estreita base genética tais como o arroz. O objetivo deste estudo foi avaliar a diversidade genética de variedades de arroz venezuelanas e de linhas elites provenientes do Plano Nacional de Melhoramento Genético do Arroz (INIA e outras instituições), da Fundação Danac e de Produtores Associados Chispa, usando cinco combinações de iniciadores AFLP. Além disso, se incluíram dois tipos de arroz vermelho. Registraram-se 220 faixas desde 25 a >300pb, das quais 60 (27,27%) resultaram polimórficas, com média de 12 faixas por combinação. O índice de diversidade de Shannon para cada combinação variou de 0,17 a 0,41 com média de 0,29 ±0,10. Dito índice mostrou um moderado nível de polimorfismo genético entre as variedades, linhas elites e arroz vermelho. A análise de agrupamento permitu identificar cinco grupos, a uma distancia de ~0,50 unidades ultramétricas. O primeiro grupo ficou conformado somente por arroz vermelho; os grupos restantes estiveram constituídos tanto por variedades como por linhas experimentais avançadas. As combinações AFLP permitiram a diferenciação de todos os materiais, observando-se associação entre os agrupamentos gerados e a constituição genética dos genótipos. Este trabalho corrobora a utilidade dos AFLP como ferramenta para discriminar entre indivíduos altamente aparentados.
PALABRAS CLAVE / AFLP/ Diversidad Genética/ Marcador Molecular/ Oryza sativa L./
Recibido: 21/03/2007. Modificado: 18/03/2008. Aceptado: 24/03/2008.
Introducción
El éxito de cualquier programa de mejoramiento genético de plantas es asegurado en gran medida por la selección de los progenitores que formarán la población base que da inicio al programa de selección. Tales poblaciones deben contener una amplia variabilidad genética, por lo que es importante conocer las relaciones genéticas entre las líneas utilizadas como progenitores (Virk et al., 2000). Este conocimiento ha tenido un impacto significativo en el mejoramiento de plantas, siendo la información generada útil tanto en la planificación de cruces para la obtención de híbridos y desarrollo de líneas, como en la asignación de líneas a grupos heteróticos y en la protección varietal (Peric et al., 1998)
Para estimar la diversidad genética de los cultivares se utilizan varios métodos, basados en la morfología, en el pedigrí y en el uso de marcadores moleculares, cuyo uso ha aumentado últimamente. La identificación basada en caracteres morfológicos, a pesar de ser ampliamente usada, ha resultado ineficiente en genotipos altamente emparentados, donde las características varietales diferenciales son escasas, además de ser altamente influenciadas por el ambiente, y requerir de evaluaciones en campo que ameritan considerable gasto de dinero, tiempo y espacio. Comparados con los análisis morfológicos, los marcadores moleculares pueden revelar diferencias entre accesiones a nivel de ADN proveyendo un método más directo, confiable, y una herramienta eficiente para la conservación y manejo del germoplasma (Ni et al., 2002; Rafalski, 2002).
En arroz, los mejoradores han reconocido en los últimos años la situación de la estrecha base genética debida a la reducción de la diversidad genética, producto del mejoramiento de los cultivares modernos, lo cual ha resultado en cultivos genéticamente vulnerables ante factores abióticos y agentes bióticos (Tang et al., 2007). En años recientes, además de los caracteres morfológicos, diversos marcadores moleculares han sido usados extensivamente para medir la variación genética a nivel del ADN en arroz. Entre estos marcadores cabe mencionar las isoenzimas, el polimorfismo en la longitud de los fragmentos de restricción (RFLP), el ADN polimórfico amplificado al azar (RAPD), los microsatélites o secuencias simples repetidas (SSR) y el polimorfismo en la longitud de los fragmentos de restricción amplificados (AFLP). El uso de estos marcadores ha permitido una estimación de las relaciones genéticas entre los individuos sin la complicación de las interacciones genotipo-ambiente, a fin de entender las razones que causan la estrecha base genética de las variedades modernas (Glaszmann, 1987; Yang et al., 1994; Caldo, et al., 1997; Ashikari y Matsuoka, 2002; Blair et al., 2002; Virk et al., 2000; Yuan et al., 2007).
Los AFLP son marcadores basados en la reacción en cadena de la polimerasa (PCR) que tienen la capacidad de detectar un gran número de loci, y presentan una mayor tasa de polimorfismo por ensayo en comparación con los RAPD y RFLP. La técnica fue desarrollada por Vos et al. (1995) y su fundamento se basa en una amplificación selectiva de un subconjunto de fragmentos de restricción provenientes de la mezcla compleja de fragmentos de ADN, obtenidos luego de la digestión del ADN genómico con dos endonucleasas de restricción. El polimorfismo es detectado por el uso de un número de bases selectivas contiguas al sitio de restricción y se visualizan por las diferencias en la longitud de los fragmentos separados en electroforesis en geles de poliacrilamida (Vos et al., 1995; Powell et al., 1996; James et al., 2003).
La tecnología de AFLP ha sido señalada como una herramienta poderosa para la detección de las relaciones genéticas, en el análisis de la evolución de colecciones de germoplasma y en la evaluación de la biodiversidad, así como en los estudios de huellas moleculares con fines de protección de los derechos de obtentor (Ovesna et al., 2007). Aguirre et al. (2005) utilizando 21 combinaciones AFLP evaluaron la diversidad genética de doce cultivares y líneas de arroz desarrollados por el Programa de Mejoramiento de Arroz en Chile. En dicho estudio, a pesar de encontrarse baja diversidad genética entre los materiales fue posible discriminar los cultivares con tres de las combinaciones AFLP utilizadas. Saker et al. (2005) realizaron el análisis de la diversidad genética de algunos cultivares de arroz en Egipto usando marcadores RAPD, SSR y AFLP; y encontraron buena discriminación de las variedades analizadas con los tres sistemas.
El objetivo del presente trabajo fue determinar la diversidad genética de algunas variedades y líneas élites de arroz venezolanas mediante marcadores AFLP.
Materiales y Métodos
Material vegetal
Se utilizaron 43 genotipos de arroz que incluyeron 10 variedades y 31 líneas experimentales desarrolladas en Venezuela, estas últimas provenientes del Plan Nacional de Mejoramiento Genético de Arroz, del programa de mejoramiento de la Fundación para la Investigación Agrícola Danac y de la empresa Productores Asociados Chispa. Las líneas experimentales evaluadas fueron originadas a partir de tres estrategias de mejoramiento: la realización de cruzamientos de dos y tres progenitores, la extracción a partir de una población de selección recurrente con androesterilidad (PCT-16), y la introducción desde otros programas de mejoramiento internacionales. Con la finalidad de evaluar la capacidad de discriminación del arroz maleza rojo, se incluyeron en el estudio dos arroces rojos provenientes de los estados de Guárico y Portuguesa, Venezuela (Tabla I).
Extracción del ADN
Hojas frescas de plantas de arroz de ~15 días de edad fueron maceradas con N2 líquido en microtubos de 1,5ml hasta la pulverización del tejido. El ADN fue extraído utilizando el kit Wizard® (Promega, EEUU). Las concentraciones de los ADN obtenidos fueron cuantificadas en un espectrofotómetro y en geles de agarosa teñidos con SYBR® Green I, (Sigma, EEUU), comparándolos con estándares comerciales de concentración conocida y ajustadas a 25ng·µl-1.
Análisis AFLP
El procedimiento de AFLP fue desarrollado según protocolo de Vos et al. (1995) con pocas modificaciones (Aguirre et al., 2005) y constó de los siguientes pasos: digestión del ADN, ligación de los adaptadores, amplificación preselectiva, amplificación selectiva y separación de los fragmentos en geles de poliacrilamida.
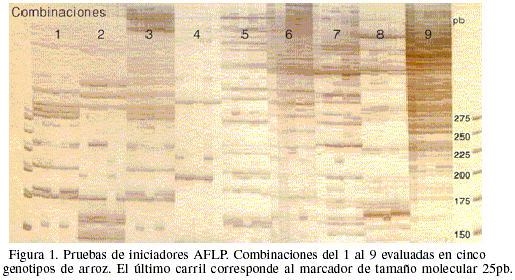
Para la amplificación selectiva se evaluaron inicialmente cinco genotipos de arroz tomados al azar con 24 combinaciones de iniciadores (Figura 1), para seleccionar las cinco combinaciones más polimórficas y de mayor resolución que serían usados para evaluar todos los genotipos de arroz.
Análisis de datos
La diversidad genética detectada a través de los marcadores AFLP en las variedades y líneas élites de arroz fue estudiada empleando el índice de Shannon, según procedimiento descrito por Shannon y Weaver (1949) y el análisis de conglomerados, basado en el análisis de clasificación jerárquica ascendente (ACJA), siguiendo el procedimiento descrito por Rohlf y Fisher (1968).
Para el ACJA las bandas polimórficas fueron genotipadas manualmente mediante un código binario de presencia (1) y ausencia (0). El análisis de agrupamiento entre las variedades y líneas élites de arroz se realizó (Rohlf y Fisher, 1968) a partir de la distancia (1-S) basada en el algoritmo de similitud de Dice (2a/(2a+b+c)). En la construcción del dendrograma se empleó el método de Ward como algoritmo de aglomeración (Crisci y López Armengol, 1983).
El criterio para seleccionar el algoritmo de Dice se fundamentó en que éste considera solo coincidencias en presencia de las bandas, generando una matriz de similitud más consistente que los algoritmos de Hamann, Jaccard y single matching (Cuadras, 1991). La matriz de distancia obtenida a partir del algoritmo de Dice presentó un coeficiente de correlación cofenética de 0,70, superior a los coeficientes cofenéticos obtenidos por los otros métodos.
En cuanto al método de aglomeración de Ward, el mismo fue empleado ya que generó grupos más compactos con varianzas dentro de grupos inferiores a las varianzas entre grupos; para ello se empleó el método de las siluetas estadísticas de Kaufman y Rouseeuw (1990) citados por Cuadras (1991), con un nivel de remuestreo (bootstrapping) de 500, donde se demostró que con cinco clases se obtiene las menores varianzas dentro de grupos (Figura 2).
Los datos fueron tabulados empleando la hoja de cálculo electrónica Office Excel© 2003 de Microsoft®. El programa Winstat v. 1.0 del ITCF-CIRAD, Francia (Winstat, 1996) fue utilizado para ejecutar los análisis estadísticos relacionados con el ACJA. El resto de los análisis se realizó con el Infostat v 1.2/Profesional de la Universidad Nacional de Córdoba, Argentina.
Resultados y Discusión
Las combinaciones 1, 2, 3, 7, y 8 fueron seleccionadas en base al número y resolución de las bandas amplificadas a partir de la evaluación inicial con cinco genotipos. Estas combinaciones produjeron un total de 220 bandas con tamaños moleculares desde 25 a > 300pb, menores que 100 pares de bases (pb) hasta mayores que 300pb. El número de bandas totales varió de 29 (Combinación 2) a 58 (Combinación 3), con un promedio de 44 bandas por combinación. Alrededor de 60 bandas (27,27%) mostraron polimorfismo, y el número de bandas polimórficas por combinación varió de 6 (Combinación 2) a 17 (Combinaciones 1 y 7). El índice de diversidad de Shannon varió de 0,17 a 0,41 con un promedio de 0,29 ±0,10. La combinación 3 presentó la mayor diversidad (Tabla II). Estos resultados mostraron un bajo nivel de polimorfismo genético detectado en las variedades, líneas élites y arroz rojo por las combinaciones de iniciadores AFLP evaluadas. Sin embargo, el nivel de polimorfismo fue superior a los obtenidos por Aguirre et al., (2005), quienes al evaluar 12 materiales chilenos (tipo Japonica) con 16 combinaciones encontraron 667 bandas, de las cuales sólo 94 (14,4%) resultaron polimórficas, y donde el porcentaje de bandas por combinación fue de 44, siendo las combinaciones 1 y 2, al igual que en nuestro caso, dos de las tres combinaciones más informativas.
El análisis de agrupamiento permitió la identificación de cinco grupos, a una distancia de ~0,5 unidades ultramétricas (Figura 3). El grupo I se formó a 0,20 unidades ultramétricas; el grupo II entre 0,10 y 0,35; el grupo III entre 0,09 y 0,28; el grupo IV entre 0,12 y 0,35; y el grupo V entre 0,02 y 0,28 unidades. El grupo I quedó conformado solo por arroces rojos. Esta separación respecto a las variedades comerciales y líneas experimentales coincide con los resultados obtenidos por Ortiz et al. (2004), quienes lograron la identificación de marcadores bioquímicos susceptibles de ser usados en la identificación de contaminación con arroz rojo durante la producción de semilla certificada de variedades comerciales de arroz.
Los grupos restantes estuvieron conformados tanto por variedades como por líneas experimentales avanzadas. Con la finalidad de evaluar la veracidad de los agrupamientos obtenidos mediante el análisis multivariado de los datos generados con los marcadores AFLP, se realizó una revisión de la genealogía de algunas variedades y líneas experimentales de diversos orígenes, incluidas en algunos de los grupos resultantes. Al evaluar la constitución genética de estos genotipos se observó relación con los agrupamientos obtenidos. La variedad Fundarroz PN1 (FUNDPN1), por ejemplo, resultó en el grupo IV, mientras que las variedades Cimarrón, Araure 1 y Fonaiap 1 son parte del grupo V. Fundarroz PN1 posee un aporte de 47,8% de su genoma proveniente de progenitores distintos al de las otras tres variedades (Tabla III) lo que pudiese explicar que Fundarroz PN1 resulte en un grupo distinto a Araure 1, Cimarrón y Fonaiap 1 (Figura 3).
Las líneas experimentales produjeron resultados similares a los obtenidos para las variedades. Las líneas FD14 y FD15 (grupo V) provienen de la retrocruza Cimarrón/Fedearroz 50//Cimarrón, mientras que FD4 y FD9 (grupo V) se originaron a partir del cruce de tres líneas: Fonaiap 1/Fundarroz PN1//CT11026. Puede apreciarse en la Tabla IV que los progenitores de FD14 y FD15 (Cimarrón y Fedearroz 50) son completamente distintos, mientras que los progenitores de FD4 y FD9 (Fonaiap 1, Fundarroz PN1 y CT11026) presentan coincidencias en su constitución genética, representadas por 8 variedades con contribuciones importantes en sus genomas (Tabla III). Esto podría explicar el hecho que FD14 y FD15 resulten más distantes entre sí que FD4 y FD9 en el análisis de agrupamiento (Figura 3). Es de destacar que aún cuando las líneas FD4 y FD9 presentan ancestros comunes y se separaron a un nivel de endocría considerable (familias F5, datos de genealogía no mostrados), pudieron ser discriminadas por las pocas combinaciones de marcadores utilizados.
Las líneas derivadas de poblaciones de selección recurrente muestran un comportamiento más variable, debido probablemente a la metodología utilizada para su extracción. Los tres pares de líneas FD2 y FD7 (grupo III), FD18 y FD19 (grupo V), y FD8 y FD6 (grupo III) fueron derivadas en un mismo ciclo de una población denominada PCT-16, a partir de tres plantas S0 distintas, respectivamente. A pesar de que la separación de cada par de líneas se produjo a un mismo nivel de endocría (familias S2), las distancias entre las líneas de cada par son distintas: 0,06 entre FD18 y FD19; 0,15 entre FD2 y FD7; y 0,25 entre FD6 y FD8 (Figura 2). Esta variación se debe probablemente a diferencias en el estado de homocigosis inicial de las plantas S0 seleccionadas para derivar las líneas: plantas S0 más heterocigotas probablemente producirían líneas más diversas, mientras que plantas más homocigotas generarían líneas con mayor semejanza genética. Aún cuando al análisis de los agrupamientos generados se realizó con base en algunas de las variedades y líneas evaluadas, los resultados muestran indicios de correspondencia entre dichos agrupamientos y la constitución genética de los genotipos. En general, todos los genotipos de arroz pudieron ser diferenciados, incluyendo aquellos con progenitores comunes y alto grado de parentesco, lo cual indica que los marcadores AFLP representan una buena herramienta para discriminar entre individuos altamente emparentados. Estos resultados evidencian la utilidad de este tipo de marcadores para la realización de estudios de diversidad genética en variedades y líneas elites de arroz, información que puede ser de gran utilidad en el diseño de métodos y estrategias de mejoramiento genético.
AGRADECIMIENTOS
Los autores agradecen al Proyecto BID-FONACIT II "Aplicación de la biotecnología como herramienta de apoyo al mejoramiento genético del arroz en Venezuela con miras a aumentar la resistencia a estrés biótico y la calidad de grano" (Subproyecto Nº 2004000369) por su apoyo financiero, al Laboratorio de Biotecnología del Instituto Nacional de Investigaciones Agropecuarias, Chile y al Laboratorio de Biología Molecular de Fundación Danac, Venezuela.
REFERENCIAS
1. Aguirre C, Alvarado R, Hinrichsen P (2005) Identificación de cultivares y líneas de mejoramiento de arroz de Chile mediante Amplificación de Fragmentos Polimórficos (AFLP). Agricultura Técnica (Chile), 65: 356-369. [ Links ]
2. Ashikari M, Matsuoka M (2002) Application of rice genomics to plant biology and breeding. Bot. Bull. Acad. Sin. 43: 1-11. [ Links ]
3. Blair MW, Hedetale V, McCouch SR (2002) Fluorescent labeled microsatellite panels useful for detecting allelic diversity in cultivated rice (Oryza sativa L.). Theor. Appl. Genet. 105: 449-457. [ Links ]
4. Caldo RA, Sebastian LS, Hernández JE (1997) Diversity of Philippines improved rice varieties and their progenitors using morphological and molecular markers. Philipp. J. Crop Sci. 22: 11–26. [ Links ]
5. Crisci JV, López-Armengol MF (1983) Introducción a la Teoría y Práctica de la Taxonomía Numérica. Serie de Biología N° 26. Organización de los Estados Americanos. Washington, DC, EEUU. 132 pp. [ Links ]
6. Cuadras CM (1991) Métodos de Análisis Multivariante. Promociones y Publicaciones Universitarias. Barcelona, España. 644 pp. [ Links ]
7. Glaszmann JC (1987) Isozymes and classification of Asian rice varieties. Theor. Appl. Genet. 74: 21-30. [ Links ]
8. InfoStat (2004) InfoStat ver. 2004. Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina. [ Links ]
9. James CM, Lesemann SS, Down GJ (2003) Modified AFLP analysis method for species with small genomes. Plant Mol. Biol. Rep. 21: 303-307. [ Links ]
10. Ni J, Peter M, Colowit M, Mackill J (2002) Evaluation of genetic diversity in rice subspecies using microsatellite markers. Crop Sci. 42: 601-607. [ Links ]
11. Ortiz A, Gómez O, Ramis C (2004) Caracterización bioquímica de poblaciones del complejo maleza arroz rojo en Venezuela. Agron. Trop. 54: 257-273. [ Links ]
12. Ovesna J, Polakova K, Lisova L (2002) DNA analyses and their applications in plant breeding. Czech. J. Genet. Plant Breed. 38: 29-40. [ Links ]
13. Peric I, Ajmone-Marsan P, Margante M, Kosumplick V, Castiglioni P, Taramino G, Motto M (1998) Comparative analysis of genetic similarity among maize inbred lines detected by RFLPs, RAPDs, SSRs, and AFLPs. Theor Appl Genet. 97: 12498-1255. [ Links ]
14. Powell W, Margenta M, Andre C, Hanfrey M, Vogel J, Tingey S, Rafalsky A (1996) The utility of RFLP, RADP, AFLP and SSR (microsatellites) markers for germoplasm analysis. Mol. Breed 2: 225-238. [ Links ]
15. Rafalski JA (2002) Applications of single nucleotide polymorphisms in crop genetics. Curr. Opin. Plant Biol. 5: 94-100. [ Links ]
16. Rohlf FJ, Fisher DL (1968) Test for hierarchical structure in random data sets. Systemat. Zool. 17: 407-412. [ Links ]
17. Saker MM, Youssef SS, Abdallah NA, Bashandy HS, El Sharkawy AM (2005) Genetic analysis of some Egyptian rice genotypes using RAPDS, SSR and AFLP. Afr. J. Biotechnol. 4: 882-890. [ Links ]
18. Savelkoul PHM, Aarts HJM, de Haas J, Dijkshoorn L, Duim B, Otsen M, Rademaker JLW, Schouls L, Lenstra A (1999) Amplified-Fragment length polymorphism analysis: the state of an art. J. Clin. Microbiol. 37: 3083-3091. [ Links ]
19. Shannon C, Weaver W (1949) The Mathematical Theory of Comunication. University of Illinois. Press. Urbana, IL, EEUU. 144 pp. [ Links ]
20. Tang SX, Wei XH, Jian YZ, Brar DS, Cruz GS (2007) Genetic Diversity bases on allozymae alleles of Chinese cultivated rice. Agricultural Sciences in China, 6: 641-646. [ Links ]
21. Virk S, Zhu J, Newbury H, Bryan G, Jackson M, Ford-Lloyd B (2000) Effectiveness of different classes of molecular markers for classifying and revealing variations in rice (Oryza sativa) germplasm. Euphytica 112: 275-284. [ Links ]
22. Vos P, Hogers R, Bleeker M, Rijans M, Van de Lee T, Hornes M, Frijters A, Pot J, Peleman J, Kuiper M, Zabeau M (1995) AFLP: A new technique for DNA fingerprinting. Nucl. Acids Res. 23: 4407-4414. [ Links ]
23. Winstat (1996) Winstat ver. 1.0 - 24/06/1996. Copyright© ITCF / CIRAD. Montpellier, Francia. [ Links ]
24. Yang GP, Saghai Maroof MA, Xu CG, Zhang QF, Biyashev RM (1994) Comparative analysis of microsatellite DNA polymorphism in landraces and cultivars of rice. Mol. Gen. Genet. 245: 187-194. [ Links ]
25. Yuan XP, Wei XH, Hua L, Yu HV, Wang YP, Xu Q, Tang SX (2007) A Comparative Study of SSR Diversity in Chinese Major Rice Varieties Planted in the 1950s and in the Recent Ten Years (1995-2004). Rice Sci. 14: 78-84. [ Links ]












 uBio
uBio