Investigación Clínica
versión impresa ISSN 0535-5133
Invest. clín v.44 n.4 Maracaibo dic. 2003
Anomalías cromosómicas en pacientes venezolanos con mieloma múltiple.
Maribel Quintero1,2, Alicia Rojas-Atencio2, Ana Ruiz1, Maczy González1, Jorge Herrera3, Fátima Atencio3, Wilmer Delgado2, Karelis Urdaneta2 y Frankie Pérez2.
1Escuela de Bioanálisis, 2Unidad de Genética Médica, Facultad de Medicina y
3Servicio de Hematología, Unidad Docente Asistencial, Hospital Central de Maracaibo. Universidad del Zulia, Maracaibo, Venezuela.
Resumen.
El estudio citogenético es un factor pronóstico fundamental en el estudio de mieloma múltiple (MM), puesto que, ha demostrado ser esencial para el asesoramiento genético en relación al diagnóstico, pronóstico y en cierta medida sugiere precozmente, el tratamiento más adecuado, hecho que representa una verdad aplicable a numerosas malignidades hematológicas. El objetivo de este trabajo fue identificar las anomalías cromosómicas en muestras de médula ósea (MO), obtenidas de pacientes con diagnóstico de MM. Los estudios cromosómicos se realizaron en cultivos de médula ósea, siguiendo la técnica descrita por Yunis en 1981; sin excepción, se llevaron a cabo previos a cualquier tratamiento con citostáticos. En la Unidad de Genética Médica de la Universidad del Zulia, se recibieron para estudio cromosómico, 22 muestras de MO, de las cuales 19 (86%) aportaron material adecuado, seis (32%) mostraron cariotipo normal, 13 (68%) anomalías cromosómicas numéricas y estructurales. De las anomalías observadas, 8 fueron numéricas (62%) y de ellas 3 casos (38%) correspondieron a hiperdiploidías, que involucraron los cromosomas 3, 5, 7, 15, 17, 18, 19. Cuatro casos (50%) mostraron hipodiploidías con pérdida de los cromosomas 8, 13, 16, 17, 18, X, Y, y se halló 1 caso de triploidía (12%). Cuatro casos correspondieron a anomalías estructurales (31%), como las deleciones 5p11, 11p14, 14q32, 17p11 y 1 caso presentó una combinación de anomalías tanto numérica como estructural (7%). El estudio demuestra la presencia de anomalías cromosómicas en la mayoría de los pacientes, los cuales presentan algunas diferencias con lo reportado en otras investigaciones.
Palabras clave: Mieloma múltiple, citogenética, anomalías cromosómicas.
Chromosomal abnormalities in venezuelan people with multiple myeloma.
Abstract.
The cytogenetic study is an important prognostic factor in Multiple Myeloma (MM). The chromosomal analysis has demonstrated to be essential for the genetic advise in relation to the diagnosis, prognosis and might suggest precociously, the most appropriate treatment for the majority of hematological malignancies. The objective of this investigation was to identify the chromosomal abnormalities in samples of bone marrow (BM) from patients with diagnosis of MM. The chromosomal studies were carried out in BM cultures, following the technique described by Yunis. Without exception the analysis was carried out previous to any treatment with cytostatics. Twenty two samples of BM were received for chromosomal studies in the Unit of Medical Genetics of the University of the Zulia (UGM -LUZ). In 19 out of 22 samples (86%) appropriate material was obtained by cytogenetic analysis; 6 (32%) showed normal karyotype and 13 (68%) presented numeric and structural chromosomal abnormalities. Eight (62%) of the chromosomal anomalies detected were numerics, three cases (38%) with hyperdiploidy involving chromosomes 3, 5, 7, 15, 17, 18, 19 and four cases (50%) with hypodiploidy involving the chromosomes 8, 16, 17, 18, X and Y. Triploidy was found in one case (12%). Structural abnormalities were present in 4 cases (31%) such as deletions 5p11, 11p14, 14q32, 17p11 and 1 case (7%) presented structural and numeric anomalies. This study shows that the majority of patients with multiple myeloma have several chomosomal abnormalities with some differences from other reports.
Key words: Multiple myeloma, cytogenetics, chromosomal abnormalities.
Recibido: 23-02-2003. Aceptado: 25-09- 2003
INTRODUCCIÓN
El Mieloma Múltiple (MM) constituye el prototipo de gammapatía monoclonal maligna y se caracteriza por la proliferación neoplásica de una clona de células plasmáticas que produce una inmunoglobulina de carácter monoclonal. Dicha proliferación da lugar a destrucción esquelética con osteoporosis y/o osteólisis, hipercalcemia, anemia y, en ocasiones, plasmocitomas extramedulares. Por otra parte, el exceso de producción de la proteína monoclonal puede conducir a insuficiencia renal, infecciones bacterianas a repetición y a síndrome de hiperviscosidad (1-3). Aunque los primeros casos bien documentados de MM se describieron en 1844 y 1850, esta enfermedad fue rara vez reconocida hasta la descripción que Kahler efectuó en 1889 (3). La incidencia anual del MM en U.S.A, es de 4 casos nuevos por año por cada 100.000 habitantes (2). El mieloma múltiple representa el 1% de todas las neoplasias y algo más del 10% de todas las hemopatías malignas. Se produce en todas las razas y áreas geográficas. No existe un claro predominio sexual. La edad media se sitúa alrededor de los 65 años (1, 3, 4). El diagnóstico de MM generalmente no ofrece muchas dudas ya que la mayoría de los pacientes presentan los criterios clínicos, radiológicos y de laboratorio establecidos previamente. Hoy es conocido, que el estudio citogenético es un factor pronóstico fundamental en el estudio de MM (4-6). En los últimos años el estudio del análisis cromosómico ha demostrado ser esencial para el asesoramiento genético con relación al diagnóstico y pronóstico, y en cierta medida sugiere precozmente el tratamiento más adecuado tanto para esta entidad como para otras malignidades hematológicas (7).
En el MM las alteraciones cromosómicas frecuentes son los cambios numéricos complejos con ganancias (especialmente 3, 5, 7, 9, 11, 15, 18, 21) y pérdidas (particularmente 8, 13 y 16), así como cambios estructurales de los cromosomas 1, 14, 16. (1-3). La presencia de translocaciones y de anomalías de los cromosomas 11, 13, 16 se asocia a formas agresivas de la enfermedad y a un pronóstico desfavorable, mientras que los pacientes con trisomías de los cromosomas 6, 9, 17 tienen una sobrevida prolongada (1, 3).
El estudio citogenético en MM aún no se ha establecido sistemáticamente en nuestro país, y el presente estudio es uno de los primeros trabajos de investigación que considera este aspecto en Venezuela, y tiene como objetivo, identificar las anomalías cromosómicas en muestras de médula ósea obtenidas de pacientes de la región occidental del país con diagnóstico de Mieloma Múltiple.
MATERIALES Y MÉTODOS
Se recibieron 22 muestras de médula ósea (MO), para estudio cromosómico, provenientes de pacientes con al menos uno de sus progenitores venezolano, en el Laboratorio de Citogenética de la Unidad de Genética Médica de la Universidad del Zulia, de ambos sexos, con diagnóstico de MM, clasificados según Durie y Salmón (4), en el período de enero 1998 a septiembre 2002.
Las muestras de MO se extrajeron mediante aspiración a nivel esternal y todas sin excepción, se cultivaron previo a la quimioterapia con citostáticos. Los cultivos para la obtención de cromosomas se realizaron siguiendo la técnica descrita por Yunis en 1981 (8), con una modificación importante representada en la utilización del Amniomax C-100 (Gibco. BRL) como medio de cultivo utilizado, suplementado con suero fetal al 20% y 1% de glutamina, sin la utilización de ningún agente estimulante (mitógeno). La incubación se realizó a 37°C durante 24 a 48 horas, y los cultivos se recogieron siguiendo esta técnica. La coloración fue realizada según el método de Bandas G, descrita por Summer y col, utilizando colorante de Wrigth (GSW). (9,10). Los cariotipos fueron analizados mediante el sistema llamado Computer-Assisted Kariotyping System que produce imágenes digitalizadas que son rápidamente reemplazadas por impresiones fotográficas.
Los cariotipos fueron descritos según versión referida en el Sistema Internacional de Nomenclatura Citogenética 1995 (ISCN.1995) (11). Fueron considerados clones anormales aquellos que se repetían 2 o más veces en el caso de anomalías estructurales y 3 o más veces en el caso de pérdidas o ganancias de cromosomas (5).
RESULTADOS
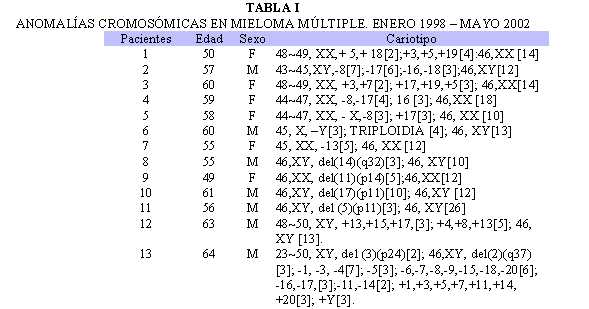
La edad de los pacientes estuvo comprendida entre 49 y 70 años, (promedio de edad 57 años), con predominio del sexo masculino, el cual estaba más frecuentemente afectado en los grupos etarios entre 51- 60 años y 61- 70 años (Tabla I).
En 19 de las 22 muestras cultivadas, se obtuvo material adecuado para análisis citogenético. En 6 (32%) se observó un cariotipo normal, y en 13 (68%) se encontraron anomalías cromosómicas tanto numéricas como estructurales. Las anomalías cromosómicas observadas estuvieron representadas por las anomalías numéricas en 8 de los casos (62%), 3 casos (38%) correspondieron a hiperdiploidías (Fig. 1), 4 casos (50%) a hipodiploidías (Fig. 2) y un caso con triplodía (12%). Las anomalías estructurales correspondieron a 4 casos (31%) (Fig. 3). Un caso presentó combinación de anomalías numéricas y estructurales (7%) (Fig. 4). Se analizaron entre 13 y 29 metafases por paciente, esto dependió de la calidad y cantidad obtenida para cada caso. La Tabla I muestra las anomalías cromosómicas encontradas en los diferentes pacientes.
DISCUSIÓN
La investigación en el área de la genética, orientada al estudio de las neoplasias, ha sido fructífera no solo porque ha permitido importantes avances en la comprensión del fenómeno de la transformación maligna, sino porque además se han descubierto alteraciones cromosómicas, tanto numéricas como estructurales, útiles en el diagnóstico y el pronóstico de las entidades neoplásicas en general y porque, con el conocimiento acerca de los oncogenes y su funcionamiento, se han abierto perspectivas para la prevención y el tratamiento del cáncer (12-16).
Nuestros resultados muestran clones citogenéticos anormales en el 68% de los pacientes con MM sin tratamiento con citostáticos, hallazgo que corresponde a un porcentaje mucho más alto de lo que hasta ahora ha sido reportado por otros autores. Sawyer y col. y Barlogie y col. reportaron en 1995 (15) y 2002 (19) frecuencias entre 35 y 52% respectivamente, frecuencias que fueron señaladas por otros autores (17-19). Esto puede deberse a la utilización de un medio de cultivo más enriquecido que ha permitido la obtención de metafases de mejor calidad (6,20), o a la variabilidad geográfica descrita por algunos autores (21, 22). Así mismo cabe destacar el hecho que de acuerdo con la revisión de la literatura nacional, este el primer reporte en Venezuela sobre alteraciones citogenéticas en esta enfermedad.
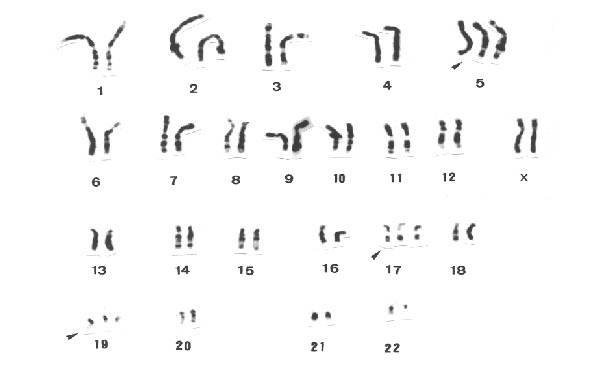
Fig 1. Muestra cariotipo hiperdiploide, las flechas señalan los cromosomas presentes en exceso.
Fig 2. Muestra cariotipo hipodiploide, las flechas señalan los cromosomas faltantes.
La naturaleza heterogénea de los cariotipos encontrados en MM es consistente con la bien conocida heterogeneidad clínica de la enfermedad, en MM se han reportado cariotipos complejos con hiperdiploidías, hipodiploidías, monosomías, trisomías y anomalías estructurales como translocaciones y deleciones. Las alteraciones más frecuentes son cambios numéricos complejos con ganancias de cromosomas, especialmente 3, 5, 9, y 11 o pérdidas de cromosomas particularmente 8, 13 y 16, así como cambios estructurales del cromosoma 1. Por otra parte se han descrito correlaciones clinico cariotípicas como del (6q) asociada a osteólisis severa; del (7q) (localización del gen MDRI) asociado a resistencia farmacológica, t (8, 14), asociada a MM IgA (3). Otra alteración descrita corresponde a adiciones en el brazo largo del cromosoma 14, las cuales generalmente provienen del cromosoma 11, ejemplo de ello lo constituye la t (11;14) (q13, q32) (1).
Fig 3. Muestra cariotipos parciales.
Fig 4. Muestra cariotipo complejo: 39, XYY, -3, -4, -6, -7, -8, -15, -16, -18, -18, -22, +1, +5, +20, +del(1)(q32), +del(3)(p24).
Zandecki y col. en el año 1994, analizando población americana, reportó en su mayoría pacientes con anomalías cromosómicas numéricas entre las cuales se incluían hiperdiploidías, hipodiploidías, pseudiploidías y cerca de la tetraploidía, siendo las más frecuentes las hiperdiploidías. (23). Los hallazgos del presente trabajo comprenden un 38% de pacientes con hiperdiploidías, éstas incluyen principalmente trisomía de los cromosomas 5, 17,19, hecho que contrasta con lo reportado por Gould y Durie y por Avet-Loiseau y col. quienes estudiando población francesa hallaron solo un 7,5% de pacientes con esta anomalía (24-26).
Clark y col. en su serie publicada en el año 1989 (27), entre otras trisomías reportó las correspondientes a los cromosomas 5 y 19, observadas en este estudio y que fueron señaladas con un patrón sugerente a posible progresión clínica.
Avet-Loiseau y col, valorando las alteraciones numéricas mediante la utilización de técnicas de Hibridación in Situ Fluorescente (FISH por sus siglas en inglés), evaluaron 15 cromosomas diferentes, que mostraron trisomía 17 en 10 de 46 pacientes analizados (26), hecho que fue interpretado por Pérez y col. como hallazgo de buen pronóstico. (28). Esta constituye una de las trisomías más frecuentemente halladas en este estudio.
En relación con las pérdidas cromosómicas, la monosomía o deleción del cromosoma 13 ha sido una de las anomalías más consistentemente encontrada y ha sido señaladas junto con las anomalías complejas con altos niveles de calcio en el suero y relacionadas con peor pronóstico (6,26, 29-31). El presente reporte incluye un caso con monosomía 13 y otro con anomalía compleja, en los cuales se evidenció este hallazgo.
Así mismo, se encontraron otras monosomías entre las que se incluyen las monosomías 8, 16 y 17. La monosomía 8 fue reportada en estudios con FISH en 2 de 39 pacientes analizados, no así el resto de las monosomías (26), pudiendo deberse a lo que otros autores han sugerido en el sentido de que es posible la existencia de variabilidad geográfica en el hallazgo de anormalidades cromosómicas, lo cual a su vez traduciría determinadas diferencias entre las razas (21, 22).
En cuanto a las alteraciones estructurales las del (13) (q12- q14) y (14) (q32), así como los rearreglos que involucran estas regiones han sido reportadas más comúnmente (16, 31). En este trabajo los hallazgos muestran 1 caso con del (14) (q32). Otras alteraciones estructurales encontradas corresponden a deleción de los cromosomas (11) (p11), 17 (p13) y 5 (p11). Konigsberg y col. (31), estudiando el papel predictivo de estos pacientes utilizando la interfase citogenética, encontró rearreglos estructurales en diferentes cromosomas, pero no reporta alteración en el cromosoma 5, y por otro lado en el cromosoma 11 se señala a nivel de la regiones del brazo largo 13 y 23, hecho que pudiera implicar la presencia de nuevos genes involucrados en esta patología. Todas estas alteraciones han sido consideradas de mal pronóstico.
En una investigación previa hecha por Schreiber y col. (32), donde realizaron un análisis de la densidad de los microvasos de la médula ósea, encontraron que aquellos pacientes con características citogenéticas desfavorables como la deleción de los cromosomas 13q14, 17q13 y 11q, presentaron mayor profusión vascular en comparación con aquellos que tenían ausencia de anomalías citogenéticas.
Los resultados reportados hasta hoy, ya son sugerentes de la importancia que representa el estudio cromosómico no solo por el valor predictivo que comienza a conocerse sino además por la identificación de las alteraciones moleculares que aún están siendo aclaradas, las cuales permitirán conocer el papel de estos cambios en la patogénesis del MM (33). Se recomienda, la inclusión del estudio cromosómico en forma rutinaria en los pacientes afectados con MM como parte de su evaluación inicial, puesto que los mismos podrían ayudar a la escogencia de una terapia más adecuada así como a identificar probables sitios de genes involucrados en esta patología, cooperando a aclarar más la patogénesis de esta enfermedad.
AGRADECIMIENTO
Al Consejo de Desarrollo Científico y Humanístico (CONDES) por el financiamiento de esta investigación. Proyecto N°: CC-0302-01 y a los pacientes sin los cuales no se hubiese hecho posible esta investigación.
REFERENCIAS
1. Beutler E, Coller B, Seligson U, Lichtman M, Kipps T. Williams Hematology. 6ta Ed. Estados Unidos de América: McGraw - Hill Medical Publising; 2001, p 1279-1291. [ Links ]
2. Lee R, Lukens J, Greer J, Foerster J, Paraskevas F, Rodgers G. Wintrobe´s Clinical Hematology. 10ma Ed. Estados Unidos: Editorial Williams & Wilkins; 1999, p 2631-2655. [ Links ]
3. Sans-Sabrafen J, Besses Raebel C, Vives Corrons J. Hematología Clínica. 4ta Ed. España: Ediciones Harcourt, S.A.; 2001, p 574-579. [ Links ]
4. Durie B. The epidemiology of multiple myeloma. Semin Hematol 2001; 38: 1-10. [ Links ]
5. Rooney D, Czepulkowski B. Human Citogenetics. A Practical Approach. 2da Ed. Estados Unidos de América: Series Editors: D. Rickwood y B. D. Hames; 1992, p 121-151. [ Links ]
6. Gutiérrez NC, Hernandez J, Garcia J, Almeida J, Mateo G, González M, Hernandez J, Hernandez-Calvo J, San Miguel J. Correlation between cytogenetic abnormalities and disease characteristic in multiple myeloma: monosomy of chromosome 13 and structural abnormalities of 11q are associated with a high percentage of S-phase plasma cells. Haematologica 2000; 85:1146-1152. [ Links ]
7. Rojas-Atencio A, Pineda L, González S, Soto M, Avila E, Urdaneta B, Prieto-Carrasquero M, González R. Anomalías cromosómicas en enfermedades hematológicas malignas. Acta Cient Venez 2000; 51:109-114. [ Links ]
8. Yunis J. New chromosome techniques in the study of human neoplasia. Hum Pathol 1981; 12:540-549 [ Links ]
9. Bernard J. Diagnóstico y tratamiento clínico por el laboratorio. 7ma Ed. España: Todd Sanford Davidsohn, Salvat Editores; 1984, p 1279-1291. [ Links ]
10. Barch M, Knutsen T, Spurbeck J. The AGT Citogenetics Laboratory Manual. 3ra Ed. Estados Unidos: Lippincott - Raven Publishers; 1997, p 269-336. [ Links ]
11. Mitelman F. An International System for Human Cytogenetics Nomenclature. S. Karger, Basel. ISCN 1995. [ Links ]
12. Avet-Loiseau H, Facon T, Godon C, Rapp MJ, Harousseau JL Grosbois B. 14q32 translocations and monosomy 13 observed in monoclonal gammapathy of undetermined significance delineated multistep process for the oncogenesis of multiple myeloma. Intergroupe Francophone du Myelome. Cancer Res 1999, 59:4546-4550 [ Links ]
13. Salamanca F. Citogenética Humana. México: Editorial Médica Panamericana; 1993, p 235-254. [ Links ]
14. Dewald GW, Kyle RA, Hicks GA, Gresp PR. The clinical significance of cytogenetic studies in 100 patients with multiple myeloma, plasma cell leukemia or amyloidoisis. Blood 1985; 66:380-390. [ Links ]
15. Sawyer J, Waldron J, Jagannath S, Barlogie B. Cytogenetics findings in 200 patients with multiple myeloma. Cancer Genet Cytogenet 1995; 82:41-49. [ Links ]
16. Barlogie B Jr, Shaughnessy JD. Early results of total therapy II in multiple myeloma: Implication of cytogenetics and FISH. Int J Hematol 2002; 1:137-139. [ Links ]
17. Cigudosa J, Calasanz M, Odero M, Prosper F, Etxaniz A, Marin J, Rifon J, Gullon A, Rocha E. Cytogenetics data in 41 patients with multiple myeloma. Karyotype and other clinical parameters. Cancer Genet Cytogenet 1994; 78:210-213. [ Links ]
18. Ankathil R, Madhavan J, Gangadharan V, Rajasekharan Pillai G, Krisman NM. Nonrandom karyotype abnormalities in 36 multiple myeloma patients. Cancer Gene Cytogenet 1995; 83:71-74. [ Links ]
19. Zandecki M, Lai JL, Facon T. Multiple myeloma; almost all patients are cytogenetically abnormal. Br J Haematol 1996, 94:217-227. [ Links ]
20. Nilsson T, Lenhoff S, Turesson I, Rylander L, Mitelman F, Westin J, Hoglund M, Johansson B. Cytogenetic features of multiple myeloma: Impact of gender, age, disease phase, culture time and cytokine stimulation. Eur J Haematol 2002; 68:345-353. [ Links ]
21. Johanson B, Mertens F, Mitelman F. Geographic heterogeneity of neoplasia associated chromosome aberration. Genes Chrom Cancer 1991; 3:1-7. [ Links ]
22. Chan L, Kwong Y, Liu H, Chan T, Tod D, Ching M. Cytogenetics analysis of hematological malignancies in Hong Kong. Cancer Genet Cytogenet 1994; 62: 154-159. [ Links ]
23. Zandecki M, Bernardi F, Luc La J, Facon T, Izydorczyk V, Bauters f, Cosson A. Image analysis in multiple myeloma at diagnosis. Correlation with cytogenetic study. Cancer Genet Cytogenet 1994; 74: 115-119. [ Links ]
24. Durie BG. The biology of multiple myeloma. Hematol Oncol 1988; 6:77-81. [ Links ]
25. Gould J, Goodacre A, Pathak S, Hecht B, Barlogie B. Plasma cell karyotype in multiple myeloma. Blood 1998, 71: 453-456. [ Links ]
26. Avet-Loiseau H, Daviet A, Brigaudeau C, Callet E, Terré Ch, Lafage M, Desangles F, Ramond S, Talmant P, Bataille R. Cytogenetic, interphase, and multicolor in situ analyses in primary plasma cell leukemia : a study of 40 patients at diagnosis, on behalf of the intergroupe Francophome du Myélome and the groupe francais de cytogenétique Hématologique. Blood 2001; 67:3 822-825. [ Links ]
27. Clark RE, Geddes AD, Whittaker JA, Jacobs A. Differences in bone marrow cytogenetic characteristics between treated and untreated myeloma. Eur J Cancer Clin Oncol 1989, 25:1789-1793 [ Links ]
28. Pérez S, García R, Tabernero M, Almeida J, González Fernández J, Moro M, Hernández J, San Miguel J, Orfao A. Pronostic value of numerical chromosome aberration in multiple myeloma: a FISH analysis of is different chromosome. Blood 1998, 91: 3366 - 3371. [ Links ]
29. Seong Ch, Delasalle K, Hayes K, Weber D, Dinopoulos M, Swantkowski J, Hun Y, Glassman A, Chaplin R, Alexanian R. Prognostic value of cytogenetics in multiple myeloma. Br J Haematol 1998; 101: 189–194 [ Links ]
30. Fassas A, Spencer T, Sawyer J, Zangari, Kee Lee CH, Anaissie E, Muwalla F, Morris Ch, Barlogie B, Tricot G. Both hypodiploidy and deletion of chromosome 13 independently confer poor prognosis in multiple myeloma. Br J Haematol 2002; 118:1041-1045. [ Links ]
31. Konigsberg R, Zoger N, Ackermann J, Kromer e, Kittler H, Fritz E, Kaufmann H, Noslinger T, Rield L, Gisslinger H, Jager U, Simonitsch I, Heinz R, Heinz l, Heinz H, Drach J. Predictive role of interphase cytogenetics for survival of patiens with multiple myeloma. J Clin Oncol 2000; 18:804 - 812. [ Links ]
32. Schreiber S, Ackermann J, Obermair A, Kaufmann H, Urbauer E, Aletaha K, Gisslinger H, Chott A, Huber H, Drach J. Multiple myeloma with deletion of chromosome 13q is characterized by increased bone marrow neovascularization. Br J Haematol 2000; 110: 605-609. [ Links ]
33. Joy Ho P. Chromosomal and genetic abnormalities in myeloma. Clin Lab Haematol 2002, 24:259-269. [ Links ]












 uBio
uBio