Revista del Instituto Nacional de Higiene Rafael Rangel
versión impresa ISSN 0798-0477
INHRR v.35 n.2 Caracas jul. 2004
Revisiones
Estructura de mosaico del cromosoma bacteriano: Islas patogénicas
Sandra Fernández F1, Guillermina Alonso2, Elsa S Toro A1.
1. Instituto Nacional de Higiene "Rafael Rangel". Departamento de Bacteriología. Ciudad Universitaria de Caracas, Apartado Postal 60412 oficina del este. Caracas. Teléfono: 58-212-2666416 Fax: 00-58-212-6934551. E-mail:sanferfi@cantv.net
2. Laboratorio de Plásmidos, Instituto de Biología Experimental, Facultad de Ciencias, Universidad Central de Venezuela.
RESUMEN
Las islas patogénicas (PAIs) son segmentos de ADN bacteriano que portan uno o más genes de virulencia, los cuales han sido adquiridos en bloque de una fuente externa. El genoma de un patógeno usualmente representa un mosaico entre estas islas recién adquiridas y un ADN relativamente antiguo. Las PAIs son identificadas por su diferencia en el porcentaje de contenido de G+C en relación a la media del cromosoma y por otras características, que sugieren su adquisición a través de elementos genéticos móviles. Genes para una amplia gama de determinantes de virulencia están asociados con PAIs, incluyendo los que codifican para ciertos mecanismos que le permiten resistir las defensas del hospedador, factores de colonización, adquisición de nutrientes y toxinas. El estudio y conocimiento de las PAIs provee evidencias importantes de la biología de las bacterias patógenas. Los productos de las PAIs podrán representar, en un futuro, blancos para la terapia antimicrobiana, vacunas y herramientas diagnósticas.
Palabras claves: Islas de patogenicidad, patogenicidad, virulencia, bacterias patógenas.
SUMMARY
The pathogenic islands (PAIs) are bacterial DNA segments that carry one or more genes of virulence, which have been acquired in block from an outsourcing. The genome of a pathogen usually represents a mosaic between these new acquired islands and a relatively old DNA. The PAIs are identified by their difference in the percentage of G+C content related to the average of the chromosome and by other characteristics, which suggest their acquisition through movable genetic elements. Genes for an wide range of virulence determinants are associate with PAIs, including which codify for certain mechanisms to resist the defenses, factors of colonization, acquisition of nutrients and toxins. The study and knowledge of the PAIs provide important evidences to the biology of the pathogenic bacteria. In a future, the products of the PAIs will be able to represent targets for the antimicrobial therapy, vaccines and diagnostic tools.
Key words: Pathogenicity island, pathogenicity, virulence, bacterial pathogens
Desde una perspectiva práctica, las bacterias patógenas son definidas como microorganismos capaces de causar una enfermedad (1). En la mayoría de los casos, la patogenicidad involucra la expresión de un gran número de productos génicos (2). En la célula se deben expresar los genes cuyos productos son requeridos para los procesos fisiológicos básicos, pero además, otras clases de genes relacionados con la virulencia, que se encuentran sólo en cepas patógenas, y sus productos juegan un rol específico en el proceso patógeno. Muchos patógenos probablemente evolucionaron por la adquisición de largas unidades de información genética, que le confieren la habilidad de colonizar nuevos nichos (3). En muchos casos, estos bloques de ADN se encuentran en plásmidos o bacteriófagos, sin embargo, muchos de los genes de virulencia se encuentran en los cromosomas bacterianos. Los proyectos genomas de varios microorganismos han permitido tener los datos de la secuencia del genoma completo, tanto de bacterias patógenas como no patógenas y el análisis de esta información ha conducido a la idea de que algunos de estos cromosomas se pueden considerar como una columna vertebral, en la cual están insertados segmentos transmitidos horizontalmente. Por ejemplo, Salmonella Typhimurium y Salmonella Typhi son considerados miembros de una misma especie, pero, 20% del genoma de S. Typhimurium no está presente en S. Typhi y viceversa (4). Estos segmentos de ADN, se denominan islas y contienen casetes genéticos que codifican diversas funciones, que van desde vías biosintéticas hasta toxinas. Un grupo importante de estas islas genómicas son las denominadas islas de patogenicidad (Pathogenicity Islands, PAIs), las cuales contienen los genes cuyos productos están involucrados en la virulencia bacteriana.
En los años 80, Goebel y colaboradores (5) analizaron cepas de E. coli uropatogénicas, capaces de producir factores de virulencia específicos, que incluían á-hemolisina (Hly). Los genes hly están localizados en regiones del cromosoma denominadas "islas de hemolisina". Estudios posteriores revelaron que genes adicionales, involucrados en la uropatogenicidad, también estaban localizados en estas islas, las cuales fueron renombradas como islas de patogenicidad. Dado que, el término PAIs se hizo popular, fue necesario desarrollar una definición precisa. Las PAIs pueden ser definidas de acuerdo con los siguientes criterios (5): (i) portan los genes de virulencia o la mayoría de estos, (ii) Están presentes en las cepas patógenas y ausentes o esporádicamente distribuidas en las cepas menos patógenas, de una misma especie o especies relacionadas, (iii) difieren en su contenido G+C en comparación con el ADN de la bacteria hospedadora, (iv) ocupan una región grande del cromosoma, usualmente mayor de 30kb, son unidades compactas, frecuentemente flanqueadas por repeticiones directas, (v) están asociadas con genes de ARNt y/o secuencias de inserción en los extremos, (vi) presentan genes de elementos móviles, usualmente crípticos, tales como: elementos de inserción, integrasas, transposasas, orígenes de replicación de plásmidos, y (vii) pueden ser inestables (6) (Figura 1).
La primera PAI fue descrita en E. coli uropatogénico (Uropathogenic E. coli, UPEC) y desde entonces, se han identificado en un rango amplio de especies. Una PAI puede portar solo un gen de virulencia, y en ese caso se les refiere como islotes de patogenicidad, por ejemplo; el islote sifA de Salmonella, que codifica una proteína secretada e involucrada en la multiplicación intracelular (7). En otros casos, pueden contener varios operones asociados con virulencia, y otras pueden ser muy grandes, por ejemplo, la PAI II de UPEC tiene un tamaño cercano a 190 kb.
La media de la composición de nucleótidos de estas regiones, de manera usual, difiere considerablemente de la media del cromosoma de la bacteria, reflejando la diferencia de uso de codones del genoma donante (8). Algunos PAIs son inestables, tienen una tasa de pérdida espontánea relativamente alta, por ejemplo, la PAI que codifica la toxina 1 del síndrome de schok tóxico de Staphylococcus aureus, es escindida del cromosoma bacteriano por bacteriófagos con una alta frecuencia (9). Sin embargo, como los genomas evolucionan, las características que diferencian las PAIs del resto del cromosoma no siempre están presentes, como es el caso del regulón vir, que codifica la proteína M de Streptococcus pyogenes, que protege a la bacteria de la fagocitosis. Esta región no contiene ninguna de las características obvias de adquisición por transferencia horizontal, pero está ausente en todas las otras especies de Estreptococos.
DETERMINANTES DE VIRULENCIA CODIFICADOS POR PAIS.
Las bacterias patógenas han adquirido PAIs que codifican un amplio rango de determinantes. Estos incluyen mecanismos para resistir las defensas del hospedador, factores de colonización, adquisición de nutrientes y toxinas. Algunas de estas actividades están mediadas por sistemas especializados de secreción.
Sistemas de secreción. Varias PAIs de patógenos Gram negativos, tales como Salmonella, E. coli, Bordetella y Pseudomonas, codifican estructuras especializadas en la exportación de proteínas, denominadas sistemas de secreción tipo III (Type Three Secretion Systems, TTSS) (10). El sistema TTSS, forma una inyectadora molecular, que inocula la proteína efectora directamente al citoplasma de la célula hospedadora a través de las membranas celulares bacterianas y eucariotas (10). También se ha demostrado en especies patógenas, la secreción de factores de virulencia mediada por otro sistema especializado asociado con virulencia y denominado sistema de secreción tipo IV (Type Fourth Secretion Systems, TFSS).
Evasión del sistema inmune: El rol de algunos de los factores de virulencia codificados por genes presentes en las PAIs es prevenir la destrucción de la bacteria por el sistema inmune del hospedador. La variación de los antígenos de superficie es un proceso común en la desviación de la respuesta y la rápida adquisición o pérdida del material genético por transferencia horizontal puede contribuir en este proceso. Un ejemplo fué, la emergencia de una epidemia de Cólera producida por la cepa V. cholerae O139, la cual emergió de una cepa preexistente O1 El Tor, que adquirió una nueva PAI, en la cual se había reemplazado parte del antígeno O en la región O1, y ahora codifica un nuevo antígeno O. La pérdida de la inmunidad cruzada contra la cepa O139, permitió el resurgimiento del Cólera en poblaciones resistentes a la infección con la cepa V. cholerae O1 (11). Las proteínas codificadas en PAIs también pueden estar implicadas en la resistencia al suero. Por ejemplo, el gen sac-4 , que codifica la resistencia a suero en Neisseria gonorrhoeae, está contenido en una PAI, presente en aislamientos de personas con infección gonococcica diseminada, mas que en aquellos casos de gonorrea no complicada (12).
Factores de Adherencia y Colonización: La habilidad de adherirse a la superficie del epitelio es esencial para la mayoría de los patógenos que interactúan con los hospederos humanos. La adherencia es usualmente mediada por moléculas que son secretadas a la superficie celular de la bacteria y que se unen a ligandos específicos en la superficie de las células del hospedador o en la matriz extracelular. Han sido identificados un gran número de factores de adherencia, divididos en dos grandes grupos; fimbrias (pili) y adhesinas. Las adhesinas son estructuras complejas en forma de barra las cuales se proyectan desde la superficie bacterial y median el contacto entre la bacteria y la célula del hospedador. En las E. coli uropatogénicas, los genes que codifican las proteínas del pilus, prf (P-related fimbriae), están localizados en una PAI, la cual también codifica otros factores de uropatogenicidad. La perdida de esta PAI ocurre naturalmente a una frecuencia relativamente alta, y resulta en la perdida de la virulencia (13). Un sistema de adhesión inusual esta codificado por el "Locus de Enterocyte Effacement" (LEE), una PAI de 35 kb, que contiene los genes eae, esp, tir y esc, presente en las E. coli enteropatogénicas y enterohemorrágicas, el cual codifica un sistema TTSS (14). Este sistema inyecta la proteína Tir (Translocated Intimin Receptor) en la membrana de la célula epitelial intestinal del hospedador, la cual actúa como receptor de otra proteína codificada en LEE, la intimina (eae) (15), resultando en una fuerte adherencia a las células epiteliales. Por otra parte, las cepas de E. coli que poseen PAIs y que pueden codificar las fimbrias S, que se unen a los receptores de ácido siálico presentes en las células endoteliales del sistema microvascular del cerebro, pueden causar sepsis o meningitis.
Alteración de la Célula Hospedadora: Muchos factores de virulencia codificados en las PAIs alteran las actividades de la célula hospedadora por la interferencia con vías de señalización eucariota. Esto produce varios efectos, como la inclusión de la bacteria por células no fagocíticas, la inhibición de la fagocitosis o la inducción de apoptosis celular. La mayoría de los patógenos que son capaces de inducir su incorporación por la célula hospedadora usan mecanismos semejantes. Por ejemplo, Salmonella y Shigella promueven su internalización por la inyección de proteínas en el citoplasma de la célula hospedadora vía un sistema TTSS codificado por PAIs. Esto activa en la célula hospedadora una cascada de señales, que conducen a un rearreglo del citoesqueleto permitiendo la invasión por la célula bacteriana (16).
Supervivencia Intracelular y Multiplicación. La supervivencia y replicación dentro de las células hospedadoras es un estado importante en la patogénesis. Posterior a la internalización, la bacteria está situada en un compartimiento rodeado de membrana, el fagosoma, y normalmente éste se fusiona con otros compartimentos que liberan toxinas. Los patógenos intracelulares deben utilizar diferentes mecanismos para escapar o prevenir el efecto de este proceso. Algunas cepas de Listeria spp. poseen un grupo genético parecido a una PAI, que codifica una toxina capaz de formar poros, la listeriolisina O. Esta enzima rompe la membrana fagosomal, liberando la bacteria al citoplasma, donde es inaccesible para las defensas de la célula y donde puede obtener nutrientes (17). Otros patógenos como, S. Typhimurium, permanecen en el fagosoma y convierten este compartimiento en el lugar para multiplicarse, mediante la prevención o resistencia a la acidificación o la liberación del contenido lisosomal (7). Se ha podido demostrar que varias proteínas codificadas en diferentes PAIs y secretadas por TTSS, pueden inducir la apoptosis de las células hospedadoras. La proteína SipB codificada en la PAI de Salmonella SPI-1, y la proteína IpaB (Invasion plasmid antigen B), codificada en el plásmido de virulencia de Shigella, inducen apoptosis en macrófagos (12). La liberación de la bacteria de los macrófagos lisados permite invadir las células epiteliales de la superficie basolateral (18).
Obtención de Nutrientes: La habilidad para competir por los nutrientes es esencial para la patogénesis. Uno de estos mecanismos es la adquisición de hierro. Muchas bacterias segregan componentes de bajo peso molecular, llamados sideróforos, que se unen a hierro. Estas bacterias tienen sistemas de captación de sideróforos unidos a hierro. Varios ejemplos son, la yersiniobactina, codificada en una PAI de Yersinia spp. (19), el transportador Sit1, codificado en la PAI SPI-1de S. Typhimurium (20), y el operón pit2 en la PPI-1, el cual codifica para los transportadores ABC requeridos en la toma de hierro y virulencia de Streptococcus pneumoniae (21).
Producción de Toxinas: Muchas bacterias patógenas portan plásmidos o bacteriófagos en los cuales están codificadas toxinas, por lo que no es sorprendente que las PAIs también porten genes que codifican para toxinas. El prototipo de las toxinas formadoras de poro codificada en una PAI son las á-hemolisinas de E. coli UPEC denominadas Hly. Estas toxinas son secretadas por un sistema de secreción tipo I y son capaces de insertarse en la membrana y lisar eritrocitos y otras células. El grupo de genes hly usualmente están localizados junto a los genes que codifican a la Fimbria-P y el locus que codifica el factor citotóxico necrotizante. Varias bacterias patógenas producen enzimas con actividades proteolíticas, muchas de las cuales juegan un rol en la patogenicidad, y los genes que codifican estas enzimas están usualmente en PAIs o en estructuras parecidas a PAIs. La PAI LIPI1 (Listeria pathogenicity island 1) de Listeria monocytogenes y L. ivanovii porta los genes que codifican para dos fosfolipasas (plcA y plcB) y el locus que codifica una metaloproteasa (mpl). Adicionalmente, se han encontrado que varias proteínas proteolíticas con actividad enterotóxica están codificadas en PAIS (22). Otra enterotoxina, la SheT1 de Shigella, la cual causa la acumulación de líquido en asa ileal de conejo, esta también codificada en la PAI SHI1 de S. flexnerii (12). Los genes que codifican las enterotoxinas de Clostridium difficile, TcdA y TcdB, las cuales tienen actividad glicosiltransferasa y modifican GTPasas, son parte del locus de patogenicidad de 19 kb, el cual muestra algunas características de PAI (12).
LOCALIZACIÓN DE LAS PAIs
La mayoría de las PAIs están localizadas en el cromosoma, sin embargo, también pueden ser parte de plásmidos y/o fagos. Las PAI de Shigella y Yersinia spp. están localizadas en plásmidos que codifican proteínas que median la internalización de las bacterias y otras respuestas en la célula hospedadora. El sistema TTSS codificado en el plásmido de virulencia de Shigella es esencial para la invasión de las células epiteliales del hospedador. El plásmido de virulencia de EHEC porta un grupo de genes hly, que codifican para las á-hemolisinas, similar a los determinantes hly localizados en una PAI de UPEC. (12).
EL ROL DE LAS PAIs EN LA EVOLUCIÓN
DE ALGUNOS PATÓGENOS BACTERIANOS
Escherichia coli: E. coli es una excelente ilustración del impacto de la transferencia horizontal de casetes de patogenicidad, tanto por plásmidos como por PAIs, en la evolución de la virulencia. La mayoría de las cepas de E. coli son no patógenas, sin embargo, un cierto número de cepas patógenas pueden causar un amplio rango de enfermedades, que van desde infecciones del tracto urinario, diarrea y síndrome urémico, hasta el shock séptico, meningitis y neumonía. En la mayoría de los casos, una cepa de E. coli en particular está asociada a una sola enfermedad. Se ha postulado que esto refleja la vía por la cual E. coli ha evolucionado y que diferentes cepas han emergido en paralelo de ancestros comensales a través de la adquisición de distintos "sets" de determinantes de virulencia (24). Un buen ejemplo del efecto de las PAIs en el fenotipo virulento de E. coli son las PAI-I y PAI-II de las cepas que causan infecciones de tracto urinario. Ambas PAIs codifican hemolisinas, mientras la PAI-II también contiene los genes para fimbria tipo Prf, necesaria para la adherencia a las células epiteliales (13, 24). Si estas PAIs se pierden del cromosoma, las cepas resultantes son incapaces de producir infecciones urinarias. Además se ha demostrado que el determinante de la cápsula de E. coli Uropatogénica cepa 536 (O6:k15:H31) está codificado en una PAI, designada PAI V536, la cual no se encuentra en el genoma de la E. coli K-12, una cepa no patógena (25).
Las cepas patogénicas de E. coli que causan infecciones intestinales también han adquirido PAIs. El fenotipo compartido por las células de EPEC y EHEC es su habilidad de adherirse intimamente a las células epiteliales e inducir la formación de lesiones de Adherencia y Borrado (Attachment/Effacement, AE), caracterizadas por la pérdida de las microvellosidades y la formación de pedestales ricos en actina, en los cuales se coloca la bacteria (26). El fenotipo AE es dependiente de la PAI LEE (Locus of Enterocyte Effacing), la cual porta los genes que codifican para un TTSS y varias proteínas secretoras. Aunque el LEE está presente en mas de una cepa patógena de E. coli, recientes análisis filogenéticos muestran que al menos dos linajes diferentes de E. coli, adquirieron el LEE en ocasiones independientes (23). Aunque la lesión AE es de importancia para la virulencia tanto para EPEC como para EHEC, las dos variedades causan enfermedades diferentes debido a la presencia de otro elemento trasmitido horizontalmente. EHEC ha adquirido un bacteriófago que codifica la toxina "Shigalike", que se piensa que causa el síndrome urémico, y una hemolisina codificada en un plásmido. Se ha propuesto que el clon de EHEC O157:H7 emergió de un progenitor EPEC parecido al clon O55:H7 que ya era capaz de producir la lesión AE, el cual fue lisogenizado con el bacteriófago que contenía los genes de la toxina "Shiga-like" (14). Cualquier cepa de E. coli tiene el potencial de convertirse en patógena, simplemente por la adquisición de nuevos determinantes de virulencia por transferencia horizontal, y se propone que esto ha pasado independientemente varias veces y en paralelo, resultando en una variedad de cepas con diferente potencial patogénico (figura 2).
Salmonella spp. Salmonella, uno de los patógenos mejor conocidos y mas estudiados, presenta sus determinantes de resistencia codificados en el cromosoma, usualmente en islas de patogenicidad, o en la región de multi-resistencia a drogas en la Isla genómica 1 (SGI1) (27). La especie S. enterica incluye un gran número de serovariedades con importancia clínica. S. Enteritidis y S. Typhimurium son causas importantes de gastroenteritis, mientras S. Typhi y S. Paratyphi son los agentes etiológicos de la fiebre tifoidea. A diferencia de E. coli, todas las Salmonellas son patógenas. La evolución de la virulencia en este patógeno, parece que ha seguido una progresión lineal. Un gran número de factores de virulencia transmitidos horizontalmente se ha identificado en Salmonella spp., incluyendo un plásmido de virulencia, al menos 5 PAIs principales y un gran número de islotes de patogenicidad, cada una portando un grupo de genes de virulencia (28). De acuerdo con la definición de PAI dada previamente, las "Salmonella Pathogenicity Islands" (SPIs) son PAIs excepcionales dentro de las bacterias Gram negativas, pues son específicas de serotipo y no específicas de cepas. Las PAI SPI-1 y SPI-2 se han encontrado en todas las cepas de S. Typhimurium probadas hasta la fecha (29). Por otra parte, las SPI no han sido detectadas en otras enterobacterias relacionadas, tales como, Yersinia, S. flexnerii y E. coli, por lo que se ha especulado que los fragmentos de DNA específicos de ambas SPI fueron transmitidos a Salmonella hace mas de 100 millones de años, cuando Salmonella y las otras enterobacterias se separaron del ancestro común (4). La adherencia y la invasión de las células epiteliales son la base del mecanismo de virulencia del género Salmonella, e involucra varios loci genéticos (30). Este incluye dos operones, lpf y fim, que codifican las adhesinas fimbriales, y el sistema TTSS codificado en la PAI SPI-1. Algunas cepas de Salmonella también causan enfermedades sistémicas, que involucran la replicación dentro de las células fagocíticas, su diseminación desde el epitelio intestinal al sistema retículo endotelial y su replicación en el hígado o bazo. En adición SPI-2 codifica un sistema TTSS, esencial para la enfermedad sistémica en ratón y para la replicación dentro del macrófago (29).
Helicobacter pylori: Hasta la fecha no se conoce el factor principal de virulencia de H. pylori, que causa la gastritis y la úlcera peptídica, pero se sugiere que una PAI juega un rol importante. Existen dos cepas predominantes de H. pylori, siendo las cepas tipo I las asociadas con la enfermedad más severa. Estas cepas poseen una PAI de 40 kb, que codifica el antígeno asociado a citotoxicidad (Cytotoxin associated antigen, CagA), el cual es traslocado dentro de las células epiteliales gástricas por un sistema TFSS también codificado en la PAI (31). Muchos de los genes localizados en la PAI Cag son requeridos para la respuesta celular del hospedador, la secreción de IL-8 por las células epiteliales gástricas, la fosforilación de tirosina en las proteínas del hospedador y, el rearreglo del citoesqueleto durante la formación del pedestal en la superficie de la célula hospedadora. Covacci y colaboradores proponen un modelo de evolución de los determinantes de virulencia en H. pylori (32), según el cual, posterior a la adquisición inicial de la PAI, la inserción de un elemento IS605 y un rearreglo ha generado las cuasiespecies de H. pylori que se observan en los aislamientos clínicos. La adquisición de la PAI generó la bacteria más virulenta que representa la subpoblación dominante, luego la inserción de IS605 conduce a la transposición y deleción de segmentos, generando un número de bacterias con fenotipos intermedios que todavía retienen virulencia. En casos raros, una deleción amplia de cagI, cagII o ambos conduciría a retornar al fenotipo no virulento.
Vibrio cholerae: Para cepas V. cholerae O1 y O139 ha sido descrita una interacción entre las PAIs y el bacteriófago convertidor de toxina colérica (CTX). Estas cepas portan una PAI de 45 kb, la cual contiene el grupo de genes tcp-acf, el gen toxT, cuyo producto está involucrado en la regulación de la toxina colérica (ctx) y también un gen que codifica para una integrasa. Interesantemente, el fago CTX usa como su receptor, una adhesina Tcp, codificada en la PAI, proveyendo una evidencia para la evolución conjunta de la PAI específica de V. cholerae y los genes asociados con el fago ctxAB (33).
Los brotes de cólera pueden transformarse en epidemias, mas aún, pueden llegar a ser pandemias de ocurrencia mundial. Análisis recientes, utilizando las técnicas de microarreglos, han demostrado que las cepas de la séptima pandemia, que se inició en 1961, tienen varios genes que no estaban presentes en las cepas aisladas con anterioridad. Se identificaron dos regiones cromosómicas que se denominaron "vibrio seventh-pandemic island". VSP-I y VSP-II muestran varias características compatibles con PAI, pero el rol en la virulencia no ha sido reportado hasta la fecha (34).
Yersinia spp.: La región HPI (High Pathogenicity Island) es responsable de la letalidad en el ratón causada por Yersinia spp. Porta el locus de pigmentación (pgm), los genes que codifican las proteínas de almacenamiento de hemina (hms) y los factores de toma de hierro (fyuA, irp2). En Y. pestis la región HPI está completa, pero en las otras especies de Yersinia solo se ha encontrado una parte. Las cepas patógenas y no patógenas de Y. enterocolítica pueden diferir en la presencia de los genes para la adquisición de hierro, pero no tienen el locus hms, especifico de Y. pestis, el agente causal de la peste (4). La comparación entre las secuencias de las HPI de las especies de Yersinia ha mostrado la posibilidad de dos líneas evolutivas de HPI, Y, enterocolitica (Yen HPI) y Y.pestis/Y.pseudotuberculosis (Yps HPI). Se piensa que la adquisición de hierro mediada por la yersiniabactina es la función principal codificada en la HPI, y los genes involucrados en la biosíntesis, transporte y regulación de esta proteína están agrupados en el "core" de HPI. Los genes del "core" están altamente conservados en ambas líneas evolutivas y se caracterizan por un contenido de G+C mayor que la media del contenido cromosomal de Yersinia. En contraste, el segundo componente de HPI es una región rica en A+T, diferente en Yen HPI y Yps HPI. Yersinia puede haber adquirido la HPI de un progenitor común con alto contenido de G+C y luego evolucionar divergentemente en dos líneas evolutivas (35). En algunas bacterias, la adquisición de una PAI puede orientar la evolución posterior, este es probablemente el caso de Y. pestis, especie que emergió recientemente (1500 – 20.000 años atrás) a partir de Y. pseudotuberculosis. En contraste con su progenitor, que utiliza la ruta oral para contaminar a su hospedador humano y animal, Y. pestis es transmitida por la picadura de una pulga y la septicemia que ocurre en el hospedador en el estado pre-morten de la peste, es un prerrequisito para la transmisión por la pulga. Por la promoción de la diseminación sistémica y la transmisión eficiente de la bacteria "in vivo", la PAI HPI presumiblemente sirvió como un factor clave de la emergencia de este microorganismo peligroso. En otras palabras, Y. pestis no hubiera evolucionado de Y pseudotuberculosis si el genoma de esta última no hubiera portado con anterioridad HPI (7).
Islas patogénicas en bacterias Gram positivas.
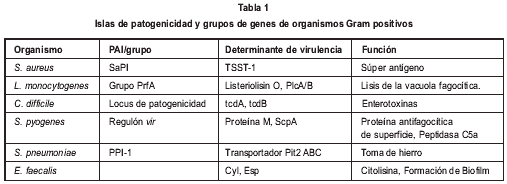
Aunque las PAIs de las bacterias Gram negativas han sido las estudiadas extensamente, también se han encontrado PAIs en especies Gram positivas (Tabla 1, (10)).
Las PAIs reportadas en las bacterias Gram positivas usualmente no encajan en la definición clásica de PAIs, ya que pueden no estar bordeadas por sitios especiales de unión, o con marcadores de inserción tales como; repeticiones directas o genes de ARNt, lo que hace más difícil su detección. Una excepción rara es la PAI de 15 kb de Staphylococcus aureus, la cual posee muchas de las características de las PAIs de Gram negativos y codifica para un sistema TTSS-1 (8). Otros patógenos Gram positivos poseen grupos de determinantes de virulencia que muestran alguna similitud con las PAIs, y han sido denominados grupos de genes de patogenicidad. Por ejemplo, las especies patógenas de Listeria poseen una región de 10 kb que codifica para la listerolisina O y sus proteínas reguladoras, que son requeridas para escapar del fagosoma dentro del macrófago. Esta región pareciera haber sido adquirida por la transducción de un bacteriófago. Listeria monocytogenes, responsable de listeriosis transmitida por alimentos, tiene una PAI de 9,5 kb, la cual porta cinco genes de virulencia. Todas las especies patógenas; L monocytogenes (patógena para humanos) y L. ivanovii (patógena para animales) poseen esta PAI, pero las cepas de las especies no patógenas (L. innocua, L. welshimeri y L. grayii) no tienen este segmento específico de ADN (4).
Algunas cepas de Clostridium difficile portan un grupo de genes en un segmento de 19 Kb que codifican dos toxinas de alto peso molecular, TcdA (una enterotoxina) y TcdB (una citotoxina), responsables de la colitis pseudomembranosa. En el cromosoma de S. pyogenes, pero no en los genomas de cualquier otra especie del género Streptococcus, se encuentra un PAI de 6 kb, también conocida cono regulón vir, en el cual se codifica el regulador principal (mga), la proteína M, proteínas relacionadas (emm, mrp, enn) y una peptidasa de C5b (scpA). En Streptococcus pneumoniae existe una región en el cromosoma con reminiscencias de PAI. Este locus se ha denominado "Pneumococcal pathogenicity island" (PPI-1), tiene 27 kb y está ausente en los otros Estreptococos. El factor de virulencia identificado en esta PAI es un sistema de adquisición de hierro codificado por pit2ABCD (21). E. faecalis tiene dos factores de virulencia conocidos, una citolisina (Cyl) y una proteína de superficie Esp. La presencia de Esp está relacionada con los aislados clínicos más virulentos. Una PAI de 154 kb, contiene el operon cyl, esp y varios genes con funciones desconocidas (36).
REGULACIÓN DE LOS GENES DE PAIS.
Una vez que son adquiridos los genes por transferencia horizontal, para que confieran alguna ventaja, su expresión debe ser regulada apropiadamente. La importancia de la regulación se puede ilustrar por el grupo genético que codifica la listerolisina O. Esta proteína media el escape de la bacteria de la vacuola del fagosoma del macrófago al citoplasma, y es un factor indispensable para la virulencia de Listeria. Sorprendentemente, el grupo genético que codifica la listerolisina O, también se encuentra en una Listeria no patógena, Listeria seeligeri, pero los genes no son expresados. Cuando se introduce el activador transcripcional de L. monocitogenes a L. seeligeri la expresión intracelular del gen de listerolisina O se induce y le confiere a esta especie la habilidad de escapar del fagosoma (37).
Los genes cuyos productos tienen funciones reguladoras pueden estar localizados dentro de las PAIs. En S. Typhimurium la SPI-2 codifica un sistema de dos componentes, SsrAB. La proteína SsrB, es el activador transcripcional, no sólo para los genes dentro de SPI-2, sino también, para cerca de otros 10 loci dentro del genoma de Salmonella, todos los cuales parecen haber sido adquiridos horizontalmente (38). Los genes en una PAI también pueden estar integrados a circuitos regulatorios existentes. Por ejemplo, la expresión de SsrA-SsrB, está regulada por el sistema OmpR/EnvZ, el cual responde a estrés osmótico, pH y temperatura. La colaboración entre el sistema OmpR-EnvZ y el sistema SsrAB conducen a una detección eficiente del ambiente intracelular y su respuesta. La proteina OmpR regula al sistema SsrASsrB y este a su vez, activa el sistema de secreción TTSS en SPI-2 (39).
MECANISMOS DE ADQUISICIÓN DE PAIs
Las PAIs usualmente contienen señales claves que indican los posibles mecanismos de adquisición. Muchas PAIs están flanqueadas por secuencias repetidas directas, usualmente entre 16 a 20 pb, las cuales fueron probablemente generadas durante la inserción de bacteriófagos, plásmidos o transposones en el genoma. También están presentes otras características de elementos genéticos móviles, incluyendo orígenes de replicación de plásmidos y transposones o genes de fagos, tales como recombinasas o integrasas. Todo lo anterior hace aparecer a las PAIs como "puntos calientes" para la recombinación e inserción de elementos genéticos móviles, algunos de los cuales podrían haber sido adquiridos posteriormente a la integración inicial de la PAI (11).
Algunas PAIs están asociadas con bacteriófagos completos, tales como las PAIs que portan genes de toxina en Corynebacterium diphtheriae, S. aureus, Pseudomonas aeruginosa, V. cholerae y E. coli (40). Algunos de estos fagos permanecen capaces de escindirse del genoma, tal es el caso de VPI, el cual puede ser movilizado entre cepas de V. cholerae. Sin embargo, los fagos asociados con otras PAIs, están reducidos a remanentes defectivos. Por ejemplo, la PAI de S. aureus, SaPI, contiene un fago defectivo, el cual solo puede ser movilizado por la infección de la bacteria con un virus "ayudador" el cual le provee en trans las funciones perdidas (8).
La integración de los fagos o transposones ocurre en sitios específicos en el genoma, por lo tanto las PAIs no se insertan al azar sino en sitos específicos. Las PAIs de bacterias Gram negativas están frecuentemente localizadas hacia la región 5´ de los genes que codifican los ARN de transferencia, posiblemente porque los loci de ARNt se conocen como sitios blancos de integración de varios fagos y plásmidos. Interesantemente, el locus de ARNt selC es el sitio de inserción de por lo menos 4 PAIs diferentes, y probablemente representa un "punto caliente" para la integración de algún elemento de transposición.
Ciertas PAIs no tienen la capacidad de moverse de una cepa bacteriana a otra, pero sin embargo, son capaces de movilizarse de un sitio a otro en el genoma de la bacteria patógena. Por ejemplo, la HPI de Y. pseudotuberculosis y Y. pestis, porta genes que codifican una integrasa, flanqueados por unos repetidos directos de 17 pb. Estas HPIs en Yersinia, usualmente están localizadas cerca del gen de ARNt específico de asparagina, asnT, pero también ha sido localizada cerca de otros genes de ARNt específicos para asparagina, asnW y asnV, indicando la movilización del elemento de un sitio a otro (11).
Algunas PAIs pueden ser el resultado de más de un evento de transferencia genética. Parece que la SPI-2 de S. Typhimurium está compuesta por lo menos de dos elementos genéticos que fueron adquiridos independientemente. Una porción de aproximadamente 15,3 kb, contiene los genes importantes para la virulencia y otra porción mas pequeña de 14.5 kb, porta 5 genes ttr (involucrados en la reducción anaeróbica de tetrationato) y 7 marcos abiertos de lectura de función desconocida. Los análisis por mutación indican que los genes en la porción de 14,5 kb no contribuyen significativamente en la infección sistémica en el ratón y el análisis de frecuencia de uso de codones revela que las dos porciones pertenecen a diferentes clases de genes (41).
¿DE DONDE VIENEN LAS PAIs?
No hay una identificación clara de la fuente original de las islas de patogenicidad y en muchos casos, el donante puede ser un organismo no cultivable o extinto. El origen de algunos factores de virulencia ha podido ser rastreado a genes relacionados que se encuentran en cepas no patogénicas. Por ejemplo, los sistemas TTSS y TFSS que se encuentran en patógenos Gram negativos son homólogos a la maquinaria para exportar y ensamblar los flagelos o los pili. También se ha sugerido, que algunos genes de eucariotas han sido incorporados en las PAIs bacterianas. Un ejemplo es YopH, una tirosina fosfatasa secretada por Yersinia, la cual es más similar a la tirosina fosfatasa de Eucariotas que a las de otras bacterias (42).
ESTADOS EVOLUTIVOS DE LAS PAIs
Cuando se describe la evolución en términos Darwinianos se habla de variabilidad genética, formación de fenotipos y selección natural. El término macroevolución se utiliza para describir el proceso de aparición de nuevas especies o tipos patogénicos dentro de un período de tiempo largo, en contraste, el término microevolución, se utiliza, cuando la generación de nuevas variantes o tipos patogénicos de alguna especie ocurre en solo días o semanas. Las PAIs contribuyen al desarrollo de nuevas variantes patogénicas en términos de macroevolución. Desde el punto de vista evolutivo, la adquisición e integración de un nuevo segmento de ADN, a través de fagos, representa la forma mas joven de PAI, como por ejemplo, VPI de V. cholerae y S. aureus. Si la PAI contribuye con el desempeño y sobrevivencia de la bacteria, con su potencial patogénico, los genes encargados de su movilidad, transferencia, deleción o escisión pueden ser inactivados o eliminados. Las PAIs HPIs de Yersinia, las de UPEC y la Cag de H. pylori pueden representar un estado intermedio, una especie de PAI adolescente. Con el transcurso del tiempo, pueden ser unos miles de años, los tamaños de las PAIs exitosas tienden a reducirse para incluir solo los genes importantes, requeridos para las funciones claves y las PAIs cambian a formas mas condensadas, por ejemplo LEE de EPEC. Así la SPI-1 de Salmonella, la cual fue introducida al ancestro cerca de 100 millones de años atrás, es parte del "core" del genoma de la bacteria. El progreso evolutivo de la PAI ha terminado cuando el elemento genético se ha adaptado al "core" del cromosoma del hospedador de tal manera que no puede ser detectado como una PAI (3).
UTILIDADES FUTURAS DE LAS PAIs
El rango de la terapia antimicrobiana disponible para el tratamiento de las infecciones bacterianas se ha estado reduciendo en los últimos años por el desarrollo y diseminación de la multi-resistencia. La identificación de "familias" conservadas de genes asociados a la virulencia, tales como, los sistemas TTSS y TFSS, abren un campo nuevo para el desarrollo de drogas que puedan inhibir este mecanismo. Estas drogas tendrían ventajas sobre las que se utilizan actualmente, pues serían activas solo sobre los patógenos, sin afectar la flora normal. Adicionalmente, la mayoría de la resistencia a los antibióticos, es debida a los productos de genes de resistencia ya existentes en las bacterias, que utilizan para defenderse de los microorganismos productores de antibióticos, como el Streptomyces, sin embargo, es poco probable que ocurran naturalmente genes que codifican para la resistencia contra drogas "anti-virulencia". Por otra parte, se conoce que es difícil producir cepas vacunales, obtenidas tradicionalmente por repetidos pases en el laboratorio, con la suficiente atenuación, pero capaces de persistir en el cuerpo lo suficiente para generar una buena respuesta inmune. Las PAIs serían buenas candidatas para ser eliminadas y producir una cepa atenuada pero potencialmente inmunogénica. Por ejemplo, las PAIs que portan los genes de producción de toxinas pueden ser removidas, sin afectar la habilidad de las cepas para colonizar superficies epiteliales.
Las PAIs son, por definición, únicas para patógenos específicos. Dado que los métodos de análisis de ADN son rápidos, la posibilidad del análisis simultáneo de varias muestras para la búsqueda de secuencias de PAIs, puede ser una herramienta diagnóstica para identificar organismos patógenos (43).
CONCLUSION:
El conocimiento cada vez mayor de la diversidad genética y de la organización del genoma bacteriano ha permitido demostrar que las PAIs representan un subtipo de otro elemento genético más general llamado Islas Genómicas (GEI), las cuales se encuentran ampliamente distribuidas entre microbios patógenos y no-patógenos. En las GEIs se encuentran determinantes que pueden ser beneficiosos para la bacteria bajo ciertas condiciones en las cuales se incremente la capacidad adaptativa a condiciones adversas. Consecuentemente esto puede contribuir a la sobrevivencia de la bacteria y la transmisión horizontal de estos determinantes (44).
Las PAIs son regiones de ADN foráneo adquiridos en forma horizontal, que llevan genes cuyos productos son necesarios para la patogenicidad de una bacteria.
La cantidad de ADN foráneo puede ser nada en genomas de parásitos intracelulares permanentes como Mycoplasma y Ritckettsia, a tanto como el 17% del genoma en cepas de E.coli y Salmonella. Una comparación reciente de la secuencia de los genomas de la E. coli K12, no patogénica, y EHEC O157:H7, enterohemorrágica, reveló un número asombroso de 1387 nuevos genes en la cepa patógena, incluyendo 9 PAIs adquiridas horizontalmente, cada una de mas de 15 kb y que codifican factores de virulencia putativos (45). El evento de transferencia de PAIs se observa raramente en el laboratorio y por ahora el proceso subyacente no esta del todo bien comprendido.
Esta revisión describe algunas características del rol de las PAIs en la patogénesis de importantes patógenos humanos, es importante señalar que el número de PAIs identificadas está en constante crecimiento. La disponibilidad de las secuencias genómicas provee una plataforma extremadamente útil para el trabajo futuro, al combinar el entendimiento de los conceptos básicos de la evolución de la virulencia bacteriana y el rol crucial de la PAIs en este proceso. De esta forma, los microbiólogos estarán en la capacidad de identificar de manera mas eficiente, nuevos patógenos emergentes o cepas patógenas.
REFERENCIAS
1. Falkow Stanley. What is a pathogen?. ASM News. 1997; 63: 359-365. [ Links ]
2. Finlay B, Falkow S. Common themes in microbial pathogenicity. Microbiol Rev. 1989; 53(2):210-230. [ Links ]
3. Groisman E, Ochman H. Pathogenicity islands: bacterial evolution in quantum leaps. Cell. 1996; 87:791-794. [ Links ]
4. Lan R, Reeves P. Gene transfer is a major factor in bacterial evolution. Mol Biol Evol. 1996; 13:47-55. [ Links ]
5. Goebel W, Oropeza-Wekerle R, Muller E, Kern P, Meyermam R. Synthesis, inactivation and localization of extracellular and intracellular Escherichia coli hemolysin. J Bacteriol. 1989; 171 (5): 2783-2788. [ Links ]
6. Hacker J, Blum-Oehler G, Mühldorfer I, Tschäpe H. Pathogenicity islands of virulent bacteria: structure, function and impact on microbial evolution. Mol Microbioly. 1997; 23:1089-1097. [ Links ]
7. Beuzon C, Méresse S, Unsworth K. Salmonella maintains the integrity of its intracellular vacuole through the action of SifA. EMBO J. 2000; 19:3235-3249. [ Links ]
8. Hacker J, Carniel E. Ecological fitness, genomic islands and bacterial pathogenicity. EMBO reports. 2001; 21:376-381. [ Links ]
9. Lindsay J, Ruzin A, Ross H. The gene for toxic shock toxin is carried by a family of mobile pathogenicity islands in Staphylococcus aureus. Mol Microbiol. 1998; 29:527-543. [ Links ]
10. Hueck C. Type III protein secretion systems in bacterial pathogens of animals and plants. Microbiol Mol Biol Rev. 1998; 62:379-433. [ Links ]
11. Bik E, Bunschoten A, Gouw R. Genesis of the novel epidemic Vibrio cholerae O139 strain: evidence for horizontal transfer of genes involved in polysaccharide synthesis. EMBO J. 1995; 14:209-216 [ Links ]
12. Hacker J, Kaper J. Pathogenicity islands and the evolution of microbes. Ann Rev Microbiol. 2000; 54:641-679. [ Links ]
13. Blum G, Ott M, Lischewski A. Excision of large DNA regions termed pathogenicity islands from tRNA-specific loci in the chromosomal of an Escherichia coli wild-type pathogen. Infect Immun. 1994; 62:606-614. [ Links ]
14. McDaniel T, Jarvis K, Donnenberg M. A genetic locus of enterocyte effacement conserved among diverse enterobacterial pathogens. Proc Natl Acad Sci USA. 1995; 92:1664-1668. [ Links ]
15. Kenny B, DeVinney R, Stein M. Enteropathogenic E. coli (EPEC) transfers its receptor for intimate adherence into mammalian cells. Cell. 1997; 91:511-520. [ Links ]
16. Galán J E, Zhou D. Striking a balance: modulation of the actin cytoskeleton by Salmonella. Proc Natl Acad Sci USA. 2000; 11:555-566. [ Links ]
17. Goebel W, Kuhn M. Bacterial replication in the host cell cytosol. Curr Opin Microbiol. 2000; 3:49-53. [ Links ]
18. Guichon A, Hersh D, Smith M, Zychlinsky A. Structure-Function analysis of the Shigella Virulence Factor IpaB. J Bacteriology. 2001; 183: 1269-1276. [ Links ]
19. Baerden S W, Fetherston J, Perry R. Genetic organization of the yersiniabactin biosynthetic region and construction of avirulent mutants in Yersinia pestis. Infec Immun. 1997; 65:1659-1668. [ Links ]
20. Zhou D, Hartd W, Galán J. Salmonella typhimurium encodes a putative iron transport system within the centisome 63 pathogenicity island. Infect Immu. 1999; 67:1974-1981. [ Links ]
21. Brown J, Gilliland S, Holden D. A Streptococcus pneumoniae pathogenicity island encoding an ABC transporter involved in iron uptake and virulence. Mol Microbiol. 2001; 40 (3): 572-585. [ Links ]
22. Perna N, Plunkett G, Bruland V. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature 2001; 409:529-533. [ Links ]
23. Reid S, Herbelin C, Bumbaugh A. Parallel evolution of virulence in pathogenic Escherichia coli. Nature. 2000; 406:64-67. [ Links ]
24. Blum G, Falbo V, Caprioli A. Gene clusters encoding the cytotoxic necrotizing factor type 1, Prs-fimbriae and alpha-hemolysin from the pathogenicity island II of the uropathogenic Escherichia coli strain J96. FEMS Microbiol Lett. 1995; 126:189-195. [ Links ]
25. Schneider G, Dobrindt U, Bruggemann H, Nagy G, Janke B, Blum G, "et al". The pathogenicity island-associated K15 capsule determinant exhibits a novel genetic structure and correlates with virulence in uropathogenic Escherichia coli strain 536. Infect Immun. 2004; 72(10):5993-6001. [ Links ]
26. Giron J, Ho A, Schoolnik G. An inducible bundle-forming pilus of enteropathogenic Escherichia coli. Science. 1991; 254:710-3. [ Links ]
27. Fluit A. Towards more virulent and antibiotic-resistant Salmonella?. FEMS Inmunol Med Microbiol. 2005; 43(1): 1-11. [ Links ]
28. Baumler A. The record of horizontal gene transfer in Salmonella. Trends Microbiol. 1997; 5:318-322. [ Links ]
29. Groisman E, Ochman H. How Salmonella became a pathogen. Trends Microbiol. 1997; 5:343-349. [ Links ]
30. Blanc A, Groisman E. The Salmonella selC locus contains a pathogenicity island mediating intramacrophage survival. EMBO J. 1997; 16:5376-5385. [ Links ]
31. Censini S, Lange C, Xiang Z. Cag, pathogenicity island of Helicobacter pylori, encodes type I-specific and diseaseassociated virulence factors. Proc Natl Acad Sci USA. 1996; 93:14648-14653. [ Links ]
32. Covacci A, Telford J, Del Giudice G. Helicobacter pylori virulence and genetic geography. Science. 1999; 284: 1328-1333. [ Links ]
33.WaldorM K, Makalanos J. Lysogenic conversion and filamentous phage encoding cholera toxin. Science. 1999. 272:1910-1914. [ Links ]
34. Dziejman M, Balon E, Boyd D, Fraser C, Heidelberg J, Mekalanos J. Comparative genomic analysis of Vibrio cholerae: genes that correlate with Cholera endemic and pandemic disease. Proc Natl Acad Sci Usa. 2002; 99:1556-1561. [ Links ]
35. Rakin A, Noelting C, Schubert S, Heesemann J. Common and specific characteristics of the high-pathogenicity island of Yersinia enterocolitica. Infect Immun. 1999; 67: 5265-5274. [ Links ]
36. Shankar N, Lockatell C, Baghdayan A, Drachenberg C, Glimore M, Johnson D. Role of Enterococcus faecalis surface protein Esp in the pathogenesis of ascending urinary tract infection. Infec Immun. 2001; 69:4366-4372. [ Links ]
37. Karunasagar I, Lampidis R, Goebel W. Complementation of Listeria seeligeri with the plcA-prfA genes from L. monocytogenes activates transcription of seeligerolysin and leads to bacterial escape from the phagosome in infected mammalian cells. FEMS Microbiol Lett. 1997; 146:303-310. [ Links ]
38.Worley M, Ching K, Heffron F. Salmonella SsrB activates a global regulon of horizontally acquired genes. Mol Microbiol. 2000; 36:749-761. [ Links ]
39. Lee A, Detweiler C, Falkow S. OmpR regulates the twocomponent system ssrA-ssrB in Salmonella pathogenicity island 2. J Bacteriol. 2000; 182:771-781. [ Links ]
40. Miao E, Miller S. Bacteriophages in the evolution of pathogen- host interactions. Proc Natl Acad Sci USA. 1999; 96: 9452-9454. [ Links ]
41. Hensel M, Matsumoto H, Ohnishi M. Molecular and functional analysis indicates a mosaic structure of Salmonella pathogenicity island 2. Mol Microbiol. 1999; 31:489-498. [ Links ]
42. Guan K, Dixon J. Protein tyrosine phosphatase activity of an essential virulence determinat in Yersinia. Science 1990; 249:553-556. [ Links ]
43. Bekal S, Brousseau R, Masson L, Prefontaine G, Fairbrother J, Harel J. Rapid identificaction of Escherichia coli pathotypes by virulence gene detection with microarrays. J Clin Microb 2003; 41:2113-2125. [ Links ]
44. Hacker J, Hochhut B, Middendorf B, Schneider G, Buchrieser C, Gottschalk G, Dobrindt U. Pathogenomics of mobile genetic elements of toxigenic bacteria. Int J Med Microbiol. 2004; 293(7-8):453-461. [ Links ]
45. McDaniel T, Kaper J. A cloned pathogenicity island from enteropathogenic Escherichia coli confers the attaching and effacing phenotype on E. coli K-12. Mol Microbiol. 1997; 23:399-407. [ Links ]












 uBio
uBio