Revista del Instituto Nacional de Higiene Rafael Rangel
versión impresa ISSN 0798-0477
INHRR v.40 n.2 Caracas dic. 2009
Colera y Vibrio cholerae
Sandra Fernandez F 1*, Guillermina Alonso 2
1. Seccion de Diagnosticos Especiales. Departamento de Bacteriologia. Instituto Nacional de Higiene "Rafael Rangel". Ciudad Universitaria de Caracas, apartado postal 60412, oficina del este, Caracas, Venezuela. Telefono: 58-212-2666416, Fax: 58-212-6934551. Correo electronico: sfernandez@inhrr.gov.ve
2. Laboratorio de Biologia de Plasmidos, Instituto de Biologia Experimental, Universidad Central de Venezuela. Calle Suapure, Colinas de Be llo Monte, Apartado 47114, Caracas 1041A, Venezuela. Telefonos: 58-212-7510766, Fax:58-212-7535897. Correo electronico: guillermina.alonso@ciens.ucv.ve
* Autor para correspondencia.
RESUMEN
El Colera es una diarrea infecciosa aguda producida por Vibrio cholerae. La transmision se produce predominantemente a traves de agua o alimentos contaminados. La administracion de antibioticos puede disminuir la severidad de los sintomas. A partir de 1977, se han caracterizado cepas de V.cholerae O1 con resistencia multiple a los antibioticos. Los determinantes de resistencia han sido reportados principalmente asociados a plasmidos e integrones. Historicamente, solo las cepas del serogrupo O1 habian sido asociadas con epidemias de Colera. En 1992, un nuevo serogrupo, O139, emergio como la cepa causante de un gran brote de Colera. La diarrea masiva es causada por la toxina colerica (CT). El operon que codifica CT forma parte del genoma de un bacteriofago lisogenizado en la bacteria el cual permite interconversion de cepas. Se ha demostrado que el "quorum-sensing" regula negativamente la expresion de los genes de viru len cia en V. cholerae. El genoma de V. cholerae El Tor consiste en dos cromosomas circulares y con una pronunciada asimetria en la distribucion de los genes, en el cual se han detectado varias islas patogenicas (PAIs). Se ha reportado la existencia de V.cholerae "viable no cultivable" en ambientes acuaticos. Este fenomeno representa una nueva perspectiva en la vision de la "sobrevivencia" de V.cholerae en el ambiente e incorpora nuevas implicaciones epidemiologicas. El Colera ha sido catalogado como una "enfermedad emergente y reemergente", que amenaza a los paises en desarrollo. Los resultados de distintos grupos de investigadores han establecido que la transferencia horizontal de genes tiene influencia en la patogenicidad y ha sido importante en la evolucion de V. cholerae. Todavia permanecen sin descifrar algunos misterios en el origen de las cepas patogenas, pero, con las nuevas tecnologias seguro se develaran datos significativos que puedan dar luces sobre el origen de la patogenicidad de este organismo, que fue en un principio una bacteria inocua de estuario.
Palabras clave: Colera, Vibrio cholerae, Colera/metabo - lismo, Colera Toxina/ fisiologia, Colera/Quorum-sensing, Colera/epidemiologia/ epidemiologia molecular, Vibrio cho lerae/patogenicidad/fisiologia, Factores de Virulencia.
Cholera and Vibrio cholerae
ABSTRACT
The Cholera is an acute infectious diarrhea produced by Vibrio cholerae. Mainly the transmission takes place through contaminated water or foods. Antibiotic administration during the acute phase of the disease can reduce the severity of the symptoms. Since 1977, strains of V. cho lerae O1 with multiple antibiotic resistances have been characterized. The resistance determinants have been reported mainly associated to plasmids and integrones. Historically, the serogrupo O1 had been associated with epidemics of Cholera. In 1992, a new serogrupo, designated O139, emerged as the strain of cause from a big Cholera outbreak. The massive diarrhea is caused by the choleric toxin (CT). Operon ctxAB, that codifies the CT, is carried in lisogenic filamentous bacteriophage in the bacterium. "Quorum-sensing" negatively regulates the expression of the virulence genes in V.cholerae. The genome of V.cholerae El Tor consists of two circular chromosomes with a pronounced asymmetry in the distribution of the genes. Several patogenics islands (PAIs) have been detected in the genome of V.cholerae. In aquatic environments the existence of "viable but non cultivable" V.cholerae, has been reported. This phenomenon represents a new perspective in the role in survival in the natural environment with new epidemiological implications. The Cho lera has been catalogued as an "emergent, re-emergent disease" that threatens developing countries. The results of different investigators groups have established that horizontal transference of genes had influence in the pathogenicity and has been important in the evolution of V.cholerae. Several mysteries in the origin of the pathogenics clones still remain, but, the new technologies will reveal significant data about the origin of this pathogen, former an estuary innocuous bacterium.
Keywords: Cholera, Vibrio cholerae, Cholera/metabolism, Cholera Toxin/pharmacology/*physiology, Cholera/Quorum-sensing, Cholera/epidemiology/molecular epidemiology, Vibrio cholerae/pathogenicity/physiology, Virulence Factors.
Recibido: 25 de junio de 2008 / Aprobado: 21 de septiembre de 2008
El Colera es una enfermedad infecciosa aguda. La etiologia bacteriana del Colera fue confirmada por Robert Koch en 1833, cuando aislo el "bacillus cholera" del agua durante un brote de Colera en Egipto (1). Durante el siglo XIX, se propagaron verdaderas oleadas de epidemias de Colera desde el sur de Asia a muchas partes del mundo (2). En 1817 se inicia la primera pandemia de Colera fuera de Asia. Desde entonces, se han producido 7 pandemias, la septima comenzo en Indonesia en 1961, desde donde se propago rapidamente al este de Asia, en 1970 llego a Africa, y en 1991 a America (3). El Colera fue la primera enfermedad para la cual se establecio un sistema de vigilancia y reporte a gran escala.
TRANSMISIÓN Y CARACTERISTICAS CLINICAS DEL CÓLERA
La transmision del Colera se produce predominantemente a traves de agua o alimentos contaminados con materia fecal. La transmision de persona a persona es extremadamente rara, probablemente debido a que el inoculo necesario para causar la enfermedad es alto (4). El cuadro clasico de Colera se caracteriza por desarrollar bruscamente una intensa diarrea acuosa, habitualmente indolora y con vomitos. Son heces muy liquidas, con presencia de mucosidad pero sin sangre, adquiriendo un aspecto caracteristico que comunmente se les denomina "heces de agua de arroz", lo cual produce una disminucion del volumen sanguineo circulante, acidosis metabolica, agotamiento del potasio y, en ultimo termino, puede provocar colapso vascular y muerte (5). No todas las personas que se infectan desarrollan el cuadro clasico de Colera, las infecciones pueden ser asintomaticas hasta en un 75% de los pacientes. El tratamiento principal para el Colera es la rehidratacion oral o intravenosa (4). Cuando ocurren brotes epi demicos, la administracion de antibioticos durante la fase aguda de la enfermedad puede disminuir la severidad de los sintomas, asi como la cantidad y tiempo de excrecion del microorganismo (6). La tetraciclina fue el primer antibiotico utilizado en el tratamiento, pero tambien se han suministrado furazolidona, eritromicina, cotrimoxazol, cloranfenicol o norfloxacina (7).
RESISTENCIA A LOS ANTIBIÓTICOS EN V. CHOlERAE
Por mucho tiempo no se reporto resistencia clinicamente significativa en Colera, pero en 1977, se caracterizaron cepas de V. cholerae O1 El Tor con resistencia multiple a los antibioticos (8), y en algunos casos se demostro que estaba mediada por plasmidos (9, 10). Existen evidencias que sugieren que la resistencia a los antibioticos de primera linea fue uno de los factores que contribuyo en la alta tasa de mortalidad durante la epidemia de Colera en zaire, en 1994, asi como, el incremento de la tasa de mortalidad durante la epidemia de Guinea-Bissau durante 1996-1997 (11, 12). Los patrones de resistencia varian dependiendo de la region, de los patrones de uso de los antibioticos y de la fecha del analisis. Se han reportado cepas de V. cholerae resistente a antibioticos en Argentina, Ecuador, Mexico, Gua temala y Brasil (13-15). En Venezuela, los aislamientos de V. cholerae desde 1991 hasta 1993 eran sensibles a todos los antibioticos ensayados. A partir del ano 1997, se empezaron a registrar las primeras cepas con resistencia a algun antibiotico. En el brote 1998-1999 se detectaron las primeras cepas con multirresistencia (16).
MECANISMOS GENÉTICOS qUE MEDIAN LA RESISTENCIA A ANTIBIÓTICOS
Entre los principales vehiculos para la diseminacion y la adquisicion de nuevos genes que codifican proteinas que confieren resistencia destacan, los plasmidos, los integrones y los transposones conjugativos. Los plasmidos son elementos de ADN extracromosomicos, que codifican diversos rasgos fenotipicos y que pueden ser movilizados por conjugacion entre celulas bacterianas. En V. cholerae, los determinantes de resistencia han sido reportados principalmente asociados a plasmidos (12, 17, 18). Estos plasmidos por lo general son gran des, entre 110-170 kb y auto transferibles. Se han reportado plasmidos que portan los genes que codifican la resistencia hasta siete determinantes de resistencia en un solo plasmido (17).
Los integrones son elementos geneticos que ad - quieren por mecanismos de recombinacion especifica, segmentos de DNA exogeno, conocidos como casetes genicos, que usualmente codifican para la resistencia a antibiotico (19). Existen los integrones de multiple resistencia, los cuales contienen varias combinaciones de casetes genicos de resistencia a antibioticos y han sido reportados en cepas de V. cholerae multirresistentes (12, 18, 20-22). Recientemente, ha sido demostrado en V. cholerae, un nuevo tipo de integron, llamado superintegron, de 126 kb, que tiene 179 casetes genicos en una sola estructura (23). Los integrones no son auto transferibles, pero se pueden encontrar en plasmidos conjugativos, asegurando su movilidad (12).
Historicamente, solo las cepas de V. cholerae que pertenecian al serogrupo O1 habian sido asociadas con epidemias de Colera. Sin embargo, en septiembre de 1992 en Bangladesh, un nuevo serogrupo, designado O139, emergio como la cepa causante de un gran brote de Colera. Esta cepa de V. cholerae O139 tenia un pa - tron de resistencia diferente a la cepa de V. choleare O1, era resistente a sulfametoxazole, trimetoprim, cloranfenicol y a niveles bajos de estreptomicina. La resistencia que mostraba esta cepa esta codificada en un elemento genetico integrado al cromosoma, auto transferible, de aproximadamente 62 kb, que es similar a los transposones conjugativos, al cual se le denomino elemento SXT (24). El elemento SXT codifica una integrasa que media su propia integracion y escision (25). Cuando en 1994 re-emergio la cepa V. cholerae O1 El Tor como la cepa pre dominante en Asia, esta cepa fue resistente a sulfametoxazole, trimetoprim, cloranfenicol y estreptomicina y los genes que le conferian esta resistencia se encontraron en un elemento SXT relacionado con el encontrado en la cepa O139 (26). Los aislamientos mas recientes de V. cholerae O139 en la India, perdieron la resistencia a sulfametoxazole y trimetoprim (27). La gran mayoria de los aislamientos clinicos de V. cholerae O1 y O139 de Asia, posteriores a 1993, contienen un elemento SXT relacionado integrado al cromosoma (28).
CARACTERISTICAS DEL GÉNERO VIBRIO
Actualmente, se reconocen 78 especies de Vibrios distribuidas en cinco grupos; Vibrio, Photobacterium, Sa linivibrio, Enterovibrio y Grimontia (29-31). Las es - pecies del genero Vibrio son bacilos Gram negativos, anae robios facultativos, de forma recta o curva, no producen esporas, presentan un diametro entre 0,5 a 0,8 mm y entre 1,4 a 2,4 mm de largo (Fig. 1). La ma yoria de las especies patogenas son moviles por medio de un unico flagelo polar (32). Cerca de la tercera parte de las especies del genero Vibrio son patogenas para el humano y la enfermedad es por lo general, el resultado de la ingestion de agua o alimentos contaminados o por la exposicion de heridas a ambientes acuaticos donde las especies de Vibrio estan presentes (33). Las especies V. cholerae, V. parahaemolyticus y V. vulnificus son los pa togenos humanos mas importantes.
CARACTERISTICAS DE LA ESPECIE VIBRIO CHOLERAE
V.cholerae es una especie bien definida sobre las bases de pruebas bioquimicas. Pero, la especie no es homogenea en lo que respecta a su potencial patogeno. La presencia de antigenos somaticos termoestables O, ha permitido determinar mas de 200 serogrupos de V. cho lerae (34). Solamente dos de ellos se reconocen como responsables de las epidemias de Colera, el O1 (32) y el O139 (35). Los serogrupos no-O1/no-O139, suelen aislarse de fuentes ambientales y pueden producir casos esporadicos de gastroenteritis y de infecciones extra-intestinales. V. cholerae O1 es considerado el serogrupo virulento y epidemico por excelencia. Sin embargo, se ha demostrado la seroconversion de V. cholerae no-O1/no-O139 a O1 (36), y la transformacion natural inducida por quitina de la cepa epidemica por la adquisicion del casete que codifica el antigeno O139 (37).
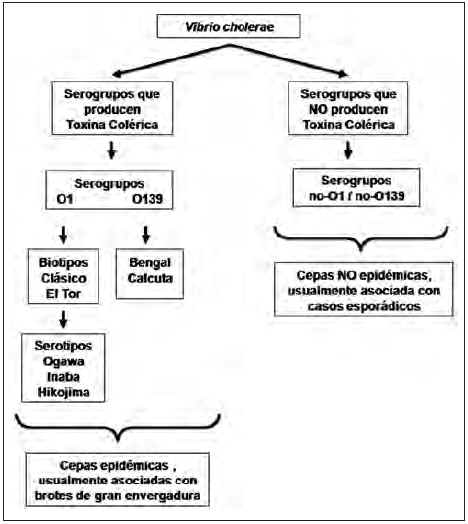
Los aislamientos del serogrupo O1 de V. cholerae se han subdividido en tres serotipos: Inaba, Ogawa e Hikojima, siendo este ultimo muy raro. Las cepas de V. cholerae O1 pueden interconvertir y producir un cambio de serotipo (36, 38, 39). Los aislamientos de V. cholerae O1 tambien pueden dividirse en dos biotipos, El Tor y Clasico, sobre la base de varias caracteristicas fenotipicas. Hasta 1936, los aislamientos de V. cholerae O1 de la epidemia de Colera eran bioquimicamente similares y pertenecian al biotipo Clasico. El Biotipo El Tor se aislo en 1905 y fue considerado no patogeno hasta 1936. La sep tima pandemia esta relacionada con el biotipo El Tor, que es el causante de todos los casos de Colera en el mundo, pues, el biotipo Clasico asociado con la sexta pandemia esta extinto (figura 2).

Figura 1. Fotografias de microscopia electronica de barrido y de transmision de V. cholerae. A: La bacteria presenta forma de coma y un flagelo polar, y habita normalmente ambientes acuaticos; B y C: Vibrios adheridos a la superficie de la mucosa intestinal.
Figura 2. Clasificacion de los serogrupos de V. cholerae en los grupos epidemico y No-epidemico. Los serogrupos O1 y O139 son por ahora los unicos serogrupos asociados con epidemias de Colera.
EL GENOMA DE VIBRIO CHOLERAE
En el año 2000 se publico la determinacion y analisis de la secuencia genomica completa de V. cholerae El Tor (40). El genoma consiste en dos cromosomas circulares de 2.961.146 pb (cromosoma 1) y 1.072.314 pb (cromosoma 2). Existe una pronunciada asimetria en la distribucion de los genes conocidos entre los dos cromosomas. La mayoria de los genes que codifican la replicacion y reparacion del ADN, transcripcion, traduccion, biosintesis de la pared celular, y una variedad de vias centrales de catabolismo y biosintesis, estan codificados en el cromosoma 1. La mayoria de los genes esenciales para la patogenicidad, como la toxina colerica, el pilus co-regulado con toxina, los lipolisacaridos y la maquinaria de secrecion de proteinas extracelulares, tambien estan localizados en el cromosoma 1. En contraste, el cromosoma 2 contienen una gran parte (59%) de marcos abiertos de lectura (ORFs) hipoteticos y ORFs de los que se desconoce su funcion. La secuencia genomica confirma la presencia de un gran sistema de captura de genes, conocido como isla de integrones. Los casetes de este superintegron aparentemente codifican funciones adaptativas y se ha determinado una gran diversidad entre las cepas toxigenicas y no toxigenicas (23, 41). Recientemente se identifico el primer gen de virulencia funcional en el superintegron (enterotoxina termo estable), lo cual sugiere que el superintegron es un mecanismo por el cual los miembros de esta especie pueden importar genes exogenos (42).
Varias evidencias sugieren que este cromosoma 2, fue originalmente un megaplasmido, capturado por una es pecie ancestral de Vibrio. La estructura de los dos cro mosomas se encuentra en otras especias de Vibrio, su - giriendo que los genes contenidos en el megaplasmido quizas proveyo al Vibrio una ventaja evolutiva en el ecosistema acuatico. Los elementos SXT parecen ser una adicion reciente al genoma de V. cholerae, pues no estan presentes en los aislamientos de la septima pandemia, como puede ejemplificarse por su ausencia en el genoma de la cepa de V. cholerae N16961, la cepa tipo utilizada pa ra la determinacion de la secuencia genetica completa del cromosoma por el Institute for Genome Research (40).
DIAGNÓSTICO DE LABORATORIO CLINICO
V. cholerae sobrevive bien en las muestras fecales si se mantiene la humedad, pero si se produce una demora de mas de unas pocas horas en el traslado al laboratorio, se debe utilizar el medio de transporte Cary-Blair. Las heces, tanto frescas como en medio de transporte, deben sembrase en agar de tiosulfato citrato sales biliares sacarosa (TCBS), que inhibe la mayoria de la flora fecal habitual, pero soporta el crecimiento de los Vibrios.
Adicionalmente, la muestra debe ser inoculada en un caldo de agua peptonada alcalina (APA), un caldo de enriquecimiento de pH alto, el cual soporta de manera preferente el crecimiento de Vibrios. Despues de 6-12 horas de incubacion, el caldo APA debe ser subcultivado en una segunda placa de TCBS. Estas placas se incuban por 18-24 horas y las colonias de V. cholerae se observaran como amarillas, brillantes, con el centro ligeramente levantado. La identificacion presuntiva se puede realizar en base a las caracteristicas de las colonias, las cuales son oxidasa positiva y aglutinan con el antisuero O1 o O139 (43). En la actualidad se estan utilizando numerosos metodos de biologia molecular tanto para la identificacion de las especies de Vibrio, como para la determinacion de genes de virulencia y la deteccion en muestras clinicas y ambientales (44-47).
FACTORES DE VIRULENCIA
A nivel molecular, la patogenesis del colera es un proceso multifactorial que involucra varios genes que codifican factores de virulencia que ayudan al patogeno para la colonizacion y la expresion coordinada de la toxina (Tabla 1). La diarrea masiva inducida por V. cholerae es causada por la toxina colerica (CT) (4), aun cuando se ha reportado que cepas que pierden los genes que codifican para la CT, todavia pueden causar diarrea.
Tabla 1. Factores de virulencia de Vibrio cholerae

TOXINA COLÉRICA
Cada molecula de CT, esta compuesta por cinco subunidades B (de enlace) y una subunidad A (activa). Las subunidades B se unen a los receptores del gangliosido GM1 en las celulas epiteliales de la mucosa intestinal. Despues de la union, se separan la subunidad A1 y el componente A2, lo cual facilita la entrada del componente A1 en la celula. El componente A1 de la toxina colerica estimula la produccion de la enzima adenilciclasa, involucrada en la produccion del monofosfato ciclico de adenosina (AMPc). Las altas concentraciones intracelulares de AMPc alteran el transporte activo de los electrolitos a traves de la membrana celular, lo cual impide la adsorcion de liquido y conduce a su secrecion en el intestino delgado.
La enterotoxina de V. cholerae esta codificada por los genes ctx. El gen ctxA codifica la subunidad A de la toxina, y el gen ctxB codifica la subunidad B. Los genes son parte del mismo operon ctxAB. El transcrito (ARNm) del operon ctx tiene dos sitios de union a ribosoma (RBS), uno en el gen A y otro en el gen B. Este ultimo es, por lo menos, siete veces mas fuerte que el sitio de la region A. De esta forma el organismo es capaz de traducir mas proteinas B que A, lo cual se requiere para ensamblar la toxina en la proporcion apropiada (1A:5B). Los componentes son ensamblados en el periplasma despues de la traduccion. Cualquier subunidad B extra puede ser excretada por la celula, pero la subunidad A debe estar adherida a 5 subunidades B, para poder salir de la celula. La subunidad A intacta no es activa enzimaticamente, y se debe fraccionar en los fragmentos A1 y A2, los cuales estan unidos por un puente disulfuro (48).
El operon ctxAB, forma parte del genoma del bacteriofago filamentoso CTXf, lisogenizado en la bacteria (40). Este genoma fagico esta compuesto por dos dominios funcionalmente distintos, la region central o "core" y la region RS2 (49). En la region central se encuentran, entre otros, los genes de la toxina colerica (ctxAB), los genes implicados en la morfogenesis del bacteriofago (psh, cep, orfU, y ace) y un gen que codifica una proteina necesaria para el ensamblaje del virion (zot). Antes de conocer este origen fagico, algunos de estos genes se consideraban que codificaban otras toxinas relacionadas con la patogenesis del Colera, ya que se habia detectado, por ejemplo que el zot (zonula occludens toxin) aumentaba la permeabilidad de la mucosa intestinal (50) y el ace (accesory cholera enterotoxin) era capaz de inducir la acumulacion de liquidos (51). La region RS2 incluye los genes necesarios para la replicacion (rstA), integracion (rstB) y regulacion (rstR) de CTXf, tambien contiene dos regiones intergenicas ig-1 y ig-2 (52). Dentro de las celulas de V. cholerae, el genoma de CTXf puede existir como la forma replicativa o co mo profago integrado dentro del cromosoma (49). Bajo condiciones apropiadas, las cepas toxigenicas de V. cho lerae pueden ser inducidas para producir particulas ex tracelulares de CTXf (49, 53). Las cepas ambientales no toxigenicas pueden convertirse por transduccion con CTXf (53).
El profago de CTXf esta usualmente flanqueado por un elemento genetico relacionado conocido como RS1, si milar genetica y funcionalmente a la region RS2 del CTXf (52). No se han identificado en aislamientos na turales de O1 El Tor y O139 profagos solitarios, no flan - queados por RS1 u otro profago (54). El elemento RS1 por ta un ORF denominado rstC que no esta presente en RS2 (52). RstC es un anti represor que controla la produccion de particulas de CTXf y la expresion de la toxina colerica (55). Todo esto sugiere una interrelacion entre los progafos RS1f y CTXf que promueve una diseminacion mas eficiente de los genes ctx, mientras, simultaneamente, aumenta el desempeno en virulencia de aquellas cepas de V. cholerae que portan los genes de TCP.
ISLA DE PATOGENICIDAD
Las islas patogenicas (PAIs) son segmentos de ADN bacteriano que portan uno o mas genes de virulencia, adquiridos en bloque a partir de una fuente externa. Las PAIs son identificadas por su diferencia en el porcentaje de contenido de G+C en relacion a la media del cromosoma y por otras caracteristicas, que sugieren su adquisicion a traves de elementos geneticos moviles (56). La Isla de patogenicidad 1 de V. cholerae (V. cholerae pathogenicity island, VPI-1) tiene un tamano de 39,5 kb. Todos los genes en VPI-1 son potencialmente importantes para producir la enfermedad, ya sea que tengan un rol directo en la patogenesis o indirecto en la transferencia y motilidad de VPI-1. El elemento VPI-1 contienen genes como tcpA que codifica un importante factor de colonizacion y el receptor para CTXO, y los genes toxT, tcpP y tcpH que codifican factores reguladores y de virulencia (57). La identificacion de genes potenciales de una integrasa y una transposasa a cada lado de VPI-1 sugieren la transferencia e integracion de VPI en cepas epidemicas y pandemicas (57).
La isla VPI-2, es una region cromosomal de 57,3 kb con caracteristicas de una isla de patogenicidad (58). Todas las cepas toxigenicas de V.choleraeO1 y O139 contienen VPI-2, mientras los aislamientos no toxigenicos no-O1/no-O139 no tienen esta region. VPI-2 codifica varios grupos de genes: un sistema de restriccion por modificacion tipo 1, el cual podria proteger a la bacteria de la in feccion por virus (59); un grupo de genes nan.nag ho - mologos a los genes involucrados al metabolismo del acido sialico, el cual podria actuar en la obtencion de fuente de carbono y nitrogeno (60); una Neuraminidasa, la cual actua en los gangliosidos de alto orden en el in - testino y convertirlos en gangliosidos GM1, los cuales sub secuentemente liberarian acidos sialicos (61); y una region con homologia al fago Mu (58).
El analisis comparativo del genoma de V. cholerae usando la tecnica de microarreglos de DNA, determino diferencias en el contenido entre la cepa de la sexta pandemia (Clasico) y la septima pandemia (El Tor). Se han identificado dos regiones designadas como: Isla 1 del Vibrio de la septima pandemia (Vibrio seventh pandemic island-I, VSP-I) y VSP-II que unicamente estaban presentes en las cepas de la septima pandemia El Tor (62). VSP-I y VSP-II muestran varias caracteristicas de isla de patogenicidad. VSP-I es una region de 16 kb y con tiene 11 ORFs, con un contenido de GC de 40%, en contraste con el promedio para todo el cromosoma del 47% (62). La region VSP-II tiene un tamano de 7.5 kb, y contiene ocho ORFs (62). Estas dos PAIs y VPI-2 se pueden escindir del cromosoma y formar una molecula in termediaria, circular, la cual es el primer paso para su transferencia horizontal (63).
MOTILIDAD
Se ha sugerido que la motilidad contribuye a la reactogenicidad de los candidatos a vacuna de una cepa atenuada, por promover la penetracion de la barrera mu cosa (64). El papel de la motilidad en la patogenesis del colera no esta completamente entendido. Los primeros estudios usando mutantes no motiles, producian resultados conflictivos del rol de la motilidad en la adherencia y enterotoxicidad, en el modelo de asa ileal de conejo (65). La inhibicion de la motilidad incrementa la trascripcion de toxT en el biotipo El Tor, pero no previene la induccion de ctxA y tcpA en el intestino de los ratones. Sin embargo, en el modelo de asa ileal de conejo, la motilidad es necesaria para la expresion completa de la enterotoxicidad (66). Esto sugiere que la relacion entre la movilidad y la expresion de los factores de virulencia es compleja.
HEMAGLUTININA / PROTEASA
V. cholerae produce una hemaglutinina/proteasa (HapA), codificada por hapA (67), tambien involucrada como factor de virulencia. HapA puede degradar proteoliticamente varias proteinas de importancia fisiologica pa ra el hospedero, incluyendo la mucina (68). HapA perturba la barrera paracelular en los cultivos de celulas de epitelio intestinal, por la accion en las proteinas asociadas a la zona ocludens (69) y se ha demostrado que se requiere la expresion de hapA para que V. cholerae penetre un gel de mucina in vitro (70). Aunque el analisis de los efectos de mutantes del gen hapA en conejos infantes y ratones lactantes no ha provisto evidencia que HapA es un factor esencial de virulencia (67), se ha probado que la proteasa HapA contribuye a la reactogenicidad en las vacunas de colera de cepas atenuadas en humanos (71). La perdida de HapA no afecto la colonizacion en el modelo de ratones lactantes, sin embargo la motilidad y HapA son necesarios para la expresion total de la enterotoxicidad en asa ileal de conejo (66).
TOXINA MARTX DE VIBRIO CHOLERAE
La secuencia de nucleotidos del genoma de V. cholerae y su analisis con tecnicas informaticas, ha revelado la presencia de otras toxinas desconocidas, entre ellas, la Toxina RTX multifuncional autoprocesada de Vibrio cholerae (multifunctional, autoprocessing RTX toxin, MARTXVc). Esta proteina es homologa a los miem - bros de la familia de las toxina RTX (Repeats in toxin), que contienen un motivo de repetidos ricos en GD. Esta codificada por un elemento genetico situado muy cerca del lugar de insercion del genoma del bacteriofago CTXf, aunque su actividad es independiente del elemento CTX (72). El gen rtxA es el marco abierto de lectura mas largo en el genoma de V. cholerae (40), y se encuentra tanto en los aislamientos clinicos como los ambientales (73, 74). La MARTXVc es secretada por un sistema T1SS atipico, compuesto por cuatro componentes (75). Esta toxina rompe los filamentos de actina del citoesqueleto de un amplio rango de tipos celulares (76). Esta disrupcion se produce por la inactivacion de las GTPasas pequenas, Rho, Rac y Cdc42 (77). Adicionalmente esta toxina posee otro dominio responsable del entrecruzamiento (actin cross-linking domain, ACD), el cual utiliza G-actina como substrato (78).
HEMOLISINA
La mayoria de los aislamientos de V. cholerae O1 biotipo El Tor y no-O1/no-O139 producen una exotoxina hemolitica, llamada El Tor Hemolisina o V. cholerae citolisina (VCC) (79-81), codificada por el gene cromosomal hlyA (40). Se ha reportado que VCC es capaz de causar severos efectos citotoxicos, tales como: lisis celular, vacualizacion e incremento de la apoptosis en celulas del epitelio intestinal (82-84). Esta hemolisina se puede encontrar como monomero con actividad hemolitica y como oligomeros que aglutinan eritrocitos. Estas diferentes formas conducen a las celulas inmunes a diferentes direcciones, los oligomeros inducen la expresion de IgM e IgA, sin embargo, la mayoria de las celulas tratadas con monomeros resultan en apoptosis (85).
HEMAGLUTININA MANOSA SENSIBLE
Una estrategia bacteriana alternativa para sobrevivir en el ambiente, es la formacion de Biopeliculas. Estas estructuras son comunidades microbianas que se forman por la adherencia de una bacteria a una superficie, a la que posteriormente se adicionan multiples capas de interacciones celulas-celulas y desarrollan una estructura tridimensional, que incluyen canales de agua a traves de los cuales entran nutrientes y salen productos de desecho (86). En los ambientes acuaticos existen diferentes superficies sobre las cuales se pueden formar biope liculas. Estas superficies incluyen particulas de minerales en suspension, plantas, y los exoesqueletos de los crustaceos y zooplancton. V. cholerae O1 El Tor se puede diferenciar del Clasico por la presencia de un pilus del tipo IV, llamado Hemaglutinina manosa sensible (mannose-sensitive hemagglutinin, MSHA), codificado por el gen mshA (87), implicado en la formacion de biopeliculas en superficies abioticas y que facilita la supervivencia de la bacteria en ambientes acuaticos (88).
TOXINA CHOLIX
Esta toxina tiene actividad de ADP-ribosiltransferasa, y es activa contra celulas de mamiferos y crusta ceos, con actividad especifica contra el factor ribosomal de elongacion tipo 2. Para su accion requiere de endo citosis mediada por un receptor, traslocacion al cito plasma hospedero, donde produce la inhibicion de la sintesis proteica por modificacion especifica del factor de elongacion. Se ha propuesto que este factor juega un rol importante en la supervivencia del microorganismo en el ambiente acuatico (89).
QUORUM-SENSING
Las bacterias son capaces de producir unas moleculas pequenas difusibles, llamadas auto-inductores. Estas moleculas se acumulan en el medio cuando ocurre un incremento de la densidad celular, y regulan la ex presion de un amplio numero de genes, que a su vez controlan una variedad de funciones fisiologicas, en un proceso que se conoce como "quorum-sensing" (90, 91). Este pro ceso permite a las bacterias comunicarse, secretando senales quimicas, habilitando asi a la poblacion bacteriana a regular de forma colectiva la expresion de ge nes, y por lo tanto su desempeno como comunidad. Este "censo-poblacional", le permite al grupo bacteriano modular la expresion de genes especificos solo en ciertos momentos de densidad de poblacion, aquellos que serian improductivos que ocurrieran por una bacteria individual, pero que son efectivos cuando se realizan en grupo. Asi, el "quorum-sensing" es un proceso que involucra la secrecion y deteccion de autoinductores que les permite a las bacterias funcionar como un organismo mul ticelular. En V. cholerae se ha demostrado que el "quorum-sensing" regula negativamente la expresion de los genes de virulencia (92, 93). Los principales autoinductores en V. choleare son: Colera autoinductor 1 (CAI- 1) y el autoinductor 2 (AI-2), que funcionan de manera sinergistica para la regulacion de los genes (94). La acumulacion de los auto-inductores modula la actividad de un regulador central, LuxO, a traves de los receptores de membrana CqsS y LuxPQ (92). Cuando hay una baja densidad celular, LuxO reprime la expresion de un regulador maestro "quorum-sensing", HapR, por la activacion de la expresion de un grupo de RNA pequenos (95). La represion de hapR permite la expresion de tcpP en la isla de patogenicidad de V. cholerae. En este punto, el resultado es la expresion de los factores de virulencia que le permiten a V. cholerae colonizar el intestino delgado, multiplicarse y producir la toxina colerica. HapR tambien reprime el operon vps (Vibrio polyssacharide synthesis), por lo que regula negativamente la formacion de biopeliculas (96). Adicionalmente, HapR directamente regula po sitivamente la expresion de hapA, lo cual produce una hemaglutinina/proteasa secretora, responsable de la adherencia de los Vibrios al epitelio intestinal (97). En resumen, HapR reprime el regulon de virulencia, los genes relacionados con la formacion de biopeliculas y activa la produccion de proteasa, que puede promover la li beracion de las celulas de V. cholerae adheridas, lo que facilita el establecimiento de un nuevo punto de in - feccion en el epitelio gastrointestinal, o alternativamente, promover la salida de V. cholerae del hospedador y asi en condiciones de alta densidad celular se inhibe la virulencia en la bacteria.
Se ha sugerido que el sistema sensorial de dos com ponentes VarsS/VarA forma parte de un sistema regulatorio dependiente de "quorum-sensing", el cual modula la expresion de genes en V. cholerae (98). Mas aun, la expresion de hapR es reprimida en alta densidad celular por si misma, aunque el significado de esta auto represion no se conoce (99). Tambien se ha identificado un regulador transcripcional, VqmA, que modula el "quo rum-sensing" de V. cholerae pues activa la expresion de hapR cuando hay baja densidad de celulas (100). Adicionalmente, se ha reportado que el "quorumsensing" aumenta la viabilidad de V. cholerae, bajo ciertas condiciones de estres, mediante la regulacion de la expresion de RpoS (factor sigma de la RNA polimerasa de fase estacionaria) (101). Por otra parte, se requiere la proteina receptora de AMPc (CRP) para la biosintesis de CAI-1, que afecta la expresion de multiples genes regulados por HapR (102), poniendose asi de manifiesto la intrincada red regulatoria que garantiza la sobrevivencia de la bacteria y del hospedero.
EL CÓLERA EN EL MUNDO
La distribucion de las enfermedades infecciosas en el mundo esta variando continuamente, debido a los cam bios que se producen en el patogeno, en el ambiente y en la poblacion hospedadora. El incremento de la resistencia a los antibioticos, el sobrecalentamiento del planeta, la globalizacion de los mercados y las migraciones de poblaciones, son los principales factores que intervienen en el desarrollo y proliferacion de este grupo de enfermedades. Dentro de este grupo de enfermedades infecciosas se encuentra el Colera, que presenta una mayor incidencia en los paises en desarrollo de clima tropical, llegando a adquirir caracteristicas epidemicas. El numero de casos de Colera reportados a la OMS durante el ano 2006 aumento dramaticamente alcanzando los niveles que se observaban durante el final de los 1990s. Un total de 236.896 casos fueron notificados de 52 paises (Figura 3), incluyendo 6.311 defunciones (tasa de mortalidad 2,66%), un incremento del 79% comparado con el numero de casos reportados durante el 2005 (103).
Figura 3. Paises/Areas que reportaron casos de Colera en el 2006.
V. cHOlERAE O 139
A finales de 1992, aparecieron epidemias de Colera en India y Bangladesh, causadas por una cepa que no se aglutinaba con los antisueros O1 y no-O1 disponibles hasta (35). Esta nueva cepa, a la que se denomino O139 Bengal (104), se propago rapidamente a los paises vecinos y desplazo los casos de Colera originados por el serogrupo O1. El cuadro clinico que producia era identico al causado por las cepas O1. Despues de la aparicion explosiva, su presencia declino paulatinamente hasta 1994, cuando un nuevo clon de V. cholerae O1 El Tor reemplazo el serogrupo O139 en Calcuta. En 1996, reaparecio en Calcuta un nuevo clon de V. cholerae O139, siendo el serogrupo dominante durante el pe - riodo 1996-1997 (105). Desde 1998 hasta la actualidad, V. cholerae O139 sigue confinado en el continente asiatico, principalmente en China.
A partir de estudios moleculares, se ha demostrado que las cepas de V. cholerae O139 estan muy relacionadas con las cepas O1 El Tor de la septima pandemia, pe ro tienen varias caracteristicas que la distinguen. Todo ello, ha contribuido a que algunos autores postulen que este nuevo serogrupo O139 se origino a partir de una cepa O1 El Tor que adquirio ADN extra cromosomico (106). Tambien se ha comprobado un cambio en la estructura y organizacion del profago CTXf en las ce - pas O139 (107). La repentina aparicion del se ro grupo O139 en 1992, su rapida propagacion por el su deste asiatico en 1993, seguido por un periodo de quies cencia durante los anos 1994 y 1995, y su reemergencia y dominacion durante los anos 1996 y 1997, son pruebas de la impredecible epidemiologia del Colera.
La pandemia actual, que comenzo en Indonesia en el ano 1961 (108), sigue su curso, y despues de cuarenta anos no muestra una tendencia al descenso. El Colera, presenta diferentes patrones epidemiologicos, segun la region que afecta. En determinados paises, en su mayoria en vias de desarrollo, la enfermedad es endemica, y en otros paises aparece en forma de brotes epidemicos esporadicos. Las dos areas endemicas mas importantes son: Africa y Asia. En cambio, en los paises desarrollados se detectan casos esporadicos, fundamentalmente importados de areas endemicas o en las que existe un brote epidemico.
EL CÓLERA EN AMÉRICA
La deteccion del Vibrio cholerae O1 en los pacientes de un pueblo costero peruano en enero de 1991, senalo la llegada de la septima pandemia del Colera a America Latina, la cual se disemino rapidamente, a razon de aproximadamente un pais por mes (109, 110). Desde 1991 a 1998, America Latina notifico 1.264.426 casos de Colera con 12.535 defunciones (111). Para el ano 2006, permanece estable el numero bajo de casos en las Americas, solo se han notificado casos en Norte America; Canada (2 casos importados) y los Estados Unidos (4 casos importados y 4 casos autoctonos) (103) (Figura 3).
EL CÓLERA EN VENEzUELA
En Venezuela, el Colera llega en diciembre de 1991 y hasta el ano 1993 se reportaron 5.420 casos, 80 de estos resultaron en muerte (112). Despues de 2 anos (1994-1995) sin haberse reportado ningun caso de Co le - ra en Venezuela, a principios de junio de 1996 se de tec ta un brote en el estado zulia, producido por V. cholerae O1 del serotipo Ogawa. El brote continuo su ex pansion y para el ano 1997 el Colera se habia extendido por 14 entidades federales, registrandose 2.557 ca sos con una tasa de letalidad de 2,3% (113). Luego de va rias semanas sin reporte de casos, a finales del ano 1998 se detecta un segundo brote de Colera en el estado Delta Amacuro (114), producido por V. cholerae O1 del serotipo Inaba, el cual se extendio a los estados, Su cre, Nueva Esparta, Monagas, Anzoategui y Miranda (115). Desde el ano 2000 no se han reportado en Ve nezuela nuevos casos de Colera (Figura 4).
Figura 4. Numero de casos de Colera reportados durante los brotes de 1996-1997 y 1998-1999 en Venezuela. En junio de 1996 se detecta un brote producido por V. cholerae O1 del serotipo Ogawa. Luego a finales del ano 1998 se detecta un segundo brote de Colera producido por V. cholerae O1 del serotipo Inaba. Desde el ano 2000 no se han reportado en Venezuela nuevos casos de Colera.
EPIDEMIOLOGIA MOLECULAR
Las investigaciones epidemiologicas del Colera han estado limitadas por los sistemas de tipificacion disponibles, tales como, identificacion bioquimica o serologica, los cuales proveian poca informacion para la discriminacion entre las cepas. Las cepas epidemicas, son por lo ge neral, del serogrupo O1, y en este serogrupo, solo existen dos biotipos, Clasico y El Tor, y solo dos serotipos Inaba y Ogawa (Figura 2). La determinacion de los biotipos requiere de pruebas que en ocasiones son dificiles de desarrollar, y algunos marcadores, como la actividad hemolitica, han cambiado con el tiempo (116). La electroforesis que discrimina enzimas de multiples lo cus (Multilocus enzyme electrophoresis, MEE) puede distinguir entre el biotipo Clasico y El Tor y tambien ha agrupado las cepas toxigenicas del biotipo El Tor en cuatro grupos clonales grandes, o tipos electroforeticos (electrophoretic types, ET), que representan grandes areas geograficas (117). Estas incluyen el clon australiano (ET1), el clon de la costa del golfo de Mexico (ET2), el clon de la septima pandemia (ET3) y el clon de Latinoamerica (ET4). Actualmente, con el desarrollo de la epidemiologia molecular se han hecho grandes avances, al utilizar diferentes tecnicas.
El analisis de los perfiles plasmidicos fue una de las primeras tecnicas de sub-tipificacion utilizada como marcador epidemiologico efectivo para muchos organismos productores de diarrea (118). Las cepas de Vibrio cholerae O1 biotipo Clasico contienen ciertos plasmidos (119), pero las cepas de Vibrio cholerae O1 biotipo El Tor rara vez se ha demostrado la presencia de plasmidos utiles como marcadores epidemiologicos. En 1982, el biotipo Clasico produjo un brote de Colera y los investigadores usaron el perfil de plasmidos para probar que 42 cepas del biotipo Clasico aisladas durante este brote fueron practicamente indistinguibles de los aislamientos obtenidos durante los anos 1916 y 1921 (119). Otro metodo para subtipificacion aplicado en Colera fue el analisis por "Southern" del ADN celular completo, utilizando una sonda dirigida a identificar el gen de la subunidad A de la Toxina Colerica. Aunque el cromosoma bacteriano contiene solo una o dos copias de los genes de toxina co lerica, se pueden diferenciar los aislamientos de acuerdo al numero y la localizacion de estos genes en el cromosoma. El uso de esta tecnica permitio determinar que los aislados de V. cholerae O1 toxigenicos del Golfo de Mexico son diferentes a los aislados de la septima pandemia, al menos en relacion al gen que codifica CT (120). La fuente del brote original desarrollado en el Pe - ru permanece sin demostrar, sin embargo, las tecnicas moleculares permitieron definir la cepa epidemica y señalar que estaba relacionada con los aislamientos de pa cientes de la septima pandemia de otras partes del mundo (121), aunque fallo en indicar la presencia de la misma cepa en otra parte del mundo.
Los genes que codifican el ARN ribosomal (ARNr) son secuencias conservadas durante la evolucion y se encuentran en copias multiples en todos los cromosomas de todas las bacterias (122), pero muestran gran diversidad, tanto en su localizacion dentro del cromosoma, como en secuencia de nucleotidos en ciertas regiones variables. Esta caracteristica ha sido utilizada para la Ribotipificacion de V. cholerae (15). Este esquema pro pone 27 Ribotipos diferentes con la enzima de restriccion BglI. La aplicacion de la Ribotipificacion permitio distinguir las cepas de la epidemia de Latino America, y los aislados de la Costa del Golfo, de las otras cepas que causaron la septima epidemia de Colera en el resto del Mundo. Estas ultimas pertenecen a los ribotipos 3 al 8. La mayoria de las cepas aisladas en la epidemia de Latino America pertenecen al Ribotipo 5, en Mexico las cepas aisladas entre 1991 y 1993, pertenecen a los ribotipos 5 y 6a (15, 123). Las cepas de Vibrio cholerae O1 resistentes a antibioticos aisladas en Mexico, Guatemala y Brazil, pertenecen al Ribotipo 6a (15), este Ribotipo no se encontro entre los aislamientos del Peru, los cuales fueron sensibles a todos los antibioticos probados (124). La Ribotipificacion de V. cholerae aisladas en di ferentes partes del mundo, desde 1941, demostro que: (i) los biotipos Clasico y El Tor son clones diferentes; (ii), la pandemia de Colera no es una epidemia de una sola cepa alrededor del mundo debida a un solo clon, sino mas bien, es la ocurrencia simultanea de varias epidemias, con varios clones involucrados; y (iii), las olas de epidemias debidas al biotipo El Tor pueden deberse a la emergencia de nuevos clones (125). El estudio de 75 cepas de V. cholerae O1, aisladas en Pe ru en el periodo 1991-1999 evidencio la existencia de tres ribotipos diferentes, observandose la aparicion cons tante y mayoritaria de uno de los ribotipos (126).
Recientemente se ha utilizado la tecnica de electroforesis en campo pulsado (Pulsed Field Gel Electropho - resis, PFGE) que permite discriminar polimorfismos en la longitud de los fragmentos de restriccion (Restriction Fragment Length Polymorphism, RFLP) para los estudios epidemiologicos a nivel molecular. La PFGE per - mite la separacion de fragmentos grandes de ADN, generados por la digestion del ADN genomico con una endonucleasa de restriccion que corta el ADN de forma infrecuente, generando pocos fragmentos. La utilizacion de esta tecnica en V. cholerae O1 con la enzima de restriccion Not-I permitio una identificacion rapida de las cepas, de manera mas efectiva que la ribotipificacion (127). En primera instancia, todos los aislamientos de la epidemia en Latinoamerica tenian un patron de PFGE identico (127). Posteriormente, encontraron 8 tipos di - ferenciados por PFGE en 50 cepas de V. cholerae O1 ais ladas en Peru, desde 1991 a 1995 (124). Los autores es timan que los cambios geneticos en la cepa responsable de la epidemia en Latinoamerica estan ocurriendo mas frecuentemente que lo que se habia reportado previamente. Se han reportado 9 genotipos diferentes en las cepas de V. cholerae aisladas en Iran, demostrando una gran diversidad en los aislamientos del serotipo O1 (128). Cuando se utilizo esta tecnica para la comparacion de V. cholerae O1 y O139, tambien se demostro que ambas cepas tienen un origen clonal (129). Esta tecnica fue utilizada para investigar el origen de los casos domesticos no relacionados epidemiologicamente en Japon, encontrando que 86,1% de los casos pertenecian a un mismo clon, a diferencia de los casos importados los cuales se pudieron separar en 13 subtipos diferentes, sugiriendo que una cepa de V. cholerae O1 se habia diseminado en varias partes de Japon y esporadicamente producia Colera (130).
La tecnica conocida como Amplificacion al Azar de ADN Polimorfico (Random Amplification of Polymorphic DNA, RAPD), utiliza diferentes juegos de iniciadores con secuencias arbitrarias y al azar, para permitir la amplificacion de regiones del ADN de la cepa en analisis. La hibridacion de los iniciadores se realiza en condiciones de baja estrictez, por lo que pueden encontrar regiones a las cuales aparearse y permitir la amplificacion de varios fragmentos del genoma. Bajo condi ciones estandarizadas, este metodo produce distintos patrones para cada muestra analizada, que pueden ser comparados con los patrones de otras muestras (131-133). Sin em bargo, se ha reportado que cuando se utilizan diferentes preparaciones de ADN (ADN de diferente calidad) y diferentes fuentes de enzimas e iniciadores, esto produce diferencias en el patron de bandas observado y una baja reproducibilidad, por lo que el metodo RAPD no es adecuado para la creacion de una base de datos para la identificacion de cepas de V. cholerae (134). Pero, debido a su sim plicidad y capacidad discriminatoria, esta tecnica es util en la deteccion de diversidad genetica entre microorganismos procesados al mismo tiempo.
Con el desarrollo de las tecnicas moleculares, han apa recido nuevas metodologias, como el tipiado se se cuencias multilocus (Multilocus Sequence Typing, MLST), que analizan la variabilidad genica mediante secuenciacion del ADN. Sobre la base del analisis de las variaciones en las secuencias de genes, se ha sugerido que las ultimas dos pandemias de Colera podrian estar causadas por clones independientes, que emergieron de V. cholerae ambientales, no toxigenicos, no- O1(135). Esta tecnica permite hacer inferencias de ca racter evolutivo de forma global, pero no es util para conocer la epidemiologia a corto plazo.
EL VIBRIO CHOLERAE Y EL AMBIENTE
En vista que la historia del Colera se ha caracterizado por oleadas epidemicas, hasta principios de 1980 se pensaba que V. cholerae era un microorganismo altamente adaptado a su huesped y que no "sobrevivia" mas de pocas horas o dias fuera del intestino humano. Se ha propuesto el termino de "viables no cultivables" para describir el estado en el cual las bacterias son detectables por contaje directo de celulas viables, pero no son cultivables (136). Se ha reportado la existencia de V. cholerae "viable no cultivable", en respuesta al agotamiento de nutrientes u otras condiciones ambientales desfavorables (137). Este fenomeno representa una nueva perspectiva en la vision de la "sobrevivencia" de V. cholerae en el ambiente, ya que permanece viable en los ambientes acuaticos y es capaz de transformarse al estado cultivable cuando las condiciones le son favorables (138).
Se ha descrito que V. cholerae puede adherirse a la quitina de los caparazones de algunos crustaceos y coloniza las superficies de algas, fitoplancton, copepodos y raices de plantas acuaticas (105, 139). Se ha sugerido que V. cholerae adherido a plancton, entra en una estado "viable no cultivable" como parte de su mecanismo para "sobrevivir" en el ambiente. Las implicaciones epidemiologicas del estado "viable no cultivable" son considerables, pues cuando es inoculado en el asa ileal de co nejo o probado en voluntarios humanos, causa acumulacion de una gran cantidad de fluido y diarrea, respectivamente (139).
VACUNACIÓN
La observacion que la infeccion natural de Colera confiere inmunidad efectiva y de largo plazo ha conducido a realizar esfuerzos para el desarrollo de vacunas. Una vacuna inyectable elaborada con organismos muertos, fue desarrollada poco despues del descubrimiento de V. cholerae durante los anos 1880 y fue am pliamente utilizada en todo el mundo, siendo un requisito para los viajeros internacionales. Sin embargo, no era costo efectiva como un sistema de intervencion en salud publica, porque la proteccion era corta, 6 meses, estaba asociada con dolor local, reaccion inflamatoria y no prevenia la diseminacion de la enfermedad (140). Tambien han sido utilizadas como vacunas diferentes fracciones purificadas de lipopolisacarido, con exitos variables. La toxina colerica puede ser convertida en toxoide en presencia de formalina y de gluraladehido, pero es un antigeno pobre y su uso ha resultado en una proteccion de bajo nivel. En la actualidad, las investigaciones mas recientes estan dirigidas al desarrollo de vacunas orales, que produzcan una proteccion substancial sin efectos adversos (141). Las vacunas orales no previenen todos los casos de colera debido a que la inmunidad local intestinal puede ser sobrepasada por un alto inoculo bacteriano, pero puede disminuir el riesgo tanto como 80% si se utiliza de forma regular. Ademas, un programa de vacunacion podria trabajar de forma sinergistica con los programas de sanitacion, por lo que el inoculo necesario para causar la enfermedad aumentaria, y el numero de organismos patogenos en el ambiente podrian descender. asi, los programas de vacunacion y sanitacion no deben ser vistos como estrategias alternativas de prevencion sino mas bien como complementarias.
EL CÓLERA UNA ENFERMEDAD EMERGENTE Y REEMERGENTE
La epidemiologia moderna comenzo con el trabajo realizado por John Snow. Su laborioso estudio le condujo a descubrir la forma de transmision de la enfermedad desde las aguas contaminadas de un pozo de agua, del cual se abastecieron los enfermos, aun antes que se conociera la existencia de la bacteria (142). Con su trabajo, Snow definio las caracteristicas epidemiologicas del Colera y establecio las bases para su prevencion y control. las caracteristicas de la epidemiologia del Colera son (i) un alto grado de agrupamiento de los casos por localidad o estacion, (ii) altas tasa de infeccion en los ninos de 1 a 5 anos en las areas donde la enfermedad es endemica, (iii) los patrones de resistencia a los antibioticos cambian frecuentemente y de ano en ano, (iv) existe una diversidad clonal de las cepas y (v) la proteccion contra la enfermedad se logra por mejoras en las condiciones sanitarias y en la inmunidad existente. el Colera ha sido catalogado como una "enfermedad emergente y reemergente" que amenaza a los paises en desarrollo (143).
Existen varios eventos que resaltan la importancia epidemiologica de esta enfermedad que incluyen la reemergencia del Colera en latinoamerica en 1991 (109, 144), el brote explosivo de Colera entre los refugiados de ruanda en Goma zaire, el cual resulto en cerca de 70.000 casos y 12.000 muertes en 1994, y la emergencia de V. cholerae o139 en el sub continente indio durante 1992-1995 (104). Cabe destacar que, las cepas de la septima pandemia estan en continua evolucion y es razonable esperar que evolucionen en nuevos clones patogenicos a traves de la adquisicion de nuevos factores de virulencia o adaptacion.
CONCLUSIÓN
Los estudios recientes han acelerado el entendimiento del Colera. los resultados de distintos grupos de in vestigadores han establecido que el genoma de V. cho lerae esta salpicado de elementos geneticos de origen gexternoh. la transferencia horizontal de genes tie ne influencia en la patogenicidad y ha sido importante en la evolucion de cepas de V. cholerae, como se evidencio en la aparicion del brote del serogrupo o139. Todavia permanecen sin descifrar varios misterios en el origen de las cepas patogenas, pero con las nuevas tecnologias de la proteomica y pos-proteomica, esperamos que se develen datos significativos que puedan dar lu - ces sobre el origen de este patogeno, que fue en un prin cipio una bacteria inocua de estuario.
REFERENCIAS
1. Koch r. an address on cholera and its bacillus. Br med J. 1884; 2:403-7. [ Links ]
2. Colwell rr. Global climate and infectious disease: the cholera paradigm. Science. 1996 dec 20;274 (5295): 2025-31. [ Links ]
3. Blake Pa. Historical perspective on pandenmic cholera. in: Wachsmuth iK, Blake Pa, olsnik o, editors. Vibrio cholerae and cholera, molecular to global perspectives. Washington d.C.: aSm Press; 1994. [ Links ]
4. Bennish ml. Cholera: Pathophysiology, clinical features, and treatment. in: Wachsmuth iK, Blake Pa, olsnik o, editors. Vibrio cholerae and cholera: molecular to global perpective. Washington, d.C.: aSm Press; 1994. p. 229-55. [ Links ]
5. Sears Cl, Kaper JB. enteric bacterial toxins: mechanisms of action and linkage to intestinal secretion. microbiol rev. 1996 mar;60 (1):167-215. [ Links ]
6. Wallace CK, anderson Pn, Brown TC, Khanra Sr, lewis GW, Pierce nF, et al. optimal antibiotic therapy in cholera. Bull World Health organ. 1968;39(2):239-45. [ Links ]
7. Grados P, Bravo n, Battilana C. Comparative effectiveness of co-trimoxazole and tetracycline in the treatment of Cholera. Bull Pan am Health organ. 1996 mar;30(1): 36-42. [ Links ]
8. rahal K, Gerbaud Gr, Chabbert Ya. [Properties of a trans ferable resistance factor in Vibrio cholerae biotype eltor]. annales de microbiologie. 1973 oct;124(3):283-94. [ Links ]
9. mhalu FS, mmari PW, ijumba J. rapid emergence of el Tor Vibrio cholerae resistant to antimicrobial agents du - ring first six months of fourth cholera epidemic in Tanzania. lancet. 1979 Feb 17;1(8112):345-7. [ Links ]
10. Towner KJ, Pearson nJ, ofGrady F. resistant Vibrio cholerae el Tor in Tanzania. lancet. 1979 Jul 21;2(8134): 147-8. [ Links ]
11. Public health impact of rwandan refugee crisis: what happened in Goma, zaire, in July, 1994? Goma epide - mio logy Group. lancet. 1995 Feb 11;345(8946):339-44. [ Links ]
12. Dalsgaard a, Forslund a, Petersen a, Brown dJ, dias F, monteiro S, et al. Class 1 integron-borne, multiple-anti - biotic resistance encoded by a 150-kilobase conjugative plasmid in epidemic Vibrio cholerae o1 strains isolated in Guinea-Bissau. J Clin microbiol. 2000 oct;38(10):3774-9. [ Links ]
13. Rossi a, Galas m, Binztein n, rivas m, Caffer mi, Corso a, et al. unusual multiresistant Vibrio cholerae o1 el Tor in argentina. lancet. 1993 nov 6;342(8880):1172-3. [ Links ]
14. Threlfall eJ, Said B, rowe B. emergence of multiple drug resistance in Vibrio cholerae o1 el Tor from ecuador. lancet. 1993 nov 6;342(8880):1173. [ Links ]
15. Popovic T, Bopp C, olsvik o, Wachsmuth K. epide mio - logic application of a standardized ribotype scheme for Vibrio cholerae o1. J Clin microbiol. 1993 Sep; 31(9): 2474-82. [ Links ]
16. Fernandez S, Toro e, Quintero W, Vargas J, Blanco J, Spa dola e. Vibrio cholerae o1 en Venezuela 1997-1998, sensibilidad a los antibioticos. rev inst nac Hig. 2002; 33:25-30. [ Links ]
17. Threlfall eJ, rowe B, Huq i. Plasmid-encoded multiple antibiotic resistance in Vibrio cholerae el Tor from Bangladesh. lancet. 1980 Jun 7;1(8180):1247-8. [ Links ]
18. Falbo V, Carattoli a, Tosini F, Pezzella C, dionisi am, luzzi i. antibiotic resistance conferred by a conjugative plasmid and a class i integron in Vibrio cholerae o1 el Tor strains isolated in albania and italy. antimicrobial agents and chemotherapy. 1999 mar;43(3):693-6. [ Links ]
19. Stokes HW, Hall rm. a novel family of potentially mobile dna elements encoding site-specific gene-integration functions: integrons. mol microbiol. 1989 dec;3 (12): 1669-83. [ Links ]
20. Dalsgaard a, mortensen HF, molbak K, dias F, Serichantalergs o, echeverria P. molecular characterization of Vibrio cholerae o1 strains isolated during cholera outbreaks in Guinea-Bissau. J Clin microbiol. 1996 may; 34(5):1189-92. [ Links ]
21. Dalsgaard a, Forslund a, Serichantalergs o, Sandvang d. distribution and content of class 1 integrons in different Vibrio cholerae o-serotype strains isolated in Thailand. antimicrobial agents and chemotherapy. 2000 may; 44(5): 1315-21. [ Links ]
22. Iwanaga m, Toma C, miyazato T, insisiengmay S, nakasone n, ehara m. antibiotic resistance conferred by a class i integron and SXT constin in Vibrio cholerae o1 strains isolated in laos. antimicrobial agents and chemotherapy. 2004 Jul;48(7):2364-9. [ Links ]
23. Rowe-magnus da, Guerout am, Ploncard P, dychinco B, davies J, mazel d. The evolutionary history of chromosomal superintegrons provides an ancestry for multiresistant integrons. Proc natl acad Sci u S a. 2001 Jan 16;98(2):652-7. [ Links ]
24. Waldor mK, Tschape H, mekalanos JJ. a new type of conjugative transposon encodes resistance to sulfamethoxazole, trimethoprim, and streptomycin in Vibrio cholerae o139. J Bacteriol. 1996 Jul;178(14):4157-65. [ Links ]
25. Hochhut B, Waldor mK. Site-specific integration of the conjugal Vibrio cholerae SXT element into prfC. mol microbiol. 1999 apr;32(1):99-110. [ Links ]
26. Hochhut B, lotfi Y, mazel d, Faruque Sm, Woodgate r, Waldor mK. molecular analysis of antibiotic resistance gene clusters in Vibrio cholerae o139 and o1 SXT cons - tins. antimicrobial agents and chemotherapy. 2001 nov; 45(11):2991-3000. [ Links ]
27. Mukhopadhyay aK, Basu a, Garg P, Bag PK, Ghosh a, Bhattacharya SK, et al. molecular epidemiology of ree - mer gent Vibrio cholerae o139 Bengal in india. J Clin microbiol. 1998 Jul;36(7):2149-52. [ Links ]
28. Amita, Chowdhury Sr, Thungapathra m, ramamurthy T, nair GB, Ghosh a. Class i integrons and SXT elements in el Tor strains isolated before and after 1992 Vibrio cholerae o139 outbreak, Calcutta, india. emerging infectious diseases. 2003 apr;9(4):500-2. [ Links ]
29. Thompson Fl, Hoste B, Vandemeulebroecke K, Swings J. Genomic diversity amongst Vibrio isolates from different sources determined by fluorescent amplified fragment length polymorphism. Syst appl microbiol. 2001;24(4): 520-38. [ Links ]
30. Thompson Fl, iida T, Swings J. Biodiversity of Vibrios. mol Biol rev microbiol. 2004;68(3):403-31. [ Links ]
31. Thompson Fl, Swings J. Taxonomy of the Vibrio. in: Thomp son Fl, austin B, Swings J, editors. The biology of Vibrios. Washington dC: aSm Press; 2006. p. 29-43. [ Links ]
32. Baumann P, Furniss al, lee JV. Vibrio. in: Krieg nr, Holt JG, editors. BergeyLs manual of systematic bacteriology. Bal timore: The Williams & Wilkins Co.; 1984. p. 518-38. [ Links ]
33. Tilson dl. Vibrio. in: murrai Pr Be, Pfaller ma, Tenover FC, Yolken rH, editor. manual of Clinical microbiology 7th ed. Washington, d.C.: american Society for microbiology 1999. p. 497-506. [ Links ]
34. Yamai S, okitsu T, Shimada T, Katsube Y. Distribution of serogroups of Vibrio cholerae non-o1 non-o139 with specific reference to their ability to produce cholera toxin, and addition of novel serogroups. Kansenshogaku zasshi. 1997 Oct;71(10):1037-45. [ Links ]
35. Albert MJ, Siddique AK, Islam MS, Faruque AS, Ansaruzzaman M, Faruque SM, et al. Large outbreak of clinical cholera due to Vibrio cholerae non-O1 in Bangladesh. Lancet. 1993 Mar 13;341(8846):704. [ Links ]
36. Colwell RR, Huq A, Chowdhury MA, Brayton PR, Xu B. Serogroup conversion of Vibrio cholerae. Canadian journal of microbiology. 1995 Oct;41(10):946-50. [ Links ]
37. Blokesch M, Schoolnik GK. Serogroup conversion of Vibrio cholerae in aquatic reservoirs. PLoS pathogens. 2007 Jun;3(6):e81. [ Links ]
38. Gangarosa EJ, Sanati A, Saghari H, Feeley JC. Multiple serotypes of Vibrio cholerae isolated from a case of cholera. Evidence suggesting in-vivo mutation. Lancet. 1967 Mar 25;1(7491):646-8. [ Links ]
39. Manning PA, Stroeher UH, Morona R. Molecular basis for O-antigen biosynthesis in Vibrio cholerae O1: Ogawa- Inaba switching. In: Wasmuth K, Blake PA, Olsvik O, editors. Vibrio cholerae and Cholera: Molecular to global perspective. Washington: Amarican Society for Micro - biology; 1994. p. 77-4. [ Links ]
40. Heidelberg JF, Eisen JA, Nelson WC, Clayton RA, Gwinn ML, Dodson RJ, et al. DNA sequence of both chromosomes of the cholera pathogen Vibrio cholerae. Nature. 2000 Aug 3;406(6795):477-83. [ Links ]
41. Pang B, Yan M, Cui z, Ye X, Diao B, Ren Y, et al. Genetic diversity of toxigenic and nontoxigenic Vibrio cholerae serogroups O1 and O139 revealed by array-based comparative genomic hybridization. J Bacteriol. 2007 Jul;189(13):4837-49. [ Links ]
42. Chen Y, Johnson JA, Pusch GD, Morris JG, Jr., Stine OC. The genome of non-O1 Vibrio cholerae NRT36S demonstrates the presence of pathogenic mechanisms that are distinct from those of O1 Vibrio cholerae. Infect Immun. 2007 May;75(5):2645-7. [ Links ]
43. (CDC). CpeCyPdE, (NCID). CNdEI, (OPS). OPdlS. Metodos de laboratorio para el diagnostico de Vibrio cholerae. Washington, D.C.; 1994. [ Links ]
44. Mendes CL, Abath FG, Leal NC. Development of a multiplex single-tube nested PCR (MSTNPCR) assay for Vibrio cholerae O1 detection. Journal of microbiological methods. 2008 Feb;72(2):191-6. [ Links ]
45. Fykse EM, Skogan G, Davies W, Olsen JS, Blatny JM. Detection of Vibrio cholerae by Real-Time Nucleic Acid Sequence -Based Amplification. Appl Environ Microbiol. 2007 Jan 12. [ Links ]
46. Mantri CK, Mohapatra SS, Ramamurthy T, Ghosh R, Col - well RR, Singh DV. Septaplex PCR assay for rapid identification of Vibrio cholerae including detection of virulence and int SXT genes. FEMS microbiology letters. 2006 Dec; 265(2):208-14. [ Links ]
47. Nhung PH, Ohkusu K, Miyasaka J, Sun XS, Ezaki T. Rapid and specific identification of 5 human pathogenic Vibrio species by multiplex polymerase chain reaction targeted to dnaJ gene. Diagnostic microbiology and infectious disease. 2007 Nov;59(3):271-5. [ Links ]
48. Kaper JB, Morris JG, Jr., Levine MM. Cholera. Clinical microbiology reviews. 1995 Jan;8(1):48-86. [ Links ]
49. Waldor MK, Mekalanos JJ. Lysogenic conversion by a fi - la mentous phage encoding cholera toxin. Science. 1996 Jun 28;272(5270):1910-4. [ Links ]
50. Fasano A, Baudry B, Pumplin DW, Wasserman SS, Tall BD, Ketley JM, et al. Vibrio cholerae produces a second enterotoxin, which affects intestinal tight junctions. Proc Natl Acad Sci U S A. 1991 Jun 15;88(12):5242-6. [ Links ]
51. Trucksis M, Galen JE, Michalski J, Fasano A, Kaper JB. Accessory cholera enterotoxin (Ace), the third toxin of a Vibrio cholerae virulence cassette. Proc Natl Acad Sci U S A. 1993 Jun 1;90(11):5267-71. [ Links ]
52. Waldor MK, Rubin EJ, Pearson GD, Kimsey H, Mekalanos JJ. Regulation, replication, and integration functions of the Vibrio cholerae CTXphi are encoded by region RS2. Mol Microbiol. 1997 Jun;24(5):917-26. [ Links ]
53. Faruque SM, Asadulghani, Alim AR, Albert MJ, Islam KM, Mekalanos JJ. Induction of the lysogenic phage encoding cholera toxin in naturally occurring strains of toxigenic Vibrio cholerae O1 and O139. Infect Immun. 1998 Aug; 66(8):3752-7. [ Links ]
54. Davis B, Waldor MK. CTX contains a hybrid genome de - rived from tandemly integrated elements. Proc Natl Acad Sci U S A. 2002;97:8572-7. [ Links ]
55. Davis BM, Kimsey HH, Kane AV, Waldor MK. A satellite phage-encoded antirepressor induces repressor aggregation and cholera toxin gene transfer. The EMBO journal. 2002 Aug 15;21(16):4240-9. [ Links ]
56. Oropeza-Wekerle RL, Muller E, Kern P, Meyermann R, Goebel W. Synthesis, inactivation, and localization of extracellular and intracellular Escherichia coli hemoly sins. J Bacteriol. 1989 May;171(5):2783-8. [ Links ]
57. Karaolis DK, Johnson JA, Bailey CC, Boedeker EC, Kaper JB, Reeves PR. A Vibrio cholerae pathogenicity island associated with epidemic and pandemic strains. Proc Natl Acad Sci U S A. 1998 Mar 17;95(6):3134-9. [ Links ]
58. Jermyn WS, Boyd EF. Characterization of a novel Vibrio pathogenicity island (VPI-2) encoding neuraminidase (nanH) among toxigenic Vibrio cholerae isolates. Micro bio - logy (Reading, England). 2002 Nov;148(Pt 11):3681-93. [ Links ]
59. Bickle TA, Kruger DH. Biology of DNA restriction. Microbiol Rev. 1993 Jun;57(2):434-50. [ Links ]
60. Vimr ER, Kalivoda KA, Deszo EL, Steenbergen SM. Diversity of microbial sialic acid metabolism. Microbiol Mol Biol Rev. 2004;68:132-53. [ Links ]
61. Galen JE, Ketley JM, Fasano A, Richardson SH, Wasserman SS, Kaper JB. Role of Vibrio cholerae neuraminidase in the function of cholera toxin. Infect Immun. 1992 Feb;60(2):406-15. [ Links ]
62. Dziejman M, Balon E, Boyd D, Fraser CM, Heidelberg JF, Mekalanos JJ. Comparative genomic analysis of Vibrio cholerae: genes that correlate with cholera endemic and pandemic disease. Proc Natl Acad Sci U S A. 2002 Feb 5;99(3):1556-61. [ Links ]
63. Murphy RA, Boyd EF. Three pathogenicity islands of Vibrio cholerae can excise from the chromosome and form circular intermediates. J Bacteriol. 2008 Jan;190(2): 636-47. [ Links ]
64. Mekalanos J, Waldor MK, Gardel CL, Coster TS, Kenner JR, Killeen K. Live cholera vaccines perspectives on their construction and safety. Bull Inst Pasteur. 1995;93:255-62. [ Links ]
65. Attridge SR, Rowley D. The role of the flagellum in the adherence of Vibrio cholerae. J Infect Dis. 1983 May; 147(5):864-72. [ Links ]
66. Silva AJ, Leitch GJ, Camilli A, Benitez JA. Contribution of hemagglutinin/protease and motility to the pathogenesis of El Tor biotype cholera. Infect Immun. 2006 Apr;74(4): 2072-9. [ Links ]
67. Hase CC, Finkelstein RA. Cloning and nucleotide se - quence of the Vibrio cholerae hemagglutinin/protease (HA/protease) gene and construction of an HA/proteasenegative strain. J Bacteriol. 1991 Jun;173(11):3311-7. [ Links ]
68. Finkelstein RA, Boesman-Finkelstein M, Holt P. Vibrio cholerae hemagglutinin/lectin/protease hydrolyzes fibro - nectin and ovomucin: F.M. Burnet revisited. Proc Natl Acad Sci U S A. 1983 Feb;80(4):1092-5. [ Links ]
69. Wu z, Milton D, Nybom P, Sjo A, Magnusson KE. Vibrio cholerae hemagglutinin/protease (HA/protease) causes morphological changes in cultured epithelial cells and perturbs their paracellular barrier function. Microbial pathogenesis. 1996 Aug;21(2):111-23. [ Links ]
70. Silva AJ, Pham K, Benitez JA. Haemagglutinin/protease expression and mucin gel penetration in El Tor biotype Vibrio cholerae. Microbiology (Reading, England). 2003 Jul;149(Pt 7):1883-91. [ Links ]
71. Perez JL, Garcia L, Talavera A, Oliva R, Valmaseda T, Ano G, et al. Passive protection of serum from volunteers ino - culated with attenuated strain 638 of Vibrio cholerae O1 in animal models. Vaccine. 2000 Sep 15;19(2-3):376-84. [ Links ]
72. Lin W, Fullner KJ, Clayton R, Sexton JA, Rogers MB, Calia KE, et al. Identification of a Vibrio cholerae RTX toxin gene cluster that is tightly linked to the cholera toxin prophage. Proc Natl Acad Sci USA. 1999 Feb 2;96(3): 1071-6. [ Links ]
73. Faruque SM, Chowdhury N, Kamruzzaman M, Dziejman M, Rahman MH, Sack DA, et al. Genetic diversity and vi - ru lence potential of environmental Vibrio cholerae popu - lation in a cholera-endemic area. Proc Natl Acad Sci USA. 2004 Feb 17;101(7):2123-8. [ Links ]
74. Chow KH, Ng TK, Yuen KY, Yam WC. Detection of RTX to xin gene in Vibrio cholerae by PCR. J Clin Microbiol. 2001 Jul;39(7):2594-7. [ Links ]
75. Boardman BK, Satchell KJ. Vibrio cholerae strains with mutations in an atypical type I secretion system accumulate RTX toxin intracellularly. J Bacteriol. 2004 Dec; 186 (23):8137-43. [ Links ]
76. Fullner KJ, Mekalanos JJ. In vivo covalent cross-linking of cellular actin by the Vibrio cholerae RTX toxin. The EMBO journal. 2000 Oct 16;19(20):5315-23. [ Links ]
77. Sheahan KL, Satchell KJ. Inactivation of small Rho GTPases by the multifunctional RTX toxin from Vibrio cho lerae. Cellular microbiology. 2007 May;9(5):1324-35. [ Links ]
78. Kudryashov DS, Cordero CL, Reisler E, Satchell KJ. Characterization of the Enzymatic Activity of the Actin Cross-linking Domain from the Vibrio cholerae MARTXVc Toxin. J Biol Chem. 2008 Jan 4;283(1):445-52. [ Links ]
79. Yamamoto K, Al-Omani M, Honda T, Takeda Y, Miwatani T. Non-O1 Vibrio cholerae hemolysin: purification, partial characterization, and immunological relatedness to El Tor hemolysin. Infect Immun. 1984 Jul;45(1):192-6. [ Links ]
80. Goldberg SL, Murphy JR. Molecular cloning of the hemolysin determinant from Vibrio cholerae El Tor. J Bacteriol. 1984 Oct;160(1):239-44. [ Links ]
81. Honda T, Lertpocasombat K, Hata A, Miwatani T, Finkelstein RA. Purification and characterization of a protease produced by Vibrio cholerae non-O1 and comparison with a protease of V. cholerae O1. Infect Immun. 1989 Sep; 57(9):2799-803. [ Links ]
82. Figueroa-Arredondo P, Heuser JE, Akopyants NS, Morisaki JH, Giono-Cerezo S, Enriquez-Rincon F, et al. Cell vacuolation caused by Vibrio cholerae hemolysin. Infect Immun. 2001 Mar;69(3):1613-24. [ Links ]
83. zitzer A, Wassenaar TM, Walev I, Bhakdi S. Potent membrane- permeabilizing and cytocidal action of Vibrio cholerae cytolysin on human intestinal cells. Infect Immun. 1997 Apr;65(4):1293-8. [ Links ]
84. Saka HA, Bidinost C, Sola C, Carranza P, Collino C, Ortiz S, et al. Vibrio cholerae cytolysin is essential for high enterotoxicity and apoptosis induction produced by a cholera toxin gene-negative V. cholerae non-O1, non- O139 strain. Microbial pathogenesis. 2008 Feb;44(2): 118-28. [ Links ]
85. Mukherjee G, Biswas A, Banerjee KK, Biswas T. Vibrio cholerae hemolysin is apoptogenic to peritoneal B-1a cells but its oligomer shepherd the cells for IgA response. Molecular immunology. 2008 Jan;45(1):266-70. [ Links ]
86. Costerton JW, Lewandowski z, Caldwell DE, Korber DR, Lappin-Scott HM. Microbial biofilm. Annu Rev Microbiol. 1995;49:711-45. [ Links ]
87. Jonson G, Lebens M, Holmgren J. Cloning and sequencing of Vibrio cholerae mannose-sensitive haemaggluti nin pilin gene: localization of mshA within a cluster of type 4 pilin genes. Mol Microbiol. 1994 Jul;13(1):109-18. [ Links ]
88. Watnick PI FK, Kolter R, . A role for the mannose-sensitive hemagglutinin in biofilm formation by Vibrio cholerae El Tor. J Bacteriol. 1999;181:3606-9. [ Links ]
89. Jorgensen R, Purdy AE, Fieldhouse RJ, Kimber MS, Bartlett DH, Merrill AR. Cholix toxin, a novel ADP-ribosylating factor from Vibrio cholerae. J Biol Chem. 2008 Apr 18;283(16):10671-8. [ Links ]
90. Fuqua WC, Winans SC, Grenberg EP. Quorum sensing in bacteria: the LuxR-Luxl family of cell density responsi ve trasncrptional regulators. J Bacteriol. 1994;176:269-75. [ Links ]
91. Waters CM, Bassler BL. Quorum sensing: cell-to-cell comunication in bacteria. Annu Rev Cell Dev Biol. 2005;21: 319-46. [ Links ]
92. Miller MB, Skorupski K, Lenz DH, Taylor RK, Bassler BL. Pa rallel quorum sensing systems converge to regulate virulence in Vibrio cholerae. Cell. 2002 Aug 9;110(3):303-14. [ Links ]
93. zhu J, Miller MB, Vance RE, Dziejman M, Bassler BL, Me kalanos JJ. Quorum-sensing regulators control virulence gene expression in Vibrio cholerae. Proc Natl Acad Sci U S A. 2002 Mar 5;99(5):3129-34. [ Links ]
94. Higgins DA, Pomianek ME, Kraml CM, Taylor RK, Semmelhack MF, Bassler BL. The major Vibrio cholerae auto - inducer and its role in virulence factor production. Nature. 2007 Dec 6;450(7171):883-6. [ Links ]
95. Lenz DH, Mok KC, Lilley BN, Kulkarni RV, Wingreen NS, Bassler BL. The small RNA chaperone Hfq and multiple small RNAs control quorum sensing in Vibrio harveyi and Vibrio cholerae. Cell. 2004 Jul 9;118(1):69-82. [ Links ]
96. Hammer BK, Bassler BL. Quorum sensing controls biofilm formation in Vibrio cholerae. Mol Microbiol. 2003 Oct;50(1):101-4. [ Links ]
97. Jobling MG, Holmes RK. Characterization of hapR, a positive regulator of the Vibrio cholerae HA/protease gene hap, and its identification as a functional homologue of the Vibrio harveyi luxR gene. Mol Microbiol. 1997 Dec; 26(5):1023-34. [ Links ]
98. Lenz DH, Miller MB, zhu J, Kulkarni RV, Bassler BL. CsrA and three redundant small RNAs regulate quorum sensing in Vibrio cholerae. Mol Microbiol. 2005 Nov;58(4): 1186-202. [ Links ]
99. Lin W, Kovacikova G, Skorupski K. Requirements for Vibrio cholerae HapR binding and transcriptional repression at the hapR promoter are distinct from those at the aphA promoter. J Bacteriol. 2005 May;187(9):3013-9. [ Links ]
100. Liu z, Hsiao A, Joelsson A, zhu J. The transcriptional regulator VqmA increases expression of the quorum-sensing activator HapR in Vibrio cholerae. J Bacteriol. 2006 Apr; 188(7):2446-53. [ Links ]
101. Joelsson A, Kan B, zhu J. Quorum sensing enhances the stress response in Vibrio cholerae. Appl Environ Microbiol. 2007 Jun;73(11):3742-6. [ Links ]
102. Liang W, Pascual-Montano A, Silva AJ, Benitez JA. The cyclic AMP receptor protein modulates quorum sensing, motility and multiple genes that affect intestinal colonization in Vibrio cholerae. Microbiology (Reading, England). 2007 Sep;153(Pt 9):2964-75. [ Links ]
103. Cholera, 2006. Wkly Epidemiol Rec. 2007 Aug 3;82(31): 273-84. [ Links ]
104. Large epidemic of cholera-like disease in Bangladesh caused by Vibrio cholerae O139 synonym Bengal. Cho lera Working Group, International Centre for Diarrhoeal Di seases Research, Bangladesh. Lancet. 1993 Aug 14; 342(8868):387-90. [ Links ]
105. Faruque SM, Albert MJ, Mekalanos JJ. Epidemiology, genetics, and ecology of toxigenic Vibrio cholerae. Microbiol Mol Biol Rev. 1998 Dec;62(4):1301-14. [ Links ]
106. Bik EM, Bunschoten AE, Gouw RD, Mooi FR. Genesis of the novel epidemic Vibrio cholerae O139 strain: evidence for horizontal transfer of genes involved in polysaccharide synthesis. The EMBO journal. 1995 Jan 16;14(2):209-16. [ Links ]
107. Basu a, mukhopadhyay aK, Garg P, Chakraborty S, ramamurthy T, Yamasaki S, et al. diversity in the arran ge - ment of the CTX prophages in classical strains of Vibrio cholerae o1. FemS microbiology letters. 2000 Jan 1; 182(1):35-40. [ Links ]
108. Glass ri, Black re. The epidemiology of cholera. in: Barua d, Greennough WBi, editors. Cholera. new York: Ple num Press, inc; 1992. p. 129-54. [ Links ]
109. Tauxe rV BP. epidemic cholera in latin america. Jama. 1992;267:1388-90. [ Links ]
110. World Health organization. Cholera in the americas. Weekly epidemiol rec. 1992;67:33-8. [ Links ]
111. Programa de enfermedades Transmisibles. Situacion del Colera en las america in: division de Prevencion y Con trol de enfermedades, editor.: organizacion Panameri cana de la Salud, organizacion mundial de la Salud. Washington, dC, 1999. [ Links ]
112. Programa de enfermedades Transmisibles. el Colera en las americas. in: division de Prevencion y Control de en - fermedades, editor.: organizacion Panamericana de la Salud, organizacion mundial de la Salud, Washington, dC, 1995. [ Links ]
113. ministerio de Sanidad y asistencia Social. Boletin epide - miologico Semanal. in: direccion General Sectorial de epidemiologia, editor.; 1998. 8-14 de marzo. no 10 Vol 49. p. 3794. [ Links ]
114. ministerio de Salud y desarrollo Social. alerTa, reporte epidemiologico semanal para el nivel gerencial. 1998;4 (45). [ Links ]
115. ministerio de Salud y desarrollo Social. alerTa, reporte epidemiologico semanal para el nivel gerencial. 1999;5 (51). [ Links ]
116. Barrett TJ, Blake Pa. epidemiological usefulness of changes in hemolytic activity of Vibrio cholerae biotype el Tor during the seventh pandemic. J Clin microbiol. 1981 Jan;13(1):126-9. [ Links ]
117. momen H, Salles Ca. enzyme markers for Vibrio cholerae: identification of classical, el Tor and environmental strains. Trans r Soc Trop med Hyg. 1985;79(6):773-6. [ Links ]
118. Wachsmuth iK, Kiehlbauch Ja, Bopp Ca, Cameron dn, Strockbine na, Wells JG, et al. The use of plasmid profiles and nucleic acid probes in epidemiologic investi - gations of foodborne, diarrheal diseases. international journal of food microbiology. 1991 Jan;12(1):77-89. [ Links ]
119. Cook Wl, Wachsmuth K, Johnson Sr, Birkness Ka, Samadi ar. Persistence of plasmids, cholera toxin genes, and prophage dna in classical Vibrio cholerae o1. infect immun. 1984 Jul;45(1):222-6. [ Links ]
120. Kaper JB, Bradford HB, roberts nC, Falkow S. molecular epidemiology of Vibrio cholerae in the u.S. Gulf Coast. J Clin microbiol. 1982 Jul;16(1):129-34. [ Links ]
121. Wachsmuth iK, evins Gm, Fields Pi, olsvik o, Popovic T, Bopp Ca, et al. The molecular epidemiology of cholera in latin america. J infect dis. 1993 mar;167(3):621-6. [ Links ]
122. Pace nr, olsen GJ, Woese Cr. ribosomal rna phylogeny and the primary lines of evolutionary descent. Cell. 1986 may 9;45(3):325-6. [ Links ]
123. Giono-Cerezo S, rodriguez angeles mG, Gutierrez-Cogco l, Valdespino-Gomez Jl. [Phenotypic and geno typic characterization of Vibrio cholerae o1]. revista lati noa - mericana de microbiologia. 1994 oct-dec;36(4): 243-51. [ Links ]
124. dalsgaard a, Skov mn, Serichantalergs o, echeverria P, meza r, Taylor dn. molecular evolution of Vibrio chole rae o1 strains isolated in lima, Peru, from 1991 to 1995. J Clin microbiol. 1997 may;35(5):1151-6. [ Links ]
125. Koblavi S, Grimont F, Grimont Pa. Clonal diversity of Vibrio cholerae o1 evidenced by rrna gene restriction patterns. research in microbiology. 1990 Jul-aug;141(6): 645-57. [ Links ]
126. Huguet TJ, arias Bl, PY m. Tipificacion molecular de Vi - brio cholerae o1 en el Peru. rev med med exp. 2000; 17:1-4. [ Links ]
127. Cameron dn, Khambaty Fm, Wachsmuth iK, Tauxe rV, Barrett TJ. molecular characterization of Vibrio cholerae o1 strains by pulsed-field gel electrophoresis. J Clin microbiol. 1994 Jul;32(7):1685-90. [ Links ]
128. Pourshafie m, Grimont F, Kohestani S, Grimont Pa. a molecular and phenotypic study of Vibrio cholerae in iran. Journal of medical microbiology. 2002 may;51(5):392-8. [ Links ]
129. Singh dV. matte mH mG, Jiang S, Sabeena F, Shukla Bn, y col. molecular analysis of Vibrio cholerae o1, o139, non o1 and non-o139 strains: clonal relationships between clinical and enviromental isolates. appl environ microbiol. 2001;67:910-21. [ Links ]
130. arakawa e, murase T, matsushita S, Shimada T, Yamai S, ito T, et al. Pulsed-field gel electrophoresis-based mole - cular comparison of Vibrio cholerae o1 isolates from do - mestic and imported cases of cholera in Japan. J Clin microbiol. 2000 Jan;38(1):424-6. [ Links ]
131. makino S, Kurazono T, okuyama Y, Shimada T, okada Y, Sasakawa C. diversity of dna sequences among Vibrio cholerae o139 Bengal detected by PCr-based dna fingerprinting. FemS microbiology letters. 1995 Feb 1;126 (1):43-8. [ Links ]
132. LEAL TCA, LEAL NC, Almeida AMP. RAPD-PCR typing of Yersinia enterocolitica (Enterobacteriaceae) O:3 se - rotype strains isolated from pigs and humans. Genet Mol Biol. 1999;22(3):315-9. [ Links ]
133. Radu S, Ho YK, Lihan S, Yuherman, Rusul G, Yasin RM, et al. Molecular characterization of Vibrio cholerae O1 and non-O1 from human and environmental sources in Malaysia. Epidemiol Infect. 1999 Oct;123(2):225-32. [ Links ]
134. Leal NC, Sobreira M, Leal-Balbino TC, de Almeida AM, de Silva MJ, Mello DM, et al. Evaluation of a RAPD-based typing scheme in a molecular epidemiology study of Vibrio cholerae O1, Brazil. Journal of applied microbiology. 2004;96(3):447-54. [ Links ]
135. Karaolis DK, Lan R, Kaper JB, Reeves PR. Comparison of Vibrio cholerae pathogenicity islands in sixth and se - venth pandemic strains. Infect Immun. 2001 Mar;69(3): 1947-52. [ Links ]
136. Roszak DB, Grimes DJ, Colwell RR. Viable but nonreco - ve rable stage of Salmonella enteritidis in aquatic systems. Canadian journal of microbiology. 1984 Mar;30(3):334-8. [ Links ]
137. Colwell R, Brayton PR, Grimes DJ, Roszak DB, Huq SA, Palmer LM. Viable, but nonculturable Vibrio cholerae and realted payhogens in the enviroment: implication for release of genetic engineered microorganisms. Bio Technology. 1985;3:2025-31. [ Links ]
138. Huq A, Colwell RR, Rahman R, Ali A, Chowdhury MA, Parveen S, et al. Detection of Vibrio cholerae O1 in the aquatic environment by fluorescent-monoclonal antibody and culture methods. Appl Environ Microbiol. 1990 Aug;56(8):2370-3. [ Links ]
139. Colwell R, Huq A. Vibrios in the environment: viable but non culturable Vibrio cholerae. In: Wachsmuth IK, Blake PA, Olsvik O, editors. Vibrio cholerae and cholera: molecu lar to global perspective. Washington: Amarican Society for Microbiology; 1994. p. 117-33. [ Links ]
140. Mosley WH, Aziz KM, Mizanur AS, Alauddin AK, Ahmed A, Fhimuddin M. Report of the 1966-67 cholera vaccine trial in rural East Pakistan. Bull World Health Organ. 1972;47:229-38. [ Links ]
141. Garcia L, Jidy MD, Garcia H, Rodriguez BL, Fernandez R, Ano G, et al. The vaccine candidate Vibrio cholerae 638 is protective against cholera in healthy volunteers. Infect Immun. 2005 May;73(5):3018-24. [ Links ]
142. Snow SJ. Commentary: Sutherland, Snow and water: the transmission of cholera in the nineteenth century. Inter - national journal of epidemiology. 2002 Oct;31(5):908-11. [ Links ]
143. Satcher D. Emerging infections: getting ahead of the cure. Emerg Infect Dis. 1995;1:1-6. [ Links ]
144. Levine MM. South America: the return of cholera. Lancet. 1991;338:45-6. [ Links ]












 uBio
uBio