Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Tropical
versión impresa ISSN 0002-192X
Agronomía Trop. v.53 n.2 Maracay mayo 2003
DIVERSIDAD GENÉTICA DE UNA COLECCIÓN DE YUCA A TRAVÉS DE MARCADORES MOLECULARES RAPDS
Asia Y. Zambrano*, J. R. Demey**, Francia Fuenmayor*, Víctor Segovia* y Zulay Gutiérrez*
*Investigadores. INIA. Centro Nacional de Investigaciones Agropecuarias.
Av. Universidad, vía El Limón. Apdo. 4521.
Maracay, 2101, estado Aragua. Venezuela.
** Consultor. TI-GC- Apdo. 4521.
Maracay, 2101.Venezuela.
Resumen
Con el objetivo de conocer la diversidad genética del Banco de Germoplasma de Yuca, Manihot esculenta Crantz, del Centro Nacional de Investigaciones Agropecuarias, INIA- Venezuela, se estudiaron los productos de amplificación de 65 entradas utilizando los iniciadores aleatorios OPA-04, OPA-07, OPB-07, OPK-03, OPK-05, OPK-15, OPM-04, OPM- 14, OPM- 18 y OPM-20. Para identificar el agrupamiento natural de los clones, las matrices de similitud o disimilitud obtenidas utilizando los coeficientes de Jaccard, Emparejamiento Simple, Dice y Rogers y Tanimoto fueron representadas gráficamente utilizando dos métodos de agrupamiento, el generado por árboles jerárquicos, bajo tres métodos de encadenamiento: simple, completo y promedio, y el generado por árboles no-jerárquicos utilizando como método de agregación el Neighbor-Joining ponderado. Los resultados obtenidos permiten seleccionar al OPB-07 como el iniciador que ofrece mayor nivel de resolución en la caracterización de los clones de la colección, la estructura generada con este iniciador garantiza la menor distorsión entre las disimilaridades iniciales y las que resultan del árbol no-jerárquico, cuantificada a través del coeficiente de correlación cofenética y el índice de diversidad de Shannon.
Palabras Clave: Manihot esculenta Crantz; RAPD; diversidad genética.
Summary
In order to study the genetic diversity of cassava collections of the CENIAP-INIA (Maracay, Venezuela) PCR products from 65 entrances were studied using the RAPD primers: OPA-04, OPA-07, OPB-07, OPK-03, OPK-05, OPK- 1 5, OPM-04, OPM- 14, OPM- 1 8 y OPM-20. With the patterns generated by primers, using similarities or dissimilarities between entrances, calculated according to Jaccard, Simple Matching, Dice and Rogers and Tanimoto coefficients, hierarchical and non- hierarchical trees aggregated according to Neighbor-Joining procedure were represented graphically in order to identify the best natural group. Results showed that best primer is OPB-07 in order to study genetic diversity with a high level of resolution. The structure generated with this primer reduces distortion between initial dissimilarities and those that are due to non-hierarchical tree measurements through the cophenetic correlation and Shannon's diversity index .
Key Words: Manihot esculenta; cassava; RAPD; genetic diversity.
Recibido: marzo 11, 2002.
Introducción
El poder de la biología molecular para examinar con alta resolución la variación del ADN a nivel de nucleótidos ha facilitado la aplicación de estas técnicas para el análisis de diferentes tipos de preguntas de la filogenia y la genética de una población.
El estudio de la diversidad genética entre el germoplasma de una especie tiene importantes aplicaciones para los mejoradores de plantas, por ejemplo, la información del grado de relación genética entre líneas o poblaciones es de gran utilidad en la torna de decisiones sobre qué individuos usar para hacer nuevas combinaciones genéticas, contribuyendo por ende a maximizar la respuesta heterótica. La determinación de las relaciones genéticas ayuda a identificar líneas o poblaciones que deben ser mantenidas para preservar el máximo de la diversidad genética en bancos de germoplasma (Beeching et al., 1994 y Chavarriaga-Aguirre et al., 1999).
Dentro de las técnicas de biología molecular más usadas para caracterizar y evaluar la diversidad genética existente en los bancos de germoplasma se encuentra la amplificación aleatoria de ADN polimórfico (RAPD). Esta metodología consiste en utilizar oligonucleótidos (iniciadores) que hibridan con secuencias homólogas en numerosos puntos del genoma, de tal forma que cuando dos de estos puntos se encuentran en cadenas opuestas del ADN, a una separación no mayor de unos cientos pares de bases el segmento comprendido entre ellos, se puede amplificar múltiples veces a través de la reacción en cadena de la polimerasa (PCR) y ser visualizado en un gel de electroforesis. Así mismo, cualquier alteración en los nucleátidos del ADN molde se evidenciará por la variación en el tamaño del locus amplificado o por la ausencia del mismo (Winter y Kahl, 1995).
Los RAPDS tienen la gran ventaja de ser utilizados sin previo conocimiento del genoma en estudio, además de su sencillez, rapidez, bajo costo, ausencia de marcaje y menor requerimiento de la cantidad de ADN (Ford y Painting, 1996).
En los RAPDS las bandas detectadas se comportan como si se tratase de la expresión fenotípica de un par de alelos, donde uno es nulo y por tanto recesivo frente al otro alelo, de tal forma que el homocigoto dominante y el heterocigoto presentarán productos de amplificación o banda, mientras que el homocigoto recesivo no. Dada las características de estos marcadores el tipo de codificación empleada para el análisis es la codificación sobre locus, donde se emplea una columna por cada locus para registrar la presencia o ausencia de productos de amplificación lo cual equivale a indicar si en un locus hay o no al menos un alelo dominante, sin especificar el número de ellos. Es esta la forma en que se analizan los datos para evaluar el grado de similitud entre genotipos en la mayoría de los trabajos publicados referentes a RAPDS, en los cuales se recurre generalmente a los coeficientes de similaridad de Jaceard, de Emparejamiento Simple o Simple Matching y Dice, entre otros (Nei, 1987; Nei y Kumar, 2000; Hall, 2001).
Entre las desventajas más importante que presentan estos marcadores dominantes con respecto a los marcadores codominantes está el bajo poder discriminante de los individuos, dada la imposibilidad o poca fiabilidad en la diferenciación del genotipo homocigoto dominante del heterocigoto cuando hay presencia de productos de amplificación (Winter y Kahi, 1995). Por esta razón, en el análisis de los datos adquiere vital importancia encontrar la mejor relación entre las medidas de similitud o disimilitud entre individuos y la técnica de agrupamiento que minimice esta pérdida de información.
En el caso de la yuca la utilización de RAPDS para caracterizar bancos de germoplasma ha sido señalada entre otros por Beeching et al (1994), Lowe et al. (1996) y Chavarriaga-Aguirre et al. (1999).
El objetivo de este trabajo fue estudiar la diversidad genética del Banco de Germoplasma de Yuca del CENIAP-INIA, Venezuela, a través de marcadores moleculares RAPDS y determinar el iniciador o grupo de estos que ofrezcan mayor nivel de resolución en la caracterización de los clones de la colección, para facilitar el procesamiento de nuevas entradas.
Materiales y métodos
Material Vegetal Se utilizó como material vegetal tejido foliar joven de plántulas de las entradas: Amacuro (AMA-130, AMA-134, AMA-135, AMA-144, AMA- 145, AMA- 166 y AMA- 168); Amazonas (AMAZ-1 88); Barinas (BAR-1); Bolívar (BOL-43, BOL-50, BOL-58, BOL-59, BOL-60, BOL-89 y BOL-98), Burrera (BUR); Brasileña (BRA-12); Chapapotera (CHMA); Cogollo Verde (COGO); Juliana (JUL); Juliana Catira (JULCAT); Lengua e pájaro (L-PAJA); Muestra (M-278, M-279, M-284, M-285, M-291, M-292, M-306, M-307, M-327, M-338, M-365, M-366, M-388, M-392, M-393, M-394, M-395, M-402, M-422, M-433, M-434, M-440, M-478 y M-479); Meven (MEV-06 1, MEV-095, MEV- 150 y MEV- 1 80); México (MEX-59); Morichalera (MOR¡); Pastorita (PASTO); Plan de hierro (P-HIERRO); Proletaria (PROLE); Querapa Blanca (QBLANCO); Remigio (REMIGIO); Rivero (RIVERO); Tempranita (TEMPRA) y Universidad Central de Venezuela (UCV-2062, UCV-2076, UCV-2105, UCV-2129 y UCV-2184) correspondientes al 40% del Banco de Germoplasma de Yuca del CENIAP-INIA, Venezuela.
Extracción y Amplificación de ADN
Se realizó la extracción de ADN de las 65 entradas según metodología señalada por Zambrano et al (2002). Una vez extraído y cuantificado el ADN se procedió a su amplificación con los iniciadores aleatorios: OPA-04, OPA-07, OPB-07, OPK-03, OPK-05, OPK-15, OPM-04, OPM-14, OPM- 18 y OPM-20, según procedimiento descrito por Beeching et al. (1994), utilizando 25 m de mezcla de reacción constituida por 2,5 m l de Buffer B 1OX, 3 mM de MgCl2, 0,2 mM de cada uno de los dNTPs, 1 µ M de iniciador, 1 U de Taq y 25 ng de ADN.
La amplificación fue realizada en un termociclador MJ Research 200 siguiendo la metodología descrita en Zambrano et al. (2002). La separación de los productos de PCR se hizo en geles de agarosa 1,2% corridos por 2 h a 80 voltios y visualizados con bromuro de etídio bajo luz UV. Para el análisis de los resultados se procedió a la cuantificación de los productos de PCR detectados para cada caso, por medio de la asociación de la movilidad relativa con su peso molecular en pares de bases (pb) por comparación con el marcador de peso (ADN del fago l doblemente digerido con Hind III y Eco RI).
Para el análisis digital de la imagen se utilizó la cámara Kodak DC-40, el filtro para bromuro de etídio (590 Df, 100 nm), un cono N° 4 (Edas Hood N° 4) y cuatro dioptrías (+4). La sensibilidad de banda utilizada fue equivalente al 75%. Para el análisis digital de la imagen se usó el ID Image Analysis Software (Eastman Kodak Company, 1997), el cual produjo los patrones para cada clon por iniciador en términos de peso molecular en pares de base (pb).
Análisis de la diversidad
Con los patrones generados para cada iniciador se calcularon las similitudes y/o disimilitudes entre individuos utilizando los coeficientes de Jaccard (Jaccard, 1908), Emparejamiento Simple o Simple Matching (Sokal y Sneath, 1963), Dice (Dice, 1945) y Rogers y Tanimoto (Rogers y Tanimoto, 1960).
Para identificar el agrupamiento natural de los clones, las matrices de similitud y/o disimilitud obtenidas para cada coeficiente por iniciador fueron representadas gráficamente utilizando dos métodos de agrupamiento, el generado por árboles jerárquicos, bajo tres métodos de encadenamiento: simple, completo y promedio o UPGMA, según Sneath y Sokal (1973) y el generado por árboles no-jerárquicos utilizando como método de agregación el Neighbor-Joining (Saitou y Nei, 1987).
Se destaca la importancia de encontrar la mejor relación entre las medidas de similitud o disimilitud y la técnica de agrupamiento a fin de minimizar la pérdida de información. Si se supone que las matrices generadas por los coeficientes para cada iniciador son en sí mismas ultramétricas, entonces la representación mediante árboles jerárquicos es exacta, pero si no lo son como ocurre en la generalidad de los casos prácticos, se estará introduciendo un error por la adecuación de una distancia no ultramétrica a un árbol ultramétrico, independientemente que el primer paso en la construcción de los árboles jerárquicos sea transformar "razonablemente" la disimilaridad original en ultramétrica a través de los métodos de encadenamiento. La desigualdad ultramétrica es una condición muy difícil de satisfacer y ninguna de las medidas de asociación presentadas la cumplen por definición, a no ser para conjuntos de datos particulares (Swofford y Olsen, 1990; Hall, 2001).
Así mismo, está comprobado que no todas las regiones del genoma evolucionan concertadamente, ya que unas están sometidas a fuertes presiones selectivas y otras a una variación prácticamente neutral (Avise, 1994; Graur y Wen-Hsiung, 2000), por lo que es inapropiado suponer únicamente un modelo de ultrametricidad; por esta razón es recomendable probar modelos aditivos que se ajusten perfectamente a árboles no-jerárquicos. Aunque la aditividad de las distancias entre individuos no es un supuesto que se cumple a priori esta condición es menos restrictiva que la de ultrametricidad. Al igual que en los métodos jerárquicos ultramétricos el primer paso de un algoritmo tendente a obtener un árbol aditivo es hacer que las distancias experimentales entre individuos sean convertidas a aditivas (Saitou y Nei,1987).
Estas ambigüedades determinan que el agrupamiento logrado que permite identificar clases existentes en relación a los individuos, dependerá no sólo de la medida de similitud o disimilitud seleccionada, del número de grupos que deben ser formados (cuando esta información existe) así como también del método de agrupación y del algoritmo de agregación elegido.
El coeficiente de correlación cofenética (Rohif y Sokal, 1981) se utilizó para seleccionar uno de varios agrupamientos alternativos, generados por iniciador. Este coeficiente indica la correlación de las distancias definidas por la métrica del árbol binario con las distancias originales entre los clones, luego se espera que el agrupamiento con mayor coeficiente sea el que mejor describa el agrupamiento natural de la colección o refleje la coherencia entre la matriz de información ingresada y el resultado de los agrupamientos logrados por alguno de los criterios aplicados.
Para una misma matriz de similitud y/o disimilitud el coeficiente de correlación cofenética variará según el método de agrupamiento empleado. Cuando el coeficiente de correlación cofenética es mayor a 0,9 este puede ser interpretado como el de un ajuste muy bueno, es decir, existe una clara estructura jerárquica entre los objetos. Valores entre 0,8 y 0,9 son considerados buenos, valores entre 0,7 y 0,8 y valores menores a 0,7 son pobres o muy pobres, respectivamente, e indican una distorsión notable entre las similaridades y/o disimilaridades iniciales y las que resultan de la representación gráfica.
El índice de Shannon (Magurran, 1988) e intervalo de confianza Bootstrap ajustado para n=1000 (Pla y Matteucci, 2000), fue utilizado para estimar la diversidad de los agrupamientos seleccionados a través del coeficiente de correlación cofenética.
Todos los análisis se realizaron utilizando el NTSYSpe ver. 2.10t (Exeter Software, 2000), InfoStat ver. 1.1 (Infostat, 2002) y Resampling Stats ver. 5,02 (Resampling Stats Inc, 1999).
Resultados y discusión
Los iniciadores de RAPDS utilizados para la amplificación produjeron un total de 92 fragmentos de amplificación reproducibles, siendo el 98% polimórficos con un tamaño entre 220-1700 bp (ver Cuadro). El menor número de fragmentos polimórficos fue generado con el iniciador OPA-04 con un total de 5, en tanto que el mayor número fue amplificado con el iniciador OPM-18 con un total de 12.
La Figura 1 muestra la distribución de los coeficientes de correlación cofenética (r) observados en las medidas de similitud y/o disimilitud para los diferentes métodos de agregación estudiados; estos resultados muestran que los dos métodos de agrupamiento, el generado por árboles jerárquicos, bajo el encadenamiento promedio (UPGMA) y el generado por árbol no-jerárquico utilizando como método de agregación Neighbor- Joining, producen estructuras de buen a muy buen ajuste, respectivamente, observándose una clara superioridad en el ajuste de las estructuras debidas al árbol aditivo generado a través del método de agregación Neighbor-Joining.
CUADRO. Iniciadores OPERON de RAPDS usados para la amplificación
de la colección de Manihot esculenta.
Iniciador Secuencia Total de Porcentajes de Rango de
Fragmentos Locus Polimórficos Ampliación
Amplificados (bp)
OPA-04 AATCGGGCTG 5 100 500-1050
OPA-07 GAAACGGGTG 11 100 480-1700
OPA-07 GGTGACGCAG 8 100 220-1350
OPB-03 CCAGCTTAGG 8 100 375-1450
OPK-05 TCTGCGAGG 8 100 350-12
OPK-15 CCCGCTACAC 11 82 255-1500
OPM-04 GGCGGTTGCT 10 100 350-1420
OPM-14 AGGGTCGTTC 10 100 308-1350
OPM-18 CACCATCCGT 12 100 374-1584
OPM-20 AGGTCTTGGG 9 100 350-1300
De los 10 iniciadores empleados, sólo OPB-07, OPM-14 y OPM-18, fueron los que produjeron las estructuras que mejor describen la diversidad natural de los clones, medida a través del coeficiente de correlación cofenética (r).
Los agrupamientos que reflejan el mejor ajuste o mejor coherencia entre la matriz de información ingresada y el resultado de los agrupamientos logrados, es decir, que permiten seleccionar el iniciador que mejor detecta las alteraciones moleculares que producen amplificaciones diferenciales en locus, fueron los siguientes: los generados con los coeficientes de similitud de Jaccard (r=0,91046) y Dice (r=0,92219) para OPM-14 y el coefleciente de Dice (r=0,92275) para OPM-18 bajo el encadenamiento medio como método de agregación y los generados con la disimilitud debidas al coeficiente de Jaecard (r=0,90854) para OPB-07 y las disimilitudes debidas a los coeficientes de Jaecard (r=0,93161) y Dice (r=0,94982) para OPM-14 y Jaccard (r=0,92764) y Dice (r=0,94990) para OPM-18, todos bajo el método de agregación Neighbor-Joining.
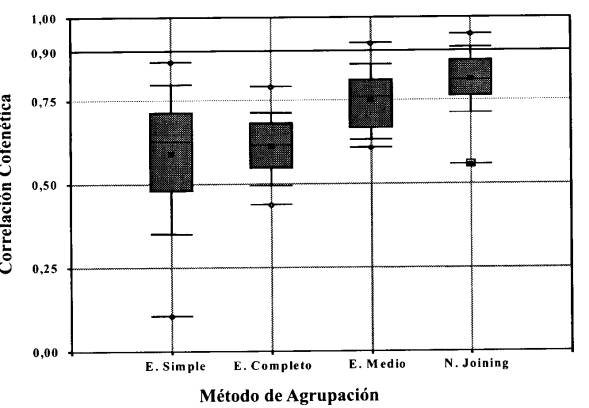
FIGURA 1. Distribución de los coeficientes de correlación cofenética observados para las diferentes medidas de similitud y/o disimilitud por cada método de agregación.
Winter y Kahi (1995) recomiendan que para garantizar una mejor resolución en las clasificaciones se debe considerar la mayor cantidad de locus; escoger un bajo número de iniciadores representa un riesgo en la explicación de la variabilidad. Los resultados obtenidos del estudio de la agrupación, medida a través del coeficiente de correlación cofenética para toda la región del genoma estudiada (92 locus y 220-1700 bp), muestran que la disimilitud debida al coeficiente de Jaccard:(r=0,85816) bajo el método de agregación Neighbor-Joining, es la estructura que mejor ajusta, sin embargo, el coeficiente obtenido es inferior al menor de los seleccionados para las estructuras generadas por los iniciadores OPB-07, OPM- 14 y OPM- 18, individualmente.
El estudio de los árboles generados muestra que el método de construcción del árbol es el que define el número de grupos que son formados, independientemente de los coeficientes de similitud y/o disimilitud estudiados; para las agrupaciones construidas bajo el método jerárquico se forman cuatro grupos y para las derivadas del método no-jerárquico se generan clasificaciones de cinco y seis grupos. La distribución de los clones dentro de los grupos varia según el coeficiente de similitud y/o disimilitud utilizado.
Los resultados obtenidos y debidos al uso del coeficiente de correlación cofenética no representan una prueba de significancia, sólo son un método que provee una regla aceptable de decisión, por esta razón se seleccionaron los agrupamientos generados mayores de r=0,90, cualquier valor en el intervalo [0,9<r<1,01] estará proporcionando información similar. Rohlf y Fisher (1968) indican que esta regla subjetiva para medir el grado de ajuste no puede ser usada para probar la significación del coeficiente de correlación cofenética, también muestran que la mayoría de las correlaciones cofenéticas encontradas son estadísticamente significativas. Lapointe y Legendre (1992) proveen las tablas de los valores críticos para las correlaciones cofenéticas en diferentes casos y concluyen que para más de 12 OTUs (Unidades Taxonómicas Operacionales) un coeficiente de correlación mayor de r=0,5 siempre será estadísticarnente significativo al 1%.
Dada la subjetividad que proporciona el coeficiente de correlación cofenética y al no disponer de estudios previos de segregación en progenies, la variación de presencia/ausencia observada para cada banda se interpreta como variación en locus individuales, entonces, la mejor interpretación genética de la diversidad estará asociada a la clasificación que produzca la mayor cantidad de grupos, es decir, el iniciador que detecte la mayor cantidad de diferencias entre clones. Esta interpretación se sustenta en el estudio de los factores que han intervenido en la construcción de la estructura genética de la yuca, donde se destacan una recurrente recombinación genética en el proceso de domesticación, alta hibridación natural con especies silvestres de Manihot y alta heterocigosidad (Second et al., 1997). Este criterio permite descartar las agrupaciones generadas por el método de agrupación jerárquica y no-jerárquica que sólo logran discriminar cuatro y cinco grupos, respectivamente.
La Figura 2 muestra el valor (H), el intervalo de confianza Bootstrap ajustado y la distribución del índice de Shannon ajustado para los agrupamientos generados por la disimilitud debida al coeficiente de Jaccard (H= 1,7964) y Dice (H = 1,7847) para los iniciadores OPB-07 y OPM-14, respectivamente.
El índice de diversidad de Shannon para seis grupos tiene un rango de variación esperado H: 0-1,7918, este intervalo teórico sin ajustar (calculado como LN (6), referido a la Figura 2, indica que a medida que la distribución se concentre más cercana al valor máximo hay menor probabilidad de seleccionar un clon y conocer el grupo a que pertenece, es decir, la distribución de los clones bajo el agrupamiento con mayor índice de Shannon será la más diversa.
Dada la cercanía de los índices de diversidad obtenidos para los dos iniciadores y al no disponer de una prueba de hipótesis que permita contrastar y garantizar que existen diferencias estadísticamente significativas entre los dos valores, se estudió las agrupaciones que producen en conjunto los iniciadores OPB-07 y OPM-14. Los coeficientes de correlación cofenética obtenidos para las disimilitudes debidas a los coeficientes de Jaccard (r=0,9134) y Dice (r=0,9147), todos bajo el método de agregación Neighbor-Joining, permiten descriminar un máximo de cinco grupos de clones para el conjunto de los 18 locus.
Estos resultados justifican la selección del iniciador OPB-07 bajo la estructura de agrupación generada por la disimilitud debida al coeficiente de Jaccard y el Neighbor-Joining como método de agregación, mejorando así la detección de las alteraciones moleculares y garantizando que la estructura jerárquica encontrada entre los clones permita una mejor comprensión de la diversidad genética del Banco de Germoplasma de Yuca del CENIAP-Maracay, con alto nivel de resolución, reducción de costos operativos y disminución del riesgo de manipulación de agentes tóxicos. Adicionalmente, la estructura generada con este iniciador garantiza la menor distorsión entre las disimilaridades iniciales y las que resultan del árbol no-jerárquico cuantificada a través del coeficiente de correlación cofenética y el uso del índice de diversidad de Shannon. El iniciador OPB-07 produjo un total de 8 fragmentos amplificados y 100"/o de locus polimórficos en un rango de amplificación de 220 a 1 350 bp.
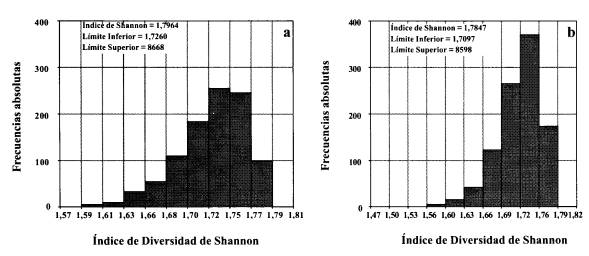
FIGURA 2. Distribución del índice de diversidad de Shannon para los agrupamientos generados por: (a) coeficiente de Jaccard para OPB-07; (b) coeficiente de Dice para OPM-14, todos bajo el método de agregación Neighbor-Joining.
La Figura 3 muestra la agrupación generada para el iniciador OPB-07 bajo la estructura seleccionada. El estudio de las relaciones genéticas de los clones de la colección permitió la formación de seis grupos. El le- de los grupos lo forman los clones cuya disimilitud es mayor a 0,34 y agrupa a: M-278, M-291, M-338, M-365, M-434, M-478, MEV-150, MEX-59, PROLE y TEMPRA. El 2do grupo lo conforman los clones: BOL-98, BRA- 12, M-284, M-327, M-392, M-402, PASTO, P-HIERRO, QBLANCO, RIVERO y UCV-2129 con disimilitudes entre 0,25 y 0,34. El 3el grupo formado por: AMA- 145, BOL-50, BOL-89, BUR, M-366 y M-388 con disimilitudes entre 0,18 y 0,25. El 41 grupo formado por: AMA-130,AMA-134,AMA-166,AMA-168,JULCAT, L-PAJA, M-394, M-395, M-479, MEV-180, MORI, UCV-2062 y UCV-2105 con disimilitudes entre 0,15 y 0,18. El 51 grupo formado por: AMA-135, AMAZ-188, BOL-58, JUL, M-285, M-292, M-306, M-422, M-440, MEV-061, MEV-095, PEMIGIO y UCV-2184 con disimilitudes entre 0,05 y 0,15, y el 61,1 grupo fon-nado por: AMA-144, BAR-1, BOL-43, BOL-59, BOL-60, CHAPA, COGO, M-279, M-307, M-393, M-433 y UCV-2076 con una disimilitud menor a 0,05.
La distribución de los individuos dentro de los grupos muestra clones con diferente denominación que conforman grupos similares, resultados que reflejan que estos individuos provienen de un ancestro común y que la denominación con la que entraron en el banco está asociada al colector de la muestra y a la zona de colecta y no a verdaderas diferencias genéticas. La variabilidad observada en la clasificación puede asociarse a la condición ex situ del Banco de Germoplasma de Yuca del CENIAP-INIA. La yuca presenta monoecia, es decir, las flores femeninas abren normalmente unos 10 a 14 días antes que las masculinas sobre una misma rama, pudiendo ocurrir la autofecundación en flores de diferentes ramas y en diferentes plantas del mismo genotipo (Jennings e Iglesias, 2002). Los bancos de germoplasma de yuca mantenidos ex situ poseen la ventaja de mantener la heterocigosis dentro de los genotipos ya que se ve favorecida la polinización cruzada, reduciendo la erosión genética (NG y NQ 2002).
La información de diversidad genética obtenida a través de los productos de amplificación RAPD permitirá la selección y mantenimiento dentro del banco de diferentes genotipos que exhiban características deseables en cuanto a resistencia a plagas y enfermedades, plasticidad a diferentes ambientes además de alto rendimiento, permitiendo así mismo la eliminación de los posibles duplicados (NG y NQ 2002, Fregene y PuontiKaerias, 2002).
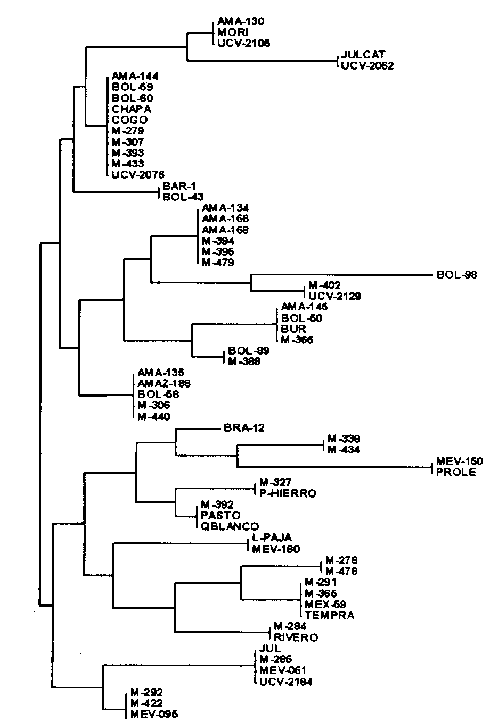
FIGURA3. Árbol generado con la disimilitud producto del coeficiente de Jaccard
y el Nighbor-Joining, como método de agregación para iniciador OPB-07.
Estos resultados rescatan las fuentes diversas que deben tomarse en cuenta en la interpretación de la diversidad genética, donde no sólo habrá que considerar los métodos estadísticos utilizados y el tipo de carácter o marcador, sino también el cómo se ha interpretado en términos genéticos la variación observada.
Bibliografía
1.AVISE, J. C. 1994. molecular markers natural history and evolution. KluwerAcademic Publishers. USA. 511 p. [ Links ]
2.BEECHINQ J. R., P. MARMEY, M. A. HUGHES and A. CHARRIER. 1994. Evaluation of molecular approaches for determining genetic diversity in cassava germplasm. In: Proceedings of the second international Scientific Meeting. The Cassava Biotechnology Network. Bogor. Indonesia. sp. [ Links ]
3.CHAVARRIAGA-AGUIRRE, P., M. MAYA, J. TOHME, M. DUQUE, C. IGLESIAS, M. BONIERBALE, S. KRESOVICH and G KOCHERT. 1999. Using microsatellites, isozymes and AFLPs to evaluate genetic diversity and redundancy in cassava core collection and to assess the usefulness of DNA-based markers to maintain germplasm collections. Molecular Breeding. 5:263-273. [ Links ]
4.DICE, L. R. 1945. Measures of the amount of ecologic assocaition between species. Ecology, 26:297-302. [ Links ]
5.EASTMAN KODAK COMPANY. 1977. ID Image analysis software. 230 p. EXETER SOFTWARE. 2000. NTSYSpc ver 2. 1 Ot. Applied biostatistics Inc. [ Links ]
6.FORD, B. and K. PAINTINIG. 1996. Measuring genetic variation using molecular markers. International Genetic Resources Institute. On Line http://www.cgian.org/ipgri. 38 p. [ Links ]
7.FREGENE, M. and J. PUONTI-KAERLAS. 2002. Cassava Biotechnology. In: Cassava: biology, production and utilization. Eds R. J. Hillocks, J. M. Thresh, A. C. Bellotti. CABI Publishing. UK. pp. 179-207. [ Links ]
8.GRAUR, D. and WEN-USIUNG. 2000. Fundamentals of Molecular Evolution. Sinauer Associates Inc. USA. 481 p. [ Links ]
9.HALL, B. G. 200 1. Phylogenetics trees made easy: A how-to manual for molecular biologists. Sinauer Assoc. USA. 179 p. [ Links ]
10.INFOSTAT 2002. Infostat software estadístico. Versión 1. 1. Córdoba, Argentina. [ Links ]
11.JACCARD, P. 1908. Nouvelles recherches sur la distribution florale. Bull. Soc. Vaud. Sci. Nat. 44: 223-270. [ Links ]
12.JENNINGS, D. L. and C. IGLESIAS. 2002. Breeding for crop improvement. In: Cassava: Biology, Production and Utilization. Eds R.J. Hillocks, J. M. Thresh, A. C. Bellotti. CABI Publishing. UK. pp. 149-166. [ Links ]
13.LAPOINTE, F. J. and P. LEGENDRE. 1992. Statistical significance of the matrix correlation coefficient for comparing independent phylogenetic trees. Systematic Biology. 41:378-384. [ Links ]
14.LOWE, A. J., 0, HANOTTE and L. GARINO. 1996. Standardization of molecular genetic techniques for the characterization of germplasm collection: the case of random amplifíed polymorphic DNA (RAPD). Plant Genetic Resources Newsletter. 107:50-54. [ Links ]
15.MAGURRAN, A. E. 1988. Ecological diversity and its measurement. Princeton Univ. Press. Prinecton. NJ. 179 pp. [ Links ]
16.NEI, M. 1987. Molecular evolutionary genetics. Columbia University Press. USA. 512 p. [ Links ]
17.NEI, M. and S. KUMAR. 2000. Molecular evolution and phylogenetics. Oxford University Press. USA. 333 p. [ Links ]
18.NG, NQ, SYC NG. 2002. Genetic resources and conservation. In: Cassava: Biology, production and utilization. Eds RJ Hillocks, JM Thresh, AC Bellotti. CABI Publishing. UK. pp. 167-177. [ Links ]
19.PLA, L. and S. D. MATTEUCCI. 2000. Bootstrap confidence intervals for Shannon biodiversity index: A simulation study. In: Proccedings of the International Biometric Conference. Berkely, LA. 117-118. [ Links ]
20.RESAMPLING STATS. 1999. Resampling Stats ver. 5,02. Resampling Stats Inc. Arlington, VA. [ Links ]
21.ROGERS, D. G and T. T. TANIMOTO. 1960. A computer program for classifying plants. Science, 132: 1115-1118. [ Links ]
22.ROHLF, F. J. and. D. L. FISHER. 1968. Test for hierarchical structure in random data sets. Systematic Zool. 17:407-412. [ Links ]
23.ROHLF, F. J. and R. R. SOKAL. 198 1. Comparing numerical taxonomic studies. Systematic Zool. 30:459-490. [ Links ]
24.SAITOU, N. and M. NEI. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4:406-425. [ Links ]
25.SECOND, Cx, A. C. ALLEN, R. A. MENDES, L. J. C. B. CARVALHO, L. EMPERAIRE, C. INGRAM and C. COLOMBO. 1997. Molecular marker (AFLP)-based manihot and cassava numerical taxonomy and genetic structure analysis in progress: Implications for their dynamic conservation and genetic mapping. African Journal of Root and Tuber Crops. 2(1-2):140-147. [ Links ]
26.SNEATH, P. H. A. and R. R. SOKAL, 1973. Numerical taxonomy. freeman. San Francisco. 573 pp. [ Links ]
27.SOK.AL, R. R. and P. H. A. SNEATH. 1963. Principles of numérical taxonomy. Freeman. San Francisco. 359 pp. [ Links ]
28.SWOFFORD, D. L. and Cx J. OLSEN. 1990. Phylogenetic reconstruction. In: Molecular systematics. Editores: Hillis, D. M. y C. Moritz C.: Sinauer Associates Inc. USA. pp. 411-501. [ Links ]
29.WINTER, P. and G KAHL. 1995. Molecular marker technologies for plant improvement. World J. Microbiology & Biotechnology. 11:438-448. [ Links ]
30.ZAMBRANO, A. Y., J. R. DEMEY, G. MARTINEZ, F. FUENMAYOR, Z. GUTIERREZ, G. SALDAÑA Y M. TORREALBA. 2002. Método rápido económico y confiable de minipreparación de ADN para amplificaciones por RAPD en bancos de germoplasma. Agronomía Trop. 52(2):235-243. [ Links ]













