Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Tropical
versión impresa ISSN 0002-192X
Agronomía Trop. v.59 n.4 Maracay dic. 2009
Evaluación de la inducción de variabilidad genética en cambur 'Manzano' (Musa AAB) a través de marcadores RAPD 1
Auris Silva*, Iselen Trujillo*, María Vidal*y Verónica Pérez*
1 Trabajo financiado por FONACIT y a la Universidad Nacional Experimental "Simón Rodríguez"
* Profesoras. Universidad Nacional Experimental "Simón Rodríguez". Instituto de Estudios Científicos y Tecnológicos (IDECYT). Centro de Estudios de Agroecología Tropical (CEDAT). E-mail: iselen03@yahoo.com
RESUMEN
En Venezuela, el cambur 'Manzano' (Musa AAB, banano tipo manzano) posee amplia aceptación por parte de los consumidores, pero su comercialización se ha visto limitada por el alto índice de plagas y enfermedades que lo atacan. En este sentido, el uso de las técnicas para el mejoramiento de la especie han tenido gran auge, por lo que en esta investigación se realizó la inducción de variación somaclonal mediante el uso de elevadas concentraciones de citocininas a través de multiplicaciones sucesivas, durante las cuales se aumentó de manera paulatina dichas concentraciones (5-10-15 mg l-1 de BA, respectivamente). Para el análisis de las plantas obtenidas de esta experiencia, se utilizó la técnica de los RAPD (ADN polimórfico amplificado al azar), la cual permitía establecer un patrón de comparación entre las plantas que se sometieron al proceso de inducción de variación somaclonal. Los resultados indican que en las 31 plantas analizadas durante la investigación con 12 iniciadores (primer) diferentes, se reflejaba un bajo porcentaje de variación al utilizar el índice de Jaccard´s como método de evaluación. Se piensa que la poca variabilidad encontrada en las plantas de Cambur Manzano (AAB) sometidas a elevadas concentraciones de citocininas, se debe a la presencia del genoma balbisiana en esta variedad, aspecto que ha sido señalado por diversos autores para otras variedades.
Palabras Clave: Variabilidad; cambur 'Manzano' (Musa AAB, banano tipo manzano); RAPD.
Evaluation of the induction of genetic variability in cambur 'Manzano' (Musa AAB) through markers RAPD 1
SUMMARY
In Venezuela, the cambur 'Manzano' (Musa AAB) it owns ample acceptance on the part of the consumers, but its commercialization has been limited by the high index of plagues and diseases attack that it. In this sense, the use of the techniques for the improvement of the species has had great height, reason why in this investigation the induction of somaclonal variation by means of the use of high concentrations of cytokinins through successive multiplications was used, during which increase of gradual way these concentrations (5-10-15 mg l-1, respectively). For the analysis of the obtained plants of this experience, the technique of the RAPD was used (at random amplified polymorphic DNA), which allowed us to establish a landlord of comparison between the plants that were put under the process of induction of somaclonal variation. The results indicate that in the 31 plants analyzed during the investigation with 12 initiators different, a low percentage of variation when using was reflected the index of Jaccard´s like evaluation method. The little found variability thinks that about the plants of cambur Manzano Musa (AAB) put under high concentrations of cytokinins, must to the presence of the balbisiana genome in this variety, aspect that has been reported by diverse authors for other varieties.
Key Words: Variability; cambur 'Manzano' (Musa AAB, banano tipo manzano); RAPD.
RECIBIDO: mayo 07, 2009 ACEPTADO: julio 20, 2009
INTRODUCCIÓN
El banano constituye uno de los cultivos más importantes en las regiones tropicales del mundo, tanto por su valor nutritivo como fruto como también su popularidad o rentabilidad, al punto que en algunos países han basado su economía en este cultivo (Escalant et al., 1989; Surga, 1988).
Así mismo, el banano constituye la principal fuente de carbohidratos para millones de personas en una amplia distribución mundial (Rowe, 1981, citado por Gawel et al., 1992).
Entre las distintas variedades que se conocen de banano en Venezuela, una de las que presenta alta demanda es el cambur 'Manzano' (AAB), debido a su sabor ya que adicionalmente representa una fuente de ingreso para los productores locales, gracias a su gran atractivo para la comercialización. Sin embargo, esta variedad es altamente susceptible a diversas enfermedades, tales como: el Mal de Panamá (Fusarium oxysporum f. cubense), Hereque o Moko (Pseudomonas solanacearum), Sigatoka Amarilla (Mycosphaerella musicola) y Sigatoka Negra (Mycosphaerella fijiensis), las cuales traen como consecuencia un bajo rendimiento de este cultivo y restando competitividad en el mercado interno, al compararlo por ejemplo con el cambur Pineo que presenta un alto índice de consumo, y por ende un alto nivel de comercialización (Haddad y Borges, 1974; Haddad y Leal, 1996).
Por esta razón, el uso de las técnicas biotecnológicas para el mejoramiento de la especie han tenido gran auge, donde destacan: selección y micropropagación de variantes naturales de cultivares seleccionados, inducción de variación somaclonal, inducción de mutaciones por agentes químicos y físicos, presión de selección por agentes bióticos y abióticos y transgénesis (Escalant et al., 1989; Trujillo, 1994; Trujillo y De García, 1996; Duncan, 1997).
El cultivo in vitro puede ser muy estresante para las células vegetales, y puede involucrar procesos mutagénicos durante el establecimiento de diversos procesos, tales como la inducción de callo organogénico y embriogénico, la formación de embriones y la regeneración de plantas. Por esta vía es posible obtener variación, de origen nuclear y/o citoplasmática, que podría ser utilizada para el mejoramiento vegetal. Este proceso, denominado variación somaclonal (Larkin y Scowcroft, 1981), involucra cambios en las plantas regeneradas que son transmitidos a la progenie.
La variación somaclonal ha sido utilizada para la producción de variedades comerciales con caracteres agronómicos superiores en diferentes especies. En Musa, su empleo se encuentra extensamente documentado. En el caso de cultivares comerciales de banana Cavendish se ha obtenido resistencia a la Fusarium oxysporum f.sp. cubense. (Cárdenas et al., 2002).
La eficacia del mejoramiento aumenta a través de la identificación de genes útiles, usando marcadores moleculares, mientras que la ingeniería genética permite la introducción de estos genes directamente en los cultivares deseados (Williams et al., 1990; Tingey et al., 1994; Vidal, 1996). El uso de técnicas de biología molecular, permite la detección de una variación genética en aquellos organismos obtenidos mediante procesos de cultivo in vitro. Métodos como los RFLP y los marcadores RAPD, junto con otras técnicas derivadas o análogas, generan marcadores útiles en el estudio de muchas especies, en este caso, especies vegetales (Fauré, 1992; Cloutier y Landry, 1994; Giménez et al., 2001; Salazar et al., 2006; Tui et al., 2006).
Los marcadores moleculares han sido usados con varios fines en especies de frutales tropicales, donde los usos más frecuentes corresponden a la estimación de diversidad genética intra e inter poblacional, sea en especies cultivadas o silvestres, para el desarrollo de mapas de ligamiento, para estudio de las relaciones filogenéticos entre especies, también como herramienta para determinar la mejor estrategia de conservación de recursos genéticos, entre otros (Azofeifa-Delgado, 2006).
En sus trabajos, Sánchez (2002) menciona que los marcadores moleculares son la principal herramienta utilizada hoy en día en microorganismos, plantas y animales para: caracterización de germoplasma, identificación de genotipos, determinación de pureza, análisis de diversidad genética, mejoramiento genético asistido por marcadores, aislamiento y caracterización de genes específicos para usar en transformación genética, construcción de mapas de ligamiento, entre otros.
Diversos autores han empleado los RAPD para estudios de diversidad genética, Crouch et al. (2000) emplearon este tipo de marcadores para el análisis de diversidad genética en variedades de Musa sp., y para comparar esta con la clasificación tradicional. Pillay et al. (2000) emplearon RAPD para identificar marcadores ligados a los genomas A y B en Musa spp., permitiendo aclarar la composición genética de todos los genotipos, facilitando la caracterización y manipulación de líneas en programas de mejoramiento. Kahangi et al. (2002) realizaron estudios de estabilidad genética en plantas regeneradas in vitro en Musa spp., donde 10 de los iniciadores utilizados generaron 69 secuencias marcadoras que permitieron la identificación de los materiales estudiados.
Los avances en la biología molecular han permitido el desarrollo de métodos de identificación y caracterización basados en el uso de marcadores moleculares que superan, en la gran mayoría de los casos, las limitaciones de los métodos tradicionales. Las técnicas de ADN recombinante hacen posible programas de mejoramiento genético eficientes y eficaces en plantas y animales, a través del llamado mejoramiento genético asistido por marcadores moleculares. Esta herramienta permite seleccionar plantas o animales que posean segmentos específicos de ADN asociados a genes de interés agronómico. De este modo, es factible la selección precoz de individuos superiores en aquellos casos en que el carácter de interés es de difícil identificación, se manifiesta muy tarde en la vida de las plantas o posee un elevado costo analítico.
Por otra parte, estas técnicas permiten la identificación inequívoca de variedades, de un modo similar al análisis del ADN que se realiza con fines forenses en el hombre. Por lo tanto, permiten no sólo identificar variedades o genotipos, sino también determinar el grado de parentesco entre especies y variedades, información de gran importancia para el mejoramiento genético y para los estudios de diversidad genética.
Este trabajo de investigación científica se orienta a la utilización de técnicas biotecnológicas que induzcan la variabilidad genética en cambur 'Manzano' (AAB), mediante el uso de concentraciones relativamente elevadas de citocininas, como una estrategia para el mejoramiento genético de esta variedad y su evaluación a través de marcadores moleculares tipo RAPD.
MATERIALES Y MÉTODOS
Material vegetal
El material vegetal fue colectado en el Banco de Germoplasma del CENIAP-INIA, estado Aragua. Hijuelos seleccionados de cambur 'Manzano' (Musa AAB) se separaron de las plantas cultivadas en campo y dicho material fue usado para iniciar la propagación in vitro de la variedad seleccionada.
Propagación in vitro de brotes de Musa
El material vegetal se sometió a un proceso de desinfección inicial, y posteriormente bajo condiciones asépticas en una cámara de flujo laminar, se realizó la iniciación de los explantes, previo tratamiento antioxidante (cisteina en una concentración de 60 mg l-1). Los explantes fueron cultivados en un medio básico con sales de Murashige y Skoog (1962) como medio de iniciación al 100% de su concentración, los cuales son suplementados con vitaminas de Morell (tiamina, 1,0 mg l-1; ácido pantoténico, 1,0 mg l-1; biotina, 0,01 mg l-1; mioinositol, 100 mg l-1), además de utilizar sacarosa (30 g l-1) como fuente de carbohidratos y agar (9 g l-1) como agente gelificante.
El pH del medio se ajustó a 5,8 con una solución de NaOH (1M), para luego ser servidos en tubos pirex de 15 cm de largo. Posteriormente, esos tubos fueron esterilizados en autoclave a una presión de 1,2 kg cm-2 y 120 ºC por un tiempo de 15 minutos. Luego de la siembra, los explantes permanecieron en oscuridad por 48 h, con el fin de disminuir la oxidación e inmediatamente pasaron a las cámaras de crecimiento. Después de este período, los explantes fueron cultivados en un cuarto de crecimiento a 30 ºC y una intensidad lumínica continúa de 70 UE/m2s de radiación fotosintética activa (RFA).
Para la iniciación del proceso de propagación in vitro e empleó una concentración de 0,5 mg l-1 de BA para inducir el proceso. La multiplicación de los explantes, se llevo a cabo utilizando el medio seleccionado en la etapa de iniciación, pero incrementando la concentración de BA en los sucesivos períodos de multiplicación de 5 mg l-1, 10 mg l-1 y 15 mg l-1, con un período de incubación de 30 d para cada subcultivo.
Para la etapa de enraizamiento se utilizó el mismo medio de multiplicación, pero modificando la concentración de sacarosa (40 g l-1) y de BA (0,5 mg l-1). Para la aclimatación de las plantas, estas fueron colocadas en propagadores, donde fueron regadas con agua corriente cada 3 d, y una vez por semana con solución de Hoagland. En un lapso de 30 d, las plantas fueron transferidas a bolsas plásticas con tierra negra abonada y se trasladaron a vivero.
Análisis molecular de la planta madre y las plantas obtenidas in vitro
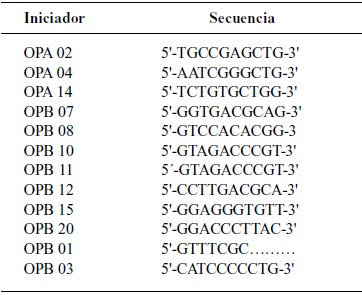
Para esta experiencia se utilizaron 31 plantas cambur 'Manzano' (planta madre y 30 plantas obtenidas in vitro). La extracción de ADN de las hojas de la planta madre y hojas de vitroplantas de los clones de banano se realizó según el método de Doyle y Doyle (1990). Para la realización del análisis molecular se usaron 10 iniciadores de la serie OPA y OPB (Operon Technologies), además, de una combinación entre varios iniciadores de la serie OPB. Para la amplificación de ADN de banano se utilizó el protocolo propuesto por Fauré et al. (1993).
La reacción de amplificación consistió de volúmenes de 25 μl que contenían: 2,5 μl 10X Buffer (500mM KCl; 100 mM tris-HCl pH 9,0; 1% tritón X-100); 0,3mM MgCl2; 200 μM dNTP's; 0,3 μM del iniciador, 2 unidades de Taq ADN polimerasa (Promega) y 200ng de ADN.
La amplificación de ADN se realizó en un termociclador marca PTC-100 Peltier Thermal Cycler MJ Research.
Las condiciones de incubación usadas fueron propuestas por Fauré et al. (1993), el programa de PCR consistió en una desnaturalización inicial con una duración de 1 min a 94 ºC, luego 40 ciclos de 1 min a 94 ºC, 1 min a 36 ºC y 2 min a 72 ºC, después un ciclo de 10 min a 72 ªC.
Los productos de la reacción de amplificación fueron separados mediante la electroforesis en gel de agarosa al 1,4% en buffer 1X TAE. La corrida electroforética se llevó a cabo inicialmente a 80 voltios, hasta que las muestras entraron en el gel, y luego a 120 voltios hasta que el cargador de muestra (0,25% azul de bromofenol + 30% glicerol) migrara aproximadamente 3 cm antes del final de la bandeja del gel, usando en la cámara el buffer 1X TAE. La tinción se realizó con bromuro de etidio para visualizar las bandas RAPD bajo luz ultravioleta y fotografiarlas.
Análisis de los datos
Para el análisis de los datos, se usó como patrón de peso molecular 1 kb DNA Ladder (Promega). Los polimorfismos detectados para cada iniciador se analizaron basado en las bandas obtenidas de la amplificación y se evaluaron como "0" cuando la banda estaba ausente en una posición particular y "1" cuando la banda estaba presente. Los datos obtenidos se analizaron mediante el programa MSVP versión 3,1b., utilizando el índice de Jaccard's para obtener un cluster análisis y se construyó un dendrograma usando el método de los promedios no ponderados (UPGMA).
RESULTADOS Y DISCUSIÓN
El proceso de micropropagación de la variedad de cambur 'Manzano', se realizó de acuerdo a la metodología empleada por Trujillo en 1994, con el fin deobtener un gran número de plantas en corto tiempo, para realizar la evaluación genética correspondiente, ya que este es el objetivo principal de la investigación.
Los resultados obtenidos de la multiplicación in vitro en la variedad cambur 'Manzano' se señalan en el Cuadro 1, donde se observa claramente que el número de brotes se incremento significativamente al aumentar la concentración de citocininas (BA).
CUADRO 1. Promedio de brotes obtenidos con la variedad cambur 'Manzano' al emplear diferentes concentraciones de citocininas.

El incremento substancial del número de brotes en cada uno de los tratamientos utilizados para el proceso de multiplicación tiene relación con las concentraciones de citocininas usadas en los medios de cultivo, tal ycomo lo demuestran autores como Orellana et al. (1991) y Pérez Ponce (1992), que relacionaron el éxito de esta etapa con los reguladores de crecimiento, ya que observaronque el uso de elevadas concentraciones de esta hormona estimula la formación de yemas adventicias,que son las responsables de la producción de nuevos explantes. El proceso de aclimatación de las plantas obtenidas in vitro resultó altamente exitoso, con un 100% de supervivencia.
Diversos autores como Tingey et al. (1994) y Munthali et al. (1996), señalan que los explantes sometidos a elevadasconcentraciones de citocininas pueden llegar a sufrir variación genética, la cual se detecta mediante el uso de marcadores RAPD's, que junto con la técnica de electroforesis en geles de agarosa y su posterior visualización mediante la tinción con bromuro de etidio y luz ultravioleta, generan una buena resolución de los fragmentos amplificados.
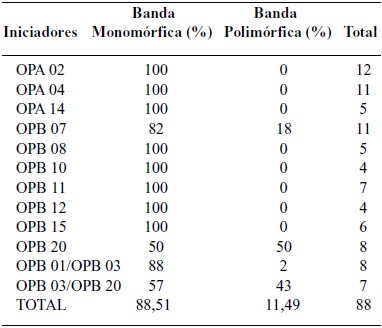
Para el análisis molecular, se utilizaron 31 plantas cambur 'Manzano' (planta madre y 30 obtenidas mediante el proceso de cultivo in vitro) y 10 iniciadores de la serie OPA y OPB (Cuadro 2), además de una combinación entre varios iniciadores de la serie OPB, de la casa Operon Technologies. Entre los iniciadores utilizados para la amplificación de ADN, sólo con 4 de ellos se pudieron obtener bandas marcadoras de polimorfismos contables, mientras que con el resto sólo se observa patrones de bandeo monomórficos (Cuadro 3).
CUADRO 2. Iniciadores utilizados en la reacción de amplificación de las plantas de cambur 'Manzano' (Musa spp.).
CUADRO 3. Porcentaje de bandas polimórficas obtenidos para cada iniciador.
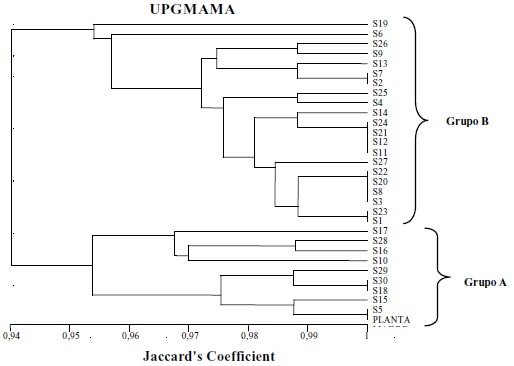
La cantidad de bandas producidas por cada iniciador en los diferentes somaclones presentó variaciones. Se analizaron un total de 80 bandas, utilizando el coeficiente de Jaccard's, por medio del cual se obtuvo un dendograma que mostró 2 grupos principales, el primero de ellos reunió a 9 somaclones junto a la planta madre y en el segundo grupo se incluyeron a los 21 somaclones restantes (Figura 1).
FIGURA 1. Análisis de agrupamiento basado en 87 marcadores RAPD obtenidos a partir de 30 somaclones de cambur 'Manzano' y la planta madre.
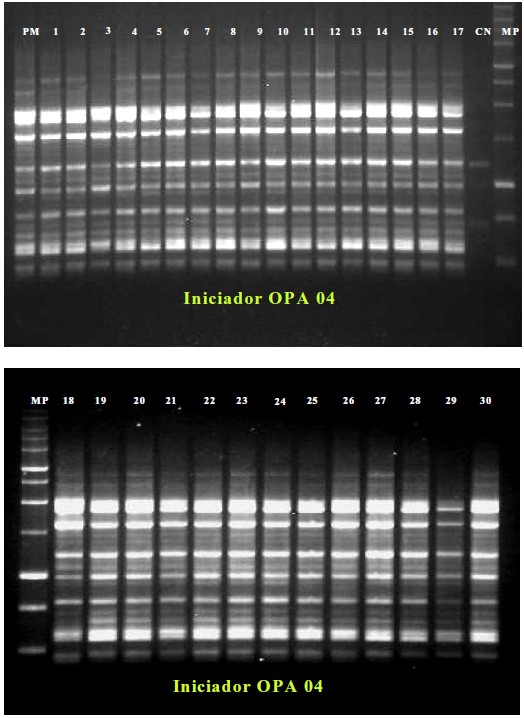
La Figura 2 muestra a manera de ejemplo la amplificación obtenida con el iniciador OPA04, donde el patrón de bandeo es totalmente monomórfico. Mientras que la amplificación obtenida con el iniciador OPB07, el cual amplificó un número máximo de 11 bandas. De los 31 somaclones usados, 25 amplificaron un total de 11 bandas; de los 6 somaclones restantes, 3 mostraron 10 bandas mientras que los 3 somaclones restantes presentaron 9 bandas, lo que permitió agrupar a las plantas en 3 grupos polimórficos entre sí. Los somaclones 2, 7 y 10 presentan 10 bandas, difiriendo en el resto de los somaclones en la banda número 3, cuyo peso molecular es aproximadamente de 1807 pb; mientras que en los somaclones 9, 13 y 18 estuvieron ausentes las bandas 2 y 3 cuyos pesos oscilan entre 1807-1655 pb, respectivamente.
FIGURA 2. Productos de amplificación de ADN de la planta madre y de los 30 somaclones obtenidos a través del proceso de cultivo in vitro, usando el iniciador OPA 04, separados por electroforesis en gel 1,4% de agarosa y teñido con Bromuro de Etidio. Las líneas corresponden: Línea MP, marcador de peso molecular; Línea PM, Planta madre, líneas de números corresponden a los somaclones ya que están enumerados de manera creciente del 1 hasta el 30.
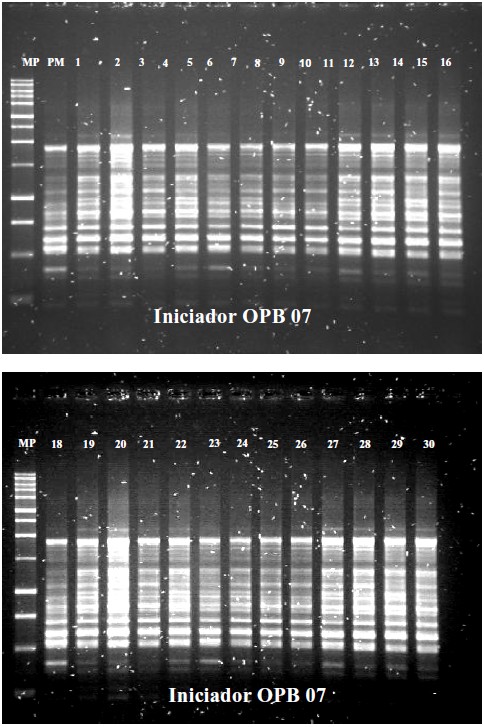
FIGURA 3. Productos de amplificación de ADN de la planta madre y de los 30 somaclones obtenidos a través del proceso de cultivo in vitro, usando el iniciador OPB 07, separados por electroforesis en gel 1,4% de agarosa y teñido con Bromuro de Etidio. Las líneas corresponden: Línea MP, marcador de peso molecular; Línea PM, Planta madre, líneas de números corresponden a los somaclones ya que están enumerados de manera creciente del 1 hasta el 30.
El iniciador OPB-20, presentó un total de 8 bandas, de las cuales 4 resultaron polimórficas con alguno de los somaclones. Con este iniciador se pudo agrupar a los 31 somaclones en 9 grupos polimórficos entre sí.
El primer grupo formado por somaclón número 6 presentó un patrón de bandeo de 7 bandas monomórficas, resultando polimórficas en la banda 6, cuyo peso molecular es de 702 pb.
Los somaclones 2, 3, 7, 8, 9 ,13, 19, 20, 22 ,26 y 27 formaban el grupo número 2 en donde se obtuvieron 7 bandas monomórficas y la banda número 7 resultó polimórfica, con un peso molecular de 617 pb. El grupo número 3 constituido por el somaclon 4 que presentó 6 bandas monomórficas y 2 bandas polimórficas, la 5 y 7 la primera con un peso de 748 y la segunda de 617 pb.
Los somaclones 11, 12, 14, 21,24 y 30 fueron colocados en el 4 grupo que mostró un patrón de 6 bandas monomórficas y 2 polimórficas, la 7 y 8 con pesos de 617 y 535 pb, respectivamente. El siguiente grupo representado por los somaclones 1 y 23 poseen 6 bandas monomórficas, resultando polimórficas las bandas 6 y 7 con pesos de 702 y 617 pb.
El grupo 6 formado por el somaclon número 25, presenta 5 bandas monomórficas y 3 polimórficas, la 5,6 y 7, de igual forma los grupos 7 y 8 mostraron un patrón de bandeo con 5 bandas monomórficas y 3 polimórficas, para el somaclon 18 resultaron polimórficas las bandas 5, 7 y 8, mientras que para los somaclones 10,16, 17, 28 y 29 dichas bandas estaban conformadas por la 6,7 y 8.
Por último, la planta madre y los somaclones 5 y 15 mostraron un total de 4 bandas monomórficas y 4 polimórficas, siendo estas las 5, 6, 7 y 8.
Los resultados indican que en las 31 plantas analizadas durante la investigación con 12 iniciadores (primer) diferentes, se reflejaba un bajo porcentaje de variación al utilizar el índice de Jaccard´s como método de evaluación.
Se piensa que la poca variabilidad encontrada en las plantas de cambur 'Manzano' (AAB) sometidas a elevadas concentraciones de citocininas, podría ser debido a la presencia del genoma balbisiana en esta variedad, aspecto que ha sido señalado por diversos autores para otras variedades (Smith, 1988; Vuylsteke et al., 1991; Trujillo, 1994; Pérez Ponce, 1998; Vendrame et al., 1999; Tui Ray et al., 2006).
Trabajos realizados por diversos autores indican que los RAPD son marcadores moleculares que han permitido el estudio de la estabilidad genética de plantas propagadas in vitro. Martín et al. (2006) obtuvieron variantes somaclonales al trabajar con concentraciones elevadas de citocininas al multiplicar in vitro la variedad de Musa Grande Naine (AAA), donde la plantas regeneradas in vitro fueron evaluadas a nivel genético empleando RAPD.
En susu trabajos, Tui Ray et al. (2006) estudiaron las relaciones y la fidelidad genética entre las variedades cultivadas y plantas micropropagadas empleando RAPD e ISSR, determinando la existencia de variantes somaclonales en el grupo estudiado. Venkatachalam et al. (2007) observaron estabilidad genética al analizar plantas micropropagadas de una variedad de Musa para postre (AAB) denominada Nanjanagudu Rasabale, obtenidas a partir de un proceso de multiplicación donde se emplearon altas concentraciones de BA.
La investigación realizada indica que altas concentraciones de citocininas en los procesos de cultivo in vitro originan variación somaclonal, sin embargo, por ser éste un proceso azaroso, los porcentajes de variabilidad pueden ser variados, y en algunas ocasiones y de acuerdo a diferentes factores como el genotipo de las plantas cultivadas in vitro, algunas veces resulta bajo, como sucedió con las plantas de cambur manzano empleadas en esta investigación.
Adicionalmente, se afirman que las características derivadas de este cambio son evaluadas a través de marcadores RAPD, los cuales resultaron ser marcadores moleculares adecuados para determinar la estabilidad o variabilidad de plantas regeneradas in vitro.
La variabilidad observada en las plantas propagadas in vitro empleando altas concentraciones de citocininas podría ser empleadas en procesos de mejoramiento genético del material estudiado, debido a que dicho cambio representa una resistencia a cierto tipo de enfermedades y plagas, lo que resultaría beneficioso, sobre todo desde el punto de vista económico para el productor, ya que podría disminuir la utilización de químicos para la eliminación de patógenos, lo que se traduce en una disminución del capital a invertir en cada cosecha y en beneficios para el ambiente.
BIBLIOGRAFÍA
1. Azofeifa-Delgado. A. 2006. Uso de Marcadores Moleculares en Plantas: Aplicaciones en Frutales del Trópico. Agronomía Mesoamericana 17(2):221-242.
2. Cárdenas, J. E, L. Pocasangre, A. S. Riveros y F. Rosales. 2002. Selección temprana de vitroplantas de Gros Michel (AAA) resistentes a la raza 1 del Mal de Panamá (Fusarium oxysporum f.sp. cubense). Memoria del II Encuentro de Investigadores en Agricultura Orgánica.
3. Cloutier. S. and B.S. Landry. 1994. Molecular Markers Applied to Plant Tissue Culture. In vitro Cell Dev. Biol. 30:32-39.
4. Crouch. H.K., J.H. Crouch, S. Madsen, D.R. Vuylsteke and R. Ortiz. 2000. Comparative analysis of phenotypic and genotypic diversity among plantain landrace (Musa spp., AAB, group). Theoretical and Applied Genetics 101(7):1056-1065.
5. Doyle. J.J. and J.L. Doyle. 1990. Isolation of plant DNA from fresh tissues. Focus 12:13-15.
6. Duncan. R. 1997. Tissue Culture-Induced variation and crop improvement. Advances in Agronomy 58:201-240.
7. Escalant, J. V., A. Tapia y J. Sandoval. 1989. Inducción de Callos Suspensión de Células y posibilidades de Regeneración en Musa sp. previa presión de Selección. In: Memorias de la IX reunión de ACORBAT, Mérida, Venezuela. p. 35-42.
8. Fauré. S., J.L. Noyer, J.P. Horry, F. Bakry, C. Lanaud and D. Gonzalez de Leon. 1993. A molecular markerbased linkage map of diploid bananas (Musa acuminata). Theor. Appl. Genet. 87:517-526.
9. Gawel. N.J., R.L. Jarret and A.P. Whittemore. 1992. Restriction Fragment Length Polymorphism (RFLP)- Based Phylogenetic Analysis of Musa. Appl.Genet. 84:286-290.
10. Giménez. C., E. de Garcia, N. Xena de Enrech and I. Blanca. 2001. Somaclonal Variation in banana: cytogenetic and molecular characterization of the somaclonal variant CIEN BTA-03. In vitro Cell. Dev. Biol.Plant 37:217- 222.
11. Haddad, O. y O. Borges. 1974. Los Bananos en Venezuela: Estudio y Descripción de Clones de Plátano y Cambur. Dirección de Investigación. Ministerio de Agricultura y Cría. 105 p.
12. Haddad, O. y F. Leal. 1996. Situación actual y perspectivas de la producción de cambur de exportación y otras Musáceas en el estado Aragua. Fundación para el Desarrollo de la Ciencia y la Tecnología en el estado de Aragua. [en línea]. Disponible en www.fundacite.arg.gov.ve./papelesf/index.html.
13. Kahangi. E.M., M.A. Lawton, C.A. Kumar. 2002. RAPD profiling of some banana varieties selected by small-scale farmers in Kenya. The Journal of Horticultural Science and Biotechnology 77(4):393-398.
14. Larkin. P. and W. Scowcroft. 1981. Somaclonal variation- a novel source of variability from cells cultures from plant improvement. Theor. Appl. Genet. 60:197-214.
15. Martin. K.P., S.K. Pachathundikandi, C.I. Zhang, A. Slater and J. Madassery. 2006. RAPD analysis of a variant of banana (Musa sp.) cv. Grande Naine and its propagation via shoot tip culture. In vitro Cell. Dev. Biol. Plant 42:188-192.
16. Munthali. M., H.J. Newbury and B.V. Ford-Lloyd. 1996. The detection of somaclonal variants of beet using RAPD. Plant Cell Rep. 15:474-478.
17. Murashige. T. y F. Skoog. 1962. A revised medium for rapid growth and bisassays with tobacco cultures. Physiology Plantarum. 15: 473-497.
18. Orellana, P, J. Pérez-Ponce, D. Agramonte, R. Gómez, E. Jiménez, S., Martínez, E. Almaguer y P. Gómez. 1991. La micropropagación del plátano a escala comercial en Cuba. Universidad central de Las Villas. Cuba 8 p.
19. Pillay. M., D.C. Nwakanma and A. Tenkouano. 2000. Identification of RAPD marker linked to A and B genome sequences in Musa L. Genome 43(5):763-767.
20. Pérez Ponce, J. 1992. Variación Somaclonal. Primer curso FAO-Francia-Cuba, sobre técnicas modernas de mejoramiento y multiplicación de especies agámicas. U.C.L.V. Cuba. S/P.
21. Salazar, E., J. G. Surga, J. A. Landínez y I. Trujillo. 2006. Uso de RAPD para la caracterización molecular de genotipos de Musa AAA propagados in vitro. Memorias de la Reunión Internacional ACORBAT 2006. Vol 2. p. 484.
22. Sánchez, I. 2002. Búsqueda y aislamiento de marcadores moleculares en Pleurotus ostreatus. (En línea). Navarra, España. (Consultado 1 ago. 2002). Disponible en www.unavarra.es/genmic/publicaciones/tfc/isabel%20sanchez.htm
23. Smith. M.K. 1988. A review of factors influencing the genetic stability of micropropagated bananas. Fruits 43:219-223.
24. Surga. J. 1988. Obtención de plantas libres de Virus de Mosaico del pepino por cultivos de ápices meristemáticos aislados in vitro de dos cultivares de banano. Fitopatología Venezolana. 1(2):69-72.
25. Tingey, S. V., J. A. Rafalski and J. G. K. Williams. 1994. Genetic Analysis with RAPD Markers. In: Aplications of RAPD Technology to Plant Breeding. Minneapolis. Minnesoto. p. 3-8.
26. Trujillo, I. 1994. Aplicación de Técnicas Biotecnológicas en el Mejoramiento del género Musa. Tesis Doctoral. Universidad Central de Venezuela. Escuela de Biología. Dto. de Botánica.
27. Trujillo. I. and E. de García. 1996. Aplicación de métodos de presión de selección en la obtención de variantes de banano resistentes a la Sigatoka amarilla. Phyton 59: 111-121.
28. Tui. Ray, D. Indrajit, S. Prasenjit, D. Sampa and S.C. Roy. 2006. Genetic stability of three economically important micropropagated banana (Musa spp.) cultivars of lower Indo-Gangetic plains, as assessed by RAPD and ISSR markers. Plant Cell, Tissue and Organ Culture. 85:11-21.
29. Vidal, M. 1996. Análisis bioquímico y genético de somaclones de Musa sp. obtenidos mediante procesos de cultivo in vitro. Universidad Central de Venezuela. Escuela de Biología. Dpto. de Botánica.
30. Vendrame. W.A, G. Kochert and H.Y. Wetzstein. 1999. AFLP analysis of variation in pecan somatic embryos. Plant Cell Rep. 18:853-857.
31. Venkatachalam. I., R.V. Sreedhar and N. Bhagyalakshmi. 2007. Genetic analyses of micropropagated and regenerated plantlets of banana as assessed by RAPD and ISSR markers. In vitro Cell. Dev. Biol. Plant 43:267-274.
32. Vuylsteke. D., R. Swennen and E. De Langhe. 1991. Somaclonal variation in plantains (Musa spp., AAB group) derived from shoot-tip culture. Fruits 46:429-439.
33. Williams. J.G.K., A.R. Kubelik, K.J. Livak, J.A. Rafalski and S.V. Tingey. 1990. ADN Polymorphisms amplified by Arbitrary Primers are Useful as Genetic Markers. Nucleic Acids Research. 18(22):6531-6535. [ Links ]