Introducción
La obesidad es una enfermedad metabólica representada por el incremento del índice de la masa corporal (IMC). En México, la prevalencia de sobrepeso es 38.8 % y la obesidad 32.4 %, 1. Como consecuencia, la obesidad y sus comorbilidades son una de las primeras causas de mortalidad prematura y evitable en el país 3, determinada por la interacción de factores genéticos y ambientales, constituye un factor de riesgo de otras enfermedades asociadas, o comorbilidades, como diabetes tipo 2, asma, enfermedades del hígado graso no alcohólico y varios tipos de cáncer 2. De conjunto, todas estas enfermedades representan un problema de salud que afecta tanto a países desarrollados como a los países en vías de desarrollo 3. Los factores de riesgo de padecer obesidad se relacionan directamente con los hábitos nutricionales. Sin embargo, la presencia de variantes genéticas originadas por mutaciones únicas y polimorfismos de un solo nucleótido (SNPs) explican entre el 40% y el 70% de la heredabilidad del IMC 5,6.
Con el acceso cada vez mayor a herramientas de diagnóstico de alta precisión para las investigaciones genéticas, se han identificado numerosos genes que influyen en el fenotipo. Los estudios genéticos especializados están ayudando a desentrañar la compleja fisiología que subyace a la regulación y el control del IMC. El mapa de genes de obesidad humana informa 127 genes candidatos para la obesidad 7, y raramente esta enfermedad ocurre en familias de acuerdo con un patrón de herencia claro causado por cambios en un solo gen.
Una descripción detallada de los genes con mayor frecuencia en los diferentes grupos étnicos debería ayudar a establecer estrategias personalizadas a nivel individual y étnico 2,3. Existe un número limitado de estudios sobre los genes que predisponen a padecer la obesidad en la población mexicana; disponer de una caracterización detallada de estos genes permitirá desarrollar estrategias más efectivas para la prevención y el control de esta enfermedad en la población mexicana. El objetivo general de este estudio es identificar variantes genéticas que predisponen el desarrollo de la obesidad en la población mexicana, y determinar las características distintivas comparadas con las variantes genéticas identificadas en otras poblaciones.
Materiales y métodos
La búsqueda bibliográfica se realizó en la base de datos Pubmed. Se incluyeron en la búsqueda los artículos publicados 2011 al 2021, utilizando como palabras clave: obesity, mexican, hispanic. Se incluyeron en el estudio los artículos publicados en español e inglés, que describen investigaciones diseñadas para determinar específicamente la asociación de factores genéticos y la obesidad tanto en niños como en adultos.
El desarrollo metodológico se realizó a partir de las consideraciones de la Declaración Prisma para el desarrollo de metaanálisis 8. Los artículos identificados se seleccionaron por inspección del Título y Resumen y se incluyeron los reportes con datos primarios o secundarios, que incluyeran estudios de asociación de variantes genéticas con el desarrollo de obesidad. La información obtenida se utilizó en un estudio comparativo, utilizando la estadística descriptiva para el análisis y la síntesis de las similitudes, diferencias y patrones en los datos de la población mexicana, en relación con otras poblaciones.
Extracción de datos
La extracción de los datos se realizó por inspección del texto completo. Para cada uno de los artículos seleccionados se extrajeron los datos de caracterización demográfica de la población en estudio. Como medida de la obesidad, se registró el IMC, calculado como el peso expresado en kilogramos por el cuadrado de la altura expresada en metros (kg/m2), y normalizado por la edad (IMC), o clasificado a partir de los percentiles (Z-SCORE) propuestos por la Organización Mundial de la Salud: IMC peso normal (18.5-24.99), sobrepeso (≥25.0), y obesidad (≥30.0); y Z-SCORE se define como normal (+ 1 a -1 DE), sobrepeso (> + 1 DE), obesidad (≥ + 2 DE) 9.
Análisis estadístico
Los datos obtenidos se procesaron mediante el programa SPSS (v.13.0), para realizar un análisis estadístico descriptivo. La caracterización de las distintas variables se realizó mediante el cálculo de la mediana y el rango, y la frecuencia, expresada como valores porcentuales. Para la determinación de los términos descriptores de los genes asociados a la obesidad en la población mexicana se utilizó el paquete de programas David versión 6.8 Oct. 2016 (https://david.ncifcrf.gov/) 10.
Resultados
Como resultado de la estrategia de búsqueda utilizada, se seleccionaron un total de 70 artículos para extraer los datos de asociación de variantes genéticas con obesidad en la población mexicana (Tabla 1). Los artículos abarcan el período 2011-2021, y muestran una tendencia general a incrementar el número por año (Tabla 1),
Tabla 1 Variantes genéticas estudiadas en la población mexicana, en su asociación con el desarrollo de la obesidad.
| SNPs asociados | SNPs no asociados | |||||
|---|---|---|---|---|---|---|
| Gen | SNPs | Ref. | Población1 | SNPs | Ref. | Población1 |
| FTO | rs1121980 rs140285 rs1421085 rs17817449 rs3751812 rs8044769 rs8057044 rs9930506 | (22) (16) (23) (5) (22) (22) (24) (22) | AM AM NM AM AM AM NM NM | rs62033400 rs6271 rs6272 rs6273 rs6274 rs6275 rs6276 rs6499640 rs7193144 rs8050136 rs8061518 rs9939609 rs9939973 | (23) (13) (13) (13) (13) (13) (13) (23) (5) (23) (23) (25) (23) | NM NM NM NM NM NM NM NM AM NM NM NM NM |
| LEPR | rs1805134 | (26) | AM | rs1137101 K109R Q223R K656N rs1892534 rs1137100 Ser492Thr rs1805094 rs1805096 | (18) (27) (28) (28) (28) (29) (26) (26) (26) (26) | AM NM NM NM NM AH AM AM AM AM |
| TCF7L2 | rs11196175 rs12255372 | (30) (30) (31) (32) | AM AM NM AH | rs7903146 rs7085532 rs4506565 rs7901695 rs6585194 rs10885406 rs290483 | (5) (33) (5) (5) (5) (5) (5) (5) | AM AM AM AM AM AM AM AM |
| ADIPOQ | rs822396 rs2241766 | (34) (18) | AM AM | rs6444174 rs2241766 rs3774262 rs1063539 rs1501299 rs266729 | (23) (33) (35) (5) (36) (5) (5) (5) (5) | NM AM AM AM AM AM AM AM AM |
| MC4R | rs6567160 rs2168708 rs28753167 rs72989246 rs6285 rs17782313 | (25) (37) (37) (37) (13) (18) | NM NM NM NM NM AM | rs11872992 rs17782313 rs571312 rs12970134 | (23) (23) (38) (39) (39) | NM NM NM NH NH |
| PCSK1 | rs1837269 rs6232 | (37) (40) | NM NM | rs6232 rs6233 rs6234 rs6235 rs271921 | (5) (5) (5) (5) (40) (5) | AM AM AM AM AM NM |
| IL-12B | rs6887695 | (41) | AM | rs1363670 rs3212220 rs3212227 rs1433048 rs2853694 rs1368439 | (41) (41) (41) (41) (41) (41) | AM AM AM AM AM AM |
| TMEM18 | rs6548238 rs7561317 | (18) (23) (23) | AM NM NM | rs6548238 rs7561317 | (38) (13) | NM NM |
| GNPDA2 | rs10938397 rs13130484 | (18) (25) | AM NM | rs16857402 rs6278 | (23) (13) | NM NM |
| KCTD15 | rs29941 | (18) | AM | rs29942 rs6281 | (23) (13) | NM NM |
| PPARG | Pro12Ala | (42) (Vázquez-Del Mercado et al. 2015) | AM AM | rs1801282 rs3856806 | (35) (33) (18) | AM AM AM |
| SLC30A8 | rs6469675 rs2464592 rs2466293 rs13266634 | (5) | AM AM AM AM | |||
| ABCA1 | rs9282541 | (43) | JM | rs2230806 | (33) | AM |
| BDNF | rs6265 | (44) (45) | AM N/JM | rs6265 | (13) | N/JM |
| FAIM2 | rs7132908 | (25) | NM | |||
| IL-15 | rs1057972 | (46) | AM | rs10833 rs3806798 rs4956403 | (46) | AM AM AM |
| MTCH2 | rs10838738 | (23) (18) | NM AM | |||
| NEGR1 | rs34305371 rs2815752 | (23) (18) (13) | NM AM N/JM | |||
| PON1 | L55M | (47) | AM | Q192R | (47) | AM |
| SEC16B | rs543874 | (25) | NM | rs10913469 | (39) (13) | NH N/JM |
| SH2B1 | rs7498665 | (18) | AM | rs4788102 rs7359397 rs7498665 | (39) (39) (13) | NH N/JM N/JM |
| CAPN10 | SNP-44 InDel-19 | (48) (48) | NM NM | rs2975760 SNP-43 SNP-63 | (33) (48) (48) | AM NM NM |
| GCK | rs1799884 rs12673242 rs2268576 | (5) | AM AM AM | |||
| KCNJ11 | rs5215 rs5210 | (5) (30) | AM AM | |||
| LRRN6C | rs10968576 | (37) | NM | |||
| MAF | rs1424233 | (18) | AM | |||
| PAI-1 | -675 4G/5G -844 G/A | (49) (50) | NM NM | HindIII C/G | (50) | NM |
| TFAP2B | rs2206277 rs987237 | (37) (13) | NM NM | |||
| UCP2 | rs660339 rs659366 | (51) (18) | A/JM AM | |||
| AHSG | rs2518136 rs4917 | (52) | AM AM | |||
| APOA5 | rs662799 rs964184 | (53) (43) | JM JM | |||
| CADM2 | rs6266 | (13) | NM | |||
| CAT | rs1001179 rs7943316 | (54) | AM AM | |||
| CD36 | rs1761667 | (55) | NM | |||
| COMT | rs933271 rs4646310 | (56) | JM JM | |||
| CRCP | rs1417938 rs3093058 | (29) | AH AH | |||
| ETS2 | rs8128155 rs7635103 | (37) | NM NM | |||
| ETV5 | rs6267 | (13) | NM | |||
| FANCL | rs6269 | (13) | NM | |||
| FLJ35779 | rs6270 | (13) | NM | |||
| GHRL | A-604G | (57) | AM | |||
| HTR2A | rs9567733 rs17069005 | (56) | JM JM | |||
| IL-10 | rs1800896 rs1800871 | (27) | NM NM | |||
| IL-6 | rs1524107 rs2069845 | (23) | NM NM | |||
| LMX1B | rs3829849 | (25) | NM | |||
| LRP1B | rs6282 | (13) | NM | |||
| MTHFR | C677T | (58) | AM | |||
| MTIF3 | rs4771122 | (13) | NM | |||
| NNMT | rs694539 rs1941404 | (16) | AM AM | |||
| OLFM4 | rs12429545 rs9568856 | (25) | NM NM | |||
| PPARA | rs1800206 | (33) | AM | rs1800206 | (35) | AM |
| PPARGC1A | rs8192678 Ser482Ser | (35) (36)(21) | AM AM | |||
| PRL | rs4712652 | (18) | AM | |||
| PTBP2 | rs1555543 | (13) | NM | |||
| SOD | rs2070424 rs4880 | (54) | AM AM | |||
| TMEM160 | rs28590228 | (37) | NM | rs3810291 | (13) | NM |
| TNFA | rs361525 | (33) | AM | 308G/A rs1800629 rs1800750 | (59) (27) (23) | AM NM N/JM |
| UCP3 | rs1800849 | (18) | AM | rs1800849 | (51) | A/JM |
| USP37 | rs4674340 | (37) | NM | |||
| ABCG2 | rs2231142 | (60) | N/AM | |||
| ACE | rs4646994 | (60) | N/AM | |||
| ACSL1 | rs9997745 | (35) | AM | |||
| ADAM23 | rs13387838 | (25) | NM | |||
| ADIPOR1 | rs2275737 | (5) | AM | |||
| ADRB1 | Arg389Gly | (61) | NM | |||
| AGT | rs699 | (33) | AM | |||
| ANKK1 | rs17115439 | (62) | AM | |||
| APOA4 | rs675 | (33) | AM | |||
| APOB | rs512535 | (33) | AM | |||
| APOE | rs405509 | (33) | AM | |||
| BCDIN3D-FAIM2 | rs7138803 | (23) | NM | |||
| CNR1 | rs10485170 | (5) | AM | |||
| COL4A1 | rs494558 | (63) | NH | |||
| DRD2/ ANKK1 | rs1800497 | (64) | AM | |||
| ELP3 | rs13253111 | (25) | NM | |||
| FAM120AOS | rs944990 | (25) | NM | |||
| FGF21 | rs11665896 | (65) | NM | |||
| FIGN | rs10192119 | (37) | NM | |||
| FOXO3/ HSS00296402 | rs9398171 | (37) | NM | |||
| GBE1 | rs3860595 | (37) | NM | |||
| GCKR | rs1260326 | (43) | JM | |||
| GIPR | rs6277 | (13) | NM | |||
| GNB3 | rs5443 | (18) | AM | |||
| GPR61 | rs7550711 | (25) | NM | |||
| GPRC5BB | rs6279 | (13) | NM | |||
| GPX-1 | rs10504050 | (55) | AM | |||
| GRP | rs1517035 | (37) | NM | |||
| GSTP1 | rs614080 | (66) | AM | |||
| HHIP | rs11727676 | (37) | NM | |||
| HMGA1 | rs6280 | (13) | NM | |||
| HMOX1 | rs2071749 | (67) | AM | |||
| HNF1A | rs7305618 | (27) | NM | |||
| HNF4 | rs1885088 | (33) | AM | |||
| HOXB5 | rs9299 | (25) | NM | |||
| HP | rs2000999 | (68) | NM | |||
| IL-1𝛽 | rs1143643 | (23) | NM | |||
| IL6R | rs2228145 | (29) | AH | |||
| INSIG2 | rs7566605 | (18) | AM | |||
| IRS2 | 10510452_139 | (69) | NH | |||
| IRX3 | rs3751723 | (16) | AM | |||
| KCNJ12 | rs5219 | (5) | AM | |||
| KLF6 | rs12253976 | (32) | AH | |||
| LEP | rs2167270 | (23) | NM | |||
| LGR4-LIN7C-BDNF | rs925946 | (23) | NM | |||
| LIPC | rs1800588 | (35) | AM | |||
| LOC285762 | rs2357760 | (37) | NM | |||
| LPL | rs320 | (33) | AM | |||
| MAP2K5 | rs6284 | (13) | NM | |||
| MGAT1 | rs4285184 | (70) | AM | |||
| MTP | rs1800591 | (53) | JM | |||
| NEGR2 | rs2815752 | (23) | NM | |||
| NPC1 | rs1805081 | (18) | AM | |||
| NQO1 | rs1800566 | (67) | AM | |||
| Nrf2 | rs2364723 | (67) | AM | |||
| NRXN3 | rs10150332 | (13) | NM | |||
| PNPLA3 | I148M | (71) | AM | |||
| POMC | rs713586 | (13) | NM | |||
| PON2 | PON1-L55M | (72) | NM | |||
| PRKCH | rs4899048 | (37) | NM | |||
| PTER | rs10508503 | (18) | AM | |||
| PTPN22 | +1858C/T | (59) | JM | |||
| RAB27B | rs8092503 | (25) | NM | |||
| RETN | rs1862513 | (27) | NM | |||
| SCARB1 | rs10846744 | (33) | AM | |||
| SLC6A3A | rs3776511 | (56) | AM | |||
| TRHR | rs7832552 | (33) | AM | |||
| TUB | rs4929949 | (13) | NM | |||
| TXNIP | rs7211 | (67) | AM | |||
| UCP1 | rs1800592 | (51) | A/JM | |||
La mayor parte de los trabajos tiene una representación balanceada de ambos sexos (Tabla 1). Solo cuatro de las investigaciones se realizan con personas de un solo sexo (5,7 % del total, sexo femenino); en los 64 trabajos restantes se incluyen ambos sexos con una frecuencia que oscila entre 19 % a 93 % del sexo femenino. El 85,7 % de los trabajos estudia población mexicana, el 51 % de los trabajos incluye población infantil (4-18 años). De conjunto, los estudios en población adulta incluyen personas desde los 19 hasta los 82 años de edad. Los trabajos tienen un número muestral variable que oscila entre 50 y 8914 personas, con un 51% con más de 500 individuos incluidos en el estudio (Tabla 1).
a. Identificación de variantes genéticas asociadas con la obesidad
Con relación al número de genes estudiados, en 6 de los 70 artículos de investigación se analiza el genoma completo. En 35 (50 %) artículos se estudian uno o varios SNPs (rango 1-7, mediana =1) de un único gen; y en 28 (40 %), se realiza la investigación en varios genes. De manera general, los estudios de múltiples genes incluyen también un mayor número de SNPs en sus análisis (rango 1-672, mediana =15).
En todos los casos, los SNPs seleccionados han sido previamente estudiados y validados en su asociación al desarrollo de la obesidad en otras poblaciones. Resulta de interés que del total de 260 SNPs estudiados en los diferentes artículos, 100 (38,4 %) se encontraron también asociados en población mexicana mientras que para 160 (61,5 %) no se confirmó la asociación en población mexicana (Tabla 1).
Otro resultado de interés es la baja frecuencia de asociación encontrada en los SNPs estudiados para los genes FTO, LEPR, TCF7L2 y ADIPOQ (Tabla 1). La relevancia de este resultado radica en que estos genes son considerados de los más importantes en los factores genéticos asociados a la obesidad 11. Por otra parte, todos los SNPs evaluados en estos estudios han sido validados en otras poblaciones, mayormente caucásicos. Sin embargo, estudios anteriores han encontrado resultados similares en cuanto a la dependencia de la proporción de ancestralidad europea en la asociación de determinados SNPs con el desarrollo de la obesidad 12.
De conjunto, estos resultados resaltan la importancia de la etnia en la heredabilidad de las variantes genéticas asociadas al desarrollo de la obesidad. A la vez, destaca la importancia de los estudios en las distintas etnias para poder conformar un sistema de predicción apropiado en cada caso.
b. Asociación de SNPs del gen FTO en niños y adultos mexicanos
En particular se aprecia un número considerable de SNPs del gen FTO que no muestran asociación en la población infantil mexicana (Tabla 1). De los 21 SNPs analizados en el gen FTO, 16 corresponden a estudios realizados en niños. De estos, solo 3 (18%) mostraron asociación con el desarrollo de la obesidad en niños mexicanos. Es de destacar que 4 de los 6 SNPs más estudiados del gen FTO si resultaron asociados también en la población mexicana en adultos mexicanos (rs17817449, rs3751812) y niños mexicanos (rs9930506, rs14210805).
Tabla 2 Asociación diferencial de variantes genéticas en niños y adultos mexicanos.
| Población mexicana (Ref.) | |||||
|---|---|---|---|---|---|
| Gen | Proteína1 | SNP | Niños | Jóvenes | Adultos |
| MC4R | Melanocortin receptor 4 | rs17782313 | - (4) | nr2 | + (29) |
| PCSK1 | Neuroendocrine convertase 1 | rs6232 | + (45) | nr | - (34) |
| TMEM18 | Transmembrane protein 18 | rs6548238 | + (29) | nr | - (9) |
| BDNF | Brain-derivedneurotrophic factor | rs6265 | - (24) | - (24) | + (42) |
| SH2B1 | SH2B adapter protein 1 | rs7488665 | - (29) | - (29) | + (42) |
Ref: Número de la referencia en la Tabla 1. 1 Descripción de la proteína de acuerdo con la base de datos UniprotKB(73). 2 nr, no relacionado
El gen FTO codifica para la Dioxigenasa dependiente de alfa-cetoglutarato (Uniprot KB: Q9C0B1). Esta enzima es conocida también comoFat mass and obesity-associated protein(Masa grasa y muscular asociada a la obesidad) por su demostrada relación con el desarrollo de la obesidad no a nivel de cambios estructurales en la proteína sino a nivel genético y epigenético 14. Aunque no se conocen en detalle los mecanismos por los cuales este gen contribuye al desarrollo de la obesidad, una de las hipótesis más aceptadas sugiere que los SNP FTO pueden estar asociados con la obesidad al regular la expresión de genes adyacentes por ejemploRPGRIP1L y IRX315,16. Estudios han demostrado que las variantes genéticas de FTO juegan un papel clave en la regulación de la ingesta de alimentos y el gasto de energía. También se ha evidenciado que los efectos de este gen en la obesidad se intensifican por factores ambientales como la actividad física reducida y una dieta alta en calorías 14.
c. Asociación diferencial de variantes genéticas en niños y adultos mexicanos
Otros estudios también evidenciaron que algunas variantes genéticas presentan una influencia diferente en niños y adultos mexicanos (Tabla 2). Estos hallazgos resultan particularmente interesantes puesto que el mismo SNPs ha sido evaluado en grupos de diferentes edades.
Los SNPs rs17782313 (MC4R), rs6548238 (TMEM18), rs6265 (BDNF) y rs7488665 (SH2B1) presentan asociación en adultos, pero no en niños o jóvenes. En cambio, el rs6232 (PCSK1) presenta asociación en niños y no en adultos. Este último resultado es consistente con el conocimiento previo. La Convertasa neuroendocrina 1 (PCSK1) es una proteasa involucrada en el procesamiento biosintético de una variedad de neuropéptidos y prohormonas en tejidos endocrinos 17. El gen que codifica para esta proteína fue uno de los primeros genes relacionados con la obesidad monogénica de inicio temprano. La proteína PCSK1 es esencial para activar la escisión de muchos precursores de hormonas peptídicas implicados en la regulación de la ingestión de alimentos, la homeostasis de la glucosa y la homeostasis de la energía 17.
Discusión
La presencia de resultados contradictorios en investigaciones las variantes genéticas en la obesidad la influencia no es diferente, pueden intervenir múltiples factores del diseño experimental y el tamaño muestral. Por una parte, los que estudian a la población infantil confirma el interés que ha tomado el tema de la prevalencia de la obesidad en la población mexicana y, por otro lado, también pueden indicar que existen factores ambientales con una influencia importante en la actividad del gen sobre la obesidad que no se están teniendo en consideración y merecen más investigación. Tal es el caso de los resultados obtenidos para el SNP rs2241766 (ADIPOQ).
Las investigaciones que dan lugar a resultados diferentes para el SNP rs2241766 (ADIPOQ) incluyen León-Mimila et al.(2013), que obtiene asociación con la obesidad y Costa-Urrutia et al. (2017), Costa-Urrutia et al. (2018), Hernández-Escalante et al. (2014) y Guzmán-Ornelas et al. (2015), estas últimas con un resultado negativo para la asociación de este SNP con la obesidad (Tabla S1). 18,19,20,5,21
Es de señalar que en el estudio de Guzmán- Ornelas et al. (2015) 18, evidenció una asociación de este SNP con la distribución de los depósitos de grasa y los marcadores de inflamación, pero no con el IMC. Resulta interesante que los estudios de Costa-Urrutia et al. (2017) y (2018), incluyen personas físicamente activas, miembros de clubes deportivos, mientras que los otros estudios incluyen adultos sin especificar el grado de actividad física. Por tanto, el resultado de no asociación del SNP rs2241766 del estudio de Costa-Urrutia (2017) 19,20 pudiera estar relacionado con la atenuación del efecto sobre la obesidad debido a la práctica del ejercicio físico. Se necesitaría un estudio diseñado intencionalmente para evaluar esta posibilidad.
Para identificar las características distintivas del conjunto de proteínas codificadas por los genes con asociación con la obesidad en población mexicana, realizamos un análisis de enriquecimiento según sus términos descriptores en las bases de datos de proteínas utilizando el programa DAVID. La muestra de análisis se conformó con los genes identificados con asociación con el IMC o la obesidad, y se comparó con la frecuencia de aparición de los términos descriptores en la lista de genes de referencia del genoma humano disponible. 10.
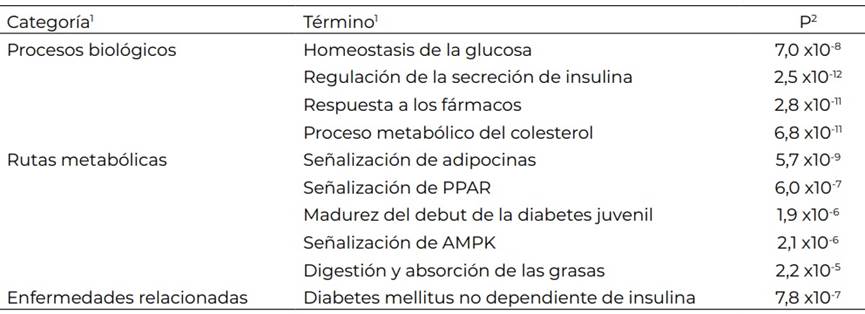
El análisis de las características distintivas del conjunto de proteínas codificadas por los genes con asociación con la obesidad en población mexicana evidenció un enriquecimiento en proteínas involucradas en los procesos y las rutas metabólicas de la homeostasis de la glucosa, incluyendo la relación con el desarrollo de la Diabetes mellitus no dependiente de insulina (Tabla 3).
Tabla 3 Resultados del análisis de enriquecimiento de términos que caracterizan a las proteínas codificadas por los genes estudiados en la relación de las variantes genéticas con el desarrollo de la obesidad.
1 Se utilizaron las herramientas de agrupamiento disponibles en la base de datos DAVID(10). 2 Significación estadística. La comparación se realizó contra la lista de proteínas humanas del DAVID.
Se destaca también el enriquecimiento de las proteínas involucradas en los procesos metabólicos del colesterol y en las vías de señalización de las adipocinas. Estos resultados coinciden con las características de la familia de genes identificados anteriormente asociados al desarrollo de la obesidad 7
Finalmente, un elemento para tener en cuenta en este análisis es que 6 de los SNPs seleccionados para evaluar en niños mexicanos estaban validados en población adulta europea 13. Por tanto, la no asociación en niños mexicanos puede responder no solo a un efecto de la diferencia en la etnia sino también en la edad.
Conclusiones
La población con obesidad y sobrepeso presenta cifras que se incrementan cada año tanto en adultos como en niños en México. Además de los hábitos de nutrición y la actividad física, se han identificado un número de SNPs que generan variantes genéticas que pueden predisponer a la persona a padecer obesidad. Estas variantes genéticas pueden tener un comportamiento dependiente de la etnia, en relación con su asociación al desarrollo de la obesidad. Numerosas investigaciones han abordado la asociación de diferentes SNPs en la predisposición a padecer obesidad en la población mexicana. La revisión de estos trabajos nos ha permitido establecer que, el conjunto de genes que presentan asociación con el desarrollo de la obesidad en población mexicana está asociado a procesos biológicos ya identificados como relevantes en el desarrollo de la obesidad, destacándose la asociación con el desarrollo de la diabetes mellitus y el metabolismo del colesterol. Sin embargo, se identifica un comportamiento diferencial en determinados SNPs en la población mexicana. Tal es el caso de los SNPs rs17782313 (MC4R), rs6548238 (TMEM18), rs6265 (BDNF) y rs7488665 (SH2B1). De particular interés resulta la identificación del SNP rs6232 del gen PCSK1 con la aparición temprana de la obesidad en la población mexicana. Los resultados sugieren además que la actividad física puede atenuar el efecto del SNP rs2241766 (ADIPOQ) en el desarrollo de la obesidad en población mexicana. De conjunto, estos resultados pueden constituir el punto de partida de una investigación dirigida al desarrollo de sistemas de alta sensibilidad para la detección de la susceptibilidad a padecer obesidad en la población mexicana, así como el desarrollo de programas personalizados para el tratamiento de esta dolencia.