Kasmera
versión impresa ISSN 0075-5222
Kasmera v.33 n.2 Maracaibo jul. 2005
Evaluación de la baciloscopia, cultivo y reacción en cadena de la polimerasa para el diagnóstico de tuberculosis pulmonar
Evaluation of Baciloscopy, Cultivation and Polimerasa Chain Reaction for the Diagnostic of Lung Tuberculosis
Nava Paz, Orlando1; Hassanhi, Manzur2 y Prieto, Lisbeth3
1Cátedra de Medicina Tropical. Facultad de Medicina. LUZ. 2Laboratorio HLA e Inmunogenética. Banco de Sangre de Maracaibo. 3Hospital General del Sur. Maracaibo, Venezuela. E-mail: onabap@cantv.net
Resumen
La utilidad de la baciloscopia, cultivo y la Reacción en Cadena de la Polimerasa (RCP) fue evaluada en 31 muestras de esputo, provenientes de pacientes de ambos sexos, sin distinción de grupo étnico y edades comprendidas entre los 17 y 80 años, con manifestaciones clínicas, hallazgos radiológicos y/o datos epidemiológicos sugestivos de tuberculosis pulmonar. A todas las muestras, se les practicó baciloscopia utilizando la técnica de coloración de Ziehl-Neelsen. Se utilizó el medio de cultivo Ogawa-Kudoh como estándar. Los objetivos para el ensayo de RCP fueron los genes que codifican la proteína de 32-KDa. y la secuencia de inserción IS6110. Luego de la amplificación, se obtuvo un fragmento de 984 pares de bases correspondiente a la secuencia de inserción IS6110 y otro de 506 pares, correspondiente al antígeno alfa 32-KDa. Del total de muestras examinadas por el método de Ziehl-Neelsen, 19 resultaron positivas y 12 resultaron negativas (61,29 y 38,71% respectivamente). De las 31 muestras sembradas 19 (61,29%) resultaron positivas y 12 (38,71%) fueron negativas al cultivo. De todas las muestras amplificadas, 22 (70,96%) mostraron la presencia de fragmentos de ADN correspondientes tanto a la secuencia de inserción IS6110, como al antígeno alfa 32 KDa., mientras que en 9 (29,04%) no se detectó la presencia de ADN micobacteriano por el método de RCP. La sensibilidad de la prueba RCP fue virtualmente de 100% y su especificidad fue del 75%. Se demostró que el RCP, basado en la amplificación simultánea del elemento de inserción IS6110 y del gen que codifica el antígeno alfa en un solo paso, puede identificar infecciones por micobacterias a partir de muestras de esputo. Se concluye que esta técnica es más sensible que los procedimientos bacteriológicos de rutina (examen directo y cultivo) y la convierten en una herramienta muy poderosa para el diagnóstico de tuberculosis pulmonar y otras enfermedades micobacterianas.
Palabras clave: Tuberculosis, RCP, Examen directo, cultivo.
Abstract
The utility of baciloscopy, cultivation and Polimerasa Chain Reaction of (PCR) was evaluated in 31 sputum samples, of patient of both sexes, without distinction of etnial grup and ages between 17 and 80 years, with clinical manifestations, discoveries radiological and/or suggestive epidemic data of lung tuberculosis. To all the samples they were practiced baciloscopy using the technique of Ziehl-Neelsen. The Ogawa-Kudoh cultivation was used like standard. The objectives for the rehearsal of PCR were the genes that code the protein 32-KDa (alpha antigen) and the insertion sequence IS6110. After the amplification was obtained a fragment of 984 couples of bases corresponding to the sequence of insert IS6110 and another of 506 couples corresponding to the alpha antigen. Of the total of samples examined by the Ziehl-Neelsen method, 19 were positive and 12 were negative (61.29 and 38.71% respectively). 19 samples (61.29%) were positive and 12 (38.71%) were negative to the cultivation. Of all amplified samples, 22 (70.69%) showed the presence of fragments corresponding to DNA so much to the sequence of insert IS6110 like the antigen alpha 32-KDa, while in 9 (20.04%) presence of DNA mycobacterium was not detected for RCP method. The sensibility of the RCP test was virtually 100% and its specificity was of 100%. It was demonstrated that RCP, based on the simultaneous amplification of the insertion element IS6110 and the gene that codes the alpha antigen in a single step, can identify mycobacterium infections. We conclude also that is more sensitive that routine bacteriological procedures (direct exam and cultivation) and can be a very powerful tool for the diagnosis of lung tuberculosis and other mycobacterium diseases.
Key words: Tuberculosis, RCP, direct exam, cultivation.
Recibido: 20-09-05 / Aceptado: 16-11-05
Introduccion
El descubrimiento de la quimioterapia efectiva para erradicar la tisis (nombre por el cual se conoció durante mucho tiempo la enfermedad) trajo consigo la creencia que la tuberculosis sería fácilmente erradicable. Sin embargo, el tiempo se encargó de demostrar lo contrario y la conjunción de factores psicosociales, epidemiológicos, administrativos, operativos han permitido que la tuberculosis continúe y se halla transformado, actualmente, en una verdadera plaga para el hombre (1).
La Tuberculosis es una de las enfermedades infecciosas que mata a más personas en el mundo entero. La mortalidad mundial por tuberculosis es aproximadamente de 2 millones de personas por año (2).
La OMS ha estimado que entre el año 2000 y el 2020, 200 millones de personas enfermarán y 35 millones morirán de tuberculosis si no se hace un riguroso control. Alrededor de 8 millones de personas en todo el mundo, enferma de tuberculosis cada año (2, 3).
Los datos obtenidos por la OMS permiten hacer las siguientes afirmaciones:
● Cada segundo una persona es infectada por primera vez por el bacilo tuberculoso.
● Cerca del 1% de la población mundial es infectada por primera vez por el bacilo tuberculoso cada año.
● Un tercio de la población mundial está actualmente infectada por el bacilo tuberculoso.
● Del 5 al 10% de las personas infectadas con el bacilo tuberculoso (pero no infectado con HIV) enfermarán de tuberculosis en algún momento de su vida (3).
Para el año 2002 se produjeron cerca de 2 millones de muertes por tuberculosis en todo el mundo, siendo la región del sur-este asiático quien aportó la mayor proporción con 625 mil, seguida por la región africana con 556 mil. Las Américas aportaron aproximadamente 6 mil muertes por tuberculosis (3).
La pandemia del VIH ha traído un gran impacto en la epidemiología de la tuberculosis. Para finales del año 2000, aproximadamente 12 millones de las 36 millones de personas infectadas con el VIH en todo el mundo, tenían coinfección con M. tuberculosis y 8.4 millones (70%) de esos coinfectados, viven en la región del África Subsahariana (4). La infección por VIH incrementa 40% el riesgo de conversión de infección a enfermedad (4,5) y por lo tanto también incrementa la transmisión de la tuberculosis a la población general. La tuberculosis es responsable de aproximadamente el 15% de las muertes por SIDA en todo el mundo (4).
El problema se combina con la emergencia de cepas de M. tuberculosis resistentes a las drogas actualmente usadas, principalmente a la Isoniazida y a la Rifampicina, las dos drogas antituberculosas más potentes, originando cepas multirresistentes, las cuales han alcanzado proporciones alarmantes en algunos países, especialmente en al antigua Unión Soviética (5, 6).
El problema diagnóstico de la Tuberculosis
El diagnóstico clínico de la tuberculosis es difícil de establecer. A tal punto que muchos autores la han catalogado como la gran simuladora (1), presentándose muchas veces bajo la forma de cuadros clínico – radiológicos completamente atípicos e inusuales, que desconciertan al médico en cuanto a la orientación diagnóstica. Por lo tanto los hallazgos clínicos y radiológicos que se observan en los pacientes tuberculosos, no aportan siempre datos patognomónicos que permitan su diagnóstico (1).
El diagnóstico bacteriológico de la tuberculosis no está exento de errores: la baciloscopia, a través del examen directo de la muestra y coloración con la técnica de Ziehl-Neelsen, no es 100% confiable, pues el bacilo no siempre es detectado en las muestras clínicas examinadas. La microscopía, utilizando la técnica de coloración de Ziehl – Neelsen, es rápida, económica y sencilla. La sensibilidad deja mucho que desear, varía dependiendo del tipo de muestra y la micobacteria involucrada, ya que como regla deben existir entre cinco mil a diez mil bacilos por ml. de expectoración para que tengan un 50% de posibilidades de ser detectados al microscopio; sólo cuando el numero de bacilos alcanza a más de 100.000 por ml. de expectoración, podemos esperar que las baciloscopias sean consistentemente positivas (7). Utilizando la tinción fluorescente, este número puede ser tan bajo como 1.000 bacilos por mL (7, 8).
Además la presencia de bacilos ácido alcohol resistentes en el examen directo de muestras clínicas, no siempre garantiza que se trate de un bacilo tuberculoso, pues puede tratarse de una micobacteria atípica o de otro microorganismo que comparta la característica de ácido alcohol resistencia (M. leprae, Actinomyces, Nocardia). Esto puede ocasionar graves problemas diagnósticos y terapéuticos. El rango de sensibilidad de la baciloscopia, oscila entre 50 – 80% y la especificidad es virtualmente del 100% (7).
El cultivo. Al lado de la baciloscopia, el diagnóstico bacteriológico de la tuberculosis, debe complementarse con el cultivo. El cultivo es una técnica que tiene mayor sensibilidad (70-90%), ya que basta que existan más de 10 bacilos/ ml., en muestras digeridas y concentradas, para que sea positivo. Recordemos que la baciloscopia sólo utiliza 0,01 ml. de la muestra, efectuando un extendido de unos 10.000 campos microscópicos, de los cuales en el mejor de los casos, sólo se leen 100 a 200 campos; en cambio, los cultivos procesan 0,1 ml. de expectoración (8).
El aislamiento de las micobacterias por cultivo es entorpecido por su lento crecimiento. Un promedio de incubación de 4 semanas en medios convencionales se requiere antes que pueda ser detectado crecimiento. Los métodos de identificación convencionales después del cultivo incluyen determinación de la velocidad de crecimiento, crecimiento a diferentes temperaturas, morfología de la colonia, producción de pigmentos y susceptibilidad a los agentes. De tal manera que en casos en los que se requiera una toma de decisiones rápidas para instaurar una terapéutica efectiva su valor es muy limitado (7, 8).
Se han hecho muchos intentos para mejorar los métodos de cultivo del bacilo tuberculoso, de modo que se pueda disponer de sus resultados en plazos más breves. Las técnicas más útiles a este respecto parecen ser las radiométricas, que permiten hacer el diagnóstico de muchas infecciones bacterianas en pocas horas y de la tuberculosis en pocos días. Además, tienen una mayor sensibilidad que los métodos bacteriológicos tradicionales. Se han descrito métodos de cultivo más rápidos como: El método radiométrico BACTEC, el sistema ESP Myco, el sistema MB/BacT Alert, el sistema de tubo MGIT, el sistema Septi-Chek AFB, la detección de microcolonias en medios sólidos (9-15). Desafortunadamente tales pruebas son costosas en su inicio, requieren de personal altamente calificado y su sensibilidad y especificidad son muy variables de uno a otro laboratorio. Por lo tanto no están disponibles en todos los países en vías de desarrollo.
Por lo tanto, las técnicas bacteriológicas tradicionales de laboratorio (baciloscopia y cultivo), a pesar que tienen una buena sensibilidad y especificidad, no son lo suficientemente útiles cuando se requiere de un diagnóstico precoz y específico (formas extrapulmonares, primoinfección tuberculosa en niños, formas cerradas, infección micobacteriana en pacientes VIH).
Las Técnicas de Biología Molecular: De las técnicas de biología molecular, la amplificación de ácidos nucleicos como la Reacción en Cadena de la Polimerasa (RCP) es una técnica muy útil para el diagnóstico bacteriológico de la tuberculosis, pues permite un diagnóstico rápido, sensible y específico, a través de la identificación del ADN o ARN presente en las muestras clínicas, pudiendo despejar los problemas derivados de los procedimientos habituales de laboratorio (13).
Los resultados del RCP igual que los de la microscopía, pueden ser obtenidos 24 horas después de recibir la muestra. La especificidad y sensibilidad excede a la de la microscopía. Estudios han reportado sensibilidades entre 74-91% y especificidades entre 95-100% (14).
Se han descrito muchos ensayos de RCP para la detectación de bacterias del Complejo M. tuberculosis. Existen dos métodos basados en la amplificación de ácidos nucleicos aprobados por la FDA (Food and Drug Administration) para la detección de M. tuberculosis, a partir de muestras respiratorias con baciloscopias positivas. Ellas son la prueba MTD (Amplified Mycobacterium tuberculosis Direct Test) de Gen-Probe®, San Diego, California; y la prueba AMPLICOR de Roche® Diagnostic Systems, Inc., Branchburg, New Jersey (15). En este sentido, la prueba de AMPLICOR desarrollada por Roche Diagnostic Systems® ha mostrado ser una herramienta muy útil para el diagnóstico precoz de tuberculosis, con una sensibilidad y especificidad de 76.4% y 99.8% respectivamente (16-18), no solo a partir de muestras respiratorias sino también a partir de muestras de fluidos y tejidos corporales (18).
Se han publicado estudios basados en la amplificación de muchas secuencias de M. tuberculosis, la secuencias de proteínas de 32 kDa (19), 65 kDa (20). Sin embargo, el objetivo más común para la amplificación es la secuencia de inserción IS6110, la cual sólo está presente en las micobacterias que pertenecen al Complejo M. tuberculosis (17, 20-24).
La IS (secuencia de inserción) IS6110 es un miembro de la familia IS3. Los miembros de esta familia son los grupos de IS bacterianas más ampliamente extendidas, siendo encontradas en más de 24 géneros de bacterias Gram positivas y Gram negativas (22). La mayoría de las cepas de M. tuberculosis tienen entre 8 y 15 copias del elemento de inserción IS6110 (22, 25). La especificidad y la naturaleza repetitiva del IS6110 lo hacen un objetivo ideal para la amplificación por RCP (26).
La secuencia de inserción IS6110 frecuentemente se encuentra en un locus único en el genoma del complejo M. tuberculosis denominado región Direct Repeat (DR) (22). La región DR consiste en secuencias repetitivas de 36pb separadas por espaciadores no repetitivos cuyas longitudes varían de 27 a 41 pb (22, 27). Esta región ha sido completamente secuenciada en M. tuberculosis. La gran mayoría de las cepas contienen uno o más elementos IS6110 en la región DR.
Herramientas de biología molecular, tales como Fingerprinting (RFLP: polimorfismo de longitud de fragmentos de restricción) o la técnica de Spoligotyping (28, 29) es altamente efectiva para tipificar las micobacterias y determinar los genotipos de los aislamientos clínicos. En tuberculosis humana la tipificación molecular se logra por el uso de la secuencia de inserción IS6110 (20, 22, 30, 31). En M.bovis, IS6110 es menos útil porque el genoma de la mayoría de las cepas contiene solo algunas copias de IS6110 (22, 32). Estudios han indicado que algunas cepas de M. tuberculosis pueden ser más fácilmente transmitidas y ultimadamente, más exitosas en la producción de infección y/o enfermedad (33).
Para incrementar nuestro conocimiento sobre la biología del bacilo tuberculoso, se ha determinado la secuencia genómica completa de la cepa más ampliamente utilizada, H37Rv (34). Análisis de bioinformática han llevado a la identificación de aproximadamente 4.000 genes en la secuencia genómica de 4.41 Mb y han provisto un gran aporte al desarrollo de la bioquímica, genética e inmunología de esta temida bacteria. La información de la secuencia genética de esta cepa está centralizada en TubercuList (http://www.pasteur.fr/Biol/Tu bercuList/) (34).
La gran ventaja de las reacciones de amplificación de ácidos nucleicos, sobre el cultivo es la velocidad con la cual el diagnóstico puede ser hecho. A diferencia de la microscopia, el Mycobacterium infectante puede ser directamente identificado.
Materiales y Métodos
Población
Se procesaron 31 muestras de esputo, de pacientes de ambos sexos, sin distinción de grupo étnico y edades comprendidas entre los 17 y 80 años, con manifestaciones clínicas, hallazgos radiológicos y/o datos epidemiológicos sugestivos de tuberculosis pulmonar. Las mismas se obtuvieron de individuos procedentes del área de hospitalización de Neumonología y de la consulta de tisiología del Hospital General del Sur de Maracaibo, el cual es Centro de Referencia Regional para el diagnóstico de esta enfermedad. Se excluyeron de este estudio individuos con diagnóstico establecido de tuberculosis o que estén recibiendo quimioterapia o profilaxia anti TBC.
Metodología
1. Examen directo: A todas las muestras, se les practicó examen microscópico directo (baciloscopia) utilizando la técnica de coloración de Ziehl-Neelsen. Para lo cual se tomó como referencia el Servicio de bacteriología del laboratorio del Hospital General del Sur de Maracaibo.
2. Cultivo: Se utilizó el medio de cultivo Ogawa-Kudoh como estándar para la siembra de las muestras de esputo. La siembra se realizó en el Laboratorio de HLA e Inmunogenética del Banco de Sangre de Maracaibo, en una campana de flujo laminar y utilizando el procedimiento de descontaminación de la muestra por el método de Kudoh. (35, 36).
Los cultivos se incubaron a 37 grados centígrados en estufa de incubación (Precision Scientific C.O) y con las tapas ligeramente flojas los dos primeros días y luego se cerraron.
Los cultivos se revisaron a los 4, 8, 15, 30 y 60 días y se hicieron registros de tiempo de crecimiento de los mismos.
No se practicó análisis e identificación bioquímica de los cultivos obtenidos.
3. Obtención de ADN a partir de esputo: Tanto la extracción del ADN como la amplificación del mismo, se practicó en el Laboratorio de HLA, Inmunología y Genética del Banco de Sangre del Estado Zulia. Las muestras fueron digeridas por el método N-acetil-L-cisterína-NaOH y concentradas por centrifugación. El ADN fue aislado de 1 ml de esputo por el método fenol-cloroformo (37).
4. Reacción en Cadena de la Polimerasa (RCP): Los objetivos para el ensayo de RCP fueron los genes que codifican el antígeno de 32-KDa (19) y la secuencia de inserción IS6110 (24). Para la amplificación del elemento de inserción IS6110, fueron desarrollados primers específicos (IS5 – IS6) utilizando el programa de computación Oligo versión 4.0. (38) IS5 (5´CGGAGACGGTGCGTAAGTGG) e IS6 (5´GATGGACCGCCAGGGCTTGC). También se desarrollaron primers específicos (MT1 y MT2) MT1 (5´TTCCTGACCAGCGAGCTGCCG) y MT2 (5´CCCCAGTACTCCCAGCTGTGC); para amplificar el gen que codifica el antígeno alfa 32-KDa (26) presente en todas las micobacterias descritas. Todas las reacciones se realizaron en un volumen final de 50 µl conteniendo 100 ng de ADN purificado, buffer de reacción 1 X, 2.5 U de Taq polimerasa, 0.2 mM de cada desoxynucleótido trifosfato, 15 pmol de IS5 – IS6, 10 pmol de MT1 – MT2. La reacción fue llevada a cabo en un termociclador automatizado MJ Research Inc. Las muestras fueron desnaturalizadas a 94°C por 5 minutos y se practicaron 35 ciclos de amplificación. Los ciclos consistieron en desnaturalización a 91°C por 1 minuto, alineación a 61°C por 90 segundos y extensión a 72°C por 2 minutos. Con una extensión prolongada de 10 minutos a 72°C. Se establecieron controles positivos y controles negativos de la reacción.
5. Verificación de la ampliación: Después de la amplificación, 10 µl de la mezcla la reacción fue analizada por electroforesis con gel de agarosa al 1%. Los productos amplificados fueron teñidos con bromuro de ethidio y fueron visualizados por transiluminación.
Resultados
Microscopía Directa:
De las 31 muestras de esputo examinadas por el método de Ziehl-Neelsen, 19 resultaron positivas (BK +) y 12 resultaron negativas (BK -). Lo cual representa un porcentaje de 61,29% y 38,71% respectivamente ( Tabla 1).
Tabla 1. Distribución de muestras de esputo. Baciloscopia.
| BK | Positivo % | Negativo % | Total % |
| Total | 19 (61.29) | 12 (38.71) | 31 (100) |
F.I.: Laboratorio de Bacteriología. Hospital General del Sur de Maracaibo.
Cultivo
Utilizando como estándar el medio de cultivo Ogawa – Kudoh, se sembraron todas las muestras de esputo. De las 31 muestras sembradas 19 (61,29%) resultaron positivas y 12 (38,71%) fueron negativas al cultivo (Tabla 2).
Tabla 2. Distribución de muestras de esputo. Cultivo.
| Cultivo | Positivo % | Negativo % | Total % |
| Total | 19 (61.29) | 12 (38.71) | 31 (100) |
F.I.: Laboratorio HLA e Inmunogenética. Banco de Sangre de Maracaibo
RCP
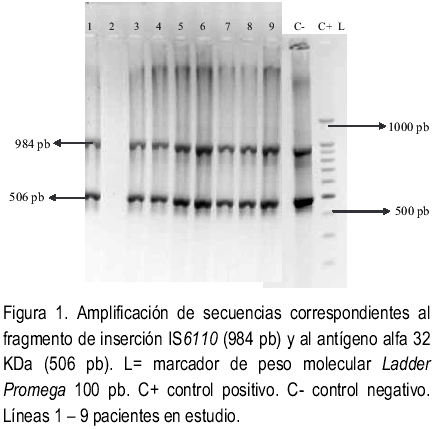
Se observaron fragmentos de 984 pares de bases y 506 pares de bases correspondientes a la secuencia de inserción IS6110 y al antígeno alfa 32 KDa respectivamente en 22 de las 31 muestras amplificadas (Fig. 1).
En la Tabla 3 se muestran los resultados del RCP. De todas las muestras amplificadas, 22 (70,96%) mostraron la presencia de fragmentos de ADN correspondientes tanto a la secuencia de inserción IS6110, como al antígeno alfa 32 KDa. Mientras que en 9 (29,04%) no se detectó la presencia de ADN micobacteriano por el método de RCP.
Tabla 3. Distribución de muestras de esputo. RCP.
| RCP | Positivo % | Negativo % | Total % |
| Total | 22 (70.96) | 09 (29.04) | 31 (100) |
F.I.: Laboratorio HLA e Inmunogenética. Banco de Sangre de Maracaibo.
Cuando se relacionaron los hallazgos obtenidos en la microscopía, cultivo y RCP, se obtuvieron los siguientes resultados plasmados en la Tabla 4.
Tabla 4. Relación Prueba BK y Cultivo.
|
| Cultivo Positivo | Cultivo Negativo | Total |
| BK Positivo | 18 | 1 | 19 |
| BK Negativo | 1 | 11 | 12 |
| Total | 19 | 12 | 31 |
F.I.: Laboratorio HLA e Inmunogenética. Banco de Sangre de Maracaibo.
En nuestro estudio se utilizó como estándar de oro el cultivo para determinar la sensibilidad y especificidad de la pruebas BK y RCP.
Del total de muestras estudiadas, se obtuvo 1 cultivo positivo proveniente de una baciloscopia negativa, el resto de los cultivos positivos (18), también fueron positivos a la baciloscopia. Sólo se obtuvo un cultivo negativo proveniente de una baciloscopia positiva, el resto de los cultivos negativos (11), también fueron negativos a la baciloscopia De esta manera, puede decirse que la sensibilidad del BK es del 94,74%, mientras que la especificad se estima en 91,67%. También es factible concluir que la capacidad predictiva de resultados positivos del BK respecto al cultivo es del 94,74% mientras que la capacidad predictiva de resultados negativos es de 91,67 %.
En forma análoga, al relacionar la prueba RCP con respecto al cultivo se tienen los resultados presentados en la Tabla 5.
Tabla 5. Relación Prueba Cultivo y RCP.
|
| Cultivo Positivo | Cultivo Negativo | Total |
| RCP Positivo | 19 | 3 | 22 |
| RCP Negativo | 0 | 9 | 9 |
| Total | 19 | 12 | 31 |
F.I.: Laboratorio HLA e Inmunogenética. Banco de Sangre de Maracaibo.
De las 22 amplificaciones positivas, 3 resultaron negativas al cultivo. Luego se puede concluir que la sensibilidad de la prueba RCP es del 100% y su especificidad es del 75%. Además, la capacidad predictiva positiva de la prueba RCP respecto al cultivo es de 86.4% mientras que su capacidad predictiva de resultados negativos es del 100%. A los mismos resultados se llega al relacionar los datos de la prueba RCP y la prueba de cultivo.
Discusión
La Tuberculosis, en los actuales momentos, sigue siendo un problema de salud pública a nivel mundial. Se requiere de un diagnóstico precoz y específico principalmente en las formas extrapulmonares, infección tuberculosa en niños infección micobacteriana en pacientes VIH y formas cerradas muy poco bacilíferas, en donde la microscopía y el cultivo tienen grandes limitantes. El rango de sensibilidad de la baciloscopia, oscila entre 50 – 80% y la especificidad es virtualmente del 100% (7). El cultivo es una técnica que tiene mayor sensibilidad (70-90%) (8). El aislamiento de las micobacterias por cultivo es entorpecido por su lento crecimiento. Un promedio de incubación de 4 semanas en medios convencionales se requiere antes que pueda ser detectado crecimiento. De tal manera que en casos en los que se requiera una toma de decisiones rápidas para instaurar una terapéutica efectiva su valor es muy limitado (7-8).
La RCP ha mostrado ser una herramienta muy útil para el diagnóstico de tuberculosis. En la actualidad se han desarrollado varios ensayos de RCP, para la detección de M. tuberculosis a partir de muestras de esputo mostrando diversos grados de sensibilidad y especificidad (15, 16, 39, 20, 21, 23, 40).
Nosotros realizamos un estudio prospectivo en donde nos propusimos evaluar la utilidad de la RCP para el diagnóstico de tuberculosis pulmonar, comparándola con las técnicas bacteriológicas habituales, como la baciloscopia y el cultivo.
Al analizar los resultados obtenidos entre la baciloscopia y el cultivo, encontramos que la sensibilidad de la baciloscopia fue del 94,74%, mucho mayor que la reportada en los trabajos publicados por Gording y colaboradores (7). Esto probablemente sea debido al hecho que los resultados de la baciloscopia dependen sustancialmente de la carga bacilar de la muestra y nuestros pacientes fueron seleccionados previamente por hallazgos clínicos y radiológicos sugestivos de tuberculosis pulmonar, por lo que es de esperar que las baciloscopias sean consistentemente positivas en estos pacientes. El mismo fenómeno se observó en el caso de los cultivos. Apreciamos una baciloscopia positiva con un cultivo negativo. Al estudiar la historia clínica de este paciente (No. 7) nos encontramos que se encontraba recibiendo tratamiento antituberculoso para el momento de la toma de la muestra. Por lo tanto este fenómeno puede deberse a la presencia de bacilos inviables o muertos que son incapaces de crecer en los medios de cultivos. Este paciente igualmente resultó positivo cuando se le practicó amplificación de ácidos nucleicos, ya que esta prueba detecta la presencia del ADN micobacteriano.
Encontramos que la sensibilidad de la RCP en nuestro estudio fue del 100% y la especificidad fue del 75%. Sin embargo algunos estudios reportan una sensibilidad relativamente más baja, como Andersen y cols. (39) y Cohen y colaboradores (16), quienes encontraron sensibilidades para sus ensayos de RCP de 63 y 73% respectivamente. La RCP es una técnica que requiere una estandarización minuciosa para evitar los problemas de la contaminación. Estas bajas sensibilidades encontradas por estos autores pudieran explicarse por condiciones subóptimas de la prueba. Los resultados de este estudio pueden compararse con los resultados obtenidos por Shah y colaboradores (18), quienes obtuvieron sensibilidades similares utilizando la prueba Amplicor de Roche.
Cuando comparamos los resultados de la amplificación de ácidos nucleicos con el con el cultivo observamos que la sensibilidad de la PCR fue mayor que las técnicas bacteriológicas tradicionales. Todos los pacientes con baciloscopias positivas y cultivos positivos, resultaron también positivos cuando se aplicaron las técnicas de amplificación de ácidos nucleicos. En tres pacientes con examen directo y cultivo negativo la RCP fue positiva. Por lo tanto la RCP puede considerarse una herramienta muy útil para el diagnóstico de tuberculosis pulmonar, con sensibilidades mayores que las pruebas bacteriológicas rutinarias.
Todas las amplificaciones positivas mostraron las bandas correspondientes a las secuencias objetivo: una banda de 506 pares de bases, que corresponde a la secuencia de bases que codifica a la proteína de 32 KDa, el cual está presente en todas las micobacterias (incluyendo Complejo M. tuberculosis y M. leprae) y que parece ser uno de los principales estimulantes de la respuesta celular y humoral contra las micobacterias (19), y otra banda de 984 pares de bases, que corresponde a los genes que codifican a la secuencia de inserción IS6110, el cual es específico para el Complejo M. tuberculosis, que incluye el mycobacterium tuberculoso humano y bovino (21-24, 40). Estos resultados coinciden con los realizados por Borremans y colaboradores (19) quienes lograron clonar la secuencia genómica de la proteína de 32 kDa de M. tuberculosis. En el mismo orden de ideas, nuestro estudio concuerda con los resultados alcanzados por Hermans (22), Kox (40), T. D. Mchugh (41), Thierry y colaboradores (24) quienes hallaron que la amplificación de la secuencia de inserción IS6110, a partir de muestras clínicas, permite identificar infecciones producidas por el Complejo M. tuberculosis, con mucha mayor sensibilidad y especificidad que el examen directo y el cultivo. Sin embargo recientes reportes han demostrado que existen algunas cepas de M. tuberculosis que no poseen la secuencia IS6110 en sus genomas (41,42). Así, el uso de métodos de RCP basados en IS6110 puede conducir a resultados falsos negativos. Por otra parte, métodos de RCP basados exclusivamente en la secuencia IS6110, no discriminan entre M. tuberculosis y otras micobacterias del Complejo M. tuberculosis (26,41).
En otras muestras clínicas la RCP ha mostrado resultados variables. En derrame pleural tuberculoso, la RCP tiene una sensibilidad de 20-80% (dependiendo de la secuencia genómica amplificada y el procedimiento utilizado en la extracción del ADN) y una especificidad de 78-100% (43). Un estudio español reciente, demostró un 81% de especificidad y una 98% de sensibilidad para la RCP cuando se utilizó un segmento de 126 pares de bases de M. tuberculosis, en la evaluación de derrame pleural (44). La RCP es positiva en el 100% de los cultivos positivos de líquido pleural y solo en 30-60% de los cultivos negativos de líquido pleural (43).
En el caso de meningitis tuberculosa, la RCP ha mostrado una sensibilidad del 75% y una especificidad del 94% en líquido cefalorraquídeo, cuando se utiliza la IS6110 como objetivo de amplificación (45, 46)
En el presente estudio, demostramos que el uso de las técnicas de amplificación de ácidos nucleicos, como la RCP, basadas en la amplificación simultánea del elemento de inserción IS6110 y del gen que codifica el antígeno alfa en un solo paso puede identificar infecciones por micobacterias a partir de muestras de esputo.
En conclusión, los resultados presentados demuestran que esta técnica es más sensible que los procedimientos bacteriológicos de rutina (examen directo y cultivo) y la convierten en una herramienta muy poderosa para el diagnóstico de tuberculosis pulmonar y otras enfermedades micobacterianas.
Según el reporte de MMWR 49(26):593-594,2000 (15), las pruebas de amplificación de ácidos nucleicos indudablemente aumentan la rapidez diagnóstica, pero ellas, hasta la actualidad, no reemplazan el examen directo del esputo o el cultivo micobacteriano, esencial para la identificación del microorganismo (las pruebas de amplificación sólo dan resultados para el Complejo M. tuberculosis), así como para la realización de pruebas de sensibilidad a antimicrobianos. Tampoco reemplazan el juicio clínico. Su especificidad es muy variable entre diferentes laboratorios. Además las pruebas de amplificación de ácidos nucleicos frecuentemente permanecen positivas después que los cultivos se hacen negativos durante la terapia y pueden permanecer positivas incluso después de finalizar la terapia antituberculosa. Por lo tanto las pruebas de amplificación de ácidos no sustituyen a las pruebas bacteriológicas tradicionales para el diagnóstico de la tuberculosis. No se recomienda utilizarlas en forma rutinaria para el diagnóstico de la enfermedad, sino dependiendo del contexto clínico, epidemiológico y radiológico del paciente. Parece ser que sus principales aplicaciones se presentan cuando las pruebas bacteriológicas tradicionales no establecen un diagnóstico concluyente, en el momento de tomar decisiones terapéuticas inmediatas (meningitis tuberculosas) y en los casos de tuberculosis infantil o asociada a SIDA.
Referencias Bibliográficas
(1) Torres C. Tuberculosis y otras micobacteriosis respiratorias. In: Velez H, Rojas W, Borrero J, Restrepo J. Fundamentos de Medicina. Neumología. 5ta. Edición. Medellín. Colombia. Editorial CIB 1998. p. 155-86. [ Links ]
(2) Guzmán-Pérez C, Vargas M. Does aging modify pulmonary tuberculosis? Chest; 1999;116: 961-967. [ Links ]
(3) World Health Organization. Fact Sheet No. 104. Revised March 2004. [ Links ]
(4) Pitchenik AE.Tuberculosis control and the AIDS epidemic in developing countries. Ann Intern Med 1990; 113: 89-90. [ Links ]
(5) Braum MM, Byers RH, Heyward WL et al. Acquired immunodeficiency syndrome and extra pulmonary tuberculosis in the United States. Arch Intern Med 1990; 150: 1913-1916. [ Links ]
(6) Dooley SW, Jarvis WR, Martone WJ, Snider Jr DE. Multidrug-resistant tuberculosis. Ann Intern Med 1992; 117: 257-259. [ Links ]
(7) Gording F, Slutkin G. The validity of acid fast smears in the diagnosis of pulmonary tuberculosis. Arch Path Lab Med 1990; 114: 1025-1027. [ Links ]
(8) Mioner H, Gebre N, Karlsson U et al.. Diagnosis of pulmonary tuberculosis. Lancet 1994; 344: 127. [ Links ]
(9) Anargyros P, Astill DJS, Lim ISL. Comparison of improved BACTEC and Lowenstein-Jensen media for culture of mycobacteria from clinical specimens. Clin Microbiol 1990; 28: 1288-1291. [ Links ]
(10) Middlebrook G, Reggiardo Z, Tigertt WD. Automatable radiometric detection of growth of Mycobacterium tuberculosis in selective media. Am Rev Resp Dis 1977; 115: 1066-1069. [ Links ]
(11) Sewell DL, Rashad AL, Rourke Jr WJ, Poor SL, McCarthy JAC, Pfaller MA.Comparison of the Septi-Chek AFB and BACTEC systems and conventional culture for recover of mycobacteria. J Clin Microbiol 1993; 31: 2689-2691. [ Links ]
(12) Welch DF, Guruswamy AP, Sides SJ, Shaw CH, Gilchrist MJR. Timely culture for mycobacteria with utilizes a microcolony method. J Clin Microbiol 1993; 31: 2178-2184. [ Links ]
(13) Erlich, H. A., ed. PCR Technology: Principles and application for DNA amplification. Stockton. New York. [ Links ]
(14) Wilson St, Mcnerney R, Nye Pm, Godfrey-Fausset PD, Stoker NG, Voller A.Progress toward a simplified polymerasa chain reaction and its application to diagnosis of tuberculosis. J Clin Microbiol 1993; 31: 776-782. [ Links ]
(15) CDC. Nucleic acid amplification tests for tuberculosis. 2000 49(26):593-594. [ Links ]
(16) Cohen RA; Muzaffar S; Schwartz D; Bashir S; Luke S; McGartland LP; Kaul K. Diagnosis of pulmonary tuberculosis using PCR assays on sputum collected within 24 hours of hospital admission. Am J Respir Crit Care Med 1998;157 (1):156-6. [ Links ]
(17) Linuma Y; Ichiyama S; Yamori S; Oohama J; Takagi N; Hasegawa Y; Shimokata K; Nakashima N. Diagnostic value of the Amplicor PCR assay for initial diagnosis and assessment of treatment response for pulmonary tuberculosis. Microbiol Immunol 1998;42(4):281-7 [ Links ]
(18) Shah S; Miller A; Mastellone A; Kim K; Colaninno P; Hochstein L; DAmato R. Rapid diagnosis of tuberculosis in various biopsy and body fluid specimens by the AMPLICOR Mycobacterium tuberculosis polymerase chain reaction test. Chest 1998; 113(5):1190-4 [ Links ]
(19) Borremans M; de Wit L; Volckaert G; Ooms J; de Bruyn J; Huygen K; van Vooren JP; Stelandre M; Verhofstadt R; Content J. Cloning, sequence determination, and expression of a 32-kilodalton-protein gene of Mycobacterium tuberculosis. Infect Immun 1989; Oct; 57(10):3123-30. [ Links ]
(20) Cousins DV, Wilton SD, Francis BR, Gow BL..Use of polymerasa chain reaction for rapid diagnosis of tuberculosis. J Clin Microibiol1992; 30: 255-258. [ Links ]
(21) Eisenach DK, Sifford MD, Cave MD, Bates JH, Crawfort JT., Detection of Mycobacterium tuberculosis in sputum samples using a polymerasa chain reaction. Am Rev Respir Dis 1991; 144: 1160-1163. [ Links ]
(22) Hermans, P. W., D. van Soolingen, J. W. Dale, A. R. Schuitema, R. A. McAdam, D. Catty, and J. D. van Embden. Insertion element IS986 from Mycobacterium tuberculosis: a useful tool for diagnosis and epidemiology of tuberculosis. J. Clin. Microbiol 1990; 28:2051–2058. [ Links ]
(23) Kolk AHJ, Schuitema ARJ, Kuijper S et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerasa chain reaction and a nonradioactive detection system. J Clin Microbiol 1992.; 30: 2567-2575. [ Links ]
(24) Thierry D, Brisson-Noel A, Vincent-Lévy-Frébault V, Nguyen S, Guesdon JL, Giequel B.Characterization of a Mycobacterium tuberculosis insertion sequence IS6110, and its application in diagnosis. J Clin Microbiol 1990; 28: 2668-2673. [ Links ]
(25) McAdam, R. A., P. W. Hermans, D. van Soolingen, Z. F. Zainudin, D. Catty, J. D. A. van Embden, and J. W. Dale. Characterization of a Mycobacterium tuberculosis insertion sequence belonging to the IS3 family. Mol.Microbiol 1990; 4:1607–1613. [ Links ]
(26) van Embden, J. D. A., M. A. Cave, J. T. Crawford, J. W. Dale, K. D. Eisenach, B. Gicquel, P. Hermans, et al.. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J. Clin. Microbiol 1993; 31:406–409. [ Links ]
(27) Groenen PM, Bunschoten AE, van Soolingen D, van Embden JD. Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method. Mol Microbiol 1993; Dec; 10(5):1057-65. [ Links ]
(28) Kamerbeek, J., L. Schouls, A. Kolk, M. van Agterveld, D. van Soolingen, S. Kuijper, A. Bunschoten, et al. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J. Clin. Microbiol 1997; 35:907–914. [ Links ]
(29) Molhuizen, H. O., A. E. Bunschoten, L. M. Schouls, and J. D. A van Embden. Rapid detection and simultaneous strain differentiation of Mycobacterium tuberculosis complex bacteria by spoligotyping. Methods Mol. Biol 1998; 101:381–394. [ Links ]
(30) Cave, M. D., K. D. Eisenach, G. Templeton, M. Salfinger, G. Mazurek, J. H. Bates, and J. T. Crawford. Stability of DNA fingerprint pattern produced with IS6110 in strains of Mycobacterium tuberculosis. J. Clin. Microbiol 1994; 32:262-266. [ Links ]
(31) Cave, M. D., K. D. Eisenach, P. F. McDermott, J. H. Bates, and J. T. Crawford. IS6110: conservation of sequence in the Mycobacterium tuberculosis complex and its utilization in DNA fingerprinting. Mol. Cell. Probes 1991;5:73-80. [ Links ]
(32) Van Soolingen, D., P. E. W. de Haas, J. Haagsma, T. Eger, P. W. M. Hermans, V. Ritacco, A. Alito, et al.. Use of various genetic markers in differentiation of Mycobacterium bovis strains from animals and humans and for studying epidemiology of bovine tuberculosis. J. Clin. Microbiol 1994;. 32:2425–2433. [ Links ]
(33) Galway, S. E., P.C. Sanchéz, T. F. Shinnick, I. Orme, T. Agerton, D. Hoy, J. S. Jones, et al. An outbreak involving extensive transmission of a virulent strain of Mycobacterium tuberculosis. N. Engl. J. Med 1998; 338: 663. [ Links ]
(34) Cole ST. What can be expected from sequencing of the Mycobacterium tuberculosis genome? Bull Acad Natl Med 1999; 183(7):41-50. [ Links ]
(35) Kudoh S, Kudoh T. A simple technique for culturing tubercle bacilli. Bull World Health Organ 1974; 51 (1): 71-82. [ Links ]
(36) Nava O.Eficacia del medio de cultivo Ogawa-Kudoh, para el diagnóstico bacteriológico de tuberculosis pulmonar. Tesis de Grado. 2001. Universidad del Zulia. Facultad de Medicina. [ Links ]
(37) Del Portillo, P., L. A. Murillo, and M. E. Patarroyo. Amplification of a species-specific DNA fragment of Mycobacterium tuberculosis and its possible use in diagnosis. J. Clin. Microbiol 1991; 29:2163–2168. [ Links ]
(38) Wojciech, R. 1992. OligoTM Version 4.0. National Biosciences, Inc., Plymouth, Minn. [ Links ]
(39) Andersen, A. B., S. Thybo, P. Godfrey-Faussett, and N. G. Stoker. Polymerase chain reaction for detection of Mycobacterium tuberculosis in sputum. Eur. J. Clin. Microbiol. Infect. Dis 1993. 12:922–927. [ Links ]
(40) Kox LFF, Rhienthong D, Medo Miranda A et al. A more reliable PCR for detection of Mycobacterium tuberculosis in clinical samples. J Clin Microbiol 1994; 32: 672-678. [ Links ]
(41) T.D. Mchugh, l. E. Newport, and s. H. Gillespie. IS6110 Homologs Are Present in Multiple Copies in Mycobacteria Other than Tuberculosis-Causing Mycobacteria. J Clin Microbiol 1997; 35: 1799-1771. [ Links ]
(42) Yuen, L.K. W., B. C. Ross, K. M. Jackson, and B. Dwyer. Characterization of Mycobacterium tuberculosis strains from Vietnamese patients by Southern blot hybridization. J. Clin. Microbiol 1993; 31:1615–1618 [ Links ]
(43) Ferrer J. Pleural tuberculosis. Eur Resp J 1997; 10: 942-947. [ Links ]
(44) Villegas W, labrada LA, Saravia NG. Evaluation of polymerase chain reaction, ADA and interferon- in pleural fluid for the differential diagnosis of pleural tuberculosis. Chest 2000; 18: 1355-64. [ Links ]
(45) Bonington, A., J. I. George-Strang, P. E. Klapper, S. V. Hood, W. Rubombora, M. Penny, R. Willers, and E. G.A. Wilkins. Use of Roche amplicor in early diagnosis of tuberculosis meningitis. J. Clin. Microbiol 1998; 36:1251-1254 [ Links ]
(46) Seth, P., G. K. Ahuja, N. Vijaya Bhanu, M. Behari, S. Bhowmik, S. Broor, L. Dar, and M. Chakraborty. Evaluation of PCR for rapid diagnosis of clinically suspected tuberculosis meningitis. Tubercle Lung Dis 1996; 77:293-388. [ Links ]












 uBio
uBio