Revista de la Sociedad Venezolana de Microbiología
versión impresa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.26 n.2 Caracas 2006
β-Lactamasas de Espectro Expandido en Enterobacterias aisladas
en Centros de Salud de Caracas
Luis Torresa, Vanesa Gagliottaa, Orlando Torresa, Minerva Beníteza,
Marisela Domíngueza, Raquel Pedrozab,*
aEscuela de Bioanálisis, Facultad de Medicina
bInstituto de Medicina Experimental, Facultad de Medicina
Universidad Central de Venezuela
Caracas - Venezuela
Resumen:
Las BLEEs son β-lactamasas producidas por una variedad de bacterias gramnegativas, que confieren resistencia a cefalosporinas de tercera y cuarta generación y aztreonam. Son principalmente producidas por K. pneumoniae y E. coli, aunque la naturaleza plasmídica de los genes que las codifican ha permitido una amplia diseminación a otros géneros bacterianos. Se realizó la detección fenotípica y molecular de BLEE a 224 aislados de Enterobacterias provenientes de ocho Centros de Salud de Caracas. El 91,1% de las cepas analizadas mediante el método de doble disco y el NCCLS 2004, fueron productoras de BLEE. El análisis de CIM para ceftazidime, cefotaxime, cefepime y aztreonam mostró una mayor proporción de cepas BLEE con actividad ceftazidimasa, compatibles con las familias SHV y/o TEM, y en menor proporción pertenecientes a la familia CTX-M. Los resultados del PCR basados en la amplificación de los genes blaSHV, blaTEM y blaCTXM, confirmaron el predominio de SHV-BLEE (72%) y CTX-M-BLEE (21,1%). Los carbapenemos con un 100% de sensibilidad. constituyen la mejor opción terapéutica. Esta investigación permitió detectar el tipo de BLEE circulante en ocho centros hospitalarios y es el primer reporte de la determinación de BLEE del tipo CTX-M, en nuestro país.
Palabras clave: β-lactamasas,
Extended-Spectrum β-Lactamases in Enterobacteriaceae isolated
in Health Centres of
Abstract:
The extended-spectrum ß-lactamases (ESBLs) are ß-lactamases produced by a variety of gram-negative bacteria that confers resistance to cephalosporins of third and quarter generation and aztreonam. They are most commonly produced by isolates of K. pneumoniae and E. coli, although the plasmidic nature of the genes that encode them has allowed a wide dissemination to other bacterial genera. A phenotypical and molecular detection of ESBLs were carried out to 224 Enterobacteria isolates from eight Health Centers of
Keywords: β-lactamases, bacterial resistance, SHV-ESBL, CTX-M-ESBL
Recibido 25 julio de 2006; aceptado 10 de septiembre de 2006
Introducción
La resistencia bacteriana a los antibióticos b-lactámicos e inhibidores de b-lactamasas constituye un problema clínico creciente en todo el mundo. En los bacilos gram-negativos el mecanismo más importante de resistencia es la inactivación por las b-lactamasas, las cuales hidrolizan el anillo b-lactámico de las penicilinas y cefalosporinas, inhibiendo así su actividad terapéutica [1].
Aunque se han descrito una gran variedad de b-lactamasas, las del tipo TEM y SHV son las observadas con mayor frecuencia entre miembros de la familia Enterobacteriaceae [2,3]. A partir de mutaciones ocurridas en los genes que codifican las TEM-1, TEM-2 y SHV-1 se originaron las b-lactamasas de espectro extendido (BLEE) [4,5].
Estas enzimas son producidas constitutivamente y transmitidas por plásmidos que pueden ser transferidos de un germen a otro. Los plásmidos que transportan BLEE son megaplásmidos (>100 KDa), que frecuentemente codifican resistencia a otros antimicrobianos, tales como aminoglicósidos, cloranfenicol, tetraciclinas y trimetoprim-sulfametoxazol. La susceptibilidad a las fluoroquinolonas es muy variable, de acuerdo con la cepa aislada y las tendencias de resistencia. El tratamiento de estos organismos multirresistentes es un verdadero reto terapéutico [5].
Los microorganismos productores de BLEE son responsables de numerosos brotes de infecciones a través del mundo y plantean cambios en las medidas para el control de las enfermedades infecciosas. Existen diferentes tipos de BLEE, es muy importante el conocimiento de su epidemiología para la utilización adecuada de los agentes antimicrobianos [6].
Se han reportado BLEE en cepas de K. pneumoniae con mayor frecuencia (70%) que en cualquier otro germen. Sin embargo, en los últimos años han sido detectadas en E. coli, P. mirabilis, Salmonella sp. y Shigella sp. Las b-lactamasas tipo AmpC son codificadas por genes localizados en los cromosomas y son comúnmente encontradas en Enterobacter cloacae, Citrobacter freundii, Morganella morganii, Providencia sp., Serratia marcescens y P. aeruginosa. Son inducidas por agentes b-lactámicos y no son afectadas por los inhibidores de b-lactamasas disponibles en la actualidad [7,8].
Los patrones de resistencia asociados con las enzimas AmpC y BLEE son similares, sin embargo, las AmpC usualmente confieren resistencia a las cefamicinas. Los organismos productores de AmpC pueden actuar como reservorios ocultos de BLEE, lo cual debe ser tomado en cuenta por los laboratorios clínicos, y así evitar reportar falsos negativos de BLEE [7].
Hoy en día existen mas de 340 BLEE entre las cuales se encuentran las de la familia CTX-M, OXA, PER, RAE, GES, BES y VEB; muchas de ellas no relacionadas con las TEM y SHV [8,9].
Existen una serie de factores que pueden conducir a una interpretación errónea de los resultados obtenidos en el laboratorio. Un aspecto importante en la resistencia asociada a las BLEE es el fenómeno de gradiente, según el cual, a medida que aumenta el inóculo, estas bacterias se hacen más resistentes. Igualmente la afinidad variable por los diferentes sustratos hace que muchas de estas enzimas sean susceptibles a cefalosporinas de tercera generación in vitro. Estos factores pueden dificultar la detección de las cepas productoras de BLEE porque afectan las Concentraciones Inhibitorias Mínimas (CIM) de los antimicrobianos, para estos gérmenes. Es importante detectar y examinar los patógenos productores de BLEE para evitar los fracasos terapéuticos clínicos [10] y así contribuir con la disminución del valor costo-beneficio generado por las infecciones bacterianas evitando su diseminación horizontal, especialmente en las Unidades de Cuidados Intensivos.
En nuestro país y en especial a nivel del área metropolitana son escasos los estudios que han permitido caracterizar en los aislamientos de Enterobacterias, los diferentes tipos de BLEE circulantes en nuestros centros hospitalarios. Desde 1999 hemos realizado diversos estudios epidemiológicos en cepas de Enterobacterias multiresistentes a los antibióticos y productoras de BLEE, aisladas en diferentes centros hospitalarios del área metropolitana [11,12,13]. En el presente trabajo se analizaron cepas de Enterobacterias productoras de BLEE, desde un punto de vista fenotípico y molecular con la finalidad de determinar las familias de b-lactamasas de espectro expandido predominantes en los Centros de Salud del área Metropolitana de Caracas.
Materiales y Métodos
Cepas Bacterianas
En este estudio se analizaron 224 cepas de Enterobacterias, presuntas productoras de BLEE, distribuidas de las siguientes manera: K pneumoniae (46%), K. oxytoca (0,9%), E coli (29,5%), P mirabilis (3,6%), Enterobacter spp. (12%), Citrobacter spp. (1,3%), S. sonnei (0,45%), Salmonella spp. (0,45%) y S marcescens (5,4%). Las muestras se aislaron de pacientes hospitalizados en los servicios de Terapia Intensiva de Adultos y Niños, Cirugía y Medicina Interna de las siguientes instituciones del área metropolitana: Hospital Universitario de Caracas, Hospital José Gregorio Hernández, Hospital Militar Dr. Carlos Arvelo, Maternidad Concepción Palacios, Policlínica Santiago de León, Hospital de Clínicas Caracas, Instituto Clínico
Pruebas de Susceptibilidad a Antimicrobianos e Identificación Fenotípica de BLEE
La detección fenotípica de BLEE se realizó mediante el método de doble difusión en disco [14] y el recomendado por
Determinación de
Aislamiento de ADN plasmídico
La naturaleza plasmídica de los determinantes de resistencia a BLEE se estableció mediante pruebas de conjugación en medio sólido y el aislamiento del DNA plasmídico se realizó mediante lisis alcalina [17], y almacenado a
Determinación molecular de BLEE
Al ADN plasmídico de las E. coli transconjugantes y de células completas [18,19,20,21] se les realizó un PCR, para determinar la presencia de genes que codifican para BLEE pertenecientes a las familias SHV, TEM, y CTX-M. Los productos de amplificación fueron separados por electroforesis en geles de agarosa 1%, en buffer TBE, teñidos con bromuro de etidio y la visualización del DNA se realizó en un equipo Gel Doc 2000Ò (BIORAD). La identificación de los determinantes que codifican para BLEE pertenecientes a la familia SHV, se realizó mediante un análisis de PCR-RFLP que consistió en la digestión del producto de amplificación con la enzima de restricción NheI [22], siguiendo las indicaciones de la casa comercial (Promega).
Resultados
Los resultados obtenidos de la distribución de 224 aislamientos de Enterobacterias provenientes de los ocho centros hospitalarios considerados se exponen en
Tabla 1. Distribución de Enterobacterias asiladas en ocho Centros de Salud de Caracas. 2001-2004.
| Microorganismo | N | % |
| K. pneumoniae | 104 | 46,4 |
| K. oxytoca | 2 | 0,9 |
| E. coli | 66 | 29,5 |
| Enterobacter spp. | 27 | 12,1 |
| S. marcescens | 12 | 5,4 |
| Citrobacter spp. | 3 | 1,3 |
| P. mirabilis | 8 | 3,6 |
| S. sonnei | 1 | 0,4 |
| Salmonella spp. | 1 | 0,4 |
| Total | 224 | 100 |
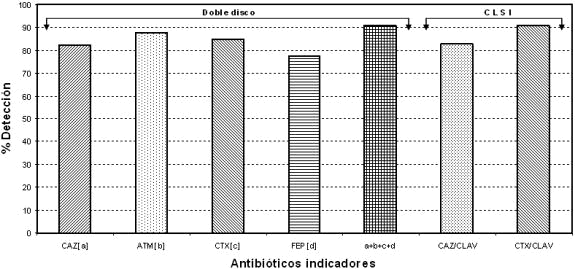
Para corroborar la producción de BLEE se aplicaron dos métodos fenotípicos. Al utilizar el de doble disco en 204 (91,1%) cepas del total se confirmó la producción de BLEE cuando se emplearon los antibióticos indicadores ATM, CTX, CAZ y FEP conjuntamente, mientras que al utilizar las drogas de manera individual la producción fue menor (Figura 1). Con el método según las Normas del CLSI, (Clinical Laboratory Standard Institute), se deben emplear las combinaciones de disco CTX/CLAV y CAZ/CLAV simultáneamente. Sin embargo, en el 91,1 % de las cepas se logró una mejor detección con la combinación CTX/CLAV, que cuando se utilizó CAZ/CLAV (82,6%), porque se confirmaron aquellas cepas (K. pneumoniae, E. coli, P. mirabilis), que mostraban una fuerte actividad cefotaximasa, las cuales frente a la combinación CAZ/CLAV, no generaron resultados positivos. Cabe destacar, que por ambos métodos en el mismo número de muestras se logro determinar la producción de BLEE.
Los resultados de las pruebas de susceptibilidad por categoría de interpretación (sensible, intermedio y resistente), realizadas frente a los 13 agentes antimicrobianos utilizados señalaron que las Enterobacterias presentan patrones complejos de resistencia múltiple. En
Figura 1. Distribución de la detección fenotípica de BLEE en 224 Enterobacterias aisladas en ocho Centros de Salud de
CAZ: Ceftazidime; ATM: Aztreonam; CTX: Cefotaxime; FEP: Cefepime;CLAV: Ácido clavulánico.
El análisis de los niveles de resistencia (CIMs) proporcionó información cuantitativa de los niveles de resistencia para CAZ, CTX, FEP y ATM e indico la prevalencia de BLEE. Sí
En el presente estudio el 67,6% de las cepas productoras de BLEE presentaron resistencia a CAZ con una CIM ≥32 mg/ml, y un 77,9% al ATM (CIM ≥32 mg/ml). Este comportamiento sugiere la presencia de BLEE de la familia SHV. Por otra parte un 24,1% de las cepas presentan alto nivel de resistencia al cefotaxime (CIM ≥ 128 mg/ml), y de estas un 17,2% muestran CIM ≥ a 16 mg/ml para el cefepime, lo que presume la existencia de BLEE tipo CTX-M.
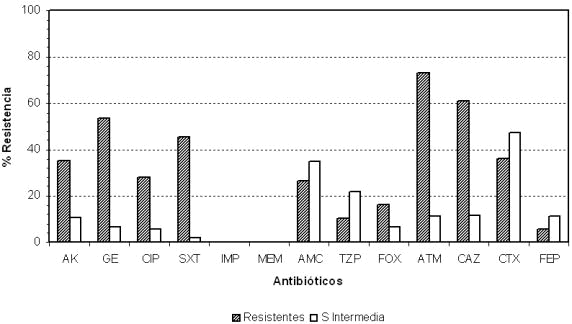
Figura 2. Resistencia (%) de las Enterobacterias aisladas en ocho Centros de Salud de Caracas. 2001-2004.
AK: Amikacina; GE: Gentamicina; CIP: Ciprofloxacina, SXT: Trimetropim/sulfametasol, IMP: Imipenem, MEM: Meropenem,
También se evaluó la transmisibilidad de los determinantes de resistencia a BLEE asociados a plásmidos. De 80 aislados clínicos que mostraron los marcadores fenotípicos diferenciales para realizar la conjugación, en 69 (86,3%) se observó la presencia de determinantes de resistencia ligados a plásmidos transferibles a cepas de E. coli K12. Para verificar que varios genes de resistencia fueron transferidos conjuntamente se reaislaron colonias de cada tipo de transconjugantes, en placas de medios selectivo con antibiótico y se determinó la presencia de marcadores. Los resultados obtenidos indican, que además, de la transferencia de genes que codifican BLEE, se observó una alta cotransferencia de determinantes de resistencia a otras familias de antibióticos como los aminoglicósidos, cotrimoxazol, etc.
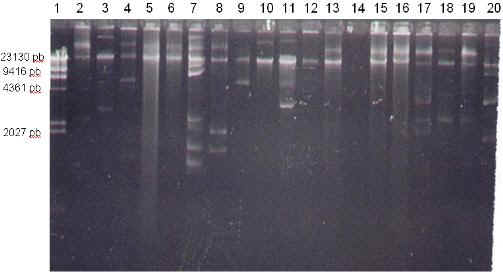
El análisis del ADN plasmídico realizado en las cepas de E coli transconjugantes mediante electroforesis en geles de agarosa, reveló la presencia de una banda común, con un tamaño mayor a 23000 pb, lo cual sugiere la presencia de plásmidos de alto peso molecular. (Figura 3). En la mayoría de las cepas transconjugantes se observo la presencia de varias bandas. Asumiendo que cada banda corresponde a un plásmido distinto se puede inferir, que en la mayoría de las cepas coexisten dos o más moléculas plasmídicas cuyos tamaños oscilan entre 2 y ≥20 Kpb.
En las 204 cepas BLEE confirmadas se realizaron ensayos de PCR con iniciadores específicos para los genes blaSHV, blaTEM y blaCTX-M para determinar las familias de BLEE circulantes entre ellas.
Figura 3. Aislamiento del ADN plasmídico obtenido de las cepas transconjugantes en gel de agarosa al 0,7 %.
Carriles: 1) λHindIII. 2) pHUC-0133. 3) pHUC-0102. 4) pHUC-0134. 5) pHUC-0135. 6) pHUC-0136. 7) pHUC-0108.
8) pHUC-0109. 9) pHUC-0110. 10) pHUC-0111. 11) pHUC-0112. 12) pHUC-0138. 13) pHUC-0115. 14) pHUC-0116.
5) pHUC-0117. 16) pHUC-0118. 17) pHUC-0119. 18) pHUC-0120. 19). pHUC-0121. 20) pHUC-0122.
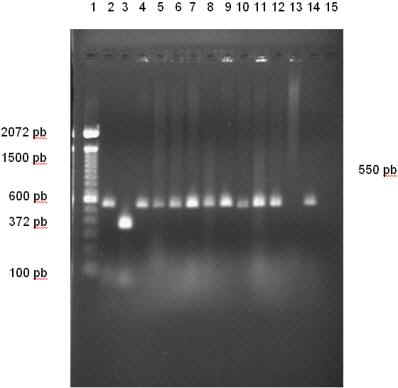
Los resultados obtenidos en el PCR señalan que en 146 (71,6%) aislados se evidenció la presencia de genes bla pertenecientes a la familia SHV (Figura 4), lo cual indica una alta prevalencía de esta familia y corrobora los análisis fenotípicos realizados. La mayoría de BLEE-SHV fueron encontradas en K. pneumoniae, sin embargo, también se reportaron en E. coli y Enterobacter spp. Con menor frecuencia (21,1%) se encontraron cepas con genes bla de la familia CTX-M. Estos aislados presentan altos niveles de resistencia a CTX y resistencia o susceptibilidad intermedia al FEP. Este comportamiento es característico de las enzimas pertenecientes a la familia CTX-M (Figura 5). Este resultado constituye el primer reporte de BLEE-CTX-M-1 en Venezuela. Además, se evidenció la combinación blaSHV blaCTX-M en 15 (7,4%) aislados.
Debe destacarse que en 52,5% de las cepas estudiadas se encontraron, además, genes bla de la familia TEM, los cuales permiten identificar tanto a las enzimas del tipo BLEE como las BLEA, por lo tanto, es necesario realizar estudios de secuenciación, que permitan determinar si se trata de BLEE derivadas de TEM-1 o TEM-2; o de enzimas del tipo BLEA. En este último caso, el fenotipo BLEE estaría determinado por el gen perteneciente a la familia SHV.
Las bacterias que producen BLEE tipo SHV, presentan generalmente una mutación en el gen blaSHV, con la sustitución de glicina en la posición 238 por serina. Esta mutación crea un sitio de reconocimiento (G x CCTAGC) para la endonucleasa NheI.
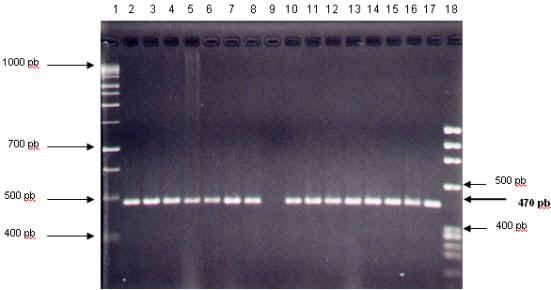
El análisis del patrón de restricción utilizando la enzima NheI mediante PCR-RFLP corroboró en 125 (61,3%) aislados la presencia de la mutación G®A en la posición 238 del gen SHV, lo cual es característico de los subtipos SHV-2, SHV-2ª, SHV-5, SHV-7, SHV-9, SHV-11, SHV-15 y SHV-34 (Figura 6). En los carriles del 2 al 7 y del 10 al 17, se observa tanto en las células completas como en su correspondientes ADN plasmídico, un producto de amplificación de 470 pb, el cual coincide con el control positivo (K. pneumoniae M-1740), portador de una BLEE tipo SHV-5.
La correlación entre los ensayos fenotípicos y moleculares permiten confirmar la importancia que tiene determinar
Figura 4. Distribución de genes bla en 204 cepas de Enterobacterias productoras de BLEE, aisladas en
Figura 5. Electroforesis del producto de amplificación de Enterobacterias empleando
Carriles: 1) Marcador de PM DNA ladder 100 pb. 2) Cepa BLEE CTX-M-1 (Control positivo).
3) E. coli ATCC 259222 (Control positivo). 4) K. pneumoniae 2105. 5) E. coli 3711. 6) E. coli 3811.
7) E. coli 3911. 8) E. coli 4111. 9) K. pneumoniae 4211. 10) E. coli 4311. 11) E. coli 4412.
12) K. pneumoniae 4512. 13) E. coli ATCC 25922 (Control negativo). 14) S. sonnei 5912.
15) Control negativo de corrida.
Discusión
Los microorganismos productores de BLEE son responsables de numerosos brotes infecciosos a nivel mundial, ofreciendo una situación de permanente cambio en la susceptibilidad de los microorganismos causantes de infecciones.
Figura 6. Electroforesis en gel de agarosa al 1%, de los productos de amplificación de células completas y el ADN plasmídico, empleando los genes blaSHV.
Carriles: 1) Marcador de PM DNA Ladder 1 Kb. 2) HUC-0104. 3) pHUC-0104. 4) HUC-0106. 5) pHUC-0106.
6) HUC-0113. 7) pHUC-0113. 8) HUC-0116. 9) pHUC-0116. 10) HUC-0124. 11) pHUC-0124. 12) HUC-0137.
13) pHUC-137. 14) HUC-0148. 15) pHUC-0148. 16) HUC-0150. 17) pHUC-0150. 18) Marcador de PM BenchtopHae II.
En esta investigación el análisis de 224 cepas de Enterobacterias sospechosas de producir BLEE, mediante dos métodos fenotípicos, demostró que el 91,1% de las cepas son productoras de BLEE. El método de doble disco [14], utilizando CAZ, ATM, CTX más el disco de cefepime, ofrece la ventaja de evidenciar claramente la producción de BLEE en los géneros de Enterobacterias (Enterobacter sp., Citrobacter spp., Serratia sp.), que portan la β-lac-tamasa cromosómica tipo AmpC, para las cuales las normas del CLSI no han establecido estándares de detección. Además, se puso en evidencia que el uso adecuado de los puntos de corte establecidos trae la ventaja, de poder detectar un alto porcentaje de cepas productoras de BLEE.
Los altos índices de BLEE son marcadores clínicos importantes que deben conducir a la toma de decisiones para intervenir y prevenir los elevados índices de morbimortalidad que los gérmenes productores de BLEE pueden ocasionar en pacientes intrahospitalarios [10].
La identificación de BLEE es un desafío que los laboratorios de todo el mundo enfrentan, debido a que los métodos para vigilar la disminución en la susceptibilidad a las oximinocefalosporinas, algunas veces no son los suficientemente sensibles para detectar los organismos productores de BLEE, especialmente en microorganismos diferentes de K. pneumoniae y E. coli [25].
El análisis de las pruebas de susceptibilidad indicó que frente al cefepime, las cepas muestran un bajo porcentaje de resistencia (5,8%) y una susceptibilidad intermedia de 11,2%. Sin embargo, debido a que las BLEE sufren del efecto inóculo, en aquellas infecciones que cursan con inóculos bacterianos elevados, el cefepime puede ser hidrolizado con mayor afinidad y velocidad, conduciendo a un fracaso terapéutico. Es por ello, que la evidencia fenotípica de la presencia de BLEE por cualquier metodología, implica extrapolar la resistencia al cefepime, independientemente de los resultados de sensibilidad obtenidos in vitro. En esta investigación, el porcentaje global de resistencia para las cefalosporinas de tercera, cuarta generación y al aztreonam fue de 91,1%. Los carbapenemos se mantienen como la mejor opción terapéutica para los microorganismos productores de BLEE. Se observó un 100% de sensibilidad para imipenem y meropenem, sin embargo, el uso indiscriminado de los carbapenemos puede conllevar a cambios en los patrones de sensibilidad de la población bacteriana frente a estos antibióticos. Con respecto a las combinaciones que usan inhibidores de β-lactamasa, la piperacilina/tazobactam, fue la que mostró un menor porcentaje de resistencia (5,4%), valores que se relacionan con los reportes internacionales, que destacan que la piperacilina/tazobactam, es una buena opción terapéutica para productores de BLEE [5,26], inclusive el tazobactam es considerado como el mejor inhibidor de las BLEE tipo CTX-M [2,6,21,25]. Sin embargo, cabe destacar, que todas las combinaciones de β-lactamicos + inhibidores de β-lac-tamasa sufren del efecto inóculo, por lo tanto, se debe evaluar adecuadamente el tipo de infección en donde pueden ser empleados, para evitar el riesgo de fracaso terapéutico.
Los resultados obtenidos en esta investigación demostraron que un 27,7% de las cepas fueron resistentes a ciprofloxacina, generalmente, la resistencia a las fluoroquinolonas surge en forma espontánea por medio de mutaciones puntuales que resultan en sustituciones de aminoácidos dentro de las subunidades GyrA, GyrB de ADN girasa y ParC o ParE de topoisomerasas IV, disminución de la expresión de la porina de membrana externa o sobrexpresión de bombas de salida para drogas múltiples. Además, la aparición reciente del gen qnr responsable de la resistencia a quinolonas mediada por plásmidos, puede producir la transferencia horizontal de la resistencia entre cepas. Es posible que la adquisición de resistencia a fluoroquinolonas y la producción de BLEE sea un proceso multifactorial. Es necesario evitar el uso excesivo de quinolonas tanto en seres humanos como en medicina veterinaria para impedir el incremento en la aparición de patógenos bacterianos zoonóticos y no zoonóticos resistentes, que podrían provocar infecciones en seres humanos o animales [5,26].
Los resultados fenotípicos y moleculares obtenidos demostraron que muchas Enterobacterias productoras de AmpC codifican concomitantemente genes BLEE, a nivel plasmídico, lo cual señala, que estas enzimas deben ser investigadas en todos los gérmenes y no solo en E. coli, Klebsiella spp. y P. mirabilis.
La presencia de plásmidos de alto peso molecular en aislados provenientes de los diferentes centros hospitalarios sugiere que la presión ejercida por el uso de antibióticos en áreas restringidas como las UTI, han favorecido la diseminación y prevalencia no sólo de plásmidos transferibles, sino también la presencia de otros elementos genéticos móviles como transposones e integrones que codifican altos niveles de resistencia asociados con varias familias de antibióticos [11,12,13]. Este estudio constituye una evidencia de la transferencia horizontal de genes de BLEE y co-transferencia de otros genes de resistencia a grupos de antibióticos diferentes de los β-lactámicos, entre cepas de bacilos Gram negativos aisladas en Centros de Salud de Caracas.
Del grupo de microorganismos analizados, el mayor porcentaje de las cepas productoras de BLEE correspondió a K. pneumoniae y E. coli, y pertenecen fundamentalmente a las familias SHV y CTX-M. Si bien es cierto que hay un predominio de las BLEE con actividad ceftazidimasa (familia SHV), el porcentaje de las BLEE con actividad cefotaximasa (familia CTX-M) muestra un incremento y se detectó en todos los centros hospitalarios estudiados. Estos tipos particulares de β-lactamasas (SHV-2, SHV-5 y CTX-M), también se han encontrado en aislamientos clínicos en diversas partes del mundo. (Argentina, Chile, Colombia, Uruguay, Francia, Japón, Bélgica, etc.) [6,27,28].
Los resultados fenotípicos fueron corroborados con los estudios genéticos de PCR, y se confirmó que los genes blaSHV amplificados correspondieron a una BLEE de esta familia, ya que al realizar un RFLP del producto de PCR con la enzima NHeI, éste fue positivo. Esto sólo ocurre si el gen blaSHV-
En los últimos años se ha visto con preocupación tanto en los hospitales públicos como privados un aumento de las bacterias productoras de BLEE, particularmente K. pneumoniae entre el 20% y el 40%, este porcentaje puede variar según el tipo de hospital, la región geográfica, la complejidad de las infecciones que allí se traten y el perfil de antibióticos utilizados [10,28].
Las bacterias han desarrollado, en su proceso evolutivo, formas cada vez más eficaces para evadir los puntos de acción del antibiótico [24]. Uno de los retos más importantes que encaran los laboratorios de microbiología es la capacidad de detectar, a tiempo, la resistencia mediada por BLEE, debido a las implicaciones clínicas y terapéuticas que pueden generar al limitar la utilidad de los antibióticos disponibles y estimular el uso de antibióticos de mayor espectro [25,29,30,31].
Los resultados obtenidos en este estudio nos permiten concluir que las cepas de Enterobacterias aisladas producen un complejo de β-lactamasas de varías familias enzimáticas, lo que podría explicar los elevados niveles de resistencia de estos microorganismos a β-lactámicos y aminoglicósidos, dificultando el tratamiento de las infecciones que ellas ocasionan. Además, nos permite aportar la primera evidencia de identificación de BLEE tipo CTX-M en Venezuela.
Al personal de los laboratorios de Bacteriología de los centros hospitalarios que gentilmente donaron las cepas de Enterobacterias utilizadas en este trabajo.
Al Lic. Oswaldo Travieso por su valiosa colaboración en la elaboración de los gráficos y revisión del manuscrito.
Esta investigación fue financiada por el Consejo de Desarrollo Científico y Humanístico (CDCH), de
2. Du Bois SK, Marriott MS, Amyes SG. TEM- and SHV-derived extended-spectrum beta-lactamases: relationship between selection, structure and function. J Antimicrob Chemother 1995; 35:7-22.
3. Jacoby GA, Medeiros AA, O´Brien TF, Pinto ME, Jiang J. Broad-spectrum transmisible beta-lactamases. N Engl J Med 1989; 319:723-4.
4. Winokui PL, Canton R, Casellas, JM, Legakis N. Variations in the prevalence of strains expressing an extended spectrum beta-lactamase phenotype and characterization of isolates from Europe, the Americas, and the Western Pacific region. Clin Infect Dis 2001; 32: Suppl 2:S94-103.
5. Paterson DL, Ko WC, Von Gottberg A, Casellas JM, Mulazimoglu L, Klugman KP, y col. Outcome of cephalosporin treatment for serious infections due to apparently susceptible organisms producing extended-spectrum beta-lactamases: implications for the clinical microbiology laboratory. J Clin Microbiol 2001; 39:2206-12.
6.
7. Pitout JD, Reisbig MD, Venter EC, Church DL,
8. Melano R, Corso, A, Petroni A, Centron D, Orman B, Pereyra A y col. Multiple antibiotic-resistance mechanisms including a novel combination of extended-spectrum beta-lactamases in a Klebsiella pneumoniae clinical strain isolated in Argentina. J Antimicrob Chemother 2003; 52:36-42.
9.
10. Martínez P, Mercado M, Máttar S. Determinación de b-lactamasas de espectro expandido en gérmenes nosocomiales del Hospital San Jerónimo, Montería. Colomb Med 2003; 34:196-205.
11. Torres L, Benítez M, Torres O, Gagliota V, Calvo A, Rodríguez N, y col. Detección de integrones clase I en cepas de Enterobacterias productoras de b-lactamasas de espectro expandido tipo SHV y CTX grupo 2. VITAE 2005; 25 Academia Biomédica Digital.
12. Torres L, Marcano D, Ramírez A, Rivero N, Pedroza R. Detección de b-lactamasas de espectro expandido en cepas de Enterobacterias aisladas de pacientes del Hospital Universitario de Caracas. XVIII Jornadas Venezolanas de Microbiología, Barquisimeto, Venezuela. 2002.
13. Torres L, Velázquez O, Pedroza R, Rodríguez-Lemoine V. Transferencia plasmídica de b-lactamasas de espectro expandido (ESbLs) en Enterobacterias aisladas de infecciones nosocomiales. XXV Jornadas Venezolanas de Microbiología, Valencia, Venezuela. 1999.
14. Jarlier V, Nicolas G, Fournier G, Philippon A. Extended broad spectrum b-lactamases conferring transferable resistance to never b-lactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis 1988; 10:867-78.
15. National Committee for Clinical Laboratory Standards (NCCLS). Performance Standards for Antimicrobial Susceptibility Testing; Twelfth Informational Supplement, January. 2002. Vol. 22 N° 1.
16. Cormican MG, Marshall SA, Jones RN. Detection of extended-spectrum b-lactamases (ESBLs) producing strains by the Etest ESBL screen. J Clin Microbiol 1996; 34:1880-4.
17. Birboim HC, Doly A. A rapid alkaline extraction procedure for screening recombinant DNA. Nucleic Acid Res 1979; 7:1511-23.
18. Chanawong A, M´Zali F, Heritage J, Lulitanond A, Hawkey P. Characterization of extended-spectrum b-lactamases of the SHV family using a combination of PCR-single strand conformational polymosfism (PCR-SSCP) and PCR-restriction fragment lenght polymorfism (PCR-RFLP). FEMS Microbiol Lett.2000; 184:85-9.
19. Petroni A, Corso A, Melano R, Cacace M, Bru A, Rossi A. Plasmidic extended-spectrum b-lactamases in Vibrio cholerae O1 E1 Tor isolates in Argentina. Antimicrob Agents Chemother 2002; 46:1462-8.
20. Mabilat C, Goussard S, Sougakoff R, Spencer C. Direct sequencing of the amplified structural gene and promotor for the extended-broad-espectrum b-lactamase TEM-9 (RHH-1) of Klebsiella pneumoniae. Plasmid 1990; 23:27-34.
21. Baurfeind A, Stemplinger I, Jungwirth R, Ernst S, Casellas JM. Sequences of b-lactamase gene encoding CTX-M-1 (MEN-1) and CTX-M-2 and relationship of their amino acid sequences with those of other b-lactamases. Antimicrob Agents Chemother 1996; 40:509-13.
22.
23. Liang Yu W, Pfaller MA, Winokur PL, Jones RN. Cefepime MIC as a predictor of the extended-spectrum b-lactamases type in Klebsiella pneumoniae,
24. Crespo María del Pilar. La resistencia bacteriana ¿estamos preparados para detectarla? Asociación Colombiana de Infectología 2005; 9:32-45.
25. Puerta H, Cantillo C, Consuegra C, Coronel W, Alvis N, Mattar S. Capacidad de los laboratorios de microbiología clínica de Cartagena para detectar microorganismos productores de betalactamasas de espectro expandido. Infectio 2005; 9(3):123-30.
26.
27. Bonnet R. Growing group of extended spectrum lactamases the CTX-M enzymes Antimicrob Agents Chemother 2004; 48:1-14.
28. Villegas MV, Correa A, Parez F, Zuluaga T, Radice M, Gutkind G, Casellas JM y col. CTXM-12 lactamase in Klebsiella pneumoniae Clinical Isolate en Colombia. Antimicrob Agents Chemother 2004; 48:629-31.
29. Galán J, Baquero M, Morosini M, Baquero F. Bacterias con alta tasa de mutación: los riesgos de una vida acelerada. Infectio 2006; 10(1):22-9.
30. Sirot D. Extended-spectrum plasmid-mediated b-lacta-mases. J Antimicrob Chemother 1995; 36:19-34.
31.
* Correspondencia:
E-mail: raquelpedroza@cantv.net












 uBio
uBio