Revista de la Sociedad Venezolana de Microbiología
versión impresa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.29 n.1 Caracas jun. 2009
Alcances y limitaciones de los métodos de epidemiología molecular basados en el análisis de ácidos nucleicos
Glenda Vílchez a, Guillermina Alonso b,*
a Escuela de Medicina Luis Razetti, Cátedra de Bioquímica, Facultad de Medicina
b Laboratorio de Biología de Plásmidos, Instituto de Biología Experimental, Facultad de Ciencias Universidad Central de Venezuela Caracas - Venezuela
* Correspondencia: E-mail: guillermina.alonso@ciens.ucv.ve
Resumen
La Epidemiología es la rama de la ciencia que se dedica al estudio del origen, la distribución y el control de las enfermedades que afectan a las poblaciones. La Epidemiología Molecular es una nueva rama de la ciencia en la cual se implementan técnicas moleculares en los estudios epidemiológicos. Diversos métodos de genotipificación molecular pueden ser empleados para clasificar a los microorganismos en grupos estrechamente relacionados o divergentes. Entre los métodos de genotipificación más usados están: la electroforesis de campo pulsado, la prueba de PCR, la secuenciación del genoma y la hibridación con sondas de DNA. Cada técnica ha ofrecido una alternativa para la investigación epidemiológica; sin embargo, también tienen aplicabilidades limitadas. El presente trabajo tiene como objetivo la revisión de las fortalezas y debilidades de éstas técnicas moleculares utilizadas para la genotipificación.
Palabras clave: Epidemiología, genotipificación, PFGE, PCR, secuenciación, arreglos de DNA
Scope and limitations of molecular methods applied to epidemiological studies
Abstract
Epidemiology is a science dedicated to the study of the origin, distribution and control of diseases that affect populations. Molecular Epidemiology is a new branch of science that uses molecular techniques in epidemiological studies. Several molecular genotyping methods can be used to classify microorganisms in closely related or divergent groups. Pulsed-field gel electrophoresis, the PCR test, genomic sequencing, and DNA probe hybridization are the most used techniques in genotyping. Each technique offers an alternative for epidemiological investigation; nevertheless, they also have limited applications. The objective of this work was to review the strengths and weaknesses of these molecular techniques used for genotyping.
Key words: Epidemiology, genotyping, PFGE, PCR, sequencing, DNA arrangements
Recibido: 23 de abril de 2008; Aceptado: 24 de abril de 2009
Introducción
La Epidemiología es una rama de la ciencia que se dedica al estudio de las enfermedades y los factores de riesgo que afectan a una población humana, su distribución y control [1]. El conocimiento derivado de los estudios epidemiológicos permite determinar la historia natural de las enfermedades, es decir, su etiología, frecuencia de aparición, distribución, vías y patrón de diseminación, reservorios o factores que incrementen el riesgo de contraerla. Adicionalmente, ofrece una alternativa para establecer mecanismos que permitan prevenir o controlar la enfermedad, así como la eficacia de dichas medidas y la magnitud del beneficio al aplicarlas sobre la población [2,3]. La Epidemiología Molecular es una disciplina que permite abordar los estudios a través de la utilización de técnicas moleculares, combinando la epidemiología analítica con métodos avanzados de laboratorio [4].
A través de la epidemiología molecular se ha incrementado el conocimiento sobre las bacterias, virus y parásitos causantes de enfermedades infecciosas. Se ha convertido en una disciplina importante en la investigación de este tipo de enfermedades, lo que implica un mejor conocimiento de su distribución y la posibilidad de diagnosticarlas con mayor rapidez y ofrecer un mejor tratamiento.
En el presente trabajo, se considerarán los alcances y limitaciones de los métodos moleculares empleados en estudios sobre epidemiología molecular haciendo especial énfasis en los resultados reportados para enfermedades infecciosas de origen bacteriano.
Métodos de tipificación: criterios de evaluación
En los estudios epidemiológicos, la identificación y caracterización de los microorganismos es indispensable. La suma de estos procedimientos se denomina tipificación, y permite establecer la identidad de los microorganismos causantes de un brote infeccioso, contribuyendo a la determinación de la fuente de la infección y de los patrones de diseminación. Además, facilitan la evaluación de las medidas de control, mediante la determinación de la prevalencia, en tiempo y espacio, del agente infeccioso [2].
La técnica de tipificación a emplear dependerá de los requerimientos y características del sistema analizado, sin embargo, cualquiera sea el método de tipificación, éste debe ser evaluado en cuanto a su capacidad para generar la información epidemiológica requerida. Struelens en 1998, establece que la pertinencia y efectividad de un método de tipificación podrá ser evaluada mediante los siguientes criterios: 1) Capacidad de identificación: proporción de aislados que pueden ser detectados por el método y asignados a un tipo particular. Idealmente, un método de detección debe permitir la identificación y tipificación de la totalidad de los aislados analizados. 2) Reproducibilidad: capacidad del procedimiento para arrojar el mismo resultado repetidas veces. 3) Estabilidad: el marcador biológico detectado por el método deberá poder ser determinado a lo largo del tiempo y no depender, de forma importante, de las variaciones en el genoma del microorganismo o de su patrón de expresión genética. 4) Poder discriminatorio: permite ubicar, con una probabilidad elevada, a individuos distintos en grupos diferentes. 5) Concordancia epidemiológica: capacidad del método para arrojar resultados similares a los obtenidos a través de otras técnicas. 6) Eficacia: estudia el balance entre los costos económicos generados por la aplicación del método y las ganancias obtenidas por la población al lograr la prevención y control de la enfermedad. Es de esperar que un método epidemiológico permita establecer una buena relación entre los logros obtenidos y el esfuerzo realizado, tanto a nivel económico como en recursos y tiempo empleado [2,5].
A esta lista de criterios deben añadirse la facilidad en la aplicación del método, la rapidez en la obtención de los resultados, la sencillez en su interpretación y la versatilidad del procedimiento para su aplicación a diversos organismos. En la práctica, para la evaluación de un método de tipificación epidemiológico deben considerarse todos los criterios descritos de forma integral. Rara vez un método cumple satisfactoriamente con todos los criterios utilizados para su evaluación, por lo cual, es frecuente y recomendable el uso combinado de diversas técnicas. Usualmente, en primera instancia, se utiliza un método de tipificación que arroje resultados rápidamente, seguido de procedimientos que ofrezcan información mas precisa, pero cuya implementación requiera de mayor tiempo [2].
Métodos moleculares para tipificación de microorganismos: fenotípicos vs. genotípicos
Los métodos para tipificación de microorganismos pueden clasificarse en dos grandes grupos: fenotípicos y genotípicos. Los métodos fenotípicos se basan en la determinación de características bioquímicas y/o fisiológicas e históricamente, constituyeron la primera herramienta que permitió la comparación de microorganismos [6,7]. Incluyen la determinación de actividades enzimáticas, capacidades metabólicas, determinantes antigénicos o susceptibilidad a agentes bactericidas, entre otros [8]. Las técnicas de tipificación genotípicas involucran el estudio del genoma del microorganismo causal de la enfermedad, permitiendo analizar propiedades, características o polimorfismos genéticos presentes en los agentes etiológicos [9].
Los métodos fenotípicos constituyen una herramienta de suma importancia para la tipificación de muchos patógenos, no obstante, el alcance de estos procedimientos puede encontrar serias restricciones. Los rasgos fenotípicos son susceptibles a la influencia del ambiente, que pueden provocar variaciones en la expresión genética, por lo cual, el resultado obtenido a través de la detección de este tipo de caracteres puede presentar poca estabilidad, reproducibilidad o poder discriminatorio. Adicionalmente, presentan la desventaja de encontrarse, en algunos casos, limitados a unas pocas especies. Resultan además, poco prácticos para el análisis de microorganismos de crecimiento lento, dificultoso o no cultivables ya que, generalmente, requieren la multiplicación de los mismos, y resultan poco apropiados para determinar las relaciones de clonalidad entre microorganismos, presentando una limitada capacidad de identificación a nivel de subespecie, subtipo o cepa [9,10].
Los métodos genotípicos están basados en la detección del material genético del organismo, por lo tanto son independientes de los cambios en el patrón de expresión genética y de las influencias ambientales, ofreciendo una alternativa con mayor estabilidad y reproducibilidad. No requieren necesariamente el aislamiento y cultivo de los microorganismos, siendo posible obtener los resultados en tiempos más cortos y, usualmente con mayor poder de resolución, sensibilidad y especificidad que los métodos fenotípicos. Los métodos de genotipificación se han constituido en una herramienta que complementa a los procedimientos fenotípicos, extendiendo los alcances de la epidemiología y permitiendo un mayor entendimiento de los problemas asociados al proceso salud-enfermedad, incrementando la posibilidad de implementar medidas efectivas de control epidemiológico [9, 10].
Procedimientos de genotipificación
¿Qué es la clonalidad? Cuándo, cómo y para qué determinar las relaciones clonales
La aparición de un cuadro infeccioso en un paciente o de un brote en una comunidad u hospital pone en marcha una serie de medidas destinadas a su control, y estas forman parte de un plan que se aplica en dos etapas. En primera instancia, se realiza el aislamiento del patógeno y su clasificación. Conocido el microorganismo responsable de la infección deben definirse con precisión su origen, la cadena epidemiológica que ha conducido a la infección y las medidas de control adecuadas [11]. En ocasiones, para identificar al patógeno, su origen o las vías y patrón de diseminación, se requiere determinar las similitudes o diferencias filogenéticas entre diversos aislados. Para realizar este análisis se trabaja bajo el supuesto que las cepas filogenéticamente relacionadas se han derivado, recientemente, de la expansión clonal de un precursor único, por lo tanto poseen un nivel de similitud entre sus genotipos, significativamente superior al que se encontraría entre cepas no relacionadas del mismo taxón, escogidas al azar. Los aislados no relacionados clonalmente, tendrán genotipos diferentes ya que provienen de ancestros distintos. Los supuestos señalados anteriormente pueden no cumplirse en aquellos casos en los cuales la diversidad genética dentro de la especie o grupo sea limitada [7].
Para la determinación de la clonalidad entre microorganismos se debe definir el nivel de relación genética necesario para diferenciar los clones y, el o los marcadores que permitan establecer dicha diferencia. El punto de corte a partir del cual se considera que dos microorganismos no se encuentran clonalmente relacionados es arbitrario y dependerá del problema concreto y de la herramienta utilizada para resolverlo. En este sentido, se debe tener especial cuidado ya que, el establecimiento incorrecto de los parámetros o la utilización de una técnica inadecuada, puede conducir a interpretaciones erradas. Finalmente debe evaluarse el procedimiento de tipificación según los criterios enumerados anteriormente, colocando especial interés en su concordancia epidemiológica.
Los métodos de tipificación fenotípicos tienen un valor discriminatorio insuficiente para definir las relaciones filogenéticas entre microorganismos de la misma especie, no permitiendo establecer la clonalidad entre los aislados. En este sentido, las técnicas de genotipicación permiten determinar si los microorganismos analizados se encuentran genéticamente relacionados y por lo tanto, pueden ser considerados como representantes de una misma cepa o clon. Por esta razón, su uso se ha popularizado con el pasar de los años, habiendo sido modificadas y perfeccionadas en el sentido de incrementar su capacidad discriminatoria, reproducibilidad y versatilidad [12].
La determinación de la clonalidad entre bacterias es necesaria en varias situaciones, por ejemplo, en los casos en los cuales se requiere la diferenciación entre infección y contaminación. Muchas de la especies causantes de infecciones son también organismos comensales de humanos, por lo cual es importante determinar si la cepa analizada es la causante de la infección o si simplemente, es un comensal que fue aislado como contaminación, durante el proceso de toma de muestra [7]. También puede ser necesario evaluar el origen clonal frente a infecciones recurrentes, y así discriminar entre re-infección por el mismo clon o infección por un clon distinto. Si el microorganismo causante del segundo cuadro infeccioso es el mismo que causó la infección original, se puede considerar la posibilidad que se esté aplicando un tratamiento antimicrobiano inadecuado o poco efectivo, siendo necesario entonces la implementación de una terapia alternativa [7].
A nivel epidemiológico, también se hace indispensable la evaluación de la clonalidad entre aislados, cuando se estudian brotes de infecciones intra o extrahospitalarias. Por ejemplo, para establecer la relación genética entre aislados de una misma fuente, pero que manifiestan diferencias a nivel fenotípico o, por el contrario, aislados provenientes de distintos pacientes pero que muestran características fenotípicas similares. La confirmación de la relación genética entre estos microorganismos permitiría determinar la fuente de la infección, el número de clones circulantes, el vehículo, la ruta y el patrón de distribución. El conocimiento que brindan las técnicas de genotipificación permite la implementación de nuevos programas que contribuyan al control y prevención de los brotes infecciosos.
Métodos moleculares para genotipificación: alcances y limitaciones
Los métodos moleculares para genotipificación son variados y pueden estar basados en el análisis de polimorfismos de secuencias repetidas, de elementos variables o del genoma completo de los microorganismos. Desde el punto de vista metodológico, las técnicas moleculares para genotipificación pueden clasificarse en cuatro grandes grupos: a) aquellas basadas en el estudio del electrocariotipo (de un segmento del genoma o del genoma completo) mediante electroforesis de campo pulsado (PFGE), b) las relacionadas con la amplificación de fragmentos de DNA por medio de la reacción en cadena de la polimerasa (PCR), c) las que involucran la secuenciación parcial o total del genoma de los microorganismos y, d) aquellas en las cuales se obtienen perfiles mediante la hibridación de ácidos nucleicos. Cada una de estas herramientas ha ofrecido una alternativa de gran utilidad para la investigación epidemiológica, sin embargo, su aplicación se encuentra limitada al caso particular en estudio.
Electroforesis de campo pulsado (PFGE): Esta es una técnica en la cual, el ADN genómico de los aislados en estudio es digerido con endonucleasas. Los segmentos generados, de peso molecular elevado, son separados en un gel de agarosa, gracias a la aplicación de campos eléctricos cuya direccionalidad cambia a intervalos predeterminados [13]. Como resultado se obtiene un patrón de bandas característico para cada aislado. Tenover y colaboradores, propusieron un criterio que permite interpretar el electrocariotipo de diversas cepas de manera tal que, se puedan establecer las relaciones genéticas y epidemiológicas entre dichos microorganismos. Según este sistema se puede clasificar a los aislados bacterianos en cuatro categorías: cepas indistinguibles, cepas estrechamente relacionadas, cepas posiblemente relacionadas y cepas no relacionadas [14].
El análisis del polimorfismo genético a través de la PFGE es la técnica que tiene el más alto poder discriminatorio, la mejor reproducibilidad y la de mayor versatilidad, permitiendo tipificar una enorme variedad de especies bacterianas. El patrón de bandas generado puede ser analizado con programas adecuados y los electrocariotipos se pueden incorporar en una base de datos de referencia que sirva como fuente de comparación [15]. Las ventajas enumeradas han convertido al PFGE en la técnica estándar de referencia para la tipificación de bacterias, hongos y parásitos de importancia clínica [16].
A pesar de sus múltiples ventajas, el uso del PFGE se encuentra limitado en ciertas situaciones debido a su laboriosidad así como por el tiempo requerido para obtener los resultados (2-3 días) [15]. Por esta misma razón, resulta inadecuado para el análisis de poblaciones numerosas o colectadas por períodos superiores a un año [14].
Reacción en cadena de la polimerasa (PCR): Las técnicas de tipificación basadas en la reacción PCR involucran la amplificación de genes o de secuencias de DNA polimórficas. Estos procedimientos son menos laboriosos, más rápidos, económicos y flexibles que el PFGE, facilitando el procesamiento de un mayor número de muestras y prescindiendo del cultivo de los microorganismos. Sin embargo, permiten analizar sólo un sector reducido del genoma del microorganismo. Por otra parte, a pesar de poseer un elevado poder discriminatorio, muestran mayor efectividad para diferenciar cepas no relacionadas clonalmente que, para establecer distinciones entre cepas relacionadas filogenéticamente. Por esta razón, se consideran que en comparación con el PFGE, presentan un menor poder de discriminación [16].
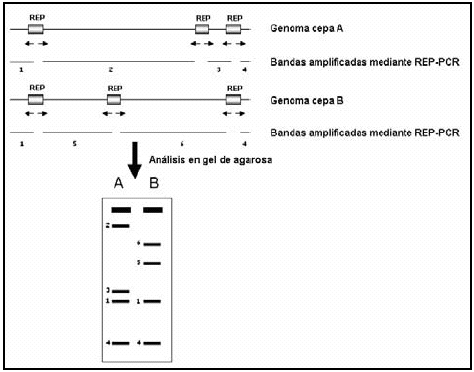
Existen diversas variantes de la prueba de PCR que han sido aplicadas con fines epidemiológicos. El AP-PCR (Arbitrarily-Primed PCR), el RAPD-PCR (Randomly-Amplified Polymorphic DNA PCR), el ERIC-PCR (Enterobacterial Repetitive Intergenic Consensus PCR) y el REP-PCR (Repetitive Extragenic Palindromic), han sido utilizadas con éxito para fines epidemiológicos y taxonómicos [16]. Actualmente se está imponiendo el uso de estas dos últimas cuando se precisa un poder de discriminación mayor. Los elementos ERIC y REP son secuencias de DNA extragénicas, cortas, repetidas y esparcidas en el genoma de las enterobacterias. Las secuencias ERIC constan de 126 pb, con un repetido invertido central conservado, mientras que las secuencias REP son elementos palindrómicos de 21-65 pb [17]. Tanto el REP-PCR como el ERIC-PCR emplean iniciadores que reconocen las secuencias conservadas REP o ERIC, y las regiones amplificadas corresponden a los segmentos que separan dichas secuencias. Así, el polimorfismo generado dependerá de la variedad en la distribución de las repeticiones y de la distancia entre las secuencias REP o ERIC dentro del genoma (Figura 1). Estas técnicas resultan sumamente sencillas, no dependen del uso de enzimas de restricción, ni de técnicas electroforéticas especiales, son rápidas, de relativo bajo costo, fáciles de analizar y de excelente reproducibilidad; todo lo cual las coloca en una posición privilegiada a la hora de seleccionar una técnica de tipificación genética [16]. Sin embargo, suelen tener una menor versatilidad y un poder de resolución menor que el PFGE.
Figura 1. Diagrama esquemático de la prueba de REP-PCR para el establecimiento de relaciones filogenéticas. Se representa la distribución hipotética de las secuencias REP en una región de los genomas de dos microorganismos A y B. Utilizando iniciadores que reconocen las secuencias REP, se amplifican las regiones espaciadoras extragénicas. Los fragmentos de DNA generados, se separan en un gel de agarosa y se comparan los patrones de bandas entre los aislados.
Secuenciación de ácidos nucleicos: La variabilidad del genoma puede ser utilizada para tipificar a los microorganismos en estudio [8]. La secuenciación parcial de los genomas ha sido de vital importancia para el análisis de situaciones en las cuales se encuentre en riesgo la salud pública, ya que ofrece una alternativa para explorar la diversidad genética de distintas cepas de una misma especie, así como también, proporciona información que contribuya al diseño de nuevas herramientas para una rápida genotipificación [18]. Dos de los protocolos empleados comúnmente para abordar estudios epidemiológicos son: SLST (Single-Locus Sequence Typing) y MLST (Multiple-Locus Sequence Typing), basados en el análisis de la secuencia de uno o varios loci genéticos, respectivamente. Ambos protocolos se aplican con la prueba de PCR, lo cual ofrece la posibilidad de amplificar directamente a partir de la muestra biológica. Estas técnicas, al poseer elevada reproducibilidad y poder ser estandarizadas con facilidad, resultan herramientas adecuadas para realizar comparaciones entre diferentes aislados [6].
En el caso del SLST se determina la variabilidad de los genes de virulencia, de patogenicidad o de aquellos que confieran resistencia a drogas, mientras que a través del MSLT se comparan regiones polimórficas de genes cuyos productos se encuentran involucrados en funciones esenciales del microorganismo. En este último caso, para realizar el análisis se considera que: a) los genes o segmentos de éstos que presenten diferencias en la secuencia de nucleótidos, pueden ser considerados alelos distintos; b) con base en el análisis de todos los genes esenciales, se podrá elaborar un perfil alélico para cada cepa y; c) las cepas que muestren perfiles alélicos idénticos, pueden considerarse como representantes de un mismo clon [6].
Los estudios con SLST han mostrado poca concordancia epidemiológica con otros métodos de tipificación como el PFGE, lo cual se ha constituido en el principal inconveniente para su aplicación en epidemiología. Por el contrario, el MLST se ha implantado exitosamente en varios sistemas, mostrando una mayor potencialidad para determinar las relaciones filogenéticas entre cepas [6]. El conocimiento de la secuencia completa del genoma del microorganismo ha contribuido a la selección apropiada de las regiones polimórficas a analizar en el estudio tipo MLST, mejorando el alcance y la capacidad de discriminación de la técnica. Sin embargo, para algunos patógenos humanos, entre los cuales se encuentra B. anthracis o M. tuberculosis, la baja variabilidad observada en los genes que codifican para funciones esenciales, ha limitado enormemente el uso de esta técnica con fines epidemiológicos [18].
El procedimiento de secuenciación con el más elevado poder discriminatorio y reproducibilidad es la secuenciación del genoma completo, sin embargo, su costo y, el tiempo requerido para la obtención de los resultados, limita enormemente su aplicación para estudios epidemiológicos. A menor escala, el SLST y el MLST, se tropiezan con estas mismas dificultades, sin embargo han sido particularmente útiles para el análisis de genomas virales [6,8].
Hibridación de ácidos nucleicos: Entre las estrategias de tipificación basadas en la hibridación tipo Southern, la más versátil y ampliamente utilizada para detectar polimorfismos asociados a los genes ribosomales, es la ribotipificación. La ribotipificación muestra excelente reproducibilidad y estabilidad, tanto in vivo como in vitro. Hoy en día, esta técnica se encuentra disponible en una presentación comercial totalmente automatizada y en un formato muy bien estandarizado [2].
El advenimiento de los avances tecnológicos que permitieron la secuenciación de genomas completos ha permitido el desarrollo de la técnica de hibridación mediante arreglos de DNA. Un arreglo consiste en un conjunto de secuencias de DNA inmovilizadas, en un orden preciso, sobre una superficie sólida. Dependiendo del número de secuencias fijadas en el soporte puede variar desde unas decenas (macroarreglos) hasta decenas de miles (microarreglos) dependiendo del soporte [19]. El DNA o el cDNA extraído de las muestras a analizar es marcado, usualmente con fluoróforos, e incubado con los segmentos inmovilizados sobre el soporte. En aquellos puntos donde se genere la hibridación molecular, se podrá visualizar una señal [20]. La técnica tiene la ventaja que permite obtener una enorme cantidad de información en un único experimento. Sin embargo, esta gran cantidad de información se convierte en un problema importante a la hora de analizar los datos manualmente, requiriéndose sistemas informáticos que permitan el análisis de todos los datos y evitar la subjetividad inherente al investigador [20].
Dentro de los formatos de los macroarreglos se encuentra el LiPA (Line Probe Assay), un sistema de detección multiparamétrico. Este sistema utiliza tiras cubiertas con sondas específicas que son hibridadas con los productos de amplificación obtenidos a partir de la muestra analizada. El sistema presenta la ventaja que en un solo análisis pueden amplificarse distintas partes de un gen, diferentes genes o DNA de diversos organismos, lo cual convierte a esta aproximación metodológica en una herramienta útil en epidemiología [20].
La identificación molecular a través de arreglos presenta en la actualidad ciertas limitaciones como su elevado cos-to, baja sensibilidad o la necesidad de cultivar la muestra o de realizar, al menos, un paso previo de amplificación para la detección de los blancos.
En el caso específico de la detección de determinantes de resistencia a los antibióticos a través de arreglos se presenta un inconveniente adicional. La enorme variabilidad asociada, tanto en mecanismos como en determinantes genéticos, hace insuficiente cualquier aproximación experimental que intente contemplar todas las alternativas en un solo modelo. Sin embargo, su aplicación racional, basada en datos epidemiológicos obtenidos por otras técnicas fenotípicas o genotípicas podría permitir abordar con rapidez los brotes de agentes infecciosos [20].
Aplicaciones de los métodos moleculares de genotipificación en estudios clínicos
Son muchas las aplicaciones de los métodos de tipificación descritos anteriormente. Han sido publicados numerosos reportes en los cuales, a través de las técnicas de genotipificación, se han obtenido resultados de importancia epidemiológica. Se ha logrado identificar toda una variedad de reservorios del foco de infección entre los que se encuentran: material médico-quirúrgico; soluciones empleadas en el tratamiento de los pacientes, bienes muebles o incluso, al personal médico-asistencial [21]. En el campo de los alimentos también han sido útiles estas técnicas. En el año 2007, se identificó a través de PFGE, la fuente de contaminación de individuos infectados con Salmonella enterica serotipo Typhimurium. Los patrones de bandas, obtenidos después de digerir el DNA genómico de las cepas aisladas de los pacientes, eran idénticos entre sí, e indistinguibles del obtenido a partir de bacterias aisladas de leche contenida en un tanque de almacenamiento. Una vez identificada la fuente de infección, las autoridades de salud realizaron una campaña para informar sobre el riesgo asociado a consumir la leche de vaca no pasteurizada [22].
Otro ejemplo interesante lo reportan Gardella y colaboradores, quienes analizaron 40 cepas de Staphylococcus aureus resistentes a meticilina (MRSA) aislados de dos hospitales universitarios. Los resultados, obtenidos mediante REP-PCR, RAPD-PCR y PFGE, demostraron que un clon de MRSA multiresistente, había sido desplazado por un clon con un perfil de resistencia a antibióticos distinto, información que contribuyó al tratamiento de pacientes con infecciones nosocomiales por MRSA [23].
La secuenciación ha resultado una herramienta particularmente útil para los estudios epidemiológicos de genomas virales. En el año 2002, tres pacientes atendidos en la Unidad de Hemodiálisis de una clínica privada, resultaron positivos para el virus de hepatitis C (HCV). El análisis de la secuencia de nucleótidos de regiones conservadas e hipervariables del genoma del HCV, confirmó que los pacientes habían sido contaminados a través del uso del equipo de hemodiálisis, y demostró dos eventos de transmisión independientes. La identificación de la fuente y vehículo de transmisión permitió incrementar las medidas de seguridad y prevenir futuras contaminaciones [24].
Como ejemplo de los procedimientos basados en arreglos de DNA que han contribuido al análisis epidemiológico, se encuentra el reporte realizado por Vora y colaboradores, al caracterizar cepas de Vibrio, patógenas de humanos. La aproximación experimental involucró la amplificación simultánea de 95 regiones del genoma del microorganismo, en combinación con un microarreglo de oligonucleótidos largos. Mediante este procedimiento se pudieron identificar los principales miembros del género patógenos de humanos: V. cholerae, V. parahaemolyticus, V. vulnificus y V. mimicus. Además, el sistema suministró información sobre la presencia de determinantes de resistencia a antimicrobianos, elementos genéticos móviles y, genes asociados a la producción de toxinas y a la patogenicidad. Este procedimiento permite obtener resultados rápidamente, proporcionando importante información epidemiológica no sólo sobre microorganismos cultivables, sino sobre formas no cultivables o de difícil crecimiento [25].
En muchos casos, la aplicación de un procedimiento de genotipificación en un estudio en particular, se deriva de un análisis previo en el cual, se comprueba su efectividad, precisión, rapidez y menor relación costo-beneficio, en comparación con los métodos fenotípicos. En relación a este punto existen innumerables reportes en los cuales se comparan ambos tipos de procedimientos. De todos ellos, se puede citar el referido a la determinación de P. aeruginosa en muestras de suelo contaminadas con hidrocarburos o en proceso de remediación [26]. En ese trabajo se analizan 43 aislados ambientales provenientes de suelos contaminados o en tratamiento, encontrándose que los métodos fenotípicos generan falsos positivos en cinco de dichas muestras. Lo anterior reduce la efectividad del procedimiento fenotípico en 3-5 % con respecto al procedimiento genotípico [26]. Adicionalmente, los autores del trabajo indican que las técnicas moleculares resultaron ser más prácticas, más precisas y requieren un menor tiempo para la obtención de los resultados, ya que los métodos de identificación fenotípicos que requieren el cultivo de los microorganismos en medios específicos, en el mejor de los casos, ofrecen resultados en 24 horas; mientras que la prueba de PCR permite conocer el genotipo de la muestra en 12 horas. Con base en los resultados obtenidos, los autores consideran apropiada la revisión de los procedimientos estándar para la identificación de P. aeruginosa con la finalidad de incorporar las técnicas moleculares en los protocolos de rutina [26].
En el campo de la salud pública, el diagnóstico molecular ha impactado positivamente, especialmente en la detección de infecciones virales, ó patógenos emergentes o en el caso de infecciones con patógenos transmitidos por vía sexual. Se ha incrementado la implementación de técnicas moleculares para el diagnóstico de patógenos asociados a infecciones transmitidas por vía sexual como Neisseria gonorrhoeae y Chlamydia trachomatis, los cuales son responsables de hasta el 70 % de éste tipo de patologías [27]. Hasta hace 10 años, la identificación de N. gonorrhoeae y C. trachomatis se realizaba a través de técnicas fenotípicas que requerían el cultivo de los microorganismos. Desde entonces, las técnicas moleculares se han impuesto como un procedimiento de rutina en los laboratorios clínicos, por superar en un 25-40 % a los procedimientos fenotípicos, al momento de realizar la detección de los patógenos. Adicionalmente, el proceso de genotipificación de éstos patógenos no es afectado por la pérdida de viabilidad del patógeno durante el proceso de toma de muestra o por su transporte hasta el sitio del análisis, pudiendo obtenerse resultados positivos hasta tres semanas después de la toma y, tampoco requiere el cultivo previo de los microorganismos, reduciendo significativamente el tiempo de obtención de los resultados [27].
Comentarios finales
Los métodos genotípicos se han convertido en elementos esenciales en los análisis epidemiológicos, siendo poderosas herramientas que complementan las estrategias utilizadas para combatir la dispersión de las enfermedades infecciosas. El alcance de los resultados dependerá, en buena medida, de la selección adecuada de la técnica, de su correcta implementación y, frecuentemente, de la aplicación combinada con diversos procedimientos. Estas técnicas no son un sustituto de los métodos convencionales, utilizados de rutina en los estudios epidemiológicos. Se plantean como un nuevo elemento que cubre las limitaciones de las técnicas fenotípicas y permiten fortalecer el campo epidemiológico, dado que los métodos fenotípicos no permiten determinar relaciones clonales entre aislados de una misma especie. De esta forma, el análisis, parcial o total, del genoma de los microorganismos debe ser incorporado como un instrumento más, cuyos resultados, sumados a los obtenidos a través de otras técnicas, permita a una ciencia social y con proyección colectiva como es la Epidemiología, ampliar la dimensión del análisis y penetrar con mayor fuerza en la realidad de la comunidad.
Referencias
1. Burton P, Tobin M, Hopper J. Key concepts in genetic epidemiology. Lancet. 2005; 366: 941-51. [ Links ]
2. Struelens M. Molecular epidemiologic typing systems of bacterial pathogens: current issues and perspectives. Mem Inst Oswaldo Cruz. 1998; 93: 581-5. [ Links ]
3. Johnson J, Russo T. Molecular epidemiology of extraintestinal pathogenic (uropathogenic) Escherichia coli. Int J Med Microbiol. 2005; 295: 383-404. [ Links ]
4. Mathema B, Kurepina N, Bifani P, Kreiswirth B. Molecular epidemiology of tuberculosis: current insights. Clin Microbiol Rev. 2006; 19: 658–85. [ Links ]
5. Epidemiología y Causalidad en Salud Ocupacional, Disponible en: http://www.estrucplan.com.ar/articulos/verarticulo.asp?IDArticulo=1196. Acceso 15 de marzo de 2008. [ Links ]
6. Zaidi N, Konstantinou K, Zervos M. The role of molecular biology and nucleic acid technology in the study of human infection and epidemiology. Arch Pathol Lab Med. 2003; 127: 1098-105. [ Links ]
7. Singh A, Goering R, Simjee S, Foley S, Zervos M. Application of molecular techniques to the study of hospital infection. Clin Microbiol Rev. 2006; 19: 512 - 30. [ Links ]
8. Foxman B, Zhang L, Koopman J, Manning S, Marrs C. Choosing an appropriate bacterial typing technique for epidemiologic studies. Epidemiol Perspect Innov. 2005; 2: 10-7. [ Links ]
9. Pfaller M. Molecular epidemiology in the care of patients. Arch Pathol Lab Med. 1999; 123: 1007-10. [ Links ]
10. Versalovic J., Lupski R. Molecular detection and genotyping of pathogens: more accurate and rapid answers. Trends Microbiol. 2002; 10: S15-21. [ Links ]
11. Sabrià-Leal M, Montoya P. Epidemiología molecular. Med Clin. 1992; 99: 137-9. [ Links ]
12. Labarca J. Utilización del antibiotipo como marcador epidemiológico en infecciones intrahospitalarias: comparación con la epidemiología molecular. Rev Chil Infect. 2002; 19: S157-60. [ Links ]
13. Olive D, Bean P. Principles and applications of methods for DNA-based typing of microbial organisms. J Clin Microbiol. 1999; 37: 1661–9. [ Links ]
14. Tenover F, Arbeit R, Goering R, Mickelsen P, Murray B. Persing D, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol. 1995; 33: 2233–9. [ Links ]
15. Fredriksson-Ahomaa M, Stolle A, Korkeala H. Molecular epidemiology of Yersinia enterocolitica infections. FEMS Immunol Med Microbiol. 2006; 47: 315-29. [ Links ]
16. Fernández-Cuenca F. Aplicaciones de las técnicas de PCR a la epidemiología molecular de las enfermedades infecciosas. Enferm Infecc Microbiol Clin. 2004; 22: 355-60. [ Links ]
17. Tobes R, Pareja E. Bacterial repetitive extragenic palindromic sequences are DNA targets for insertion sequence elements. BMC Genomics. 2006; 7: 62. [ Links ]
18. Fournier P, Drancourt M, Raoult D. Bacterial genome sequencing and its use in infectious diseases. Lancet Infect Dis. 2007; 7: 711-23. [ Links ]
19. Garaizar J, Rementeria A, Porwollik S. DNA microarray technology: a new tool for the epidemiological typing of bacterial pathogens? FEMS Immunol Med Microbiol. 2006; 47: 178-89. [ Links ]
20. Doménech-Sánchez A, Vila J. Fundamento, tipos y aplicaciones de los arrays de ADN en la microbiología médica. Enferm Infect Microbiol Clin. 2004; 22: 46-54. [ Links ]
21. Richards C, Jarvis W. Lessons from recent nosocomial epidemics. Curr Opin Infect Dis. 1999; 12: 327-34. [ Links ]
22. Lind L, Reeser J, Stayman K, Deasy M, Moll M, Weltman A, et al. Salmonella typhimurium infection associated with raw milk and cheese consumption--Pennsylvania, 2007. MMWR Morb Mortal Wkly Rep. 2007; 56: 1161-4. [ Links ]
23. Gardella N, Picasso R, Predari SC, Lasala M, Foccoli M, Benchetrit G, et al. Methicillin-resistant Staphylococcus aureus strains in Buenos Aires teaching hospitals: replacement of the multidrug resistant South American clone by another susceptible to rifampin, minocycline and trimethoprim-sulfamethoxazole. Rev Argent Microbiol. 2005; 37: 156-60. [ Links ]
24. Bracho M, Gosalbes M, Blasco D, Moya A, González-Candelas F. Molecular epidemiology of a hepatitis C virus outbreak in a hemodialysis unit. J Clin Microbiol. 2005; 43: 2750-5. [ Links ]
25. Vora G, Meador C, Bird M, Bopp C, Andreadis J, Stenger D. Microarray-based detection of genetic heterogeneity, antimicrobial resistance, and the viable but nonculturable state in human pathogenic Vibrio spp. Proc Natl Acad Sci USA. 2005; 102: 19109-14. [ Links ]
26. Atzél B, Szoboszlay S, Mikuska Z, Kriszt B. Comparison of phenotypic and genotypic methods for the detection of environmental isolates of Pseudomonas aeruginosa. Int J Hyg Environ Health. 2008; 211: 143-55. [ Links ]
27. Miller M. The Impact of Molecular Diagnostics on Womens Health. Clinical Microbiology Newsletter. 2008; 30: 15-21. [ Links ]












 uBio
uBio