Revista de la Sociedad Venezolana de Microbiología
versión impresa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.30 n.1 Caracas jun. 2010
Estandarización de una PCR para la detección del gen invA de Salmonella spp. en lechuga
Luz Chacón*, Kenia Barrantes, Cristina García, Rosario Achí
INISA, Universidad de Costa Rica. Costa Rica
* Correspondencia: E-mail: luz.chacon@ucr.ac.cr
Resumen:
Salmonella spp. es un patógeno bacteriano muy importante causante de diarreas, que es transmitido tanto por la vía fecal-oral, como por alimentos y agua contaminados. En este trabajo se estandarizó una técnica de PCR en lechuga para la detección del gen invA de Salmonella spp.; dicho gen se relaciona con el proceso de invasión al epitelio intestinal. Con la PCR desarrollada en este trabajo se logró estandarizar un método que permite la amplificación del gen invA con una detección de 102 UFC/25 g. Este método acorta los tiempos de respuesta de los resultados presuntivos y brinda información complementaria al cultivo tradicional del patógeno. El estudio del gen invA establece el potencial patógeno del microorganismo presente en la muestra, lo que puede ser de utilidad para la salud pública.
Palabras clave: Salmonella spp., gen invA, PCR, lechuga
Standardization of a PRC method for the detection of the Salmonella spp. invA gene in lettuce
Abstract:
Salmonella spp. is a very important bacterial pathogen that causes diarrhea and which is transmitted both through the fecal-oral pathway, as by contaminated food and water. In this study we standardized a PCR method in lettuce for the detection of the Salmonella spp. invA gene. This gene is related to the invasion of the intestinal epithelium process. With the PCR method developed in this study we were able to standardize a method which permits the amplification of the invA gene with a 102 CFU/25 g detection. This method shortens the response times of the presumptive results and gives complementary information to the traditional culture of the pathogen. The study of the invA gene establishes the pathogenic potential of the microorganism present in the sample, which can be useful for public health purposes.
Keywords: Salmonella spp., invA gene, PCR, lettuce
Recibido 25 de septiembre de 2009; aceptado 27 de abril de 2010
Introducción
Salmonella spp., es la causa principal de enteritis bacteriana de transmisión por alimentos a nivel mundial, según los datos de la Organización Mundial de la Salud [1]. En Austria se presentaron 103 casos por cada 100.000 habitantes durante el año 2003 [2]. En Estados Unidos, se estima que ocurren cerca de 1,4 millones de infecciones no tifoideas, que generan 168.000 visitas al médico, 15.000 hospitalizaciones y 580 muertes anualmente [3]. Los datos de World Health Organization Global Salm-Surv country data bank entre el 2000 y 2002 para Costa Rica muestran 49 casos de salmonelosis asociada a humanos y 60 asociada a animales, y para Venezuela 691 casos de salmonelosis asociados a humanos y 162 asociados a animales [4].
Salmonella spp. produce una amplia variedad de cuadros clínicos; el más frecuente es la gastroenteritis caracterizada por diarrea y vómitos. Generalmente su transmisión se produce por ingestión de alimentos contaminados o contacto con animales infectados. La fiebre tifoidea, causada principalmente por la ingesta de alimentos contaminados con Salmonella enterica serovariedad Typhi, es la forma más severa de fiebre entérica. Se caracteriza por fiebre prolongada, bacteremia y diseminación a múltiples órganos. El cuadro puede llegar tener un curso de tres semanas. La bacteremia no tifoidea se da principalmente en niños pequeños y adultos mayores y se caracteriza por ser intermitente posterior a un cuadro de gastroenteritis. Existe además el estado portador, posterior a una infección con esta bacteria, ya que coloniza la vesícula biliar y se mantiene por largos períodos de tiempo [5].
En países desarrollados la transmisión de Salmonella ocurre principalmente por consumo de productos derivados de animales, particularmente carne fresca y huevos. Por el contrario, en países en vías de desarrollo las principales fuentes de contaminación las constituyen los vegetales frescos, el agua y la transmisión persona a persona [6].
Un factor importante son los reservorios naturales de la bacteria; en tanto que S. enterica serovar. Typhi y Paratyphi son serovariedades exclusivas del ser humano, otras como Typhimurium y Litchfield, pueden ser consideradas como zoonóticas, debido a que tienen entre sus hospederos a animales como aves y ganado [7-9].
Un gen importante en la virulencia de Salmonella spp. es el invA. Este gen es un factor de virulencia relacionado con el proceso de invasión al epitelio intestinal y utilizado por otros enteropatógenos como Shigella spp. durante el proceso de infección. El gen invA es común en todas las variedades invasoras, lo que significa que se puede asociar con posibles cuadros virulentos. Se encuentra codificado en el cromosoma bacteriano lo que le confiere mayor estabilidad, en una región conocida como isla de patogenicidad 1 de Salmonella (SPI1). Esta isla codifica, entre otros, proteínas del sistema de secreción tipo III [10-13]. El gen invA ha sido ampliamente utilizado en estudios para la detección de Salmonella spp. en muestras de alimentos, debido principalmente a la estabilidad que presenta desde el punto de vista genético [14-16].
La detección de Salmonella por cultivo convencional es considerada el método de referencia. Sin embargo, esta técnica presenta desventajas como la baja sensibilidad y el tiempo para la obtención de un resultado. Actualmente surgen otras alternativas diagnósticas, como los métodos de biología molecular. Uno de los más utilizados es la Reacción en Cadena de la Polimerasa (PCR por sus siglas en inglés), que permite la detección de microorganismos en los alimentos en menor tiempo si se compara con las técnicas de cultivo convencional. Entre los factores limitantes de la PCR están las numerosas sustancias inhibitorias que pueden encontrarse en la matriz del alimento (hemoglobina, lactoferrina, polisacáridos y grasas); además, la detección del ADN de un patógeno, no asegura la viabilidad del mismo en la muestra al momento del análisis, lo que representa una desventaja con respecto al cultivo convencional. [17-19]. Por otra parte, el aislamiento por cultivo en alimentos no asegura que el microorganismo aislado posea los determinantes de virulencia necesarios para producir infección y enfermedad en un hospedero susceptible [20].
El objetivo principal de este estudio fue la obtención de un método de PCR capaz de identificar de forma directa genes asociados a la virulencia de Salmonella spp., como herramienta adicional al diagnóstico tradicional de este patógeno, y que además brinde información importante en términos de salud pública en un corto plazo.
Materiales y métodos
Bacterias utilizadas: Para el procesamiento de las muestras de lechuga y la estandarización de la PCR se utilizó una cepa de Salmonella enterica serovariedad Enteritidis (ATCC 13076). Para las pruebas de especificidad de la PCR se utilizaron las siguientes bacterias: Escherichia coli ATCC 25922, E. coli (LT-/ST-), E.coli (LT+/ST-), E.coli (LT-/ST+), E.coli (LT+/ST+), E.coli enteropatógena, Enterobacter cloacae, Citrobacter freundii, Moraxella phenylpyruvica, Proteus mirabilis, Vibrio cholerae, Pseudomonas aeruginosa, Brevundimonas diminuta, Plesiomonas shigelloides, Aeromonas hydrophila, Staphylococcus aureus ATCC 22923, Shigella flexneri, S. flexneri (pINV) y S. sonnei las cuales pertenecen a la colección bacteriana del Instituto de Investigaciones en Salud de la Universidad de Costa Rica.
Descripción de muestra: En el presente estudio se utilizaron 13 muestras de 25 g, analizadas por triplicado (39 submuestras totales) de lechugas variedad mantequilla (Lactuca sativa var. capitata), adquiridas en supermercados del Valle Central.
Procesamiento de muestras: Las porciones de 25 g de lechuga fueron contaminadas intencionalmente con Salmonella ATCC 13076 (diluciones seriadas de 107 UFC/mL a 101 UFC/mL). Adicionalmente se utilizó una porción de 25 g. con 1 mL de agua peptonada estéril, como control negativo. Estas muestras se sometieron a un pre-enriquecimiento agregando 225 mL de caldo lactosado y caldo lactosado suplementado con novobiocina y se incubaron por 24 horas a 35ºC. Luego de la incubación se agregó 1 mL de este pre-enriquecimiento en un tubo cónico, estéril, tipo Eppendorf, para la extracción del ADN del sedimento de células, y otro mililitro se colocó en caldo tetrationato y selenito-cistina, que se incubó por 24 horas a 35ºC. Por último se realizó un pasaje de este último a los medios desoxicolato citrato (DC) y xilosa lisina desoxicolato (XLD), de donde se escogieron colonias con morfología sospechosa de Salmonella spp., para los análisis por métodos bioquímicos tradicionales.
Identificación bioquímica: Se realizaron pruebas para urea, movilidad, indol, citrato y TSI (agar triple azúcar hierro, por sus siglas en inglés). Luego se utilizó el API 20E BioMérieux® para la identificación completa. Los aislamientos con perfil de Salmonella spp. fueron confirmados serológicamente con el antisuero Salmonella O antiserum poly-A-I & Vi (DIFCO).
Extracción del ADN: Se separó una fracción de 1 ml del caldo de pre-enriquecimiento, tanto de las lechugas contaminadas con concentraciones conocidas de Salmonella spp., como las que se mezclaron con agua peptonada estéril, que se centrifugó por 10 minutos a 34.000 g. El sobrenadante se decantó, y el sedimento celular se resuspendió en 1 mL de agua destilada estéril. Este procedimiento de lavado con agua destilada se repitió 3 veces para eliminar posibles inhibidores de la PCR. La extracción del ADN se realizó por choque térmico, resuspendiendo el botón en 1 mL de agua destilada y colocándolo en un recipiente con agua a 95°C por 10 minutos. El tubo se centrifugó por 10 minutos a 33.000 g, y el sobrenadante con el ADN se separó en otro tubo estéril, que fue almacenado a -70ºC. Para obtener el ADN utilizado como control positivo de la PCR, se inoculó 1 mL de agua destilada estéril con una colonia de Salmonella spp. y se repitió el procedimiento anterior de centrifugado, lavado y extracción del ADN por calor.
Selección de los iniciadores: Se utilizaron los iniciadores Salm3 5GCTGCGGCGCAACGGCGAAG-3 y Salm4 5- TCCCGGCAGAGTTCCCATT-3 [12, 15]. Su especificidad con el genoma de Salmonella enterica fue corroborada por medio del programa BLAST (Basic Local Aligment Search Tool), mostrando para Salm3 una identidad igual a 20/20, (E-value=0,006); y para Salm4 una identidad=19/19 (E-value=0,017).
Técnica de PCR: Se siguió el protocolo propuesto por Struber [12] con modificaciones. Dentro de las modificaciones destaca la utilización de PCR touchdown; este constó de 10 ciclos con una temperatura de hibridación inicial de 65ºC, disminuyendo 1ºC cada ciclo; seguido de 30 ciclos con una temperatura de hibridación estable a 55ºC. Además se agregó dimetil sulfóxido como adyuvante. Los protocolos de trabajo ensayados y las condiciones de trabajo se detallan en la tabla 1. Las reacciones se realizaron en un Termociclador Gene Amp PCR System 2400, Perkin Elmer®.
Tabla 1. Pruebas realizadas en la estandarización de la técnica de PCR para la detección del gen invA en Salmonella spp.
| Reactivos | Protocolos de trabajo (µl) | ||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | |
| Buffer 10X | 1,0 | 1,0 | 1,0 | 1,0 | 1,0 | 1,0 | 1,0 |
| MgCl2 mM/µl | 1,5 | 2,0 | 2,0 | 2,7 | 1,3 | 3,3 | 2,0 |
| DMSO | - | - | - | - | 0,15% | 0,25% | 0,25% |
| dNTPs mM/µl | 0,5 | 0,5 | 0,5 | 0,5 | 0,5 | 0,5 | 0,5 |
| Taq polimerasa UI/ µl | 0,025 | 0,025 | 0,025 | 0,025 | 0,025 | 0,025 | 0,025 |
| Iniciadores µg/ µl | 4,0 x 10-5 | 8,0 x 10-5 | 2,0 x 10-4 | 4,0 x 10-4 | 4,0 x 10-4 | 4,0 x 10-4 | 1,0 x 10-4 |
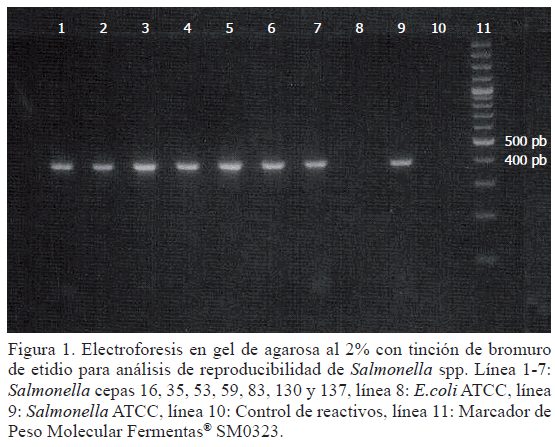
Visualización de los amplicones: Se realizó por medio de una electroforesis en gel de agarosa al 2% teñido con bromuro de etidio, cuya concentración en el gel fue de 0,5 μg/mL. La corrida se realizó a 140 V por 45 minutos en una solución amortiguadora TBE con pH de 8,3.
Purificación y secuenciación: La banda obtenida de 389 pb se purificó a partir del gel de agarosa utilizando un kit comercial (Qiakit de Qiagen®). Este producto fue secuenciado utilizando los mismos iniciadores que el PCR touchdown. Para la secuenciación se utilizó un equipo 310 ABI (Applied Biosystems®) del Centro de Investigación en Hemoglobinas Anormales y Trastornos Afines (CIHATA) de la Universidad de Costa Rica.
Reproducibilidad de la PCR: Se realizó extracción del ADN de 30 aislamientos de Salmonella spp. obtenidas del Laboratorio de Microbiología de Alimentos y Aguas de la Facultad de Microbiología y de la colección de microorganismos del Instituto de Investigaciones en Salud (INISA), de la Universidad de Costa Rica. Estos se detallan en la tabla 2. Para la extracción se aplicó la técnica de choque térmico, descrita anteriormente.
Tabla 2. Cepas utilizadas para la estandarización de la técnica de PCR para la detección del gen invA en Salmonella spp.
| Salmonella 009 | Salmonella 122 |
| Salmonella 016 | Salmonella 123 |
| Salmonella 021 | Salmonella 130 |
| Salmonella 023 | Salmonella 135 |
| Salmonella 031 | Salmonella 137 |
| Salmonella 033 | Salmonella 146 |
| Salmonella 035 | Salmonella 150 |
| Salmonella 036 | Salmonella enterica subsp. enterica ATCC 6539, originalmente Typhi |
| Salmonella 041 | Salmonella enterica subsp. enterica ATCC 14028, originalmente Paratyphi |
| Salmonella 049 | Salmonella enterica subsp. enterica ATCC 13076, originalmente Enteritidis |
| Salmonella 050 | Escherichia coli ATCC 25922 |
| Salmonella 053 | Shigella flexneri 397 |
| Salmonella 059 | Shigella flexneri (OP) |
| Salmonella 073 | Shigella sonnei 433 |
| Salmonella 082 | Enterobacter cloacae 296 |
| Salmonella 083 | Citrobacter freundii 294 |
| Salmonella 087 | Moraxella phenylpyruvica 136 |
| Salmonella 095 | Proteus mirabilis 13 |
| Salmonella 112 | Pseudomonas aeruginosa 28 |
| Salmonella 114 | Aeromonas hydrophila 35 |
| Plesiomonas shigelloides | Escherichia coli 64111 |
| Brevundimonas diminuta | Escherichia coli 286 |
| Escherichia coli 75C88 | Escherichia coli 34420 |
| Staphylococcus aureus |
|
Especificidad de la PCR: Se realizó extracción del ADN de las bacterias indicadas anteriormente utilizando la técnica ya descrita. El aislamiento por cultivo se realizó en agar sangre, agar tergitol 7 y agar DC incubando por 24 horas a 35ºC. Las enterobacterias fueron identificadas con el Sistema API 20E y las cepas no entéricas (S. aureus) se identificaron según protocolo de referencia [22].
Resultados
Estandarización de la técnica de PCR: A fin de determinar la concentración óptima de reactivos para la obtención de una amplificación adecuada del producto de 389 pb del gen invA, fue necesario realizar varias PCR. Se inició con las concentraciones de reactivos propuestas por Struber et al [12]. Debido a la que la amplificación no se logró bajo estas condiciones, fue necesario modificar las temperaturas y los tiempos en el termociclador, así como la realización de variaciones en concentración de MgCl2. Las distintas condiciones ensayadas en cuanto a concentración de reactivos se aprecian en la tabla 1.
Las variaciones con respecto al protocolo de amplificación fueron dirigidas a cambios en la temperatura de hibridación de los iniciadores con el fin de obtener un acoplamiento adecuado a la secuencia diana, así como la óptima polimerización del ADN. Los resultados no fueron óptimos, por lo que se procedió a utilizar una variación del PCR tradicional, conocida como PCR touchdown. La concentración de reactivos y temperaturas de la amplificación se indican en la tabla 1, procolo 7: Buffer Tris-HCl pH 8,3 1X, MgCl2 2,0 mM/µL, 0,2 mM/µL dNTPs, 1,0 x10-3 µg/µL, Taq 0,024 UI/µL y DMSO al 0,25%. Al evaluar el producto en geles de agarosa al 2%, se observó una banda bien definida de 389 pb (Figura 1).
Para garantizar que la banda observada realmente correspondiera al gen invA de Salmonella enterica, se secuenció el producto obtenido, el cual mostró alta identidad (281/282 o 99%, E-value=2X10-143) con el gen invA en los genomas de Salmonella enterica serovar Gallinarum, Salmonella enterica serovar Paratyphi, Salmonella enterica serovar Typhi, Salmonella enterica serovar Typhimurium y Salmonella enterica serovar Enteritidis, (base de datos del National Center for Biotechnology Information-NCBI), números de ingreso AM933173.1, FM200053.1, AL627276.1, EU348365.1, y AM933172.1, respectivamente).
Reproducibilidad de la técnica de PCR: Se analizó el ADN de 30 aislamientos de Salmonella spp. (identificadas bioquímica y serológicamente) conforme al protocolo de PCR estandarizado. En todos los casos se observó la banda correspondiente al gen invA (Figura 1).
Especificidad de la técnica de PCR: Se analizó el ADN de bacterias diferentes a Salmonella spp., sin observarse la banda de 389 pb (Figura 2).
PCR vs cultivo convencional: Se analizaron por cultivo varias porciones de 25 g de lechuga contaminadas con concentraciones conocidas de Salmonella spp. y se logró evidenciar por cultivo convencional y por PCR concentraciones de hasta 102 UFC/25 g. Los ADN extraídos del caldo lactosado suplementado con novobiocina no mostraron amplificación del producto de 389 pb.
Discusión
Luego de varias modificaciones del protocolo base [12] se logró la detección de un producto de 389 pb, del gen invA, específico de Salmonella spp., al utilizar la variante de PCR touchdown. Esta técnica de PCR mostró ser reproducible, específica y sensible para la detección de Salmonella spp. Es recomendable que la estandarización de una PCR se realice en cada laboratorio y para cada matriz utilizada; en nuestro caso, el protocolo original tuvo que modificarse considerablemente para que se adaptara a las condiciones del laboratorio.
La adición de DMSO permitió separar estructuras secundarias ricas en guanina-citosina durante los últimos ciclos de una reacción de PCR, así como está reportado en la literatura [21, 22]. Adicionalmente, con el aumento de la astringencia de la reacción, producido por el incremento en la temperatura de anillamiento durante la reacción de amplificación, se logra una acción sinérgica con la que se obtiene una amplificación óptima del ADN blanco al utilizar estos iniciadores. La utilización de adyuvantes de la PCR para mejorar su eficiencia y el conocimiento teórico de la temperatura de hibridación de los iniciadores, son puntos indispensables en la estandarización de un protocolo de PCR, por lo que los análisis teóricos previos al respecto son fundamentales para el trabajo de laboratorio [25].
La comparación cualitativa del método de PCR con el cultivo convencional, mostró una mayor eficiencia del PCR en cuanto a tiempo de procesamiento de muestra y obtención de resultados (36 a 48 horas), comparado con la técnica de cultivo (5-6 días). El nivel de detección fue el mismo para ambos métodos (102 UFC/25 g), marcando la diferencia básicamente el tiempo necesario para la obtención de resultados. Estos resultados se pueden explicar debido a la utilización del antibiótico novobiocina, recomendado para inhibir flora de competencia presente en la muestra, sin embargo, otros factores que posiblemente afectaron los resultados son las condiciones propias obtenidas para el PCR estandarizado.
Cabe destacar que la detección por PCR se realizó a partir del caldo de pre-enriquecimiento, lo cual es recomendado en la literatura para eliminar el efecto de inhibidores presentes en la matriz, además de aumentar la posibilidad de detectar microorganismos viables en el alimento [26].
La secuenciación del producto obtenido corrobora que el método de PCR detecta el gen invA de diferentes serovariedades de Salmonella enterica, lo cual garantiza la especificidad del método estandarizado y además es un requisito para la validación de cualquier método molecular para el diagnóstico de patógenos en alimentos, conforme a lo recomendado a nivel internacional [24].
Como conclusión, la técnica estandarizada, en nuestro laboratorio, es una herramienta base para la detección de Salmonella enterica que podría utilizarse a futuro para diferentes matrices, pues la transmisión de este patógeno se asocia no sólo con vegetales crudos sino con otros alimentos y agua contaminada. La detección de factores de virulencia, como el gen invA, brinda información que va desde la identidad de una bacteria determinada hasta su potencial virulento, ya que la producción de cuadros clínicos depende, en gran medida, de los genes de virulencia que posea la bacteria y, como beneficio adicional permite un resultado presuntivo en menor tiempo [20].
A su vez, en el Instituto de Investigaciones en Salud (INISA), entre otros trabajos, se ha desarrollado la metodología de detección simultánea de Salmonella spp. y Shigella spp. en muestras de lechuga (manuscrito en preparación) utilizando como base el presente protocolo, lo que evidencia la versatilidad de la técnica para detectar diferentes patógenos bacterianos de forma simultánea.
Referencias
1. World Health Organization. 2007. Fact sheet Nº 237: Food safety and foodborne illness. En: http://www.who.int/mediacentre/factsheets/fs237/en/. Acceso 17 de setiembre 2009. [ Links ]
2. Hein I, Flekna G, Krassning M, Wagner M. Real-time PCR for the detection of Salmonella spp. in food: An alternative approach to a conventional PCR system suggested by the FOOD-PCR project. J Microbiol Methods. 2006; 66:538-47. [ Links ]
3. World Health Organization. 2005. Fact sheet Nº 139: Drug-resistant Salmonella. En http://www.who.int/mediacentre/factsheets/fs139/en/index.html. Acceso 17 de setiembre 2009. [ Links ]
4. Galanis E, Lo Fo Wong D, Patrick M, Binsztein N, Cieslik A et al. Web-based surveillance and Global Salmonella distribution, 2000-2002. Emerg Infect Dis.2006; 12:381-8. [ Links ]
5. Mahon C, Manuselis G. Textbook of diagnostic Microbiology. 2da ed. Estados Unidos de Norte América: W.B. Saunders Company; 2000. [ Links ]
6. Wegener H, Hald T, Wong D, Madsen M, Kosgaard H, Bager F, et al. Salmonella control programs in Denmark. Emerg Infect Dis. 2003; 9:774.80. [ Links ]
7. Pérez-Ciordia I, Ferrero M, Sánchez E, Abadías M, Martínez-Navarro F, Herrera D. Enteritidis por Salmonella en Huesca. 1996-1999. Enferm Infecc Microbiol Clin. 2002; 20:16-21. [ Links ]
8. OBrian S. Foodborne zoonoses: Food poisoning can be serious, and doctors and vets have key roles in tackling it. BJM. 2005; 331:1217-8. [ Links ]
9. Baum, S. Zoonoses-with friends like this, who needs enemies?. Trans Am Clin Climatol Assoc. 2008; 119:39-52. [ Links ]
10. Zhang S, Kingsley A, Santos R, Andrews-Polymenis H, Raffatellu M, Figueiredo J et al. Molecular pathogenesis of Salmonella enterica serotype Typhimurium-induced diarrhea. Infect Immun. 2003; 71:1-12. [ Links ]
11. Ginocchio C, Galán J. Functional conservation among members of the Salmonella Typhimurium InvA family of proteins. Infect Immun. 1995; 63:729-32. [ Links ]
12. Stuber K, Frey J, Burnens A, Kuhnert P. Detection of type III secretion genes as a general indicator of bacterial virulence. Mol Cell Probes. 2003; 17:25-32. [ Links ]
13. Malorny B, Hoofar J, Bunge C, Helmuth R. Multicenter validation of the Analytical Accuracy of Salmonella PCR: towards in an International Standard. Appl Environ Microbiol. 2003; 69:290-6. [ Links ]
14. Daum L, Barnes W, McAvin J, Neidert M, Cooper L, Huff W, et al. Real-Time PCR detection of Salmonella in suspect foods from a gastroenteritis outbreak in Kerr County, Texas. J Clin Microbiol. 2002; 40:3050-2. [ Links ]
15. Ferretti R, Mannazzu I, Cocolin L, Comi G, Clementi F. Twelve-hour PCR-based method for detection of Salmonella spp. in food. Appl Environ Microbiol. 2001; 67:977-8. [ Links ]
16. Li Y, Mustapha A. Simultaneous detection of Escherichia coli O157: H7, Salmonella, and Shigella in apple cider and produce by a Multiplex PCR. J Food Prot. 2004; 1:27-33. [ Links ]
17. Vantarakis A, Kamininou G, Venieri D, Papapetropoulou M. Development of a multiplex-PCR detection of Salmonella spp. and Shigella spp. in mussels. Lett Appl Microbiol. 2000; 31:105-9. [ Links ]
18. Pouch-Downes F, Ito K. Compendium of methods for the microbiological examination of food. 4ta ed, Estados Unidos de Norte América: American Public Health Association (APHA), 1998. [ Links ]
19. González T, Rojas R. Enfermedades transmitidas por alimentos y PCR: prevención y diagnóstico. Salud Pública Mex. 2005; 47:388-90. [ Links ]
20. International Atomic Energy Agency (IAEA). Determination of human pathogen profiles in food by quality assured microbial assays. Proceedings of a final Research Coordination Meeting held in Mexico city, 2002. January 2005, IAEA-TECDOC-1431, 83-95. [ Links ]
21. FDA-CFSAN. Bacteriological analytical manual. 2007. En: http://www.fda.gov/Food/ScienceResearch/LaboratoryMethods/BacteriologicalAnalyticalManualBAM/ucm070149.htm. Acceso 17 de setiembre del 2009. [ Links ]
22. Soriano V, González-Lahoz J. III Curso de Biología molecular para clínicos. Barcelona: Publicaciones Permanyer; 2002. [ Links ]
23. Harris, E. A low-cost approach to PCR. Appropriate transfer of biomolecular techniques. New York: Ed. Oxford University Press; 1998. [ Links ]
24. ISO/FDIS 20838 Microbiology of food and animal feeding stuffs-Polimerase chain reaction (PCR) for the detection of food-borne pathogens-Requeriments for amplification and detection of qualitative methods. 2006. [ Links ]
25. AOAC. AOAC International Methods Committee Guidelines for validation of qualitative and quantitative food microbiological official methods of analysis. 2002. En: http://www.aoac.org/vmeth/Manual_App_X.pdf. Acceso 11 de agosto del 2009. [ Links ]
26. Lauri A, Mariani P. Potentials and limitations of molecular diagnostic methods in food safety. Genes Nutr. 2009; 4:1-12. [ Links ]












 uBio
uBio