Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Comunidad y Salud
versión impresa ISSN 1690-3293
Comunidad y Salud vol.14 no.1 Maracay jun. 2016
PERFIL GENÉTICO HLA EN FAMILIAS VENEZOLANAS: ÚTIL HERRAMIENTA BIOMÉDICA.
Normig Zoghbi1, Paolo Tassinari2, María del Pilar Fortes2, María Paredes2, Ligia Gámez2, Marina Palacios2
1 Departamento de Bioquímica y Fisiología, Escuela de Medicina "Dr Witremundo Torrealba", Universidad de Carabobo-Núcleo Aragua.
2 Sección de Inmunogenética, Instituto de Inmunología, Facultad de Medicina. Universidad Central de Venezuela. Correspondencia: normig@hotmail.com
RESUMEN
El sistema de antígenos leucocitarios Humano (Human Leukocyte Antigen, HLA) regula la respuesta inmune mediante su unión a moléculas como el receptor de células T, participando en la presentación de antígenos y el reconocimiento de lo propio en el organismo. La caracterización genética del sistema HLA en determinada población es de gran utilidad para la comprensión de mecanismos asociados a susceptibilidad o resistencia a enfermedades y en la selección de donantes/receptores en trasplantes de órganos. En este estudio se planteó determinar la frecuencia de los Haplotipos HLA de clase I presentes en individuos sanos, relacionados familiarmente y venezolanos de tercera generación y su correspondiente desequilibrio de ligamiento. Se incluyeron 765 individuos pertenecientes a 218 familias a los cuales se les realizó tipificación HLA A y HLA B por PCR-SSOP (polymerase chain reaction-sequence specific oligonucleotide probe) en baja resolución. Se identificaron 265 haplotipos de los cuales los más frecuentes fueron HLA A*24 B*35 (11,98 %), A*02 B*51 (9,7%) y A*02 B*35 (8,6 %).Para los cálculos de desequilibrio de ligamiento se consideraron las frecuencias mayores al 1% (28,7%) y no arrojaron valores estadísticamente significativos el 6,78% de estos haplotipos. Los resultados obtenidos corroboran la composición triétnica históricamente conocida de nuestra población, en la cual predominan genes caucásicos, amerindios y afrodescendientes; y su porcentaje marca la diferencia con otras poblaciones americanas estudiadas. Estos resultados representan una aproximación de la conformación genética establecida en Venezuela y aporta datos que podrán ser usados como referencia en programas de salud para la población.
PALABRAS CLAVE: Frecuencia de Haplotipos, Haplotipos HLA A-B, Repuesta inmune
HLA GENETIC PROFILE IN VENEZUELAN FAMILIES: BIOMEDICAL USEFUL TOOL
ABSTRACT
The system of Human leukocyte antigens (HLA) is the most polymorphic in humans. Its function is performed by regulating the immune response by binding to molecules such as T-cell receptor, involved in antigen presentation and recognition of the same in the body. Genetic characterization of HLA system in a given population is useful for understanding the mechanisms associated with susceptibility or resistance to various diseases and selection of donors and recipients in organ transplants, among others. The objective of the present study is to determine the frequency of HLA Class I Haplotypes present in healthy individuals, family relationships and third-generation Venezuelan and their corresponding linkage disequilibrium. We included 765 individuals belonging to 218 families who underwent HLA typing HLA A and B by PCR-SSOP (polymerase chain reaction-sequence specific oligonucleotide probe) in low resolution. 265 haplotypes were identified of which the most frequent were HLA A * 24 B * 35 (11.98%), A * 02 B * 51 (9.7%) and A * 02 B * 35 (8.6%). Calculations of linkage disequilibrium were considered frequencies above 1% (28.7%) and did not show statistically significant the 6.78% of these haplotypes. The results support the historically known tri-ethnic composition of our population, in which genes predominantly Caucasian, Mongoloid and Negroid, and make a difference with other American populations studied. These data can be used as reference to applications of benefit to this population.
KEY WORDS: Haplotype frequency, HLA haplotypes AB, Inmune response.
Recibido: 05/02/2015 Aprobado: 20/01/2016
INTRODUCCIÓN
Las moléculas del sistema HLA (Antígenos Leucocitarios Humanos, acrónimo en inglés de Human Leucocyte Antigen), pertenecen al llamado Complejo Principal de Histocompatibilidad. Su función principal se resume como la regulación de la respuesta inmune mediante una unión al receptor de células T durante la presentación de antígenos, o el reconocimiento de lo propio en el cuerpo, o a receptores en las células NK (natural Killer). Son moléculas encargadas de dirigir funciones muy importantes dentro de la homeostasis del organismo, que van desde la regulación de la respuesta inmune celular y humoral, pasando por la presentación de antígenos de patógenos intracelulares a los linfocitos T citotóxicos (LTc) encargados de la respuesta inmune efectora, hasta ser la fuente de adaptación más importante con los microorganismos que nos rodean e interactuamos a diario y cuya gran diversidad y cambio constante ha inducido una enorme variabilidad en este sistema genético. Toda esta gama de funciones fisiológicas que cumplen las moléculas HLA hacen que se vean asociadas con diversas enfermedades, especialmente con las de tipo autoinmune, de tal manera que la respuesta del sistema inmune a la infección es, al menos en parte, dependiente de las diferentes combinaciones de la herencia del sistema.
Los genes del complejo HLA se encuentran en el brazo corto del cromosoma 6 (6p21) y su región comprende 3600 kb; se divide en tres regiones denominadas HLA-I, - II y -III (Figura 1). La región del HLA-I comprende los genes del HLA-A, -B, y -C cuyos productos, las cadenas, son expresados en la amplia mayoría de las células nucleadas y plaquetas, con la excepción de los espermatozoides y las células trofoblásticas .Dicha cadena α se une no covalentemente a la ß-2 microglobulina codificada por un gen no polimórfico localizado en el cromosoma 15, para formar el complejo proteico maduro. La región del HLA-II incluye los genes de los productos HLA-DR, DQ y DP, los cuales están constituidos por 2 cadenas, una α y otra ß, y son expresados únicamente en las células presentadoras de antígenos, linfocitos B y linfocitos T activados y células endoteliales 3. Por su parte, la región HLA-III codifica otros componentes del sistema inmune, como proteínas del complemento y el factor de necrosis tumoral. La región HLA-I se encuentra telomérica, mientras que la HLA-II es más centromérica, con la HLA-III entre éstas. El conjunto de HLA-DR posee un gen para cadena α y 4 genes para cadenas ß (DRB1, 3, 4, 5), mientras que para el HLA-DQ y -DP solamente hay un gen productor de cadenas α y otro para cadenas ß funcionales.1-3
Figura 1. Organización de los genes HLA en el cromosoma 6 región P21. Disponible en: http://www.scielo.org.ve/imp/fbpe/ic/v5on3/art13fig1.9i
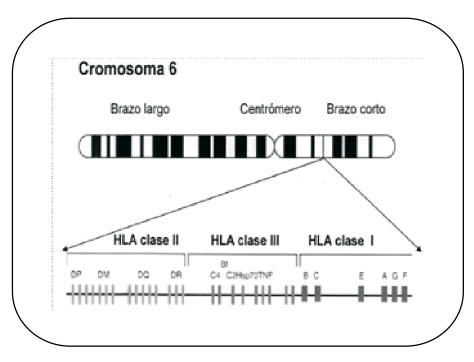
Los genes del HLA poseen un patrón de herencia mendeliano, con la presencia de haplotipos con frecuencias específicas en cada población humana y un ligamiento genético importante con alrededor de un 1% de recombinación. El sistema HLA debido a su extremo polimorfismo, cuyo conocimiento se ha incrementado con el análisis molecular, constituye una poderosa herramienta en la investigación de una serie de problemas relacionados con la biología molecular, la inmunogenética, trasplante de órganos y tejidos, susceptibilidad a las enfermedades y variabilidad inter e intrapoblacional.4-8
Actualmente se han identificado 3285 variedades de genes HLA-A, 4077 de HLA-B, entre otros genes (IMGT (the international ImMunoGeneTics information system)/HLA data base, Enero 2016), es decir, las diferentes poblaciones se distinguen por las frecuencias en que se encuentran los haplotipos HLA y por ello es fundamental analizar el perfil genético poblacional, con primordial utilidad en los programas de trasplante de órganos y tejidos en los cuales se hace difícil encontrar un individuo con perfil HLA compatible con otro.
Por la complejidad del sistema HLA y por razones de costo y tiempo, en América Latina la mayoría de las poblaciones estudiadas con el sistema HLA son preferentemente indígenas, aisladas o escasamente mestizadas. Los datos provenientes de poblaciones urbanas mestizas latinoamericanas son menos abundantes y estos corresponden a los siguientes países: Brasil, México, Uruguay, Argentina, Cuba y Colombia, entre otros. En Venezuela son escasos los estudios poblacionales en este sentido, y la mayoría de los trabajos publicados hasta el momento muestran frecuencias alélicas basadas en técnicas menos precisas (serología) o han sido realizados en poblaciones indígenas,9-18 lo que explica que características bioantropológicas y genéticas de la población Venezolana son limitadas.
Venezuela posee una población de 30.620.404 habitantes para el año 2015.19 La mayor parte de los venezolanos descienden de inmigrantes europeos, de indígenas amerindios venezolanos y de africanos traídos al país como esclavos en la época colonial y todos han contribuido en diversos grados a la composición étnica y cultural de Venezuela. Se ha reportado que tenemos un 50% de genes amerindios, 40% europeos y 10% negroides, basados en estudios del grupos sanguíneos, de distribución de apellidos, entre otros. La población restante está constituida por indígenas amerindios en una proporción estadística mucho menor (primordialmente tribus indígenas Wayúus, Timoto-cuicas y Yanomamis).20 Estudios de perfil genetico HLA en Venezuela Con relación a la población mestiza venezolana los estudios poblacionales que han empleado el sistema HLA han utilizado técnicas serológicas y moleculares y estimación de haplotipos de Clase I y Clase II empleando programas de genética estadística computarizada. Cuando se plantea estudiar haplotipos del sistema HLA es importante destacar que el mismo presenta una fuerte tendencia al desequilibrio genético (Desequilibrio de ligamiento, D), por lo que sus genes se heredan en bloque en una frecuencia muy alta (esto aunado al alto grado de polimorfismo, produce una gran especificidad).13
Los alelos que se heredan de manera conjunta en un cromosoma constituyen un haplotipo, los cuales se determinan (como en la mayoría de las investigaciones) por estimación a partir de los alelos encontrados en individuos no relacionados a través de programas genéticos estadísticos o de forma más fidedigna mediante el análisis de la segregación de alelos en familias. Es por ello y dada la importancia genética e inmunológica del sistema HLA, se planteó determinar las frecuencias haplotípicas HLA de Clase I en familias venezolanas y sus respectivos desequilibrios de ligamiento. Conocer los Haplotipos HLA más frecuentes en individuos venezolanos es de gran utilidad para los programas de trasplantes, para el estudio del origen y la evolución de los diferentes grupos étnicos y en muchas especialidades médicas relacionadas con enfermedades genéticamente asociadas al sistema HLA debido a que estos genes pueden actuar como determinantes de susceptibilidad o resistencia a enfermedades. 21-25
MATERIALES Y MÉTODOS
Se realizo un estudio descriptivo mediante el cual se analizaron muestras sanguíneas de 764 individuos de una lista de posibles donantes para trasplante de médula ósea, relacionados familiarmente (familiares del afectado que acudieron al Instituto de Inmunología de la Universidad Central de Venezuela IDI-UCV para realizar pruebas de compatibilidad genética basado en su haplotipo HLA requerida para trasplante), sanos, venezolanos de tercera generación y distribuidos en 218 familias. Estos individuos pertenecían a distintos estratos sociales de nuestra población y provenían de diferentes zonas geográficas del país, por lo que la posibilidad de tener un sesgo social en la muestra fue mínima. Las muestras de sangre fueron colectadas previo consentimiento informado entre Enero de 2.005 hasta Agosto de 2.008. A cada muestra se le realizó la tipificación HLA en el Laboratorio de Inmunogenética IDI-UCV. La tipificación HLA inicial (receptor y posibles donantes) para verificación de compatibilidad se realizó según lo convencional para los locus HLA-A y HLA-B en baja resolución (dos dígitos para cada alelo). El estudio fue aprobado por el Comité de Bioética del IDIUCV.
La extracción de ADN se realizó usando la técnica de precipitación salina del material genético presente en glóbulos blancos aislados de muestras de sangre anticoaguladas con EDTA,26 mediante un kit comercial de la compañía Qiagen (QIAmp DNA Mini Kit). La determinación de los alelos de Clase I HLA A-B se realizó mediante técnica de amplificación por reacción en cadena de la polimerasa con hibridación con sondas oligonucleotídicas de secuencia específica marcadas, y revelado enzimático, denominada técnica PCR-SSOP.27 La asignación alélica se realizó mediante el análisis de los patrones amplificados empleando la tabla de clasificación suministrada por el fabricante (Dynal Reli SSO HLA-A /HLA-B typing kit).
Análisis Estadístico
Se realizó previamente en la base de datos la homologación del grado de resolución de la tipificación HLA a dos dígitos. La frecuencia de haplotipos de dos loci (HLA A-B) se logró por contaje directo determinando los haplotipos de cada familia (dividendo el número de veces que aparece un haplotipo entre el total de Haplotipos por cien); dichas frecuencias obtenidas se corroboraron por el método de máxima verosimilitud usando el programa Arlequin v.3.0. El cálculo de desequilibrio de ligamiento (D) para cada haplotipo se realizó según la fórmula postulada por Lewontin y Kojima.
[Dij = Pij - Pi Pj; donde tanto i como j son alelos distintos que conforman un haplotipo]
El cálculo de desequilibrio de ligamiento estandarizado (D') por el valor máximo que pueda tener d se realizó según la fórmula postulada por Lewontin. 28
D'ij = Dij min (Pi Pj (1-Pi) (1-pP)) si Dij < 0
Dij max min ((1-Pi)Pj- , Pi(1-Pj)) si Dij > 0
Además se calcularon los coeficientes r2. El rango de valores de desequilibrio de ligamiento va de +1 a -1, un desequilibrio de ligamiento de 0 indica equilibrio de enlace genético, mientras que un desequilibrio de +1 indica una asociación de un par de alelos presentes en un haplotipo y un valor de -1 indica una asociación no significativa o ausencia de asociación entre esos dos alelos.
RESULTADOS
El sistema HLA ha sido ampliamente estudiado debido a la gran cantidad de alelos que posee cada locus, así como también por la variación que presentan estos alelos en las diferentes poblaciones y por sus implicaciones en la respuesta inmunológica. Diversos autores han reportados datos de frecuencia génica para el sistema HLA en los grupos étnicos mayoritarios, caucasoides, negroides y amerindios. Como se puede apreciar, los alelos HLA A*01, A*03, B*07, B*44 presentan una mayor frecuencia en los grupos caucasiodes; por otra parte, los negroides exhiben una mayor frecuencia de los alelos HLA A*23, A*30, B*53 y B*58. Finalmente los grupos amerindios exhiben una mayor frecuencia de los alelos HLA A*02, A*24, A*28, B*05, B*15, B*35, B*39 y B*40, entre otros.29 A partir de los resultados obtenidos en el presente estudio se observa que en las 218 familias analizadas se encontraron 265 haplotipos para los locus HLA A-B siendo los más frecuentes HLA A*24-B*35 (11,98%), HLA A*02-B*51 (9,81%), HLA A*02-B*35 (8,30%), HLA A*02-B*39 (5,66%), HLA A*02-B*44 (5,28%), HLA A*11-B*35 (5,28%), entre otros (Figura 2); siendo estos datos confiables, al ser obtenidos por su segregación en la familia.
La alta frecuencia obtenida del haplotipo HLA A*24-B*35 se puede explicar por la influencia de la población amerindia, ya que estos alelos son predominantes en este grupo étnico venezolano y refleja la posible mezcla que ocurrió con la población nativa. Los haplotipos HLA A*02-B*44 y HLA A*02-B*07 cuyas frecuencias se destacaron entre las más altas, son de los más característicos en la población europea; reflejando la gran influencia de los genes de origen caucásico en nuestros mestizos.
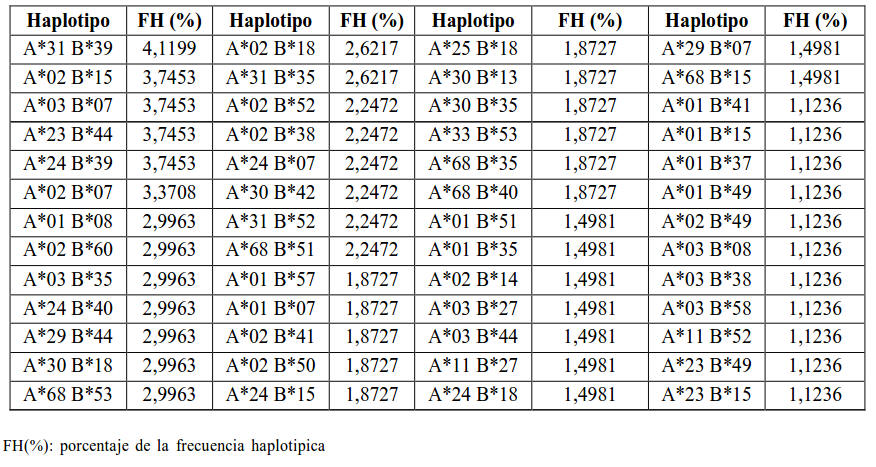
El polimorfismo HLA brinda al individuo un mejor patrón de respuesta inmunológica frente a los agentes infecciosos. Las variantes nuevas pudieran ser de particular ventaja evolutiva ya que frente a estas los patógenos aún no han logrado evolucionar para evitar la presentación de sus péptidos al sistema inmune por estas variantes HLA. Para el análisis de los desequilibrios de ligamientos solo se consideraron los haplotipos con frecuencias iguales o superiores al 1%, lo cual representó 28,7% haplotipos HLAA-B (Tabla 1). Los valores de desequilibrio de ligamiento fueron significativos (p<0,0001) en 71 de los 76 haplotipos HLA A-B. De estos los valores no significativos (haplotipos en equilibrio genético Hardy Weinberg) representaron 6,58 % de los haplotipos HLA-A-B (recordando que tan solo se consideraron haplotipos con frecuencias mayores al 1% para el cálculo de D, datos no mostrados) y resultaron ser: A*01 B*35, A*02 B*14, A*24 B*51, A*24 B*44, A*24 B*53.
Estos datos también arrojaron una diferencia con la población vecina de Colombia donde reportaron como valores de desequilibrio de ligamiento sin significancia estadística a los haplotipos A*02 B*44, esta investigación, se encuentran dentro de los 15 más frecuentes en población caucásica, siendo los haplotipos A*02 B*35, A*24 B*35 los más comunes en el grupo de latinos, y A*02 B*44 uno de los más comunes en caucásicos.23
Los venezolanos somos producto de un mestizaje entre aborígenes mongoloides, inmigrantes europeos caucasoides e individuos de raza negra principalmente del oeste de África. Estos tres grupos, representan el modelo de población existente en los países Iberoamericanos, que han tenido la influencia de las poblaciones Europeas y Africanas sobre el sustrato Amerindio (nativo) inicial.24
Al comparar los distintos haplotipos observados en este estudio con otros realizados en poblaciones clasificadas según el grupo étnico, se encontró que estos corresponden a los haplotipos más frecuentes en población hispánica, latinoamericana y caucásica. Es así como los haplotipos A*01 B*08, A*24 B*35, A*02 B*44, A*03 B*07, A*29 B*44, A*03 B*35, A*02 B*51, A*11 B*35 y A*02 B*35, presentes en población latinoamericana y en frecuencia alta en esta investigación, se encuentran dentro de los 15 más frecuentes en población caucásica, siendo los haplotipos A*02 B*35, A*24 B*35 los más comunes en el grupo de latinos, y A*02 B*44 uno de los más comunes en caucásicos.23
DISCUSIÓN
La población venezolana comparte la mayoría de sus alelos y Haplotipos con otras poblaciones intracontinetales, sin embargo, aunque se observan
Figura 2. Haplotipos HLA A-B más frecuentes en familias venezolanas, n= 764
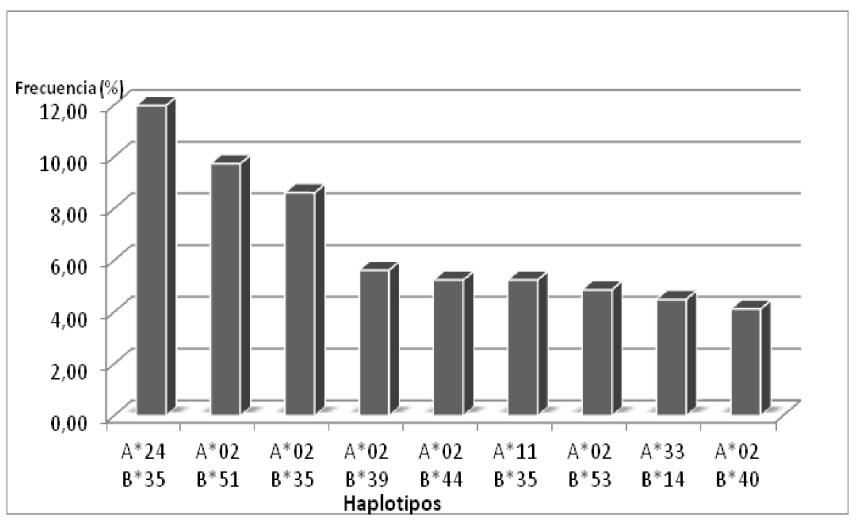
Tabla 1. Frecuencia de haplotipos encontrados en 218 familias venezolanas
A*02 B*18, A*02 B*61, A*24 B*07 y A*11 B*35.Los venezolanos somos producto de un mestizaje entre aborígenes mongoloides, inmigrantes europeos caucasoides e individuos de raza negra principalmente del oeste de África. Estos tres grupos, representan el modelo de población existente en los países Iberoamericanos, que han tenido la influencia de las poblaciones Europeas y Africanas sobre el sustrato Amerindio (nativo) inicial.24 Al comparar los distintos haplotipos observados en este estudio con otros realizados en poblaciones clasificadas según el grupo étnico, se encontró que estos corresponden a los haplotipos más frecuentes en población hispánica, latinoamericana y caucásica. Es así como los haplotipos A*01 B*08, A*24 B*35, A*02 B*44, A*03 B*07, A*29 B*44, A*03 B*35, A*02 B*51, A*11 B*35 y A*02 B*35, presentes en población latinoamericana y en frecuencia alta en frecuencias similares, algunos Haplotipos están muy aumentados y otros disminuidos, lo cual confiere características propias a cada una de las poblaciones americanas; esto puede explicarse por el origen de los flujos inmigratorios hacia la República Bolivariana de Venezuela.
Los dos grupos inmigratorios más importantes en la conformación de la población venezolana entre los siglos XV y XVIII, fueron los españoles, los negros africanos y los indígenas, quienes poblaron nuestro territorio e influyeron decisivamente nuestro perfil genético.30 A partir de las dos primeras décadas del siglo XIX comienzan a establecerse en el país algunas casas comerciales extranjeras alemanas, inglesas, francesas e italianas; y luego de la independencia, entre 1830 y 1900, se puede estimar que ingresaron al país unos 67.000 extranjeros, principalmente de las Islas Canarias y la España peninsular, seguidos, muy de lejos, por italianos, alemanes y franceses; con un flujo inmigratorio hacia Venezuela relativamente pequeño en comparación con algunos otros países de la región.20 Dicho panorama cambió en el siglo XX, a consecuencia de la dramática situación de guerras que vivió Europa. Durante ese período Venezuela se benefició de los movimientos inmigratorios como fuerza de trabajo para la sociedad. En el lapso comprendido entre 1948 y 1961 ingresaron al país cerca de 800.000 extranjeros, cuyo 78% correspondían a españoles (de las Islas Canarias y Galicia), italianos (del sur y del centro), portugueses (principalmente de la isla de Madeira), colombianos y estadounidenses; hasta que a principios de la década de 1970, con el incremento de los precios del petróleo, volvió a crecer la inmigración, a un ritmo mayor que el experimentado anteriormente.
Esta segunda oleada inmigratoria del siglo XX fue diferente a la primera: esta vez la inmigración procedente de los países sudamericanos superó a la europea. A ella se agregaron argentinos, chilenos y uruguayos, expulsados por las dictaduras militares y, luego, los inmigrantes peruanos, ecuatorianos y dominicanos, en procura de empleo y mejores condiciones de vida.31 Dichos inmigraciones han marcado el acontecer nacional en muchos sentidos de la vida social, cultural, económica, política, deportiva y han dejado una profunda huella en nuestro patrón genético.
En el presente estudio fueron hallados 265 haplotipos para los locus HLA A-B en 218 familias analizadas, a partir de los cuales en 28,7 % de los haplotipos se obtuvieron frecuencias superiores a 1%; siendo el haplotipo HLA A*24-B*35, el de mayor frecuencia en la muestra analizada, y el mismo ha sido reportado también como el de mayor frecuencia en otras investigaciones realizadas en la población venezolana, pero en diferentes porcentajes; los individuos en los que se identifique este haplotipo tendrían más oportunidad para un trasplante de medula ósea. El haplotipo A*02-B*51 arroja la segunda frecuencia predominante y difiere de lo reportado para la población colombiana (3,42%)16 y se asemeja a lo publicado anteriormente en la población venezolana24 (10,79%) al igual que el haplotipo A*02-B*35 (8,30%), el cual fue también uno de los más frecuentes en Amerindios de México, Guatemala y Bolivia. Los haplotipos caucásicos A*0101-B*0801 (Técnica empleando alta resolución que permite caracterización por cuatro dígitos), A*2902- B*4403 y A*3301-B*1402 presentes en varias poblaciones europeas están dentro de los más frecuentes en el estudio.32
Cabe mencionar que aunque los resultados de este estudio están de acuerdo con lo encontrado en otras poblaciones, la distribución de las frecuencias haplotípicas y sus valores de desequilibrio de ligamiento fueron diferentes, lo cual evidencia la variación a nivel interpoblación existente y corrobora la mezcla racial en nuestra población, con un alto grado de influencia caucásica y enriquecida con la contribución africana y europea (principalmente, aunque no exclusivamente). El mapeo de la estructura genética de haplotipos HLA de un país, simultáneo a la difusión e incentivo hacia la cultura sanitaria de donación de tejidos y órganos por parte de sus habitantes, es de utilidad inmediata en el cálculo de la probabilidad de encontrar un donante compatible para determinado receptor; en nuestro caso particular a través de la Organización Nacional de Trasplante de Venezuela (ONTV) y permite entre otros beneficios establecer políticas sanitarias de intercambio de tejidos y órganos con otros países, lo que ayudaría a mejorar la presente crisis deficitaria del sistema de salud nacional, porque se invierten muchos recursos en terapias de mantenimiento (un ejemplo es cuando existe disfunción renal y se aplica tratamiento de hemodiálisis) a muchos pacientes a quienes con un trasplante se pudiera aumentár su probabilidad de vivir más y con calidad de vida y para quienes es difícil o nula la probabilidad de encontrarles un donante.
La caracterización poblacional nos permite adentrarnos no solamente en el estudio de las causas, sino además en el de los mecanismos de inmunidad dependiente de la frecuencia. Obviamente habría que hacer un estudio genético de Haplotipo HLA relacionado con determinada enfermedad, estudiando con que enfermedades están relacionados estos haplotipos más frecuentes, determinar cuáles son los haplotipos que confieren resistencia a una patología y cuales confieren predisposición genética, y para el desarrollo de nuevas vacunas y fármacos efectivos. Esta investigación aporta información de gran utilidad para estudios antropogenéticos, de asociaciones entre HLA y el desarrollo de ciertas condiciones de salud tales como: celiaquía, artritis reumatoide, hepatitis, HIV, aborto, entre otras25; aclarando que en las investigaciones de las últimas décadas se ha demostrado que nuestro destino y estado de salud no depende solo de nuestros genes, porque la información genética es más que secuencia de unidades continuas en los cromosomas; somos el resultado de un intercambio entre la información que heredamos de nuestros padres y la que incorporamos desde el ambiente, de la interacción compleja entre señales físicas, moléculas, microorganismos internos y externos, de la relación con nuestros congéneres, de nuestros actos sociales, de nuestras emociones e historia de vida,33 intercambio que nos define como sujetos más que cualquier patrón de genes y que determinará nuestro estado de salud. Los datos aportados en este estudio pueden considerarse como característicos de la muestra estudiada y solamente pueden ser extendidos a la población asumiendo la hipótesis de homogeneidad en la distribución de los haplotipos en la misma, resultando interesante y de mucha utilidad continuar el análisis de nuestro perfil genético HLA.
AGRADECIMIENTOS
Al personal del laboratorio de Inmunogenética del Instituto de Inmunología de la Universidad Central de Venezuela quienes con su apoyo incondicional facilitaron todos los requerimientos para realizar este estudio.
REFERENCIAS BIBLIOGRÁFICAS
1) Robinson J, Halliwell J.,Hayhurst J., Flicek P., Parham P. y Marsh S. The IPD and IMGT/HLA database: allele variant databases. Nucleic Acids Research. 2015; 12(1):423-431. [ Links ]
2) The IMGT/HLA Database of HLA allele as named in the WHO Nomenclature Committee Reports. Disponible en:
http://www.ebi.ac.uk/imgt/hla/allele.html. Consultado el 12 Enero de 2016.
3) Alper C., Larsen C., Dubey D., Awdeh Z., Yunia E. The haplotype structure of the human mayor histocompatibility complex. Hum. Inmunol. 2006; 67: 73-84. [ Links ]
4) Chang A., Bencomo A., Morera L., Ustáriz C., de la Guardia O. Evolución de la nomenclatura de los factores del sistema de antígenos leucocitarios humanos Revista Cubana de Hematol, Inmunol y Hemoter. 2014; 30(1):11-20. [ Links ]
5) Claas F., Roelen D., Mulder A., Doxiadis I., Oudshoorn M. y Heemskerk M. Differential immunogenicity of HLA Class I alloantigens for the humoral versus the cellular immune response: "Towards tailor-made HLA mismatching". Humn Inmunol. 2006; 67:424-9. [ Links ]
6) Caputo M., Cerrone G., López A., Gonzalez C., Mazza C., Cedola N. Genotipificación del gen HLA DQB1 en diabetes autoinmune del adulto (LADA) Medicina Buenos Aires. 2005; 65: 235-40 [ Links ]
7) Garavito G., Iglesias A., Egea E., Jaraquemada D., Martínez P.y Egea E. Una aproximación al significado biológico del polimorfismo del complejo mayor de Histocompatibilidad. El modelo de la asociación HLA y ARJ. SaludUninorte. 2002; 16: 53-72. [ Links ]
8) Domínguez M., Lorenzo N., Barbera A., Hernández M., Torres A., Nazabal M. Caracterización de moléculas HLA tipo II y evaluación de citocinas en pacientes cubanos con artritis reumatoide. Rev Cub Reumatol. 2006; 8: 9-10. [ Links ]
9) Layrisse Z., Heinen H., Balbas O., Garcia E., Stoikov Z. Unique HLA-DR/DQ associations revealed by family studies in Warao Ameridians. Haplotype and homozygisity frequencies. Human Inmunol. 1988; 23-45. [ Links ]
10) Gorodesky C., Loon J., Molitermo R., Torres E. y Pelegrino J. HLA in some latin American populations: Mexicans, brazilans, venezuelans and uruguayans. Proceddings of the eleventh International histocompatibility Workshop and conference. Vol 1 Oxford, UK, Oxford University Press. 1991. [ Links ]
11) Vullo C., Delfino L., Angelini G., Ferrara G. HLA polymorphism in a Mataco South American Indian tribe: serology of Class I and II antigens. Hum Immunol. 1992; 35: 209-214. [ Links ]
12) Cerna M., Falco M., Friedman H., Raimondi E., Maccagno A., Fernández M., Stasny P. Differences in HLA Class II alleles of isolated South American indians populations from Brazil and Argentina. Hum Immuno. 1993; 37: 213-220. [ Links ]
13) Bengochea M., Álvarez I., Hidalgo P., Cabrera A., Senatore O., Toledo R., et al . HLA-A, -B, -DR en receptores de trasplante de médula ósea en Uruguay. Sistema nacional de registro, tipificación y búsqueda de donantes de médula ósea y progenitores hematopoyéticos (SINDOME). Rev. Méd. Urug. 2003; 19(2): 149-158. [ Links ]
14) Layrisse Z., Guedez Y., Dominguez E., Herrera F., Soto M. Extended HLA haplotypes among de Bari Ameridians of the Perija range: Relationship to other tribes base don tour-loci haplotype frecuencies. Human Inmunol. 1995; 44:228. [ Links ]
15) Alfaro E., Dipierri J., Gutiérrez N. y Vullo C. Frecuencias génicas y haplotípicas del sistema HLA en el Noroeste argentino. Antropo, 6, 15-23. Disponible en: www.didac.ehu.es/antropo . 2004. [ Links ]
16) Rodríguez L., Giraldo M., García N., Velásquez L., París S., Álvarez C., García L. Frecuencias alélicas, genotípicas y haplotípicas HLA-A, HLA-B, HLA-DRB1 en donantes fallecidos, Medellín, Colombia. 2007. Biomédica. 2007; 27 (4): 537-47. [ Links ]
17) Echeverría M., Rivera S., Hassanhi M., Fuenmayor A., Márquez G., González M. y Zabala W. Alelos del complejo principal de histocompatibilidad clase II DRB1*/DQB1* de la población wayúu de la Guajira venezolana. Opción; 2008; 56: 44 - 66. [ Links ]
18) Ustáriz C., Morera L., Hernández P., Estrada M., Bencomo A., García M., et al. Origen y composición genética de la población cubana. Rev Cubana Hematol Inmunol Hemoter; 2011; 27(3): 273-282. [ Links ]
19) Instituto Nacional de Estadística de la República Bolivariana de Venezuela. Disponible en: http://www.ine.gov.ve/ . Consultado el 07 de Marzo de 2015. [ Links ]
20) Curcio M. Venezolanos heredan enfermedades genéticas de sus ancestros indígenas. Boletín Informativo Semanal, BIS, Nº 132. 2004. [ Links ]
21) Makhatadze N., Franco M. y Layrisse Z. HLA Class I and Class II Allele and Haplotype Distribution in the Venezuelan Population. Human Inmunol; 1997; 55: 53-58. [ Links ]
22) Kunze-Schumacher H., Blasczyk R. y Bade-Doeding C. Soluble HLA technology as a strategy to evaluate the impact of HLA mismatches. Journal of Immunology Research, vol. 2014, Article ID 246171. 2014. [ Links ]
23) Fortes M, Tassinari P., Machado I.. Genética en hepatitis autoinmune. Gen, 62(4), 318-322. Recuperado en 07 de enero de 2016, de http://www.scielo.org.ve/scielo.php?script=sci_arttext&pid=S0016-35032008000400014&lng=es&tlng=es. 2008 [ Links ]
24) Fortes M., Gil G., Paredes M., Gámez L., Palacios M., Tassinari P. Distribución de los alelos y haplotipos de los antígenos leucocitarios clase I y Clase II en la población venezolana. Rev. Ven Alerg Asma Inmunol. 2008; 8 (1): 12-16. [ Links ]
25) González-Sánchez L. Aplicación de HLA en el diagnóstico de enfermedades crónico-degenerativas. Revista electrónica Medicina, Salud y Sociedad. 2014; 5 (1); 70-76. Disponible en: http://cienciasdelasaluduv.com/site/. [ Links ]
26) Miller .S, Dykes D. y Polesky F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Reserch. 1988; 16 (3): 1215. [ Links ]
27) Middleton D. y Williams F. PCR-Sequence-Specific Oligonucleotide Probe Typing for HLA-A, -B, and -DR. Libro de Protocolo. MHC Protocols. Vol 210 of the series Methods in Molecular Biology. 2003; 67-112. [ Links ]
28) Single R., Martin M., Meyer D.,Gao X. y Carrington M. Methods for assessing gene content diversity of KIR with examples from a global set of populations. Immunogenetics. Dec. 2008; 60(12): 711-725. [ Links ]
29) Rothhammer F. y Llop E. Poblaciones chilenas: cuatro décadas de investigaciones bioantropológicas. Universidad de Chile. Programa de Genética Humana. Editorial Universitaria. Capitulo 10: Sistema Mayor de Histocompatibilidad Humano, HLA. 2004. [ Links ]
30) Rodríguez-larralde A., Castro D, González-Coira M y Morales J. frecuencia génica y porcentaje de mezcla en diferentes áreas geográficas de Venezuela, de acuerdo a los grupos RH y ABO. Inverciencia. 2001; 26(1): 8-12. [ Links ]
31) Cartay R. Aportes De Los Inmigrantes A La Conformación Del Régimen Alimentario Venezolano En El Siglo XX. Agroalimentaria. 2005; 10(20): 43-55. [ Links ]
32) Middleton D., Williams F., Meenagh A., Daar A., Gorodezky C., Hammond M., et al. Analysis of the distribution of HLA-A alleles in populations from five continents. Hum inmunol. 2000; 61(10):1048-52. [ Links ]
33) Heredia D. El mito del gen: genética, epigenética y el bucle organismo ambiente. Medicina naturista.2012; 6(1): 39-42. [ Links ] 26













