Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Agronomía Tropical
versión impresa ISSN 0002-192X
Agronomía Trop. v.53 n.3 Maracay jul. 2003
DIVERSIDAD MORFOLÓGICA E ISOENZIMÁTICA DE 22 PROGENITORES DE UNA POBLACIÓN BÁSICA INDEHISCENTE DE AJONJOLÍ
Antonio Díaz*, Alfredo Layrisse*, Damaris Laya* y Luis Tovar*
*Profesores. Universidad Central de Venezuela
Facultad de Agronomía Instituto de Genética
Apdo. 4653. Maracay 2101, estado Aragua. Venezuela
RESUMEN
Se estudiaron 14 descriptores morfológicos y 2 isoenzimáticos para caracterizar la diversidad genética de los posibles 22 progenitores que formarán la población básica indehiscente de ajonjolí, Sesamum indicum L., PUCV4. Las frecuencias relativas de cada categoría para cada descriptor fueron procesadas para estimar dos índices de diversidad, el índice de Shannon y Weaver (H) y el índice de uniformidad (E). Con los datos electroforéticos se calculó el índice de diversidad de Nei (h). El índice H fue empleado para obtener un índice de información (I), el cual permitió cuantificar la variabilidad total y la contenida dentro y entre las agrupaciones generadas por un árbol Neighbor Joining y el Análisis de Coordenadas Principales. Se encontró una alta diversidad genética, expresada como descriptores polimórficos con estimados promedios de E y H igual a 0,71 y 0,54, respectivamente. Los caracteres más polimórficos fueron pubescencia de la cápsula, pubescencia del tallo, color de la semilla, color del labio inferior de la flor y número de cápsulas por nudo. El análisis multivariado generó dos grupos; G1 y G2, con cinco y nueve progenitores, respectivamente; el resto de la colección se presentó dispersa, representada por los genotipos más divergentes, grupo designado como GR, La diversidad total se descompuso en dos fuentes principales: la primera contenida dentro de GR y la segunda entre G 1, G2 y GR. Se concluye que los progenitores presentan suficiente variabilidad para formar la población básica PUCV4 con una amplia diversidad genética, necesaria para garantizar ganancias con la selección durante el proceso de mejoramiento genético.
Palabras Clave: Sesamum indicum; indehiscencia; diversidad; morfología; izoenzimas.
SUMMARY
In order to characterize the genetic diversity of the possible 22 progenitors of the sesame indehiscent basic population PUCV4, 14 morphological and 2 isoenzymatic descriptors were studied. The relative frequencies of each category for each descriptor were processed to estimate two diversity indexes, the Shannon and Weaver index (H) and the Eveness index (E). Also, the Nei genetic diversity index was calculated from electrophoretic data. An information index (I) was generated from H to quantify the total variability, inside and among the groups generated by a Neighbor-Joining tree and a principal coordinate analysis. A high genetic diversity was found, expressed as polymorphic descriptors with H and E estimates of 0.71 and 0.54, respectively. The most polymorphic characters were capsule hairiness, stem hairiness, seed color, lower lip color of flower and capsule number per node. Multivariate analysis generated two groups of genotypes (G1 and G2), with five and nine elements, respectively; the rest of the collection was diffused, formed by the most divergent genotypes, designated as GR. Total diversity was partitioned in two main sources: the first one contained within GR, and the second one among G1, G2 and GR. As a conclusion, it may be state that the progenitora show enough variability to generate the basic population PUCV4 with the broad genetic diversity needed to guarantee genetic gains with selection during breeding.
Key Words: Sesamum indicum; indehiscence; morphological diversity; morphology; isozymes.
RECIBIDO: Junio 25, 2002
INTRODUCCIÓN
El ajonjolí, Sesamum indicum L., es una especie oleaginosa anual con abundante formación de flores, cada una de las cuales origina un fruto con 40 a 50 semillas, aproximadamente. La domesticación del cultivo es parcial, pues sus frutos abren una vez secos, a través de suturas que facilitan la dispersión de la semilla, como en las especies silvestres. Debido a que la cosecha se hace cuando el fruto está seco, las pérdidas de granos en campo pueden originar una reducción de los rendimientos de un 15 a 25% (Montilla y Mazzani, 1966), bajo el sistema de cosecha mecanizado de dos etapas (corte y trilla) que predomina en Venezuela.
Como alternativa para minimizar la pérdida de granos durante la cosecha, se propone el empleo de cultivares indehiscentes de ajonjolí. Mazzani (1983) señala otras ventajas de la utilización de los mismos a gran escala: a) menor urgencia de la cosecha, b) menor duración de las operaciones en campo, y c) menor costo, ya que la cosecha se haría en una sola operación. A pesar de ello, la aceptación de materiales indehiscentes a nivel comercial no ha sido posible, debido a que presentan rendimientos por debajo de los dehiscentes como producto de una reducida fertilidad.
Sin embargo, este tipo de variedades presentan baja fertilidad, tal como lo señala Berlingeri (2000) con respecto a la variedad Morada indehiscente, lo cual es ocasionado principalmente por una carga reducida de polen sobre el estigma, un menor número de tubos polínicos en el estilo e incapacidad del tejido femenino de promover el crecimiento de los tubos polínicos, en comparación con Morada dehiscente. El cultivar indehiscente tiene además una baja eficiencia reproductiva, expresada como un menor número de semillas por fruto y un menor número de flores por axila.
Este conjunto de efectos negativos en los indehiscentes se debe a la acción de un gen (id) pleiótropo recesivo (Langham, 1946), que además produce deformación en las hojas y flores, desarrollo de neoplasias en ambos órganos, deformación de las cápsulas y engrosamiento del mesocarpio (Ashri y Ladijiski, 1964; Day, 2000; Mazzani y Horovitz, 1952
El efecto del gen id está afectado por genes modificadores (Mazzani y Horovitz, 1952). Estos autores consiguieron materiales experimentales con grandes niveles de indehiscencia y buena fertilidad, lo cual les permitió plantear la hipótesis de que los modificadores son de efecto simple, afectando cada uno de ellos una característica en particular, pero no todas en conjunto y, en consecuencia, podrían acumularse los efectos relacionados con la fertilidad, sin perder la indehiscencia de los frutos. Los modificadores pueden alterar sustancialmente la fertilidad de los indehiscentes, al punto que se ha observado una ausencia de correlación entre la apertura de la cápsula y el rendimiento de semillas en algunos casos (Delgado et al., 1994).
De lo anterior se deriva que puede obtenerse una selección hacia plantas fértiles en poblaciones idid sin modificar apreciablemente la condición indehiscente de la población (Mazzani y Horovitz, 1952). En tal sentido, en el Instituto de Genética "Salomón Horovitz" de la Facultad de Agronomía de la Universidad Central de Venezuela (IG-FAGRO), se inició la formación de una población básica indehiscente (PUCV4), que será utilizada en un programa de selección recurrente para incrementar los rendimientos. Para ello se realizan los cruces de 21 progenitores dehiscentes con el padre indehiscente UCR 82-13. De estos cruzamientos se están derivando, mediante autofecundación, un número equivalente de familias indehiscentes endocriadas. Finalmente, estas familias se recombinarán al azar para obtener 'el ciclo cero (C0) del programa, el cual técnicamente se considerará como la PUCV4.
La selección recurrente es una de las principales líneas de investigación de mejoramiento poblacional en el ajonjolí llevada a cabo por el IG-FAGRO. Partiendo de una población dehiscente suficientemente variable, conformada por el cruzamiento de 25 materiales comerciales y exóticos, se consigue una respuesta favorable en dos ciclos de selección para rendimiento en grano, con un incremento del rendimiento de 14,5% por ciclo (Laurentín et al., 2000)
Algunos elementos adicionales reafirman la efectividad del programa, por ejemplo: la mejor línea del C1 rindió por encima de la mejor línea del C0. A pesar de que la media del C0 fue significativamente inferior a los testigos, la media del C2 fue igual. De acuerdo a la distribución normal, es probable que existan líneas con rendimientos superiores a la media del C2 y, por lo tanto, a la media de los testigos. También se ha demostrado que dos poblaciones básicas indehiscentes, una de origen africano y otra de origen chino, presentan suficiente variabilidad como para iniciar un programa de selección recurrente, ambas con estimados importantes de la varianza genética aditiva (Acevedo y Monteverde, 1998; Silva y Monteverde, 1998).
Se espera que la población PUCV4 posea suficiente variabilidad genética para garantizar un avance significativo en la selección en los ciclos derivados. En este sentido, es prioritario conocer la diversidad presente en los progenitores que formarán a la PUCV4, por lo que esta investigación tuvo como objetivo caracterizar la diversidad genética observada en dichos progenitores, desde el punto de vista morfológico e isoenzimático.
MATERIALES Y MÉTODOS
Material vegetal
Las denominaciones de los 22 progenitores se presentan en el Cuadro 1. Los cultivares Fonucla y UCLA-1 y las líneas experimentales 90-1, H-65, 249-42, H-83, Exp-295 y 37-1 fueron cedidos por Hernán Laurentín de la Universidad Centro Occidental Lisandro Alvarado (UCLA), Barquisimeto, Venezuela y las accesiones Felicidad, China amarillo, Turén, Venezuela 51, 98-280 y UCR 82-13 por Elena Mazzani del Instituto Nacional de Investigaciones Agrícolas (INIA), Maracay, Venezuela. El resto de los materiales se obtuvieron de la Colección de Trabajo del IG-FAGRO.
Caracterización morfológica
La evaluación morfológica se realizó en Maracay, en el campo experimental del IG-FAGRO, durante el período enero-abril de 2001. Cada material se sembró en una hilera de 5 m de longitud, espaciadas a razón de 80 cm con 15 plantas por metro lineal, aproximadamente. A 10 plantas tomadas al azar dentro de cada hilera, se les registraron 14 descriptores cualitativos (Cuadro 2), sugeridos en la lista de descriptores de ajonjolí del IBPGR (1981). Estos descriptores son altamente heredables, de fácil registro visual y con expresión constante en todos los ambientes. Otros caracteres evaluados resultaron invariables en la colección, como el hábito de ramificación y el color de la foveola, por lo que fueron descartados al momento de realizar los análisis estadísticos.
Caracterización isoenzimática
En un principio, la colección se caracterizó para los sistemas isoenzimáticos isocitrato deshidrogenasa (IDH, E.C. 1.1.1.42), siquimato deshidrogenasa (SKI), E.C. 1.1.1.25), malato deshidrogenasa (MDH,
| CUADRO 1. Diversidad morfo-isoenzimática de los 22 progenitores de la población indehiscente de ajonjolí PUCV4. | ||||||||||||||||
| | ||||||||||||||||
| Progenitores | Descriptores* | |||||||||||||||
| | ||||||||||||||||
| ptall | coho | puho | psho | aiho | ccor | clin | pubc | cnod | fcap | ccap | pcap | csem | tsem | idha | idhb | |
| 1. UCV-1 | 9 | 2 | 3 | 1 | 3 | 1 | 1 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 2. Felicidad | 9 | 2 | 3 | 1 | 5 | 1 | 1 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 0 | 1 |
| 3. China Amarillo | 9 | 2 | 3 | 1 | 3 | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 0 | 1 |
| 4. 37-1 | 3 | 1 | 3 | 1 | 5 | 2 | 2 | 1 | 1 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 5.UCR 82-13 | 9 | 1 | 9 | 1 | 3 | 2 | 1 | 0 | 2 | 1 | 2 | 2 | 3 | 2 | 1 | 0 |
| 6. Inamar | 9 | 2 | 3 | 1 | 3 | 2 | 2 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 0 |
| 7. Acarigua | 3 | 2 | 3 | 1 | 3 | 1 | 1 | 0 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 |
| 8.DV-1 | 9 | 2 | 3 | 1 | 3 | 2 | 2 | 1 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 1 |
| 9. UCV-2 | 9 | 2 | 3 | 1 | 3 | 2 | 1 | 1 | 2 | 2 | 2 | 3 | 1 | 1 | 0 | 1 |
| 10. 90-1 | 3 | 1 | 3 | 3 | 3 | 2 | 2 | 1 | 1 | 2 | 2 | 3 | 1 | 1 | 1 | 0 |
| 11. Turén | 9 | 2 | 3 | 1 | 3 | 2 | 2 | 0 | 1 | 2 | 2 | 1 | 1 | 1 | 1 | 1 |
| 12. UCV-3 | 3 | 2 | 3 | 3 | 3 | 2 | 2 | 0 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 |
| 13. H-65 | 9 | 2 | 3 | 1 | 3 | 2 | 2 | 1 | 2 | 1 | 2 | 2 | 2 | 1 | 0 | 1 |
| 14. Venezuela 51 | 9 | 2 | 9 | 1 | 3 | 2 | 2 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 15. Caripucha | 9 | 2 | 9 | 1 | 5 | 2 | 2 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 16. Maporal | 3 | 2 | 3 | 1 | 3 | 2 | 2 | 0 | 2 | 3 | 2 | 2 | 2 | 2 | 1 | 1 |
| 17. Fonucla | 3 | 2 | 3 | 1 | 3 | 2 | 2 | 1 | 2 | 2 | 1 | 1 | 2 | 1 | 1 | 0 |
| 18. 249-42 | 3 | 1 | 3 | 3 | 3 | 2 | 2 | 1 | 1 | 2 | 2 | 2 | 1 | 1 | 1 | 0 |
| 19. H-83 | 9 | 2 | 9 | 1 | 3 | 1 | 2 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 20. Exp-295 | 3 | 1 | 3 | 3 | 3 | 2 | 2 | 1 | 1 | 2 | 2 | 2 | 1 | 1 | 1 | 0 |
| 21. UCLA-1 | 3 | 1 | 3 | 3 | 3 | 2 | 2 | 1 | 1 | 2 | 2 | 2 | 1 | 1 | 1 | 1 |
| 22. 98-280 | 9 | 2 | 9 | 1 | 5 | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 1 | 0 | 1 |
| | ||||||||||||||||
| * Ver Cuadro 2 para mayor detalle. | ||||||||||||||||
E.C. 1.1.1.37), fosfogluconato deshidrogenasa (PGD, E.C. 1.1.1.44), glutamato deshidrogenasa (GDH, E.C. 1.4.1.2), fosfoglucoisomerasa (PGI, E.C. 5.3.1.9), fosfoglucomutasa (PGM, E.C. 5.4.2.2) y glutamato oxaloamino transaminasa (GOT, E.C. 2.6.1.1). No obstante, sólo IDH resultó polimórfico, por lo que los otros sistemas fueron descartados.
Para cada material se colectaron 200 g de tejido foliar en expansión, provenientes de 4 plantas (50 g cada una) con dos a tres semanas de emergidas. Se hizo un compuesto equitativo de las cuatro plantas y se maceró con un tampón de extracción constituido por polivinil pirrolidona-40 3%, 2-mercaptoetanol 0,9%, tris 151 mM, albúmina de suero bovina 3%, ácido cítrico 19 mM y polietilénglicol 3% (Díaz, 1997).
Los extractos se colocaron en papeles de filtro Whatman N° 1 y fueron cargados en un gel de almidón al 12%. E1 sistema gel-electrodo y las condiciones de corrida utilizados fueron Morfolina-Citrato pH 6,1 a 30mA por 6 h. La fórmula de tinción fue la siguiente: 50 ml solución tris-HCl (605,5 mg tris; enrasar con agua a 50 ml; ajustar pH a 8,0 con HC1), 2 ml MgCl2 10%, 30 mg ácido isocítrico, 3 mg PMS, 10 mg MTT, 8 mg NADP Se registró la presencia o ausencia de los alelos a y b del locus Idhl para cada material, según Díaz y Layrisse (2002) codificados como los descriptores Idha e Idhb, respectivamente (Cuadro 2).
Análisis estadístico
Se obtuvieron por conteo directo las frecuencias de aparición de cada categoría para cada descriptor. Con ellas se estimaron dos índices de diversidad estrechamente relacionados. El índice de Shannon y Weiner (H) se obtuvo a través de la ecuación H = - ∑ pi ln pi, en la que pi. es la frecuencia relativa de la i-ésima categoría (Sneath y Sokal, 1973) y el índice de uniformidad E, calculado como E = H / log S, donde S es el número de categorías presentes.
El índice H cuantificó el grado de entropía o grado promedio de incertidumbre en predecir a cuál categoría iba a pertenecer un individuo escogido al azar de la colección. El índice E midió la proporción de la diversidad observada con relación a la máxima diversidad esperada. Con el valor de H se obtuvo además una medida de información (I) del sistema, estimada como I = n H, donde n es el número de progenitores. La sumatoria de I para los distintos descriptores es una medida de entropía total del grupo. Por otra parte, se estimó el índice "h" de diversidad de Nei (1978), el cual se calculó como h =1-∑ p2, donde p es la frecuencia del i-ésimo alelo isoenzimático del locus IdhL
| CUADRO 2. Descriptores y sus categorías empleados para determinar la variabilidad genética de los 22 progenitores de la población indehiscente de ajonjolí PUCV4. | |
| | |
| Carácter | Categorías |
| | |
| Pubescencia del tallo (ptall) | 3 = escasa; 9= abundante |
| Color de hoja a inicios de la floración (coho) | 1 = verde; 2 = verde amarillento |
| Pubescencia de la hoja (puho) | 3 = escasa; 9 = abundante |
| Posición de la hoja en el tallo (psho) | 1= opuesta; 3= opuestas y alternas |
| Ángulo inserción de hojas en el tallo (aiho) | 3 = agudo; 5 = recto |
| Color exterior de la corola (ccor) | 1 = blanco;2 = blanco con tonos violeta-purpura |
| Color de labio inferior de la flor (clin) | 1 = sin color; 2 = con color |
| Pubescencia de Brácterias y colores (pubc) | 0 = ausente; 1 = presente |
| Número de cápsulas por nudo (cnod) | 1 = una; 2 = más de una |
| Forma de la cápsula (fcap) | 1 = alargada; 2 = oblonga; 3 = cuadrada |
| Color de la cápsula seca (ccap) | 1 = verde; 2 = marrón |
| Pubescencia de la cápsula (pcap) | 1 = ausente; 2 = corta; 3 = larga |
| Color de la cubierta de la semilla (csem) | 3 = marrón |
| Textura de la cubierta de la semilla (tsem) | 1 = lisa; 2 = rugosa |
| Alelo isoenzimático Idhl-a (idha) | 0 = ausente; 1 = presente |
| Alelo isoenzimático Idhl-b (idhb) | 0 = ausente; 1 = presente |
Para establecer las semejanzas entre los 22 progenitores se construyó un árbol no enraizado mediante el método "Neihgbor-Joining" (NJ), a partir de la matriz de distancias calculadas de la raíz cuadrada del complemento del coeficiente de similitud general de Gower (1 - SG)1/2, donde SG = número de descriptores coincidentes / total de descriptores (Sneath y Sokal, 1973). Los cálculos de este procedimiento se consiguieron con el programa NTSYS (1990) y visualizados con el programa TREEVIEW (Page, 1996). Como complemento, se realizó un análisis de coordenadas principales (ACOP) partiendo de la matriz de similitud entre progenitores utilizándose el índice SG. Los cálculos del ACOP se obtuvieron a través del programa NTSYS (1990).
RESULTADOS Y DISCUSIÓN
Semejanzas genéticas
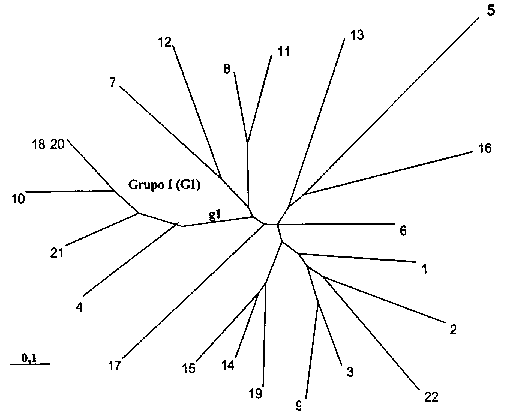
En la Figura 1 se muestra el árbol no enraizado NJ. La topología del árbol muestra ramas externas más largas que las internas, a excepción del ramal gl que permitió diferenciar del núcleo del árbol a un grupo designado como G 1. Dentro de este grupo se encontraron los progenitores 4, 10,18, 20 y 21, de los cuales dos (18 y 20) no pudieron ser diferenciados mediante los 16 descriptores.
|
FIGURA 1. Árbol no enraizado NJ de los 22 progenitores de la población indishescente de ajonjolí PUCV4. Para la codificación de los genotipo ver Cuadro 1. |
Todos los elementos de este grupo procedieron de la colección de trabajo de la UCLA. La topología del árbol no permitió definir otros grupos y sugiere que los progenitores en general presentan una alta divergencia entre sí. Debido a que el método NJ permite expresar las distancias entre los materiales en función de la longitud de las ramas que los separan, puede inferirse que UCR 82-13 (5), H-65 (13), Maporal (16) y Fonucla (17) fueron los más divergentes de la colección.
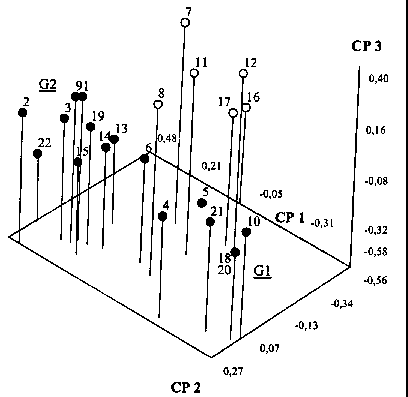
Con el ACOP se distinguió un grupo adicional que no se observó con el árbol NJ, designado como G2 (Figura 2), constituido por los materiales 1, 2, 3, 6, 9, 14, 15, 19 y 22. El resto de los progenitores (5, 7, 8, 11, 12, 13, 16 y 17) se presentaron dispersos y separados de G 1 y G2, sobretodo en las Coordenadas Principales (CP) 2 y 3.
|
FIGURA 2. Gráfico de las tres primeras coordenadas principales de los 22 progenitores de la población indehiscente de ajonjolí PUCV4. Para la codificación de los genotipo ver Cuadro 1. |
Este conjunto de progenitores con amplia divergencia genética se designó como GR. Los valores propios asociados con las tres primeras CP se presentan en el Cuadro 3, con un acumulado del 55% de la variabilidad original. El otro 45% fue distribuido en las siguientes 14 CP, con incrementos despreciables a partir de la octava CP. Similar a lo generado por el árbol NJ, los materiales 5, 16 y 17 fueron los más divergentes, cada uno separado ampliamente del otro en las 3 primeros CP.
Diversidad genética
Las variantes conseguidas en los 16 descriptores se presentan en el Cuadro 1. Para cuantificar la diversidad en cada uno ellos se empleó el índice E, el cual refleja la similitud entre las frecuencias relativas de sus categorías. La variabilidad máxima posible se hubiese conseguido con un valor de E igual a 1, en cuyo caso todas las categorías hubiesen sido igualmente frecuentes.
Los descriptores que más se ajustaron a esta condición fueron los de mayor diversidad en los materiales evaluados (Cuadro 4). Entre ellos se señalan a ptall, clin y cnod, cuya categoría menos frecuente no fue menor que 0,30. Los descriptores menos diversos fueron ccap, tsem y fcap, con una categoría prácticamente ubicua y las restantes presentes en no más de tres genotipo. Fonucla fue el único material que presentó un ccap verde, Maporal y UCR 8213 una tsem rugosa, H-65 y UCR 82-13 una fcap alargada y Maporal una fcap cuadrada.
| CUADRO 3. Valores propios de las tres primeras coordenadas principales (CP), obtenidas de una matriz de semejanzas según el coeficiente de similitud general de Gower, para los 22 progenitores de la población indehiscente de ajonjolí PUCV4. | |||
| CP | Valor Propio | Porcentaje | Acumulado |
| 1 | 2,2016 | 29,20 | 29,20 |
| 2 | 1,0138 | 13,45 | 42,65 |
| 3 | 0,9509 | 12,61 | 55,26 |
| CUADRO 4. Estimados de diversidad de los descriptores morfo-isoenzimáticos para los progenitores de la población indehiscente de ajonjolí PUCV4. | |||||||||
| Descriptor | Toda la colección | G1(5) | |||||||
| Frecuencias | H(2) | E(3) | I(4) | Frecuencias | H | I | |||
| | |||||||||
| Ptall(1) | 3=0,41; | 9=0,59 | 0,68 | 0,98 | 14,88 | 3=1,00 | 0 | 0 | |
| coho | 1=0,27; | 2=0,73 | 0,59 | 0,85 | 12,89 | 1=1,00 | 0 | 0 | |
| puho | 3=0,77; | 9=0,23 | 0,54 | 0,77 | 11,79 | 3=1,00 | 0 | 0 | |
| psho | 1=0,77; | 3=0,23 | 0,54 | 0,77 | 11,79 | 1=0,20; | 3=0,80 | 0,50 | 2,50 |
| . | . | . | . | . | ... | . | . | ||
| aiho | 3=0,82; | 5=0,18 | 0,47 | 0,68 | 10,43 | 3=0,80; | 5=0,20 | 0,50 | 2,50 |
| ccor | 1=0,18; | 2=0,82 | 0,47 | 0,68 | 10,43 | 2=1,00 | 0 | 0 | |
| clin | 1=0,32; | 2=0,68 | 0,63 | 0,90 | 13,76 | 2=1,00 | 0 | 0 | |
| pubc | 0=0,23; | 1=0,77 | 0,54 | 0,77 | 11,79 | 1=1,00 | 0 | 0 | |
| . | . | . | . | . | ... | . | . | ||
| cnod | 1=0,32; | 2=0,68 | 0,63 | 0,90 | 13,76 | 1=1,00 | 0 | 0 | |
| fcap | 1=0,09; | 2=0,86 | 0,49 | 0,44 | 10,68 | 2=1,00 | 0 | 0 | |
| 3=0,05 | |||||||||
| ccap | 1=0,05; | 2=0,95 | 0,19 | 0,27 | 4,07 | 2=1,00 | 0 | 0 | |
| pcap | 1=0,23; | 2=0,68 | 0,82 | 0,74 | 17,95 | 2=0,80; | 3=0,20 | 0,50 | 2,50 |
| 3=0,09 | |||||||||
| . | . | . | . | . | . | . | . | ||
| csem | 1=0,77; | 2=0,18; | 0,65 | 0,59 | 14,30 | 1=1,00 | 0 | 0 | |
| 3=0,05 | |||||||||
| tsem | 1=0,91; | 2=0,09 | 0,30 | 0,44 | 6,70 | 1=1,00 | 0 | 0 | |
| idha | 0=0,23; | 1=0,77 | 0,54 | 0,77 | 11,79 | 1=1,00 | 0 | 0 | |
| idhb | 0=0,27; | 1=0,73 | 0,59 | 0,85 | 12,89 | 0=0,60; | 1=0,40 | 0,67 | 3,37 |
| Promedio | . | 0,54 | 0,71 | . | . | . | . | ||
| Sumatoria | . | . | . | 189,9 | . | . | 10,9 | ||
| %(7) | . | . | . | (100) | . | . | (5) | ||
| (1) Ver Cuadro 2 para la codificación de los descriptores y sus categorías; (2)Índice de diversidad de Shannon y Weaver; (3)Índice de Uniformidad; (4)Cantidad de Información; (5)G1, G2 y GR corresponden a subconjuntos de genotipo según se comenta en el texto;(6) Obtenido como la diferencia; del I de toda la colección con las de G1, G2 y GR; (7) Porcentaje en función del I de toda la colección.../....continúa | |||||||||
| ../...continuación CUADRO 4 | |||||||||
| Descriptor | G2 | GR | Entre grupos | ||||||
| Frecuencias | H | I | Frecuencias | H | I | I(6) | |||
| | |||||||||
| Ptall(1) | 9=1,00 | 0 | 0 | 3=0,50; | 9=0,50 | 0,69 | 5,55 | 9,34 | |
| coho | 2=1,00 | 0 | 0 | 1=0,13; | 2=0,87 | 0,38 | 3,01 | 9,88 | |
| puho | 3=0,56; | 9=0,44 | 0,69 | 6,18 | 3=0,87; | 9=0,13 | 0,38 | 3,01 | 2,60 |
| psho | 1=1,00 | 0 | 0 | 1=0,87; | 3=0,13 | 0,38 | 3,01 | 6,28 | |
| . | |||||||||
| aiho | 3=0,67; | 5=0,33 | 0,64 | 5,73 | 3=1,00 | 0 | 0 | 2,20 | |
| ccor | 1=0,33; | 2=0,67 | 0,64 | 5,73 | 1=0,13; | 2=0,87 | 0,38 | 3,01 | 1,69 |
| clin | 1=0,56; | 2=0,44 | 0,69 | 6,18 | 1=0,25; | 2=0,75 | 0,56 | 4,50 | 3,08 |
| pubc | 1=1,00 | 0 | 0 | 0=0,63; | 1=0,37 | 0,66 | 5,29 | 6,50 | |
| . | |||||||||
| cnod | 2=1,00 | 0 | 0 | 1=0,25; | 2=0,75 | 0,56 | 4,50 | 9,26 | |
| fcap | 2=1,00 | 0 | 0 | 1=0,25; | 2=0,62 | 0,90 | 7,20 | 3,47 | |
| 3=0,13 | |||||||||
| ccap | 2=1,00 | 0 | 0 | 1=0,13; | 2=0,87 | 0,38 | 3,01 | 1,06 | |
| pcap | 2=0,89; | 3=0,11 | 0,35 | 3,14 | 1=0,63; | 2=0,37 | 0,66 | 5,29 | 7,02 |
| . | |||||||||
| csem | 1=0,89; | 3=0,11 | 0,35 | 3,14 | 1=0,50; | 2=0,37; | 0,97 | 7,80 | 3,36 |
| 3=0,13 | |||||||||
| tsem | 1=1,00 | 0 | 0 | 1=0,75; | 2=0,25 | 0,56 | 4,50 | 2,20 | |
| idha | 0=0,44; | 1=0,56 | 0,69 | 6,18 | 0=0,13; | 1=0,87 | 0,38 | 3,01 | 2,60 |
| idhb | 0=0,11; | 1=0,89 | 0,69 | 3,14 | 0=0,25; | 1=0,75 | 0,56 | 4,50 | 1,89 |
| | |||||||||
| Promedio | . | . | . | . | . | . | . | ||
| | |||||||||
| Sumatoria | . | . | 39,4 | . | . | 67,2 | 72,4 | ||
| %(7) | . | . | (20,8) | . | . | (35,4) | (38,1) | ||
| | |||||||||
| (1)Ver Cuadro 2 para la codificacion de los descriptores y sus categorias; (2)Índice de diversidad de Shannon y Weaver; (3)Índice de uniformidad; (4)Cantidad de informacion; (5)G1, G2 y GR corresponden a subconjuntos de genotipos segun se comentan en el texto; (6)Obtenido como la diferencia; del I de toda la coleccion con las de G1, G2 y GR; (7)Porcentaje en funcion del I de toda la coleccion. | |||||||||
Aparte de serpubs un indicador de la variación, entropía o desorden, el índice H sugirió el peso de los descriptores en la definición de la topología del árbol NJ y de las coordenadas de los progenitores en las 3 primeras CP. Al ordenar los estimados de H de mayor a menor, en el cuartil superior se ubicaron los de las variables pcap, ptall, csem, clin y cnod (Cuadro 4).
Como consecuencia, se presentaron distintas combinaciones de categorías predominantes para estos descriptores en G1, G2 y GR. El grupo G1 mostró una alta proporción de cápsulas con pubescencia corta y la totalidad de las plantas con escasa pubescencia en el tallo, semillas color blanco o crema, flores con labio inferior con color y una cápsula por nudo. El grupo G2 estuvo caracterizado por una predominancia de cápsulas con pubescencia corta, la totalidad de plantas con abundante pubescencia en el tallo, una alta proporción de semillas color blanco o crema, leve predominancia de flores con labio inferior sin color y la totalidad de las plantas con una cápsula por nudo.
En GR se encontró una leve predominancia de cápsulas sin pubescencia sobre pubescencia corta, igual proporción de plantas con tallos de abundante y escasa pubescencia, leve predominancia de semillas color blanco o crema sobre marrón claro, y una alta proporción de plantas con flores con color en el labio inferior y de más de una cápsula por nudo.
La aditividad de los estimados de I permitió calcular la diversidad total de la colección y la diversidad contenida dentro de G1, G2 y GR (Cuadro 4). La variabilidad interna de los grupos G1 y G2 representó un 26,5% de la diversidad total y no superó a la contenida en GR, con un 35,4%. En total, la diversidad contenida dentro de los tres grupos representó un 61,9 % de la diversidad total. El 38,1 % restante correspondió a la diversidad genética entre G1, G2 y GR. Un 75% de los descriptores no mostró variabilidad dentro de G 1 y un 50% no la mostró dentro de G2; mientras que los elementos de GR fueron invariables solamente para aiho.
En relación con el locus Idh1, los progenitores presentaron un h de Nei igual a 0,499, por lo que existe igual probabilidad de que dos alelos extraídos al azar de la colección sean distintos. Según Nei (1978) esto se traduciría como la diferencia promedio en codones de dos alelos tomados al azar para el locus Idh1.
Como una aproximación al estudio de la diversidad con fines de mejoramiento genético se ha planteado el uso de métodos multivariados para cuantificar la variabilidad y semejanzas de un conjunto de materiales.
Estos métodos brindan la oportunidad de combinar gran parte de la variabilidad presente en los descriptores, transformándola en estimados y representaciones gráficas, cuya sencillez facilita la interpretación de los resultados. Diversos estudios en el ajonjolí han abordado el análisis de la diversidad genética por esta vía. Bedigian et al. (1986), por ejemplo, evaluaron cultivares provenientes de 20 países distintos para un total de 32 caracteres morfológicos, mediante el análisis de factores, de grupos ("cluster"), discriminante y de componentes principales, consiguiendo 8 grandes grupos con variantes morfológicas exclusivas para cada uno de ellos.
Los métodos multivariados facilitan la creación de colecciones nucleares ("core collections") que representen la variabilidad contenida en toda la colección (Bisht et al., 1998; Shen et al., 1995; Xiurong et al., 2000) o para aumentar la eficiencia en la escogencia de progenitores ampliamente divergentes al momento de formar las poblaciones básicas para mejoramiento (Singh et al., 1997; Swain y Dikshit, 1997; Wei et al., 1994).
De acuerdo a la topología del árbol NJ, así como a la distribución espacial de los puntos en el ACOP, los progenitores de la PUCV4 mostraron una amplia diversidad genética, contenida principalmente en dos fuentes de variabilidad. La primera se encontró dentro de GR, constituido por ocho materiales aislados, de los cuales UCR 82-13, Fonucla, Maporal y H-65 fueron los más divergentes de la colección; mientras que la segunda correspondió a las diferencias entre G1, G2 y GR.
Por lo tanto, la selección de materiales pertenecientes a grupos contratantes en los programas de cruzamientos para formar la PUCV4 garantizaría una población básica con alta diversidad genética (Wei et al., 1994; Swain y Dikshit, 1997) y sugiere que existe una baja probabilidad de que los progenitores posean alelos idénticos en los loci que controlan las características de interés agronómico (Ferreira et al., 2000). Esto se vió reflejado indirectamente mediante descriptores altamente variables.
Las variables estudiadas presentaron en promedio un 71 % del valor teórico máximo E, lo que indica que fueron muy polimórficas con variantes distribuidas en combinaciones individuales que favorecieron la identificación de 20 de los 22 progenitores estudiados. Debido a que el estudio se hizo en una sola localidad, una serie de caracteres cuantitativos con posible interacción genotipo-ambiente no se incluyeron.
Formalmente en el análisis. No obstante, es interesante mencionar los rangos de variación para estos caracteres, pues amplían la visión sobre el grado de variabilidad de los progenitores. Por ejemplo, días a floración varió entre 23 y 43 d, peso de mil semillas entre 2,40 y 3,93 g, altura de la planta entre 51 y 112 cm y longitud de la cápsula entre 2,24 y 3,92 cm.
De acuerdo con Idh1, la diversidad genética de los progenitores es comparable a la que presentó una colección de 40 accesiones de ajonjolí procedentes de seis centros de diversidad (Díaz et al., 1999), en el cual el índice de diversidad "h" de Nei (1978) para este mismo locus varió entre 0,245 para Asia Central y 0,499 para Asia Occidental, con una media de los seis centros de 0,369. El "h" de los progenitores fue igual a 0,499, superior a cinco de los seis centros de diversidad y específicamente al de India, Corea, África, China-Japón y Asia Central. Esto indica que los progenitores presentan tanta o más diversidad que la encontrada en los centros de diversidad del cultivo, en función de las frecuencias relativas de los alelos de Idh1.
Las semejanzas entre algunos progenitores concuerda con su origen. En este sentido, Maporal y Fonucla divergieron en mayor grado del resto de los materiales estudiados, probablemente porque ambos comparten germoplasma africano (Nava y Layrisse, 1991); la estrecha semejanza entre `Turén' y `DVI' se explicaría porque este último provino de la aplicación de mutaciones inducidas en embriones inmaduros y posterior regeneración in vitro de plantas de Turén (Mazzani, 1999) y la proximidad de Venezuela 51 con `Inamar', porque ésta se derivó de una planta seleccionada individualmente en la descendencia del híbrido Venezuela 51 X `45.1138'.
Por otra parte, otros agrupamientos no pudieron explicarse por el origen de los progenitores. Es probable que la variación de los descriptores morfológicos empleados estaría sujeta a una selección convergente por mejoramiento genético, en el que predominó la búsqueda de ideotipos para ciertas variantes morfológicas de la planta, lo que generaría que linajes distintos presentasen fenotipos idénticos. Este efecto quizás fue atenuado al incluirse dos aloenzimas del locus Idh1, considerados hasta cierto punto selectivamente neutrales (Gepts, 1995).
En un estudio realizado con el cultivar Turén, el locus Idhl presentó una variabilidad no compatible con la acentuada homogeneidad morfoagronómica del cultivar (Díaz y Layrisse, 2000), lo cual sugiere que la selección aplicada para uniformizar las características agronómicas no afectó la diversidad isoenzimática en el locus Idh1.
Para incorporar la indehiscencia a la PUCV4, se cruzaron los 21 progenitores dehiscentes con UCR 82-13 como padre, por lo que éste aportará el 50% del germoplasma de la población. UCR 82-13 fue el material más divergente según el árbol NJ, lo que aumenta la posibilidad de que se formen nuevas recombinaciones en las generaciones descendientes de cada cruce.
Este sistema de cruzamiento se empleó en el estudio llevado a cabo por Silva y Monteverde (1998) en una población de líneas F4 de origen chino, obtenidas de los respectivos cruces de UCR 82-13 con 10 líneas élites dehiscentes. Para rendimiento en grano de 10 plantas, número de frutos por planta, longitud de carga y número de semillas por fruto se consiguió que tanto la acción génica de tipo aditivo, como la no aditiva, determinan la expresión fenotípica. Sin embargo, los estimados de la varianza aditiva superaron en magnitud a los de la varianza de dominancia, llegando a ser seis veces más alta para el número de frutos por planta.
Los autores concluyeron que la variabilidad genética presente en la población y la alta heredabilidad mostrada en todas las características evaluadas hacen predecir grandes progresos en la selección. Se espera que se pueda repetir esta condición en la PUCV4, más aun si se considera que se están incluyendo un mayor número de progenitores dehiscentes, que los empleados por Silva y Monteverde (1998).
CONCLUSIONES
-
Los estimados promedio de diversidad conseguidos en los progenitores fueron altos. Esta diversidad se distribuyó en un componente intragrupal, con un 61,9% de la diversidad total, y una fuente intergrupal, con un 38,1 °/o de la misma. Esto sugiere que la escogencia de estos progenitores garantizaría una población PUCV4 con suficiente variabilidad genética para iniciar un programa de selección recurrente.
-
La identidad morfológica e isoenzimática de 249-42 y Exp-295 indica que ambos materiales están estrechamente relacionados, por lo cual se recomienda la exclusión de uno de ellos de la formación de la PUCV4.
AGRADECIMIENTO
Los autores agradecen a Julio Viera (UCV) y Elena Mazzani (INIA) por sus comentarios constructivos sobre el manuscrito. De igual forma agradecen a E.M. y a Hernán Laurentín (UCLA) por la generosa donación de algunos genotipos empleados en este trabajo.
BIBLIOGRAFÍA
1.ACEVEDO, M. y E. MONTEVERDE. 1998. Heterosis y heredabilidad en una población indehiscente de ajonjolí (Sesamum indicum L.) de tipo africano. Rev. Fac. Agron. (Maracay, Ven.) 24:11-23. [ Links ]
2.ASHRI A. and G. LADIJISKI. 1964. Anatomical effects of the capsule dehiscence alleles in sesame. Crop Sci. 4: 136-138. [ Links ]
3.BEDIGIAN, D., C. SMYTH and J. HARLAN. 1986. Patterns of morphological variation in Sesamum indicum. Econ. Bot. 40: 353-365. [ Links ]
4.BERLINGERI, C. 2000. Posibles causas de la baja fertilidad del ajonjolí indehiscente (Sesamum indicum L.). Tesis de Maestría. Maracay, Ven. Universidad Central. 81 p. [ Links ]
5.BISHT, I., R. MAHAJAN, T. LOKNATHAN and R. AGRAWAL. 1998. Diversíty in indian sesame collection and stratification of germplasm accessions in different diversity groups. Genetic Resources and Crop Evolution 45:325-335. [ Links ]
6.DAY, J. 2000. Anatomy of capsule dehiscence in sesame varieties. J. Agric. Sc¡. 134:45-53. [ Links ]
7.DELGADO, N., A. LAYRISSE y P. QUIJADA. 1994. Herencia de la indehiscencia del fruto del ajonjolí Sesamum indicum L. Agronomía Trop. 44(3):499-512. [ Links ]
8.DÍAZ, A. 1997. Metodología electroforética, interpretación genética y diversidad isoenzimática en el ajonjolí (Sesamum indicum L.). Tesis de Maestría. Maracay, Ven. Universidad Central. 162 p. [ Links ]
9.DÍAZ, A. y A. LAYRISSE. 2002. Control genético de los sistemas isoenzimáticos isocitrato deshidrogenasa y siquimato deshidrogenasa en el ajonjolí. Acta Científica Venezolana 53 (3):176-182. [ Links ]
10.DÍAZ, A. y A. LAYRISSE. 2000. Variabilidad interna isoenzimática de los cultivares de ajonjolí Turén y Arawaca. Agronomía Trop. 50(3):391-400. [ Links ]
11.DÍAZ A., A. LAYRISSE y T. PUGH. 1999. Análisis de la diversidad genética en el ajonjolí mediante electroforesis de isoenzimas. Agronomía Trop. 49(2):169-186. [ Links ]
12.FERREIRA, M., M. PENTEDO, C. BRONDANI, A. BELÓ, M. FERREIRA y P RANGEL. 2000. Caracterización y uso de marcadores RAPD y microsatélites (SSR) en el monitoreo del programa de mejoramiento poblacional en arroz. In: Avances en el mejoramiento poblacional en el arroz. Guimaraes E. (Ed.). EMBRAPA, Brasil. p. 37-62. [ Links ]
13.GEPTS, P. 1995. Genetic markers and core collections. In: Core collections and plant genetic resources. Hodking T., A. Brown, T. van Hintum y E. Morales (Eds). Sinauer Associates, Inc. p. 127-146. [ Links ]
14.IBPGR. 1981. Descriptors for sesame. Rome, Italy. 19 p. [ Links ]
15.LANGHAM D. 1946. Genetics of sesame. III. "Open Sesame" and mottled leaf. J. Hered. 37:149-152. [ Links ]
16.LAURENTIN, H., A. LAYRISSE y P. QUIJADA. 2000. Evaluación de dos ciclos de selección recurrente para altos rendimientos de semilla en una población de ajonjolí. Agronomía Trop. 50(4):521-535. [ Links ]
17.MAZZANI, B. 1999. Investigación y tecnología del cultivo del ajonjolí en Venezuela. Caracas. Ediciones del CONICIT. ISBN 980-6020-54-5. p. 50-51. [ Links ]
18.MAZZANI, B. 1983. Cultivo y mejoramiento de plantas oleaginosas. FONAIAP Caracas. 283 p. [ Links ]
19.MAZZANI B. y S. HOROVITZ. 1952. Mejoramiento de Sesamum indicum L., de frutos indehiscentes. Agronomía Trop. 2(3):197-205. [ Links ]
20.MONTILLA, D. y B. MAZZANI. 1966. Estimación de pérdidas de semillas en la cosecha mecánica del ajonjolí. Agronomía Trop. 16:209-211. [ Links ]
21.NAVA, A. y A. LAYRISSE. 1991. Variedades comerciales de ajonjolí en Venezuela. FUNDESOL. Caracas. 56 p. [ Links ]
22.NEI, M. 1978. Estimation of average heterozigosity and genetic distance from a small number of individuals. Genetics 89:583-590. [ Links ]
23.NTSYS-pc. 1990. Version 1.60. Applied Biostatistics, Inc. [ Links ]
24.PAGE, R. 1996. TREEVIEW: An application to display phylogenetic trees on personal computers. Computer Applications in the Biosciences 12:357-358. [ Links ]
25.SHEN, J., X. ZHANG, Y. ZHAO, X. FENG, H. CHENG and X. WU. 1995. Cluster analysis of sesame germplasm in China. Journal of Huazhong Agricultural University 14:532-536. [ Links ]
26.SILVA, R. y E. MONTEVERDE. 1998. Estimación de la heterosis y heredabilidad en una población indehiscente de ajonjolí tipo chino. Agronomía Trop. 48(2):177-192. [ Links ]
27.SINGH, R., A. SINGH, K. PARKASH, N. THAKRAL and P. KUMAR. 1997. Genetic divergence in sesame. Annals of Biology Ludhiana 13:41-45. [ Links ]
28.SNEATH, P. and R. SOKAL. 1973. Numerical taxonomy. San Francisco. Freeman and Company. 573 p. [ Links ]
29.SWAIN D. and U. DIKSHIT. 1997. Genetic divergence in rabi sesame (Sesamum indicum L.). Indian Journal of Genetics and Plant Breeding 57:296-300. [ Links ]
30.WEI W., H. ZHANG, F. LU and S. WEI. 1994. Principal components analysis and genetic distance estimation and their application in sesame breeding programme. Acta Ágriculturae Boreali Sinica 9:29-33. [ Links ]
31.IURONG, Z., Z. YINGZHONG, C. YOUNG, F. XIANGYUN, G. QUINGYUAN, Z. MINGDE and T. HODGKIN. 2000. Establishment of sesame germplasm core collection in China. Genetic Resources and Crop Evolution 47:273-279. [ Links ]