Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Archivos Latinoamericanos de Nutrición
versión impresa ISSN 0004-0622versión On-line ISSN 2309-5806
ALAN v.56 n.2 Caracas jun. 2006
Comparación del perfil de sensibilidad a antibióticos de cepas de Listeria monocytogenes y Salmonella spp. aisladas a partir de alimentos con cepas de origen clínico
Juan Manuel Zamora, Carolina Chaves y María Laura Arias
Facultad de Microbiología, Universidad de Costa Rica. Centro de Investigación en Enfermedades Tropicales
Resumen
El uso actual de los antibióticos no se da únicamente con fines terapéuticos, sino que se ha extendido hacia la prevención de enfermedades y como promotores del crecimiento en animales. Esta prácticas han llevado a la propagación de resistencia a antibióticos, lo cual representa un riesgo en Salud Pública. En el presente estudio, se evaluó el perfil de sensibilidad a antibióticos de 20 cepas de Listeria monocytogenes y 40 cepas de Salmonella spp. aisladas a partir de alimentos y se comparó con los perfiles de sensibilidad de 20 cepas de L. monocytogenes y 100 de Salmonella sp de origen clínico.
El 95% de las cepas de L. monocytoges aisladas a partir de alimentos fue sensible a ampicilina, comparado con el 65% de las cepas de origen clínico. De la misma manera, el 100% de las cepas alimentarias mostraron sensibilidad a la gentamicina, comparado en el 85% de las cepas clínicas. El 95% de ambas mostró sensibilidad a tripetropin sulfametoxazol y el 100% a ciprofloxacina.
Con respecto a Salmonella spp., para los antibióticos trimetoprim sulfametoxazol, gentamicina, ciprofloxacina, ácido nalidíxico y amoxicilina/ácido clavulánico, los porcentajes de sensibilidad fueron similares, sin embargo, las cepas de origen alimentario mostraron un 97,5% y un 82.5% de sensibilidad a la tetraciclina y cefalexina respectivamente, comparado con un 83 y 90% obtenido a partir de las cepas de origen clínico.
Los resultados obtenidos ponen de manifiesto el riesgo potencial que representan las cepas bacterianas aisladas de alimentos en la transmisión de resistencia a los antibióticos.
Palabras clave: Listeria monocytogenes, Salmonella, perfil de sensibilidad a los antibióticos
Comparison of the antibiotics sensibility pattern of Listeria monocytogenes and Salmonella spp. strains isolated from food with clinical origin samples.
Summary
The actual use of antibiotics includes, not just its therapeutic cases, but also for disease prevention and as a growth promoter in animals. These practices have resulted in the propagation of resistance to antibiotics, representing a threat for Public Health. In this work, the antibiotic sensibility pattern of 20 Listeria monocytogenes and 40 Salmonella spp. strains, isolated from foodstuff was studied and compared with the antibiotic sensibility patterns of 20 L. monocytogenes and 100 Salmonella strains of clinical origin.
95% of the L. monocytogenes strains isolated from food were sensible to ampicillin, compared with the 65% of the clinical origin strains. Same way, 100% of food strains were sensible to gentamicin, compared with 85% of clinical origin strains. 95% of both showed sensibility to trimethoprim sulfametoxazole and 100% to ciprofloxacin.
For Salmonella spp., the sensibility patterns for trimethoprim sulfametoxazole, gentamicin, ciprofloxacin, nalidixic acid and amoxicilin/clavulanic acid from both origins were similar. Nevertheless, food origin strains showed a 97,5% and 82,5% sensibility for tetracycline and cephalosporin respectively, compared with a 83 and 90% sensibility shown by clinical origin strains.
The results obtained demonstrate the potential risk that bacterial strains isolated from food represent in the transmission of antibiotics resistance.
Key words: Listeria monocytogenes, Salmonella, sensibility pattern to antibiotics.
RECIBIDO: 05-12-2005
ACEPTADO: 06-06-2006
Introducción
Las enfermedades de transmisión alimentaria han presentado, en los últimos años, un aumento significativo en su frecuencia. Entre los principales patógenos involucrados, se encuentran Salmonella spp. y Listeria monocytogenes, entre otros. Estos agentes producen una morbilidad y mortalidad importante, aunado a altos costos para el sector salud (1).
Estas infecciones requieren, generalmente, tratamiento con agentes antimicrobianos, con el fin de reducir la mortalidad y morbilidad asociadas. (2).
El uso indiscriminado de antibióticos en diversas prácticas, incluyendo las médicas, veterinarias y agrícolas resulta en una presión selectiva sobre las bacterias, contribuyendo a la selección de microorganismos con patrones de resistencia a antibióticos (3-6). La presencia y persistencia de bacterias resistentes a antibóoticos ha sido descrita en diferentes ambientes, incluyendo suelos (7), agua superficial y potable (8,9) y alimentos (10) y representa un creciente problema de Salud Pública. En el caso de Listeria monocytogenes, se describe una creciente incidencia, especialmente en alimentos listos para consumir y que son conservados en refrigeración por tiempo prolongado (11). Al mismo tiempo, se describe un aumento en la transferencia de factores de resistencia por conjugación de plásmidos y transposones a partir de Enterococcus y Streptococcus (12). Para Salmonella spp., se ha descrito una rápida y creciente emergencia de cepas multirresistentes provenientes de animales y humanos (2,13-15) lo cual incide en un creciente número de fracasos terapéuticos (16). La resistencia a algunos antibióticos, incluyendo β lactámicos, tetraciclina, cloranfenicol y trimetoprim sulfa es cada vez mayor (8,14). Además, en los últimos tiempos se ha observado, a nivel mundial, la emergencia de cepas de S. typhimurium multirresistentes, procedentes de animales y humanos (2).
Dado lo anterior, el propósito del presente trabajo es comparar la susceptibilidad a antibióticos de cepas de L. monocytogenes y Salmonella spp aisladas a partir de alimentos con cepas de origen clínico de Costa Rica.
Material y métodos
El estudio se realizó entre los meses de enero y abril del 2005 en el Laboratorio de Microbiología de Aguas y Alimentos de la Facultad de Microbiología de la Universidad de Costa Rica.
Población estudiada
Se analizó el perfil de sensibilidad a antibióticos de 20 cepas de Listeria monocytogenes y 40 cepas de Salmonella sp aisladas e identificadas a partir de alimentos en el Laboratorio de Microbiología de Aguas y Alimentos de la Facultad de Microbiología de la Universidad de Costa Rica entre los años 2002 y 2004.
Para la comparación con cepas de origen clínico se utilizaron registros de sensibilidad a antibióticos de 100 cepas de Salmonella y 20 cepas de Listeria monocytogenes aisladas en el Laboratorio de Bacteriología del Hospital Nacional de Niños entre los años 2002 y 2004.
Prueba de sensibilidad a antibióticos:
La susceptibilidad a los antibióticos de las cepas aisladas de muestras de alimentos se determinó por difusión en agar usando discos impregnados con antibióticos de la casa comercial Oxoid, según la técnica Kirby Bauer (17). El diámetro de los halos de inhibición fue medido e interpretado según las recomendaciones del National Comittee for Clinical Laboratory Standard (NCCLS) (18). Como control de calidad, se emplearon las cepas E.coli ATCC 25922, para Gram negativos y S. aureus ATCC 25923 para Gram positivos.
Para Listeria monocytogenes, se probaron los antibioticos ampicilina (AMP 10 μg), penicilina (P 10 μg), tetraciclina (TE 30 μg), trimetoprim-sulfametoxazol (STX 25 μg), cloranfenicol (C 30 μg.), gentamicina (CN 10 μg), ciprofloxacina (CIP 5 μg), clindamicina (CLI 5μg) y rifampicina (RIF 5 μg). Para Salmonella spp, se incluyó tetraciclina (TE 30 μg), trimetoprim-sulfametoxazol (STX 25 μg), gentamicina (CN 10 μg), ciprofloxacina (CIP 5 μg), neomicina (N 30 μg), αcido nalidíxico (NA 30 μg), amoxicilina (AM10 μg), amoxicilina/ αcido clavulánico (AMC 10μg), cefalexina (Cl 30 μg) y cloranfenicol (C 30 μg.)
Prueba de sensibilidad a antibióticos de cepas aisladas de muestras clínicas:
La prueba de sensibilidad a antibióticos de las cepas de Listeria monocytogenes se realizó con el método microdilución, usando el sistema Vitek con la tarjeta de sensibilidad GPS 107. Se determinó la sensibilidad de los aislamientos a 4 antibióticos de uso clínico: ampicilina, trimetoprim-sulfametoxazol, gentamicina y ciprofloxacina.
La prueba de sensibilidad de los aislamientos clínicos de Salmonella sp se realizó también usando el sistema Vitek con la tarjeta de sensibilidad GNS 204. Se probaron los antibióticos tetraciclina, trimetoprim-sulfametoxazol, gentamicina, ciprofloxacina, ácido nalidíxico, amoxicilina/ácido clavulánico y cefalexina.
Resultados y Discusión
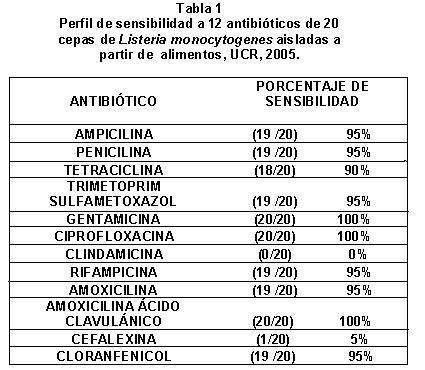
Los resultados de la prueba de sensibilidad para los 20 cultivos de origen alimentario de Listeria monocytogenes se muestran en la Tabla 1. De los 20 cultivos, el 95% presentó sensibilidad a ampicilina, penicilina, trimetoprim sulfametoxazol, rifampicina, amoxicilina y cloranfenicol. El 100% de los cultivos fue sensible a gentamicina, ciprofloxacina y amoxicilina- ácido clavulánico, el 90% fue sensible a tetraciclina. Los resultados para estos 10 antibióticos muestran una buena sensibilidad a antibióticos de primera elección, betalactámicos como ampicilina y penicilina y aminoglucósidos como gentamicina y otros de segunda elección como trimetoprim-sulfametoxazol y ciprofloxacina. Sin embargo, los cultivos mostraron una resistencia de 95% a cefalexina. El alto porcentaje de cultivos resistentes a cefalosporinas es un evento bastante descrito en la literatura (19). El 100% de los cultivos mostró resistencia a clindamicina. Este hallazgo concuerda con la resistencia descrita de Listeria monocytogenes a este antibiótico, como lo demuestra los estudios de Saldar en el 2002, donde describió 96.2% de resistencia a clindamicina en 84 aislamientos de pacientes con listeriosis sistémicas en un centro para pacientes con cáncer (10).
Es importante destacar que una de las cepas de L. monocytogenes aislada a partir de alimentos (UCR Listeria monocytogenes 20) presentó resistencia a 8 de los 12 antibióticos probados, exceptuando gentaminicina, ciprofloxacina, cefalexina y amoxicilina-ácido clavulánico.
La presencia de cepas multirresistentes a antibióticos en Listeria monocytogenes es un fenómeno relativamente reciente. Los estudios de Charpentier, 1999, demuestran la adquisición de resistencia a cloranfenicol, tetraciclinas, macrólidos y trimetoprim-sulfametoxazol por transmisión de "clusters" de genes de Enterococcus y Streptococcus hacia Listeria monocytogenes.(12,20).
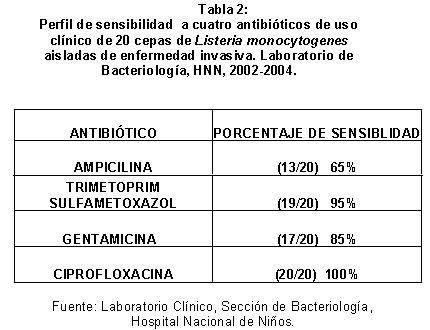
El perfil de sensibilidad a antibióticos de los cultivos de Listeria monocytogenes realizados a partir de muestras de origen clínico se muestra en la Tabla 2. Es importante resaltar la resistencia a los antibióticos de primera elección ampicilina y gentamicina. De los 20 cultivos, el porcentaje de resistencia a ampicilina fue de 35% y el de gentamicina fue de 15%. Los cultivos mostraron una buena sensibilidad a antibióticos de segunda elección como trimetoprim-sulfametoxazol, 95% de sensibilidad, y ciprofloxacina 100% de sensibilidad.
Al comparar los cultivos de origen alimentario con los clínicos, el perfil de sensibilidad a ampicilina y gentamicina fue diferente en los primeros. El 95% de las cepas de origen alimentario fue sensible a ampicilina, mientras que sólo el 65% de las cepas de origen clínico fue sensible a este antibiótico. El 100% de las cepas aisladas de muestras de alimentos fue sensible a gentamicina, comparado con el 85% de los aislamientos clínicos.
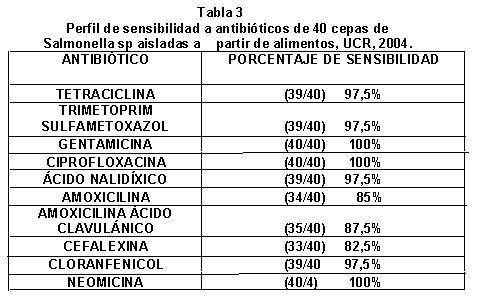
El perfil de sensibilidad a antibióticos para Salmonella spp aisladas a partir de muestras de origen alimentario se presenta en la Tabla 3.
Los aislamientos mostraron un perfil de resistencia a antibióticos betalactámicos. Los porcentajes de resistencia variaron entre 12.5% para amoxicilina-ácido clavulánico, 15% para amoxicilina y un 17.5% para cefalexina.
Cabe resaltar que una única cepa de Salmonella spp, (UCR Salmonella 01) aislada de una muestra de alimentos, presentó resistencia a tetraciclina, trimetoprim-sulfametozaxol, ácido nalidíxico, amoxicilina, amoxicilina- ácido clavulánico, cloranfenicol y ampicilina. Este hallazgo representa el primer caso documentado de multirresistencia en Salmonella en Costa Rica. La multirresistencia en Salmonella es un fenómeno reciente que cada vez logra mayor importancia en salud pública. Así lo demuestran los aislamientos de Salmonella serotipo typhimurium portadora del fago DT104, el cual transmite resistencia a 5 antibióticos (2,17) La cepa UCR Salmonella 01 presentó resistencia a 7 antibióticos, 4 de los cuales son inactivos frente a Salmonella typhimurium DT104. Es importante resaltar el riesgo que representa la presencia de una cepa multirresistente a nivel alimentario y la posibilidad de transmisión de genes de resistencia mediada por integrones (8).
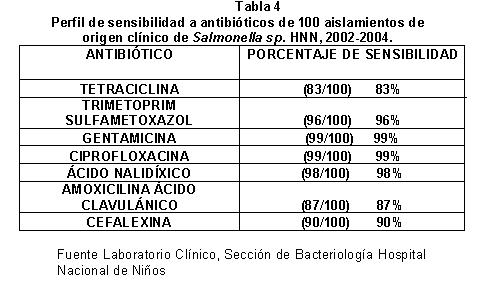
En la Tabla 4 se presenta el perfil de sensibilidad de los aislamientos clínicos. Se puede observar que el comportamiento de las cepas bacterianas de ambos orígenes fue similar, excepto en dos antibióticos: tetraciclina (97.5% muestras de alimentos, 83% de muestras clínicas) y cefalexina (82.5% muestras de alimentos, 90% de muestras clínicas). La mayor resistencia descrita para cefalexina a partir de las muestras de origen alimentario comparadas con las de origen clínico, resulta una llamada de atención para el sector productor del país y sugiere la necesidad de evaluar la alimentación dada a animales de engorde o al uso indiscriminado de antibióticos en los sectores agrícolas y ganaderos nacionales.
La selección y diseminación en la naturaleza de cepas resistentes a los antibióticos es una práctica que se debe controlar, con el fin de mantener un balance ecológico que favorezca el predominio de bacterias susceptibles y asegurar el tratamiento efectivo de las enfermedades infecciosas humanas.
Referencias
1. Reuben A, Treminio H, Arias ML y C. Chaves. Presencia de Escherichia coli O157H7 Listeria monocytogenes y Salmonella spp. en alimentos de origen animal en Costa Rica. Arch Lat Nut. 2003; 53: 389-392. [ Links ]
2. Aidara-Kane A. Dual emergence in food and humans of a novel multiresistant serotype of Salmonella in Senegal: Salmonella enterica subsp. enterica Serotype 35:c:1,2. J Clin Microb. 2001; 39: 2373-2374. [ Links ]
3. Andersson D, Levin B. The biological cost of antibiotic resistance. Current Opinion in Microbiology. 1999; 2: 489-493. [ Links ]
4. Aarestrup FN, Wegener HC. The effects of antibiotic usage in food animals on the development of antimicrobial resistance of importance for humans in Campylobacter and Escherichia coli. Microbes Infect. 1999; 1: 639-644. [ Links ]
5. Al Jebouri M. A note on antibiotic resistance in the bacterial flora of raw sewage and sewage polluted river Tigris in Mosul, Iraq. J Appl Bacteriol. 1985; 58: 401-405. [ Links ]
6. Van den Bogaard AE & Stobberigh E. Antibiotic usage in animals: impact on bacterial resistance and public health. Drugs. 1999; 58: 589-607 [ Links ]
7. Galland J, Hyatt D, Crupper S & Acheson D. Prevalence, antibiotic susceptibility and diversity of Escherichia coli O157:H7 isolates from a longitudinal study of beef cattle feedlots. Appl Environ Microbiol. 2001; 67: 1619-1627. [ Links ]
8. Gallardo F, Ruiz J, Marco F, Towner KJ, Vila J. Increase in incidence of resistance to ampicillin, chloramphenicol and trimethoprim in clinical isolates of Samonella serotype typhimurium with investigation of molecular epidemiology and mechanisms of resistance. J Med Microbiol 1999; 48: 367-74. [ Links ]
9. Sayah R, Kaneene J, Johnson Y & Miller R. Patterns of antimicrobial resistance observed in Escherichia coli isolates obtained from domestic and wild animal fecal samples, human sewage and surface water. Appl Environ Microbiol. 2005; 71: 1394-1404. [ Links ]
10. Saldar A & Armstrong D. Antimicrobial activities against 84 Listeria monocytogenes isolates from patients with sistemic listeriosis at a comprensive cancer center (1955-1997). J Clinical Microbiol. 2003; 49: 483-485. [ Links ]
11. Center for Food Safety and Applied Nutrition, Food and Drug Administration; Food Safety and Inspection Service, US Department of Agriculture and Center for Disease Control and Prevention. Quantitative assessment of relative risk to public health from foodborne Listeria monocytogenes among selected categories of ready to eat foods. Online httP: www.foodsafety.gov/dms/lmr2.toc.html 2003. [ Links ]
12. Gray M, Zadoks R, Fortes E, Dogal B, Cai S, Chen Y, Scott V, Gombas David, Boor K & Wiedmann M. Listeria monocytogenes isolates from foods and humans form distinct but overlapping populations. Appl Environ Microbiol. 2004; 70: 5833-5841. [ Links ]
13. Martel JL, Chaslus-Dancla E, Coudert M, Lafont JP. Evolution de la sensibilité aux antibiotiques des Salmonelles d'origine bovine en France. Med Mal Infect 1996; 26: 415-9 [ Links ]
14. Breuil J, Berger N, Dublanchet A et le collège BVH. Sensibilité aux antibiotiques de 2800 souches de Salmonelles et Shigelles isolées en France en 1994. Med Mal Infect 1996; 26: 420-5 [ Links ]
15. Yang H, Chen S, White D, Zhao S, McDermott P, Walker R & Meng J. Characterization of multiple antimicrobial resistant Escherichia coli isolates from diseased chickens and swine in China. J Clin Microbiol. 2004; 42: 3483-3489. [ Links ]
16. Aarestrup, FM. Association between the consumption of antimicrobial agents in animal husbandry and the occurrence of resistant bacteria among food animals. Inter J Antimicrob Ag 1999; 12: 279-85. [ Links ]
17. Anonymous. Clinical Microbiology Procedures Handbook. ASM, Washington, 1995, p 460-539. [ Links ]
18. National Committee of Clinical Laboratory Standard. 1997. Performance standard for antimicrobial disk susceptibility test, 6th ed. Approved Standard Documents M2. National Committee of Clinical Laboratory Standard, Wayne, Pa. [ Links ]
19. Rocourt J, BenEmbarek P, Toyofuju H & Schlundt J. Quantitative risk assessment of Listeria monocytogenes in rady to eat foods: the FAO/WHO approach. FEMS Immunol Med Microbiol. 2003; 35: 263-267. [ Links ]
20. Lorber B. Listeriosis. Clin Infect Dis. 1996; 24: 1-11. [ Links ]