Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Obstetricia y Ginecología de Venezuela
versión impresa ISSN 0048-7732
Rev Obstet Ginecol Venez vol.72 no.4 Caracas dic. 2012
Diseño de un sistema de PCR para la detección del virus del papiloma humano mediante el uso de oligonucleótidos degenerados de la región E6
Lcda. Danmarys Hernández, Mg.Scs. Jhon Cruz, Militza Quintero Vega, Marco Bastidas, Dr. Juan Puig Pons
Laboratorio de Biología y Medicina Experimental (LABIOMEX), Facultad de Ciencias, Departamento de Biología, Universidad de Los Andes. Mérida, Estado Mérida, Venezuela.
RESUMEN
Objetivo: Diseñar y estandarizar un sistema de PCR para detectar un amplio espectro de tipos de VPH en una sola reacción utilizando como blanco la región E6 del genoma viral. Métodos: Utilizando secuencias del gen E6 de distintos tipos de VPH del NCBI y las herramientas informáticas Mult Alin versión 5.4.1. y Oligo Analyzer 1.1.2 se diseñaron oligonucleótidos degenerados que permitían la amplificación de un fragmento de aproximadamente 214 pb. Las muestras seleccionadas incluyen: 25 muestras de ADN, cada una positiva para un solo tipo de VPH tipificados por el sistema PCR-RFLP MY11/MY09 del gen L1. Resultados: Se obtuvieron amplificados de aproximadamente 214 pb de 25 tipos distintos de VPH, se detectaron amplificados de E6 en muestras negativas para el sistema MY09/MY11. Conclusiones: Se pudo determinar que el diseño de los oligonucleótidos degenerados fue altamente eficiente para la amplificación de por lo menos 25 tipos distintos de VPH lo que facilitaría la detección en muestras clínicas.
Palabras clave: VPH, Gen E6, PCR, MY09/MY11.
SUMMARY
Objective: Design and standardize a PCR system to detect a broad spectrum of HPV types in a single reaction using target the E6 region of the viral genome. Methods: Using E6 gene sequences of different HPV types and NCBI tools Mult Alin Oligo Analyzer version 1.1.2 5.4.1. Degenerate oligonucleotides were designed that allowed amplification of a fragment of approximately 214 bp. Selected samples include: 25 DNA samples, each positive for a single HPV type typified by PCR-RFLP system MY11/MY09 L1 gene. Results: We obtained approximately 214 bp amplified from 25 different HPV types were detected in samples amplified from E6 negative MY09/MY11 system. Conclusions: It was determined that the design of degenerate oligonucleotides was highly efficient for amplification of at least 25 different HPV types which would facilitate detection in clinical samples.
Key words: HPV, E6 gene, PCR, MY09/MY11.
INTRODUCCIÓN
Estudios epidemiológicos apuntan a la infección por VPH como el principal factor de riesgo para desarrollar cáncer cervical, las proteínas E6 y E7 de los virus de alto riesgo son las principales mediadoras de la carcinogénesis, debido a su interacción con varios blancos celulares (1). La proteína E6 activa telomerasas y kinasas SRC e inhibe p53 y BAK, actuando como represor de la apoptosis y mediador de la supervivencia de las células que poseen daños significativos (2). Las proteínas que son blanco de E6 intervienen en la apoptosis, regulación de la transcripción, estabilidad cromosómica, diferenciación y organización epitelial, adherencia célula-célula, polaridad y control de la proliferación de células infectadas (3). Durante la progresión a cáncer, se generan cambios importantes en la expresión de los genes virales, estos cambios son diferentes y dependen del tipo viral (4). Normalmente, durante la infección con VPH el genoma viral se mantiene en bajo número de copias como episoma y durante la diferenciación del epitelio se incrementa la expresión de las proteínas E1, E2, E4 y E5 así como L1 y L2 produciendo el ensamblaje y la liberación de los viriones (5). En la progresión a cáncer cervical, la producción de viriones se restringe a pequeñas áreas en la superficie del epitelio y la integración del genoma del virus en el genoma de la células va acompañado de la pérdida de la regulación de la expresión de E6 y E7, así como la pérdida de los genes E2, E1 y L1 (5,6). Debido a la interrupción del ORF de E1 y E2 se genera sobreexpresión de los oncogenes E6 y E7 que favorece la transformación maligna (7,8).
En cuanto a la detección molecular y la tipificación, dos métodos de PCR son relevantes para la detección del ADN de VPH: la PCR tipo específico y la PCR general o de amplio espectro (9). El método de PCR general más popular es el que amplifica el gen L1 utilizando los oligonucleótidos MY09/MY11, GP5+/ GP6+, la desventaja de este sistema de PCR es que el ORF del gen que se usa como blanco puede perderse cuando el virus se integra en el genoma de la célula hospedera (lo que sucede con frecuencia en los tipos oncogénicos), por lo que se desarrollaron sistemas de PCR tipo específica; que utilizan oligonucleótidos para los genes E6 y E7, los cuales se mantienen intactos durante la integración viral, del mismo modo, estos genes admiten el empleo de sistemas de PCR como el PCR tiempo real, que permite determinar carga viral (10,11).
Park y col. (12) evaluaron la eficiencia de los oligonucleótidos MY09/MY11 y de oligonucleótidos específicos para E6 en la detección de VPH y reportaron una tasa de detección significativamente alta con los oligonucleótidos para E6 de 54 % en comparación con un 26 % con los oligonucleótidos para L1, no obstante, a pesar de los resultados obtenidos, apoyan el uso de los oligonucleótidos consenso L1 por su capacidad para detectar simultáneamente varios tipos de VPH. Resnick y col. (13) así como también Yoshikawa y col. (14) diseñaron sistemas para la detección de VPH que emplean oligonucleótidos consenso para E6, tipificando por SouthernBlot y RFLP respectivamente, diez años más tarde, Sasagawa y col. (15) amplificaron todo el gen E6 y parte N-terminal del gen E7 de más de 34 papilomavirus, estos amplificados, fueron analizados mediante RFLP para tipificación. De igual modo, Sotlar y col. (16) diseñaron un sistema PCR nested multiplex del gen E6 que permite tipificar por diferencias en el tamaño de los amplificados. Estrategias dirigidas a la región E6/E7 del genoma del virus, son prometedoras para el diagnóstico del VPH, ya que, entre los tipos de alto y bajo riesgo estas secuencias difieren y su uniformidad en cada tipo de papilomavirus significa menor probabilidad de alineamientos no específicos durante la PCR (11). La cantidad de tipos virales infectantes detectados en la población merideña es considerable, estudios realizados por Quintero y col. (17), reportan como tipos virales más frecuentes para la población merideña: 6, 16, 31, 53, 59, 61 y 68. A nivel mundial, los tipos de VPH 6, 11, 16 y 18 son los más frecuentes, siendo 16 y 18 de alto riesgo oncogénico, el VPH tipo 18 es el segundo tipo más común en mujeres infectadas (18).
MÉTODOS
Muestras biológicas. Para la estandarización del sistema PCR del gen E6 de VPH se utilizaron muestras de ADN extraídas a partir de cepillados y biopsias del área genital de pacientes que asistieron a consulta pública y privada de la población merideña y otras regiones del país durante el período comprendido entre junio-2007/ mayo-2008 a los cuales se les practicó, previo consentimiento informado, un examen de tipificación de VPH.
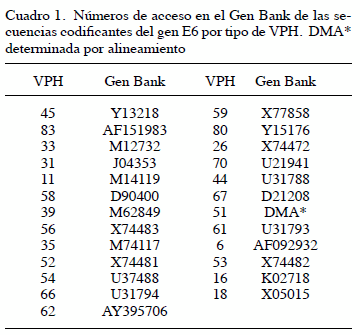
Diseño de oligonucleótidos consenso degenerados. Se realizó la detección y tipificación del virus en las muestras seleccionadas mediante la técnica PCR-RFLP MY09 /MY11 del gen L1 (17). Fueron detectados y tipificados 25 tipos de papilomavirus (6, 11, 16, 18, 26, 31, 33, 35, 39, 44, 45, 51, 52, 53, 54, 56, 58, 59, 61, 62, 66, 67, 70, 80, 83). Se ubicaron las secuencias del gen E6 de estos 25 tipos de VPH en la base de datos del NCBI (National Center for Biotechnology Information) (Cuadro 1) (19). Se realizó el alineamiento de las 25 secuencias en el programa MultAlin versión 5.4.1. (20). Una vez obtenida la secuencia consenso, se seleccionó la región, que mediante el uso de los programas OligoAnalyzer 1.1.2 (21) y el programa BLAST (22) del NCBI cumpliera con las condiciones del diseño de oligonucleótidos, en el Cuadro 2 se presentan las secuencia de los oligonucleótidos consenso degenerados diseñados para el presente estudio.
Sistema PCR general del gen E6 de VPH. Los componentes de la mezcla de PCR incluyen: Tris-HCl pH 8.4 20 mM, KCl 50 mM, MgCl2 3 mM, dNTPs 25 mM, Platinium® Taq ADN polimerasa (Invitrogen) 0.5U, (DAM-E6F/ DAM-E6R) 25pmol, 0.5-1 μl de ADN molde (entre 50-100 ng) para un volumen final de 25 μL. Se utilizó un termociclador Eppendorf Mastercycler ep gradient S® con el programa: Temperatura inicial de desnaturalización 94 ºC por 3 min, 35 ciclos (desnaturalización 94 ºC por 1 min, alineamiento 56 ºC por 1 min, extensión 72 ºC por 1 min), Temperatura de extensión final 72.ºC por 1 min. Los amplificados fueron visualizados en geles horizontales de poliacrilamida 29:1 al 6% en tampón TAE pH 8 0,1 M, teñidos con Bromuro de Etidio (Promega) 0,0006 % y fotografiados en un equipo de foto documentación SyngeneBio-Imagyn®.
RESULTADOS
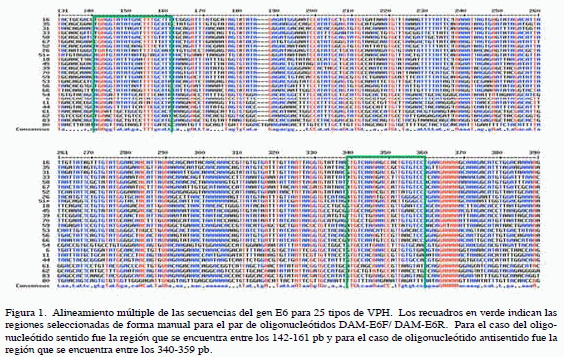
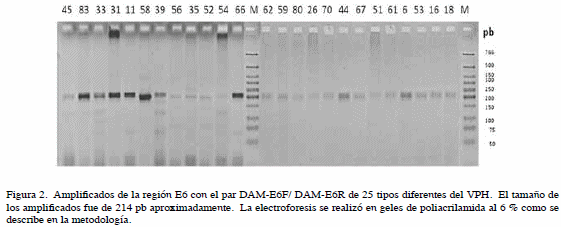
Las secuencias codificantes del gen E6 para los 25 tipos de VPH que poseen los números de acceso en el Gen Bank que se resumen en la Cuadro 1 se alinearon utilizando MultAlin versión 5.4.1. (20) (Figura 1). Obtenida la secuencia consenso; la región que presentó la mayor homología para el caso del oligonucleótido sentido fue la región que se encuentra entre los 142- 161 pb (Figura 1). Para el caso del oligonucleótido antisentido se seleccionó la región entre 340-359 pb (Figura 1). Para determinar el tamaño de los fragmentos amplificados para cada tipo de VPH se realizó el alineamiento del par de oligonucleótidos con cada una de las secuencias del gen E6 de los 25 tipos VPH, resultando una predicción informática de 214 pb de longitud a excepción de los VPH 61, 62 y 80 para los cuales se predijeron longitudes de 286, 87 y 143 pares de bases respectivamente. Todas las muestras para este estudio fueron utilizadas en la estandarización del sistema de PCR general del gen E6 con los oligonucleótidos diseñados DAM-E6F/ DAM-E6R. Tal y como se describe en la sección Materiales y Métodos resultaron positivas para VPH por el sistema de PCR MY09/ MY11 y mediante la técnica RFLP presentaron un patrón característico para un solo tipo de VPH. Al someter las 25 muestras de los diferentes tipos de VPH al sistema de PCR general del gen E6 se obtuvo para todas ellas una banda que se encuentra entre los 200-250pb (aproximadamente 214pb) (Figura 2). Al someter 16 muestras negativas para VPH por el sistema MY09/ MY11 al sistema de PCR general del gen E6 bajo las condiciones establecidas, 9 de ellas presentaron la banda de 214 pb (Figura 3) correspondiente al tamaño del amplificado esperado con los oligonucleótidos diseñados DAME6F/ DAM-E6R.
DISCUSIÓN
Debido a que numerosos estudios epidemiológicos y moleculares han demostrado la asociación del VPH con el desarrollo de lesiones preneoplásicas y neoplásicas del tracto anogenital, las infecciones localizadas en el cuello uterino constituyen el principal foco de atención por presentar mayor susceptibilidad a la transformación maligna (23). Estudios epidemiológicos realizados en Brasil, Colombia, México, Perú y Costa Rica, entre otros países, ratifican la importancia del cáncer del cuello uterino en Latinoamérica (24). En Venezuela, desde la década de los años cuarenta, el cáncer del cuello uterino es una de las principales causas de muerte por cáncer en la mujer. El cáncer cervical es responsable aproximadamente del 20 % del total de las muertes por cáncer en Venezuela; los estados Trujillo, Guárico, Yaracuy, Zulia, Lara, Aragua, Mérida, Táchira, Bolívar y Falcón son las entidades más afectadas por esta patología, con tasas de mortalidad superiores al promedio nacional (24). El gran número de tipos, subtipos y variantes del VPH descritos hasta el momento, solo admite el desarrollo de técnicas moleculares que permitan el estudio de tal diversidad genética (25). Considerando la importancia clínica que posee el conocimiento del tipo viral presente para el seguimiento de mujeres con tratamiento de las lesiones y posterior al mismo, así como el desarrollo de estudios epidemiológicos detallados; generar nuevas metodologías que aborden dicho asunto reviste de gran importancia (25,16). Actualmente en nuestro país no existe una metodología reportada de detección y tipificación que utilice la región E6 como blanco; con el diseño de oligonucleótidos iniciadores consenso degenerados para esta región se pretende iniciar una línea de investigación que apunte a la estandarización de una técnica que detecte un amplio espectro de tipos de VPH y mejore el diagnóstico y seguimiento de los pacientes, disminuyendo la posibilidad latente de reportar falsos negativos cuando se utiliza la región L1. El tamaño de los productos de PCR esperado según los alineamientos del juego de oligonucleótidos con la secuencia de cada tipo de VPH correspondieron con el tamaño de la banda observada luego de la amplificación en la mayoría de los casos, el tamaño de dicha banda se encuentra entre los 200-250 pb del marcador de peso molecular BioLabs (Figura 2). Para el caso de los papilomavirus tipo 61, 62 y 80 el tamaño del amplificado obtenido no correspondió con el tamaño que se determinó virtualmente mediante los alineamientos, no obstante podría decirse que de igual modo en el estudio realizado por Márquez (26), estaríamos en presencia de subtipos o variantes del virus. Para la estandarización del sistema PCR general se ensayaron distintas condiciones que permitieron establecer las concentraciones óptimas de los componentes así como los parámetros del sistema de amplificación. Los oligonucleótidos diseñados hibridan con pequeñas regiones consenso dentro del gen E6 en 25 papilomavirus permitiendo la amplificación de tipos de alto y bajo riesgo como se puede observar en la Figura 2; estos resultados se corresponden con el estudio realizado por Resnick y col. (13) en California donde dos regiones conservadas en la región E6 identificadas por medio de alineamientos permiten el diseño de oligonucleótidos consenso que amplifican un fragmento de 240-250 pb para los VPH 6, 11, 16, 18, 31 y 33. Años más tarde la reproducibilidad con respecto al diseño de oligonucleótidos consenso degenerados para la región E6/E7 sigue en aceptación gracias a estudios como los de Sasagawa y col. (15) en Japón y Sotlar y col. (16) en Alemania. Se sometieron al sistema de PCR general del gen E6 16 muestras que resultaron negativas para VPH por el sistema MY09/MY11 (escogidas al azar y sin distinciones de ningún tipo). Nueve de estas muestras presentan la banda de 214 pb correspondiente al amplificado del fragmento del gen E6 mediante los oligonucleótidos diseñados (Figura 3). De las nueve muestras positivas para E6, 6 correspondieron a pacientes con LIEBG y 3 no presentaron lesiones por lo que se apoya el hecho de que la amplificación de regiones del gen E6 permiten la detección del virus en estado integrado donde la región L1 podría perderse (12). Sin embargo, esta es solo una pequeña prueba exploratoria de la eficiencia de los oligonucleótidos diseñados. Para comprobar el argumento referido en la literatura por muchos autores y que se ha expuesto previamente en este trabajo; donde la elección del gen E6 resulta ventajosa como sitio blanco para el diseño de oligonucleótidos iniciadores consenso degenerados, es necesario un estudio estadísticamente significativo en nuestra población que permita evidenciarlo, ya que este gen no se pierde a consecuencia de la integración del genoma viral en el genoma de las células y su expresión continua ha demostrado ser necesaria para la transformación de las células hospederas.
El alineamiento de las secuencias codificantes del gen E6 para los 25 tipos de VPH seleccionados en este estudio permitió determinar las regiones consenso que hicieron posible el diseño de los oligonucleótidos iniciadores consenso degenerados DAM-E6F/ DAM-E6R, de la misma manera, la estandarización del sistema de PCR general del gen E6 permitió amplificar un fragmento del gen de 214 pb. Los oligonucleótidos diseñados demostraron una cualidad útil en la detección de los VPH en aquellos casos de posible pérdida de la región L1 por integración del cromosoma del virus ya que al amplificar la región E6 es posible descartar falsos negativos generados por la utilización de los oligonucleótidos MY09 /MY11.
Agradecimientos:
A todos los médicos que contribuyeron con la recolección de las muestras en sus respectivas consultas, en especial a la Dra. Adriana Rodríguez. Al personal de la cátedra de Citología de la Facultad de Farmacia y Bioanálisis de la Universidad de los Andes, que evaluaron las citologías, y al personal de LABIOMEX por su valiosísima colaboración y apoyo para la realización de este trabajo.
REFERENCIAS
1. Zur Hausen H. Papillomaviruses causing cancer: Evasion from host–cell control in early events in carcinogenesis. J Natl Cancer Inst. 2000;92:690-698. [ Links ]
2. Boulet G, Horvath C, Vanden Broeck D, Sahebali S, Bogers J. Human papillomavirus: E6 and E7 oncogenes. Int J Biochem Cell Biol. 2007;39:2006- 2011. [ Links ]
3. Mantovani F, Banks L. The Human Papillomavirus E6 protein and its contribution to malignant progression. Oncogene. 2001;20:7874 -7887. [ Links ]
4. Dell G, Gaston K. Contributions in the domain of cancer research: Review Human papillomaviruses and their role in cervical cáncer. CMLS, Cell. Mol. Life Sci. 2001;58:1923-1942. [ Links ]
5. Doorbar J. The papillomavirus life cycle. J Clin Virol. 2005;32(Suppl):7-15. [ Links ]
6. Doorbar J. Molecular biology of human papilomavirus infection and cervical cancer. Clinical Science. 2006;110:525-541. [ Links ]
7. Yu T, Ferber M, Cheung T, Hung Chung T, Wong Y, Smith D. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 2005;158:27-34. [ Links ]
8. Cricca M, Morselli-Labate A, Venturoli S, Ambretti S, Gentilomi G, Gallinella G, et al. Viral DNA load, physical status and E2/E6 ratio as markers to grade HPV16 positive women for high-grade cervical lesions. Gynecol Oncol. 2007;106:549-557. [ Links ]
9. Molijn A, Kleter B, Quint W, Van Doorn L-J. Molecular diagnosis of human papillomavirus (HPV) infections. J Clin Virol. 2005;32(Suppl):43-51. [ Links ]
10. Boulet G, Horvath C, Berghmans S, Bogers J. Human Papillomavirus in cervical cancer Screening: Important Role as Biomarker. Cancer Epidemiol Biomarkers Prev. 2008;17(4):810-817. [ Links ]
11. Morris BJ. Cervical human papilloma virus screening by PCR: Advantages of targeting the E6/E7 region. Clin Chem Lab Med. 2005;43(11):1171-1177. [ Links ]
12. Park J-S, Namkoong S-E, Han S-K, Nha D-J, Lee H-Y, Kim S-J. Comparison of L1 Consensus Primers with E6 Type Specific Primers for Detection of Human Papillomaviruses in Paraffin Sections of Cervical Neoplasia. J Korean Med Sci. 1993;8(1):60-67. [ Links ]
13. Resnick RM, Cornelissen MTE, Wright DK, Eichinger GH, Fox HS, ter Schegget J, et al. Detection and typing of Human Papilloma virus in Archival Cervical Cancer Specimens by DNA Amplification with Consensus Primers. J Natl Cancer Inst. 1990;82(18):1477-1484. [ Links ]
14. Yoshikawa H, Kawana T, Kitagawa K, Mizuno M, Yoshikura H, Iwamoto A. Detection and typing of multiple genital human papilloma viruses by DNA amplification with consensus primers. Jpn J Cancer Res. 1991;82:524-531. [ Links ]
15. Sasagawa T, Minemoto Y, Basha W, Yamazaki H, Nakamura M, Yoshimoto H, et al. A new PCR based assay amplifies the E6-E7 genes of most mucosal human papillomaviruses (HPV). Virus Res. 2000;67(2):127-139. [ Links ]
16. Sotlar K, Diemer D, Dethleffs A, Hack Y, Stubner A, Vollmer N, et al. Detection and Typing of Human Papilloma virus by E6 Nested Multiplex PCR. J Clin Microbiol. 2004;42(7):3176-3184. [ Links ]
17. Quintero M, Cruz J, Bastidas M, Márquez L, Puig J. Detección y tipificación de virus de papiloma humano (VPH) mediante PCR-RFLP. Rev Obstet Venez. 2008;68(1):25-31. [ Links ]
18. de Sanjosé S, Diaz M, Castellsagué X, Clifford G, Bruni L, Muñoz N, et al. Worldwide prevalence and genotype distribution of cervical human papilloma virus DNA in women with normal cytology: A meta-analysis. Lancet Infect Dis. 2007;7:453-459. [ Links ]
19. NCBI. Gen Bank. Base de datos en Internet. En: http://www.ncbi.nlm.nih.gov/. [ Links ]
20. Corpet, F. Multiple sequence alignmet with hierachical clustering. Multialin Nucl.Acids Res. En: http://bioinfo.genotoul.fr/multalin/. [ Links ]
21. Kuulasmaa T. Oligo Analyzer -1.1.2. En: www.uku.fi/~kuulasma/OligoSoftware. [ Links ]
22. NCBI. BLAST. Programa de alineamiento de secuencias en Internet. En: http://blast.ncbi.nlm.nih.gov/Blast.cgi. [ Links ]
23. Alonio L, Dalbert D, Picconi M, Cervantes G, García A, Distefano A, et al. Mutaciones en genes Ha-ras y p53 detectadas mediante PCR-SSCP en lesiones premalignas y malignas de cuello uterino asociadas con virus papiloma humano. Medicina. 2000;60(6):895- 901. [ Links ]
24. Molina J, Guzman C, Mendez V, Blasco E, Tamayo J. Alteraciones cromosómicas en el cáncer de cuello uterino. VITAE Academia Biomédica Digital. 2005;25. [ Links ]
25. Gómez MA, Abba MC, Golijow CD. Detección y genotipificación del papilomavirus humano (HPV) por PCR-LIS-SSCP. Rev Argent Microbiol. 2001;33:22. [ Links ]
26. Márquez L. Análisis de la región MY09/MY11 del gen L1 para la identificación de variantes de los papilomavirus humano tipo 6, 11, 16, 18, 31 y 58. Tesis de maestría. Facultad de Ciencias. Posgrado en Biología Molecular. Laboratorio de Biología y Medicina Experimental (LABIOMEX). 2009; Universidad de Los Andes [ Links ]