Revista Científica
versión impresa ISSN 0798-2259
Rev. Cient. (Maracaibo) v.18 n.4 Maracaibo ago. 2008
Diversidad genética de la raza criollo limonero utilizando marcadores de adn Microsatélites
Genetic diversity of limonero creole breed using Microsatellites molecular markers
Yenen Villasmil-Ontiveros1, Rafael Román Bravo1, Luis Yáñez-Cuéllar 1, Gloria Contreras 2, Jordi Jordana 3 y José Aranguren-Méndez 1
1Unidad de Investigaciones en Producción Animal, Facultad de Veterinaria, Universidad del Zulia. Maracaibo, Venezuela. Correo-e: jaaranguren@luz.edu.ve.
2Instituto de Investigaciones Agrícolas, INIA-Zulia. Venezuela.
3Facultad de Veterinaria, Universidad Autónoma de Barcelona, España.
RESUMEN
Con la finalidad de estudiar la variabilidad genética de la raza Criollo Limonero considerada patrimonio Nacional y orientada a la producción de leche, se analizaron 95 animales puros utilizando 14 marcadores moleculares de ADN del tipo microsatélites. Los animales pertenecen al rebaño de la estación local Carrasquero, adscrita al Instituto Nacional de Investigaciones Agrícolas del estado Zulia (INIA-Zulia) y ubicado al noroeste del Estado. Para medir la diversidad genética se estimaron y discutieron los valores de heterocigosis observada (HO) y esperada (HE) total y entre familias (HMO, HME), probabilidad de exclusión (PE), Índice de Contenido Polimórfico (PIC) y número de alelos por locus. El número promedio de alelos por locus, HE y PIC fueron: 8,7; 0,689 y 0,651, respectivamente. Las HE variaron desde 0,355 hasta 0,787 y el PIC fluctuó de 0,302 a 0,757. El locus menos polimórfico fué el ILST5 y el más polimórfico fue el CSSM66. La PE con los 14 marcadores fue de 0,9962 y 0,9999 para uno y dos padres conocidos, respectivamente. Los resultados indicaron que este grupo de marcadores resultaron ser eficientes para realizar pruebas de paternidad en esta raza, así mismo se muestran que los niveles de heterocigosis indican la existencia de una alta diversidad molecular en la población estudiada, la cual deberá mantenerse como estrategia para la conservación del Criollo Limonero como recurso genético bovino de Venezuela para la producción animal en la región tropical.
Palabras clave: Criollo Limonero, variabilidad genética, microsatélites.
ABSTRACT
In order to study the genetic variability of the Criollo Limonero Breed, a dairy breed considered National Patrimony of Venezuela, 95 purebred animals were analyzed using 14 DNA microsatellites. The animals belonged to the local Carrasquero station, assigned to the National Institute of Agriculture Research of Zulia (INIA-Zulia) and located in the northwest of Zulia State. Average values of observed and expected heterocigocities (HO, HE), between families (HMO, HME) respectively, exclusion probability (PE), Polymorphic Information Content (PIC) and number of alelles by locus were considered and discussed to measure the genetic diversity. The average of alleles by locus, HE and PIC were: 8.7; 0.689 and 0.651, respectively. HE ranged from 0.355 to 0.787 and PIC fluctuated from 0.302 to 0.757. The least polymorphic locus was ILST5 and the most polymorphic was the CSSM66. PE with the 14 markers was of 0.9962 and 0.9999 for one and both known parents, respectively. The results indicate that this set of markers are efficient to make paternity tests in this breed, it is also evident that there are heterozigosity levels indicating the existence of a high molecular diversity in this population, which should be maintained as a strategy for the conservation of the Criollo Limonero breed, a bovine genetic resource of Venezuela for animal production in the tropical regions.
Key words: Criollo Limonero Breed, genetic diversity, microsatellite.
Recibido: 25 / 01 / 2007. Aceptado: 18 / 09 / 2007.
INTRODUCCIÓN
En Venezuela como en otros países tropicales, la reducción del censo y la pérdida de las poblaciones bovinas autóctonas son eventos de causas multifactoriales, donde la razón fundamental es la sustitución de las razas criollas por razas bovinas especializadas para la producción de leche o carne, tanto a través de programas de cruzamiento como por la incorporación de animales puros.
La raza bovina Criollo Limonero es uno de los recursos genéticos más importantes de Venezuela, tiene su asentamiento principal cerca de la población de Carrasquero al norte del estado Zulia, Venezuela. La raza está orientada hacia la producción de leche y su origen se remonta a la época de la colonia como un producto de los animales traídos por los españoles. Durante ese tiempo ha permanecido en interacción con las condiciones tropicales por lo cual ha desarrollado gran rusticidad y adaptación que se combina con un gran potencial lechero [13, 18], lo que le permite aventajar a otros genotipos, ya que pueden ser utilizados en programas de mejoramiento puros o cruzados, destinados a la producción de leche o en explotaciones de doble propósito.
En la actualidad, el inventario de esta raza no sobrepasa los 600 animales puros, lo que según los indicadores propuestos por la Organización de las Naciones Unidas para la Agricultura y la Alimentación (FAO) cataloga a esta raza como en peligro de extinción [32], aunado a este censo reducido, gran parte de los semovientes se encuentran ubicados en una sola explotación por lo que los riesgos de pérdida de la raza por desastres naturales o epidemias se incrementan notablemente [37].
En estas poblaciones de censo reducido, el incremento del valor promedio en las relaciones de parentesco y el apareamiento entre individuos relacionados conduce a un incremento del número de loci homocigotos [31], lo cual conduce inevitablemente a la depresión endogámica, que se expresa con la disminución de los valores fenotípicos de características de producción, reproducción o de resistencia a enfermedades, como consecuencia de la expresión de genes recesivos desfavorables.
La variabilidad genética en las poblaciones pueden ser medidas mediante varias metodologías, destacándose entre ellas el uso de marcadores moleculares de ADN, y en especial el uso de los Microsatélites o STR´s, los cuales son secuencias de repeticiones continuas de 2 a 6 nucleótidos que se encuentran distribuidas a lo largo del genoma de los eucariotas y que se caracterizan por ser altamente polimórficos, de herencia mendeliana simple, codominantes y fácilmente automatizables, de allí que hayan sido utilizados ampliamente para caracterizar razas de diferentes especies, analizar la estructura de la población, y establecer relaciones filogenéticas entre especies diferentes y razas [2-5, 10, 15, 16, 21-23, 27].
El presente trabajo es el primer estudio a gran escala que se realiza en Venezuela para caracterizar un recurso genético animal, a través de la caracterización genética de un grupo de 14 marcadores de ADN del tipo microsatélite y se pretende contribuir en gran medida, al conocimiento del genotipo de la raza Criollo Limonero.
MATERIALES Y MÉTODOS
Se analizaron 95 animales puros Criollo Limonero, a los cuales les fue extraída muestras de sangre utilizando tubos al vacío con EDTA. Se incluyeron individuos de ambos sexos, adultos y jóvenes de las diferentes familias con el objeto de obtener una visión general de la población ubicada en la Estación Local Carrasquero, específicamente en el sector Playa Bonita del municipio Mara del estado Zulia, Venezuela (N10°5813; W72°0722). Este rebaño mantiene un sistema de apareamientos rotacional donde los machos de una familia sirven a las hembras de la familia que le antecede y las becerras se ubican en la familia del padre y los becerros en la familia de la madre [37].
El ADN genómico se extrajo de los leucocitos contenidos en las muestras de sangre con el método que incluye el uso de cloroformo y precipitación con alcohol etílico descrito por Ausubel y col. [6]. Para la extracción se utilizaron 400 µL de sangre y luego de la deshidratación con el alcohol etílico se resuspendió el pellet en 200 µL de una solución de TE (Tris-HCl; 10mM y 1 mM EDTA, pH=8,4).
Los marcadores utilizados han sido descritos en la literatura: CSSM66 [7], ETHE y ETH10 [33], ETH225 [34], HEL1, HEL5 y HEL9 [24], ILST5 [14], INRA5, INRA23, INRA35, INRA37 e INRA63 [36] y TGLA44 [19], las secuencias de los microsatélites y la ubicación se presenta en la TABLA I. Para la amplificación se prepararon 5 reacciones diferentes que abarcaron los 14 marcadores, las reacciones fueron nombradas A1, A2, B1, B2 y C. En la amplificación de la reacción A se incluyeron los marcadores INRA35, HEL5, INRA23, CSSM66 y ETH225. Para la reacción B se incluyeron los marcadores ETH3, INRA37, HEL1, TGLA44, ILST5 y ETH10, por último la reacción C los marcadores HEL9, INRA5 e INRA63. La designación de los marcadores en cada una de las reacciones se realizó tomando en cuenta el fluorocromo utilizado y el rango de los alelos, con la finalidad de evitar que alelos de diferentes loci y marcados con el mismo color se solapen durante la lectura.
TABLA I. MARCADORES UTILIZADOS EN LA AMPLIFICACIÓN / MARKERS USED IN AMPLIFICATION
| Número | Nombre | Cro | Secuencias de los cebadores | Múltiplex | Rango de los alelos |
| 1 | CSSM66 | 11 | F: ACACAAATCCTTTCTGCCAGCTGA | A2 | 177-199 |
|
|
| R: AATTTAATGCACTGAGGAGCTTGG |
|
| |
| 2 | ETH3 | 13 | F: GAACCTGCCTCTCCTGCATTGG | B1 | 105-133 |
|
|
| R: ACTCTGCCTGTGGCCAAGTAGG |
|
| |
| 3 | ETH10 | 5 | F: GTTCAGGACTGGCCCTGCTAACA | B2 | 207-223 |
|
|
| R: CCTCCAGCCCACTTTCTCTTCTC |
|
| |
| 4 | ETH225 | 7 | F: GATCACCTTGCCACTATTTCCT | A2 | 207-223 |
|
|
| R: ACATGACAGCCAGCTGCTACT |
|
| |
| 5 | HEL1 | 9 | F: CAACAGCTATTTAACAAGGA | B1 | 139-157 |
|
|
| R: AGGCTACAGTCCATGGGATT |
|
| |
| 6 | HEL5 | 8 | F: GCAGGATCACTTGTTAGGGA | A1 | 101-117 |
|
|
| R: AGACGTTAGTGTACATTAAC |
|
| |
| 7 | HEL9 | 11 | F: CCCATTCAGTCTTCAGAGGT | C | 149-167 |
|
|
| R: CACATCCATGTTCTCACCAC |
|
| |
| 8 | ILST5 | 3 | F: GGAAGCAATGAAATCTATAGCC | B2 | 143-169 |
|
|
| R: TGTTCTGTGAGTTTGTAAGC |
|
| |
| 9 | INRA5 | 6 | F: CAATCTGCATGAAGTATAAATAT | C | 182-186 |
|
|
| R: CTTCAGGCATACCCTACACC |
|
| |
| 10 | INRA23 | 8 | F: GAGTAGAGCTACAAGATAAACTTC | A2 | 131-147 |
|
|
| R: TAACTACAGGGTGTTAGATGAACTC |
|
| |
| 11 | INRA35 | 5 | F: ATCCTTTGCAGCCTCCACATTG | A1 | 201-215 |
|
|
| R: TTGTGCTTTATGACACTATCCG |
|
| |
| 12 | INRA37 | 8 | F: GATCCTGCTTATATTTAACCAC | B1 | 100-110 |
|
|
| R: AAAATTCCATGGAGAGAGAAAC |
|
| |
| 13 | INRA63 | 6 | F: ATTTGCACAAGCTAAATCTAACC | C | 118-136 |
|
|
| R: AAACCACAGAAATGCTTGGAAG |
|
| |
| 14 | TGLA44 | 11 | F: AACTGTATATTGAGAGCCTACCATG | B2 | 175-185 |
|
|
| R: CACACCTTAGCGACTAAACCACCA |
|
|
Cro: Cromosoma.
Para realizar la Reacción en Cadena de la Polimerasa (PCR), se utilizó un termociclador 9700 (GeneAmp PCR System Perkin Elmer, EUA). El programa de amplificación se inició con una etapa de desnaturalización de 10 minutos a 95°C, seguido de 30 ciclos constituidos por una etapa de desnaturalización de 1 minuto a 95°C, una etapa de anillado de 1 minuto a 55°C y una etapa de elongamiento de 1 minuto a 72°C, para finalizar se realizó una etapa de elongación de 72°C por 20 minutos. La PCR se llevó a cabo en una reacción final de 15 µL, constituida por 50 a 150 ng de ADN genómico, 200 µM de desoxirribonucleótidos (dNTP´s), 0,3 µL de Taq ADN polimerasa (AmpliTaq-Gold, 5 UI/µL) y se utilizaron 60 ng de los cebadores INRA23, 30 ng de los cebadores CSSM66, INRA5, INRA35, ETH3, ETH10, ILST5 y TGLA44, 25 ng de los cebadores HEL1 y 15 ng de los cebadores HEL5, HEL9, ETH225, INRA37 e INRA63.
Para la lectura del producto amplificado y la cuantificación de cada uno de los alelos se usó electroforesis capilar a través de un secuenciador automático ABI3100 (Applied Biosystems 3100 DNA sequencer, EUA), dicha lectura y verificación de los genotipos se realizó con el programa Genescan (Applied Biosystems, EUA).
Las frecuencias alélicas fueron determinadas por conteo directo, los valores medios de heterocigosis esperada (HE) y observada (HO) para cada uno de los loci fueron obtenidos usando el programa Biosys-2 [35]. La heterocigosis esperada asumiendo un equilibrio Hardy-Weinberg se realizó utilizando la fórmula siguiente:
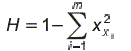
donde m es el número de alelos en el locus y xi es la frecuencia del alelo i en el locus 1.
El índice de contenido polimórfico (PIC) se calculó mediante la metodología de Botstein y col. [11], mediante la ecuación:
![]() , i = 1, j = i – 1
, i = 1, j = i – 1
donde pi pn son las frecuencias de los n alelos.
La probabilidad de exclusión (PE) de cada uno de los marcadores fue estimada según el método de Jamieson [20], utilizando la fórmula siguiente:
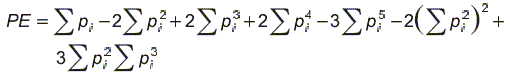
ó
![]()
cuando se refiere a un locus y con n número de alelos, donde i > j y pi pn son las frecuencias de los alelos.
![]() ;
;
para la probabilidad global del conjunto de marcadores.
RESULTADOS y DISCUSIÓN
Todos los marcadores utilizados en este ensayo amplificaron y mostraron alto polimorfismo, detectándose 114 alelos en los 14 loci de los 95 animales estudiados, lo que corresponde a un promedio de 8,14 alelos/locus. El número de alelos por locus varió desde un máximo de 13 alelos en el locus ETH3 hasta 3 alelos que correspondió al locus ILST5 (TABLA II). Cabe destacar que más del 64% (9 de 14) de los marcadores presentaron 8 ó más alelos diferentes, lo que se refleja indiscutiblemente sobre el promedio de alelos tan alto en la población, en los niveles de heterocigosis y en el PIC obtenido. El número promedio de alelos encontrados resultó ser relativamente superior al 5,56 reportado para la raza Criolla Colombiana Casanare [9], al 6,6-8,11 de bovinos autóctonos mexicanos [30], al 6,2 y 7,8 para razas bovinas argentinas y bolivianas, respectivamente [25], al 5,2-6,5 para razas autóctonas de la India [28], al 4,4 para la raza española Mallorquina [3] y al 6,5 reportado para razas autóctonas españolas, francesas y portuguesas [15], sin embargo, resultó ser inferior a lo citado por Barrera [8] en otras razas colombianas que alcanzaron un promedio de 11,58 alelos por marcador. Este alto valor podría explicarse por el alto número de animales muestreados, ya que en general a mayor número de animales muestreados existe mayor posibilidad de detectar mayor cantidad de alelos en la población. El locus que presentó mayor número de alelos en este grupo de marcadores fue el ETH3, coincidiendo de alguna manera con los resultados presentados en la raza española Mallorquina, de igual forma el locus con menos alelos fue el ILST5 [3], lo que sugiere un comportamiento similar de los marcadores en diferentes poblaciones.
TABLA II. CARACTERÍSTICAS FÍSICO-QUÍMICAS DE LOS QUESOS DE MANO EN CUATRO CENTROS DE MAYORES VENTAS EN EL MUNICIPIO GIRARDOT EDO. ARAGUA / PHYSIO-CHEMICAL CHARACTERISTICS OF HAND CHEESE IN THE FOUR BEST SALES CENTERS IN THE GIRARDOT MUNICIPALITY, ARAGUA STATE
| Parámetros estadísticos* | Humedad (%) | Grasa (%) | Proteína (%N×6,38) | pH | Acidez (% Ac. láctico) | NaCl (%) | |||
| bh | bs | bh | bs | bh | bs | ||||
| Media | 51,31 | 24,05 | 49,40 | 22,17 | 45,52 | 4,51 | 0,76 | 1,82 | 3,74 |
| Desviación estándar | 2,39 | 1,63 | 1,68 | 1,46 | 1,67 | 0,49 | 0,16 | 0,57 | 1,29 |
| Valor mínimo | 46,67 | 20,4 | 38,25 | 19,71 | 36,95 | 3,5 | 0,53 | 1,09 | 2,04 |
| Valor máximo | 55,20 | 27,6 | 61,61 | 24,97 | 55,74 | 5,11 | 0,99 | 3,29 | 7,34 |
| Coeficiente de variación | 4,7 | 6,8 | 3,4 | 6,6 | 3,7 | 11 | 21 | 31 | 35 |
* Tamaño de la muestra n=32. bh = base humedad; bs =base seca.
Por la dependencia de la cantidad de alelos del número de individuos muestreados, se recomienda utilizar la HO y HE para medir la variabilidad, tal como su nombre lo indica la HO es el número relativo de individuos heterocigotos para cada locus que se encuentra en la población estudiada, y la HE es la frecuencia relativa que se debería observar luego de apareamientos panmícticos con las mismas frecuencias génicas observadas en la población. En cuanto a la heterocigosis observada, el promedio fue de 0,602 con el valor superior en el locus HEL9 con 0,787 y el valor inferior en el locus ILST5 con 0,278, siendo además muy importantes los loci ETH225, CSSM66 y TGLA44 los cuales presentaron heterocigosis por encima de 0,700.
La HO en la población de Criollo Limonero fue de 0,602 valor superior al 0,350 reportado en animales de las raza Gyr, Nellore, Guzerat y Holstein [26] y muy similar al 0,584 reportado en animales criollos uruguayos [5], al 0,59 en bovinos nativos de la India [28], al 0,608 de la raza española Mallorquina [3] y a los valores obtenidos en bovinos criollos argentinos [38]. Lirón y col. [25] reportan valores de HO, desde 0,615 hasta 0,851 en animales criollos argentinos y bolivianos y animales de las razas Aberdeen Angus, Hereford, Holstein, Nellore, Brahman y Retinta de Andalucía; de igual forma otros autores reportan valores por encima de 0,630 para bovinos criollos mexicanos y para la raza colombiana Casanare [9,30].
La HE promedio en los 14 loci resultó ser de 0,689, oscilando entre 0,355 en el locus ILST5 y 0,870 en el locus CSSM66. Es importante hacer notar, que 8 de los 14 loci presentaron valores superiores a 0,700 lo que demuestra nuevamente la alta variabilidad de la población estudiada; valores similares fueron reportados por Armstrong y col. [5] que encontraron un valor de 0,623 para una muestra de toros criollos uruguayos, Lirón y col. [25] observaron valores entre 0,637 y 0,786 de una muestra de animales criollos bolivianos y argentinos, la raza española Retinta y las razas Nellore, Brahman, Aberdeen Angus y Hereford de rebaños argentinos y bolivianos, y también es superior a lo reportado en la raza bovina Jersey aislada desde los años 1700 en una isla del Reino Unido con el mismo nombre [17]. Los valores obtenidos de HE y HO se encuentran en niveles aceptables, sobretodo si se comparan con valores obtenidos en razas comerciales sometidas a altas presiones de selección (0,35-0,48) entre las que se puede citar a Machado y col. [26], en las razas Gyr, Nellore, Guzerat y Holstein; Peelman y col [29] para la raza Azul de Bélgica y MacHugh y col. [27] para razas cárnicas y lecheras europeas. Tales diferencias refuerzan la tesis que a mayor presión de selección, menor debería ser la variabilidad genética, más aún cuando se utiliza la metodología del modelo animal, ya que con ésta las probabilidades de mantener en el rebaño animales emparentados son superiores a situaciones donde no existen programas de mejora genética establecidos con la inclusión de la matriz de parentescos en la estimación.
El PIC promedio fue de 0,651 y con la excepción de los loci ILST5 e INRA35 que tuvieron valores inferiores a 0,5 todos los demás fueron altamente polimórficos, destacándose de forma individual el locus CSSM66 que presentó un valor de 0,853. Estos valores son coincidentes con los obtenidos en las razas Mallorquina, Criollo uruguayo, Nellore, Criollo argentino, Criollo boliviano, Sahiwall, Hariana y Deoni, en los cuales se reportaron valores que oscilaron entre 0,55 y 0,65 utilizando entre 10 y 20 marcadores microsatélites [3, 5, 25, 28]. El locus CSSM66 fue uno de los más informativos y junto a los loci ETH3, ETH10, ETH225, HEL1, HEL5, HEL9 e INRA23 son los marcadores que mayor contribución aportan al mencionado índice, todos estos marcadores se encuentran con un valor superior a 0,70 producto de un mínimo de 7 alelos y una buena distribución de las frecuencias. Los 8 loci antes mencionados también presentaron valores de PIC superiores al 0,5 en la raza Mallorquina [3] y los marcadores ETH3, ETH10, ETH225 e INRA23 tuvieron un comportamiento similar en la raza Nellore [16], indicando que estos marcadores contribuyen solidamente al conocimiento de la raza en estudio debido a que son altamente informativos. El locus CSSM66 a pesar de no ser el marcador con más alelos si tiene el mayor PIC por la distribución de las frecuencias, tal como lo señalan Zambrano y col. [38], los sistemas más informativos son aquellos que, teniendo un número suficiente de alelos, sus frecuencias están equilibradas. Este elevado nivel de polimorfismo permitió obtener una probabilidad de exclusión con este grupo de microsatélites de 0,9962 cuando se considera solo uno de los progenitores y de 0,9999 cuando se conocen ambos.
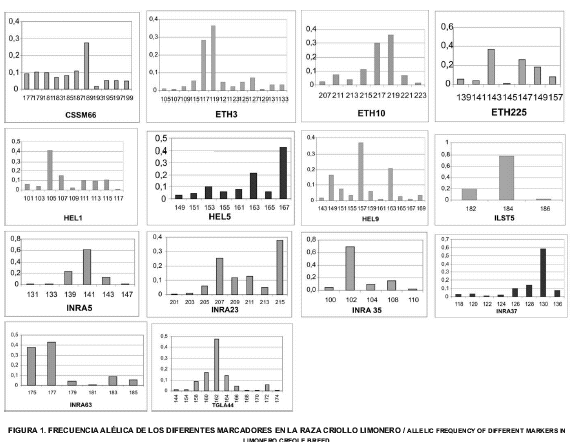
Las frecuencias de los alelos para cada uno de los locus se presentan en la FIG. 1. Con excepción de los alelos 184 en el locus ILST5, 102 en el INRA35, 141 en el INRA5 y 130 en el INRA37, las frecuencias génicas de todos los alelos fueron inferiores al 50%, indicando una distribución importante de los genes en cada uno de los loci restantes. La frecuencia más alta de todos se observó en el locus ILST5, donde el alelo 184 presentó una frecuencia de 77,8% y su alelo 182 con un 20% hacen de este marcador sea casi bialélico en la población, ya que el alelo restante (186) obtuvo una frecuencia inferior al 2%.
Aunque los alelos privados se estudian principalmente para diferenciar razas, 3 alelos privados se encontraron dentro de las familias de ganado Criollo Limonero, no obstante ninguno alcanzó una frecuencia superior al 10% y se presentaron en las familias Bonita, Fundadoras y Compradas y en la familia Joaquín Reyna (TABLA III). Solo el alelo 167 del locus HEL9 sobrepasó el 5%, mientras que, los otros alelos 117 (HEL1) y 181 (INRA63) solo estuvieron entre el 2 y el 3% lo que limita su utilización para la identificación familiar cuando esté presente en un individuo. A pesar de que podría existir una diferencia genética leve en la familia Joaquín Reyna por la introducción de genes de animales centroamericanos, el análisis de los alelos privados indica que, el esquema de los apareamientos y las generaciones transcurridas contribuyen a distribuir los genes a través de las familias, de allí que no existan diferencias importantes entre los grupos familiares estudiados.
TABLA III. ALELOS PRIVADOS Y FRECUENCIA EN FAMILIAS DE LA RAZA CRIOLLO LIMONERO / PRIVATE ALLELES AND FREQUENCY IN FAMILIES FROM LIMONERO CREOLE BREED
| Locus | Familia | Alelo * | Frecuencia en la Familia (%) |
| HEL1 | BONITA | 117 | 2,9 |
| HEL9 | FUNDADORAS | 167 | 5,3 |
| INRA63 | JOAQUIN REYNA | 181 | 2,2 |
* Alelo: tamaño expresado en número de bases.
En la TABLA IV se presenta la variabilidad genética por familia, las heterocigosis observadas (HMO) y esperadas (HME) con sus respectivos errores estándar para cada una de las familias analizadas. Puede observarse que los valores de HMO resultaron ser bastantes similares y oscilaron entre 0,572 ± 0,034 en la familia Fundadoras y Compradas hasta 0,648 ± 0,061 en la familia Bella Vista (P<0,05). En cuanto a la HME los valores oscilaron entre 0,673 ± 0,045 (Hachote) y 0,695 ± 0,042 (Fundadoras y Compradas) (P<0,05).
TABLA IV. VARIABILIDAD GENÉTICA POR FAMILIA, NÚMERO DE ALELOS POR LOCUS, HETEROCIGOSIDAD MEDIA OBSERVADA Y ESPERADA PARA LAS 5 FAMILIAS DE LA RAZA CRIOLLO LIMONERO / GENETIC VARIABILITY BY FAMILY, NUMBER OF ALLELES BY LOCUS, MEAN OBSERVED HETEROCIGOCITY AND EXPECTED FOR THE FIVE FAMILIES FROM LIMONERO CREOLE BREED
| Familia | Individuos / locus | Media de alelos/locus | HMO ± EE | HME ± EE |
| Bella Vista | 15,8 ± 0,3 | 5,4 ± 0,5 | 0,648 ± 0,061 | 0,680 ± 0,039 |
| Bonita | 15,6 ± 0,5 | 6,3 ± 0,6 | 0,602 ± 0,054 | 0,683 ± 0,039 |
| Fundadoras | 17,4 ± 0,6 | 6,5 ± 0,5 | 0,572 ± 0,034 | 0,695 ± 0,042 |
| Hachote | 18,7 ± 0,5 | 6,4 ± 0,7 | 0,594 ± 0,047 | 0,673 ± 0,045 |
| Joaquín Reyna | 20,0 ± 0,5 | 6,8 ± 0,7 | 0,597 ± 0,053 | 0,680 ± 0,045 |
EE = Error Estándar. HMO = Heterocigosis Media Observada. HME Heterocigosis Media Esperada. Individuos/Locus = Número promedio de individuos muestreados por locus.
Tal como sucede con otros grupos raciales en Latinoamérica y Europa, que a pesar de tener un censo muy reducido se mantienen firmes en la idea de no incluir genes de otras razas, los resultados aquí presentados demostraron que existe gran variabilidad genética en esta población y que son el resultado de más de 500 años de interacción con el trópico. Una tesis similar sostiene Chikhi y col. [17] en relación a la raza insular Jersey, señalando que la utilización de semen de Jersey mejorado de otras poblaciones podría rápidamente afectar el pool de genes de la isla, tal como sucedió con la raza Hereford en las Islas Británicas [12] y que no contribuirían de alguna manera si se quieren conservar estos genes. Además se debe considerar que apenas se comienzan a conocer aspectos moleculares en estos genotipos, que en el futuro podrían ayudar a entender los procesos fisiológicos que permiten la resistencia a las altas temperaturas, a las enfermedades y parásitos, y otros elementos que le permiten tener éxito bajo las condiciones estresantes del trópico.
Estos argumentos no implican para nada que la raza no debería estar sometida a un programa de mejoramiento genético, sino que, es necesario un amplio conocimiento de las potencialidades de la raza, que permita tomar decisiones ajustadas a criterios científicos, que determinen un mejoramiento sistemático sin detrimento de las futuras oportunidades que tendría la misma para cubrir algunas exigencias del mercado o de los sistemas de explotación en los cuales se desenvuelve. En este particular, los programas de conservación de algunas de las razas bovinas criollas colombianas, incluyen convenios entre entes de desarrollo agropecuario y criadores con la finalidad de promover centros de multiplicación que prevengan la extinción [1]. Propuestas como éstas deben considerarse, ya que la raza formaría parte del entorno ganadero y sentaría las bases para una iniciativa impactante de su conservación, como podrían ser los beneficios adicionales de comercializar productos con denominación de origen, que han dado resultados en otras latitudes.
CONCLUSIONES
Una consecuencia negativa y limitante para la conservación de poblaciones de censo reducido es la pérdida de variabilidad, no obstante, la diversidad genética obtenida fue alta y comparable a la reportada en otros grupos criollos latinoamericanos que presentan censos mayores, lo que determina que este genotipo sea considerado un valioso recurso genético para Venezuela y Latinoamérica. Adicionalmente se puede señalar que este grupo de marcadores microsatélites son adecuados para el estudio y contribuyen al conocimiento genético de la raza Criollo Limonero, con excepción del locus ILST5, que en esta población resultó ser prácticamente bialélico. La Probabilidad de Exclusión con este grupo de marcadores fue de 99,62 y 99,99% cuando uno o los dos padres son conocidos, por lo que son idóneos para realizar pruebas de paternidad en la población que confirmen o descarten relaciones de parentesco entre animales. Esta práctica garantiza la planificación adecuada de los apareamientos e incide directamente en los planes de conservación.
AGRADECIMIENTOS
Los autores quieren expresar su más sincero agradecimiento al Consejo de Desarrollo Científico y Humanístico de la Universidad del Zulia (CC-0243-04) por el financiamiento de esta investigación, a la Unidad de Mejora genética y de los alimentos de la Universidad Autónoma de Barcelona, España y al Instituto Nacional de Investigaciones Agrícolas del Zulia por el apoyo prestado en el desarrollo de la investigación.
REFERENCIAS BIBLIOGRÁFICAS
1. ANZOLA, H.; MARTÍNEZ, R.; NEIRA, J.; OSSA, G. Desde la conservación a un programa nacional de fomento de los bovinos criollo colombianos. En: VII Simposio Iberoamericano sobre conservación y utilización de recursos zoogenéticos. Cochabamba, 12/5-7. Bolivia. 44-46 pp. 2006. [ Links ]
2. ARANGUREN-MÉNDEZ, J.; GÓMEZ, M.; JORDANA, J. Hierarchical analysis of genetic structure in Spanish donkey breeds using microsatellite markers. Hered. 89: 207-211. 2002a. [ Links ]
3. ARANGUREN-MÉNDEZ, J.; JORDANA, J.; AVELLANET, R.; TORRENS, M. Estudio de la variabilidad genética de la raza Mallorquina para propósitos de conservación, Rev. Cientif. FCV-LUZ. XII(5):358-366. 2002b. [ Links ]
4. ARANGUREN-MÉNDEZ, J.; ROMÁN, R.; ISEA, W.; VILLASMIL, Y.; JORDANA, J. Los microsatélites (STR´s), marcadores moleculares de ADN por excelencia para programas de conservación. Arch. Latinoam. Prod. Anim. 13(1):1-6. 2005. [ Links ]
5. ARMSTRONG, E.; POSTIGLIONI, A.; MARTÍNEZ, A.; RINCÓN, G.; VEGA-PLA, J. Microsatellite analysis of a sample of Uruguayan Creole Bulls (Bos taurus). Genet. and Molec. Biol. 29:267-272. 2006. [ Links ]
6. AUSUBEL, F.; BRENG, R.; KINGSTON, R.; MOORE, D.; SEIDMAN, G.; SMITH, J.; STRUHL, K. Currents protocols in molecular Biology. Green Publishing Associates and Wiley-Interscience, New York. 243 pp.1987. [ Links ]
7. BARENDESE, W.; ARMITAGE, S.; KOSSAREK, L.; KIRKPATRICK, B.; RYAN, A.; SHALOM, A.; CLAYTON, D.; LI, L.; NEIBERGS, H.; NAN, Z.; GROSSE, M.; CREIGHTON, P.; MCCARTHY, F.; RON, M.; SOLLER, M.; FRIES, R.; MCGRAW, R.; MOORE, S.; TEALE, A.; GEORGES, M.; WOMACK, J.; HETZEL, D. A preliminary genetic map of the bovine genome. Nat. Genet. 6:227-235. 1994. [ Links ]
8. BARRERA, G.; MARTÍNEZ, R.; PÉREZ, J.; POLANCO, N.; ARIZA, F. Evaluación de la variabilidad genética en ganado Criollo Colombiano mediante 12 marcadores microsatélites. AGRI. 38: 35-45. 2006a. [ Links ]
9. BARRERA, G.; SASTRE, H.; MARTÍNEZ, R.; ORTEGÓN, Y. Estado genético de la raza criolla colombiana Casanare. En: VII Simposio Iberoamericano sobre conservación y utilización de recursos zoogenéticos. Cochabamba, 12/5-7. Bolivia. 41-43 pp. 2006 [ Links ]
10. BEJA-PEREIRA, A.; ALEXANDRINO, P.; BESSA, I.; CARRETERO, Y.; DUNNER, S.; FERRAND, N.; JORDANA, J.; LALOE, D.; MOAZAMI-GOUDARZI, K.; SÁNCHEZ, A.; CAÑON, J. Genetic characterization of southwestern european bovine breeds: A historical and biogeographical reassessment with a set of 16 microsatellites. J. of Hered. 94:243-250. 2003. [ Links ]
11. BOTSTEIN, D., WHITE, R.; SKOLNICK, M.; DAVIS, R. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. Am. J. of Hum. Genet. 32: 314-331. 1980. [ Links ]
12. BLOTT, S.; WILLIAMS, J.; HALEY, C. Genetic variation within the Hereford breed of cattle. Anim. Genet. 29: 202–211. 1998. [ Links ]
13. BRACHO, I.; CONTRERAS, G.; PIRELA, M.; ZAMBRANO, S. La raza Criollo Limonero: Una realidad para la ganadería de doble propósito. En: Avances en la ganadería de doble propósito. González-Stagnaro, C.; Soto Belloso, E. (Eds). Ed. Astro Data S.A. Maracaibo (Venezuela). Cap. I. 9-25pp. 2002. [ Links ]
14. BREZINSKY, L.; KEMP, S.; TEALE, A. ILST005: a polymorphic bovine Microsatellite. Anim. Genet. 24: 73-75. 1993. [ Links ]
15. CAÑON, J.; ALEXANDRINO, P.; BESSA, I.; CARLEOS, C.; CARRETERO, Y.; DUNNER, S.; FERRAN, N.; GARCIA, D.; JORDANA, J.; LALOE, D.; PEREIRA, A.; SÁNCHEZ, A.; MOAZAMI-GOUDARZI, K. Genetic Diversity of local European beef cattle breeds for conservation purpouses. Genet. Sel. Evol. 33: 311-332. 2001. [ Links ]
16. CERVINI, M.; HENRIQUE-SILVA, F.; GORTARI, N.; MATHEUCCI, E. Genetic variability of 10 microsatellite markers in the characterization of brazilian Nellore cattle (Bos indicus). Genet. and Molec. Biol. 29:486-490. 2006. [ Links ]
17. CHIKHI, L., GOOSSENS, B.; TREANOR, A.; BRUFORD, M. Population genetic structure and inbreeding in an insular cattle breed, the jersey, and its implications for genetic resource management. Hered. 92: 396-401. 2004. [ Links ]
18. CORFONLUZ. Ganado Criollo Limonero. Convenio de cooperación técnica. Edición Comité Ejecutivo CORFONLUZ Boletín Técnico. 2-3 pp. 1997. [ Links ]
19. GEORGES, R.; MASSEY, J. Polymorphic DNA markers in bovidae. World Intellectual Property Org. Geneva. Wo. Publi. No/013102.1992. [ Links ]
20. JAMIESON, A. The effectiveness of using codominant polymorphic allelic series for (1) checking pedigrees and (2) distinguishing full-sib pair members. Anim. Genet. 25: 37-44. 1994. [ Links ]
21. JORDANA, J.; ALEXANDRINO, P.; BEJA-PEREIRA, A.; BESSA, I.; CAÑON, J., CARRETERO, Y.; DUNNER, S.; LALOE, D.; MOAZAMI-GOUDARZI, K.; SÁNCHEZ, A.; FERRAND, N. Genetic structure of eighteen local south European beef cattle breeds by comparative F-Statistics analysis, J. Anim. Breed. Genet. 120: 73-87. 2003. [ Links ]
22. JORDANA, J.; PIEDRAFITA, J. The Bruna dels Pirineus: a genetic study of a rare cattle breed in Catalonia (Spain). Biochem. Systemat. and Ecol. 24:485-498. 1996. [ Links ]
23. KANTANEN, J.; OLSAKER, I.; HOLM, L.; LIEN, J.; VILKKI, K., BRUSGAARD, E.; EYTHORSDOTTIR, E., DANELL, B., ADALSTEINSSON, S. Genetic diversity and population structure of 20 north European cattle breeds. J. of Hered. 91:446-457. 2000. [ Links ]
24. KAUKINEN, J.; VARVIO, S. Eight polymorphic bovine microsatllites. Anim. Genet. 24:148-152. 1993. [ Links ]
25. LIRÓN, J. P.; PERAL-GARCÍA, P.; GIOVAMBATTISTA, G. Genetic characterization of Argentine and Bolivian creole cattle breeds assessed through microsatellites. J. of Hered. 97(4):331-339. 2006. [ Links ]
26. MACHADO, M.; SCHUSTER, I.; MARTÍNEZ, M.; CAMPOS, A. Genetic diversity of four cattle breeds using microsatellite markers. R. Bras. Zoot. 32:93-98. 2003. [ Links ]
27. MACHUGH, D.; SHRIVER, M.; LOFTUS, R.; CUNNINGHAM, P.; BRADLEY, D. Microsatellite DNA variation and the evolution, domestication and phylogeography of Taurine and Zebu cattle (Bos taurus and Bos indicus). Genet. 146:1071-1086. 1997. [ Links ]
28. MUKESH, M.; SODHI, M.; BHATIA, S.; MISHRA, B. Genetic diversity of Indian Cattle breeds as analysed with 20 microsatellite loci. J. Anim. Breed. Genet. 121:416-424. 2004. [ Links ]
29. PEELMAN, L.; MORTIAUX, F.; VAN ZEVEREN, A.; DANSERCOER, A.; MOMMENS, G., COOPMAN, F.; BOUQUET, Y.; BURNY, A.; RENAVILLE, R., PORTETELLE, D. Evaluation of the genetic variability of 23 bovine microsatellite markers in four Belgian cattle breeds. Anim. Genet. 29:161-167. 1999. [ Links ]
30. QUIROZ-VALIENTE, J.; MARTÍNEZ, A.; ULLOA, R.; VEGA-PLA, J. Caracterización genética de las poblaciones bovinas criollas de México. En: VII Simposio Iberoamericano sobre conservación y utilización de recursos zoogenéticos. Cochabamba, 12/5-7. Bolivia. 220-222 pp. 2006. [ Links ]
31. ROCHAMBEAU, H.; FOURNET-HANOCQ, F.; VU THIEN KHANG, J. Measuring and managing genetic variability in small populations. Ann Zoot. 49: 77-93. 2000. [ Links ]
32. SCHERF, B. Lista Mundial de Vigilancia para la Diversidad de los Animales Domésticos. (2a Ed.). Roma: Organización de las Naciones Unidas para la Agricultura y la Alimentación. 222 pp. 1997. [ Links ]
33. SOLINAS-TOLDO, S.; FRIES, R.; STEFFEN, P.; NEIBERGS, H.; BARENDSE, W.; WOMACK, J.; HETZEL, D.; STRANZINGER, G. Physically mapped, cosmid-derived microsatellite markers as anchor loci on bovine chromosomes. Mamm Genom. 4:720-727. 1993. [ Links ]
34. STEFFEN, P.; EGGEN, A.; DIETZ, A.; WOMACK, J.; STRANZINGER, G.; FRIES, R. Isolation and mapping of polymorphic microsatellites in cattle. Anim. Genet. 24:121-124. 1993. [ Links ]
35. SWOFFORD, D. L.; SELANDER, R. B. BIOSYS-2: A computer program for the analysis of allelic variation in population genetic and biochemical systematics. (Release 2.0). University of Illinois. Urbana. Champaign. 1999. [ Links ]
36. VAIMAN, D.; MERCIER, D.; MOAZAMI-GOUDARZI, K.; EGGEN, A.; CIAMPOLINI, R.; LE´PINGLE, A.; VELMALA, R.; KAUKINEN, J.; VARVIO, S.; MARTIN, P.; LEVEZIEL, H.; GUERIN, G. A set of 99 cattle microsatellites: Characterization, synteny mapping and polymorphism. Mamm. Genom. 5:288-294. 1994. [ Links ]
37. VILLASMIL-ONTIVEROS, Y. Caracterización genética de bovinos de la raza Criollo Limonero utilizando marcadores moleculares de ADN del tipo microsatélites. Universidad del Zulia. Tesis de Maestría. 97 pp. 2007. [ Links ]
38. ZAMORANO, M.; GÉNERO, E.; RODERO, A.; VEGA-PLA, J.; RUMIANO, F. Caracterización genética de ganado bovino criollo argentino utilizando microsatélites. Arch. Zoot. 47: 273-277. 1998. [ Links ]












 uBio
uBio