Revista Científica
versión impresa ISSN 0798-2259
Rev. Cient. (Maracaibo) v.18 n.5 Maracaibo oct. 2008
Comparación de modelos sigmoidales aplicados al crecimiento de lactococcus lactis subsp. Lactis.
Comparison of Sigmoid Models Applied to the Growth of Lactococcus lactis subsp. Lactis.
Gustavo Castro1*, Emiro Valbuena1, Edgar Sánchez2, Wilfido Briñez1, Henry Vera1 y Merlis Leal1
1 Unidad de Investigación en Ciencia y Tecnología de los Alimentos. Venezuela. E-mail: gcastro@luz.edu.ve.
2 Unidad de Bioestadística. Facultad de Ciencias Veterinarias, Universidad del Zulia. Maracaibo, Venezuela.
RESUMEN
La microbiología predictiva es una herramienta útil para describir y predecir el crecimiento bacteriano en los alimentos. Para comparar la aplicación de diferentes modelos sigmoidales aplicados al crecimiento de Lactococcus lactis subsp. lactis, se analizaron muestras de leches tomadas del total del ordeño matutino de dos rebaños, bufalino (n=6) y vacuno (n=6), manejados bajo las mismas condiciones, ubicados en el municipio Mara del estado Zulia, Venezuela. Se determinaron algunos parámetros, tales como pH, acidez titulable y presencia de inhibidores. Se preparó una serie de tubos de ensayo con leche esterilizada a 110 ± 2°C por 10 min. y se inocularon con Lactococcus a 1 x 104 ufc/mL. Posteriormente, fueron incubados a 36 ± 0,5°C y cada dos horas se prepararon diluciones y siembra en placas con agar M-17. Se determinó el Log10 de ufc/mL para las 0; 2; 4; 6; 8 y 10 horas de incubación. Se realizó un análisis de regresión con los modelos de Gompertz, Logístico, Stannard y Richards, por medio del algoritmo de Marquardt. Se encontró similitud en el ajuste de los modelos aplicados. Los modelos de Stannard y Richards resultaron ser iguales en la estimación de los datos y fueron los que presentaron mayor dificultad para alcanzar el criterio de convergencia. El modelo de Gompertz de tres parámetros presentó diferencia significativa con los modelos de cuatro parámetros. Se considera los modelos de Gompertz y Logístico (con cuatro parámetros), modificados por Gibson, como los mejores para modelar y predecir el crecimiento del microorganismo en estudio.
Palabras clave: Lactococcus lactis, microbiología predictiva, Gompertz, búfala.
ABSTRACT
Predictive Microbiology is a useful tool to describe and predict the bacterial growing in food. Milk samples from total morning milking of two herds, buffalo (n=6) and cattle (n=6), managed under similar conditions, located in Mara Municipality, Zulia State, Venezuela, were analyzed to compare the application of different sigmoid models to the growth of Lactococcus lactis subsp. lactis strain. Sanitary quality was determined by means of pH, acidity titration and detection of inhibitors. A series of assay tubes were prepared with sterilized milk at 110 ± 2°C for 10 min. The milk in the assay tubes was inoculated with Lactococcus at 1 x 104 ufc/mL. Then, the assay tubes were incubated at 36 ± 0.5°C and every two hours dilutions and culture were prepared in Petrie dishes with M-17 agar. Log10 of ufc/mL was determined for 0; 2; 4; 6; 8 y 10 hours post incubation. A regressions analysis with Gompertz, Logistic, Stannard and Richards models through Marquardt algorithm was used. Similarity was found when models were adjusted. The Stannard and Richards models were equal for the data estimation and they presented the greatest difficulty to reach the convergent criterion. The three parameters Gompertz model showed significant difference compare to the four parameters models. The Gompertz and Logistic models (with four parameters) modified by Gibson are considered the best to model and predict the growing of the microorganism under study.
Key words: Lactococcus lactis, predicitive microbiology, Gompertz, buffalo.
Recibido: 23 / 03 / 2006. Aceptado: 13 / 12 / 2007.
INTRODUCCIÓN
El estudio de los microorganismos ocupa una atención especial en la ciencia de los alimentos, de ellos dependen muchas de las características organolépticas de los mismos, a la vez que afectan su vida comercial. Son de interés los microorganismos patógenos, por los problemas que puedan causar en el consumidor y por otro lado, los cultivos iniciadores, por los beneficios que aportan a la industria de los alimentos. Sean beneficiosos o no, es necesario conocer sus características de crecimiento, los factores que los afectan y como se pueden controlar.
La microbiología predictiva es una ciencia multidisciplinaria (abarca las áreas de la microbiología, ingeniería, estadística), que basada en el estudio del crecimiento de los microorganismos, especialmente las bacterias, ha desarrollado o adaptado modelos matemáticos para describir y predecir el comportamiento de los microorganismos bajo diferentes condiciones [11, 12]. En la ciencia de los alimentos, la microbiología predictiva es útil para el estudio de la inocuidad de los alimentos, tiempo de vida útil y en la optimización o desarrollo de los procesos de producción y comercialización [22].
Existen en la literatura, diferentes modelos matemáticos utilizados para describir el crecimiento bacteriano, algunos toman en cuenta el consumo de sustrato, tal como el de Monod y otros como el de Gompertz, Richards, Standard, Schnute, Brody y el modelo Logístico, no lo toman en cuenta [11, 12]. En el presente estudio, el consumo de sustrato no fue tomado en cuenta, ya que se considera que en la leche existen, en tales concentraciones, como para permitir el crecimiento máximo de los microorganismos. Igualmente se han aplicado las redes neurales para predecir el crecimiento microbiano [5-8, 13, 15, 20].
Las fases de crecimiento de mayor interés en alimentos corresponden a las tres primeras (adaptación, exponencial y estacionaria), ya que es en ellas donde ocurren los mayores problemas microbiológicos (producción de metabolitos importantes, cambios en las características de los alimentos, producción de toxinas, etc.). Tomando esas tres fases de la curva, el crecimiento presenta una forma sigmoidal, de allí que varios modelos sean desarrollados para ajustarse a dicha forma [1].
El Modelo de Gompertz fue primero descrito en la ciencia actuarial para ajustar datos de mortalidad humana [11] y se considera que es la ecuación primaria más ampliamente utilizada para describir el crecimiento microbiano en alimentos bajo condiciones ambientales controladas y definidas [21]. Esta función está basada en una relación exponencial entre la velocidad de crecimiento específico y la densidad de la población. A partir de este modelo se pueden desarrollar modelos secundarios [10, 11, 19]. En la literatura se encuentran algunas variantes para escribir esta ecuación, una con tres parámetros y otra forma modificada por Gibson y col., con cuatro [10, 12].
El modelo logístico, también muy utilizado en estudios predictivos, presenta algunas variantes en la literatura e igualmente una forma modificada por Gibson y col. [10, 12]. El Modelo de Stannard, descrito por dicho autor en 1985, fue reparametrizado por Zwietering y col. [11, 22]. Otro modelo sigmoidal que se encuentra en la literatura, no solo aplicado al crecimiento microbiano, es el modelo de Richards [1, 10, 22], el cual ha sido considerado como un modelo flexible comparado con algunos modelos de tres parámetros, gracias a que posee un parámetro de forma que le permite modelar las curvas con mayor precisión. Sin embargo, ha sido también criticado, ya que dicho parámetro no tiene significado biológico y en ciertas ocasiones genera problemas para ajustar los datos experimentales [12].
No existen en la literatura consultada, bases claras para la selección de cual modelo utilizar o para decir cual es el ideal para casos particulares. Algunos de los principios que se han considerados deben cumplir dichos modelos y que permiten de algún modo seleccionarlos son: parsimonia (simplicidad): los modelos deben contener la mínima cantidad posible de parámetros; parametrización: deben usarse parámetros con las mejores propiedades de estimación; rango de aplicabilidad: el modelo debe poder ajustar todos los datos observados; especificación estocástica: el error también debe ser modelado; interpretabilidad: se prefieren parámetros con interpretación física [15]. Zwietering y col., hicieron énfasis en la importancia de la parsimonia e interpretabilidad de los parámetros [22].
El Lactococcus lactis subsp. lactis es una bacteria ácido láctica, capaz de producir nisina, una bacteriocina ampliamente usada en la industria alimentaria como preservativo biológico [16,17,19]. Diferentes trabajos han sido publicados reportando la eficacia de este microorganismo en inhibir el crecimiento de bacterias patógenas, entre ellas Listeria monocytogenes y Staphylococcus aureus [16,17], razón por la cual puede ser empleado como biopreservativo en la industria láctea; pero para ello, previamente es necesario estudiar su crecimiento en los productos lácteos.
El objetivo del presente estudio fue comparar la aplicabilidad de varios modelos predictivos sigmoidales al crecimiento del Lactococcus lactis subsp. lactis en leche estéril de vaca y búfala.
MATERIALES Y MÉTODOS
Durante un período de tres meses, se recolectaron un total de 12 muestras de leche (250 mL c/u, 6 por especie), del total del ordeño matutino de un rebaño de búfalos (Bubalus bubalis) y vacunos (Bos taurus-indicus) establecidos en la hacienda Cordero, ubicada en la parroquia Luis Vicente, municipio Mara del estado Zulia, Venezuela, a las márgenes del río Limón.
Las muestras tomadas asépticamente, fueron envasadas en recipientes de vidrio estériles y colocadas en refrigeración, para luego ser transportadas hacia el laboratorio de Ciencia y Tecnología de la Leche de la Facultad de Ciencias Veterinarias de la Universidad del Zulia, para su análisis y ejecución del estudio. A las muestras se les determinaron algunos parámetros, tales como el pH a 25°C, la acidez titulable y la presencia de inhibidores.
La determinación del pH se hizo empleando un potenciómetro marca Orión modelo 420-A (Orion Research INC, EUA), el cual posee un electrodo marca Orión modelo 91-57B, calibrado con dos soluciones buffer, una de pH 7 ± 0,1 y otra de pH 4 ± 0,1 a 25°C, distribuidos por la misma casa comercial.
La acidez titulable se realizó mediante el procedimiento descrito en la norma COVENIN 658-86 [3], expresando los resultados en mL de NaOH 0,1N/100 mL de leche.
La detección de inhibidores se realizó con el fin de asegurar que las muestras estuviesen libres de sustancias que pudiesen afectar el crecimiento del microorganismo en estudio. Se llevo a cabo mediante la técnica del DELVOTEST SP® (DSM Food Specialties, Delft, Holanda), el cual contiene varias ampollas con un medio sólido, donde se encuentran células del Bacillus stearothermophilus var. calidolactis, bacteria muy sensible a cantidades trazas de inhibidores en la leche como penicilina, tetraciclinas, sulfamidas, etc.
Posterior a los análisis previos, se procedió a esterilizar aproximadamente 100 mL de leche de ambas especies, en autoclave a 110 ± 2°C por 10 min, parámetros bajo los cuales se evita el pardeamiento no enzimático, a la vez que se asegura una eficiente reducción de la carga bacteriana.
De las muestras esterilizadas se preparó una batería de 12 tubos de ensayo, conteniendo 9 mL de leche estéril, los cuales se inocularon con 1 mL de un cultivo conteniendo aproximadamente 1 x 104 ufc/mL de Lactococcus lactis subsp. lactis (ATCC 11454), mantenido en leche descremada. Con ello se esperaba que por efecto de dilución, la población microbiana inicial en cada tubo estuviese en aproximadamente 1 x 103 ufc/mL. Las muestras se incubaron a 36 ± 0,5°C en un baño maría y cada dos horas (de 0 a 10 horas de incubación) se retiraba un tubo de muestra de cada especie, para proceder a realizar diluciones seriadas y siembra en profundidad, en placas con agar M-17 (Himedia® Laboratories Limited; Bombai, India), las cuales fueron incubadas en estufa a 37 ± 1°C por 24 horas. Periodo después del cual se hizo el conteo de colonias, según la norma COVENIN 902-87 [4] y determinó el crecimiento bacteriano en unidades formadoras de colonias por mililitro (ufc/mL).
Los datos en ufc/mL fueron convertidos a logaritmo base 10 y modelados con las ecuaciones presentadas en la TABLA I; aplicando un análisis de regresión no lineal, usando para ello el módulo NLIN del paquete estadístico SAS 8,1 [18]. El método para la estimación de los parámetros (método Marquardt) y el criterio de convergencia utilizado fue predeterminado por el programa estadístico señalado.
| TABLA I. MODELOS PREDICTIVOS DE CRECIMIENTO UTILIZADOS EN EL ESTUDIO/ PREDICTIVE MODELS OF GROWTH USED IN STUDY | ||
| Modelo | Expresión | Nomenclatura en el texto |
| Gompertz | Log N(t)= A × exp (-exp (B-M × t)) | Gompertz-1 |
| Log N(t)= A+D × exp(-exp(-B × (t-M)) | Gompertz-2 | |
| Logístico | Log N(t)= |
Logístico-1 |
| Log N(t)= |
Logístico-2 | |
| Stannard | Log N(t)= |
Stannard |
| Richards | Log N(t) =A(1 + v × exp ( k ( t - t))(–1/v) | Richards |
| LogN: logaritmo común de poblaciones bacterianas (UFC/mL). t: tiempo de incubación (horas). A: logaritmo común de la población inicial en UFC/mL (inoculo). D: logaritmo común de la diferencia entre la población inicial y final en la fase estacionaria. B: representa la pendiente de la curva y describe la tasa de crecimiento. M: tiempo en el cual la tasa de crecimiento es de mayor magnitud. k; p; t; v: parámetros de ajustes (sin significancia biológica). | ||
La comparación de los diferentes modelos ajustados, se realizó tomando en cuenta la facilidad de los mismos para alcanzar el ajuste, determinada por el número de iteraciones y el tiempo necesario para alcanzar el criterio de convergencia [9, 14]. Los valores para los coeficientes de determinación (R2), se emplearon para comparar entre los modelos con igual número de parámetros.
Para comparar entre los modelos de tres parámetros y cuatro parámetros se utilizó la prueba de falta de ajuste (lack to fit), asumiendo como modelo de referencia aquel que presentó un menor error, medido por la suma de cuadrado de las residuales (SCR), y que en el estudio le correspondió a los Modelos de Richard y Stannard. En esta prueba, la diferencia entre las SCR del modelo de tres y cuatro parámetros se compara con la SCR del modelo de cuatro parámetros [11, 22]. Esta relación es calculada mediante la siguiente expresión:
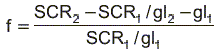
donde:
SCR2 = suma de cuadrados del modelo de tres parámetros
SCR1 = suma de cuadrados del modelo de cuatro parámetros (referencia)
gl2 = grados de libertad del modelo de tres parámetros
gl1 = grados de libertad del modelo de cuatro parámetros (referencia) y probada frente al valor tabulado de Fgl1, gl2-gl1.
Esta prueba no es de completa validez estadística cuando se aplica a modelos sigmoidales, ya que su normal aplicación es para modelos lineales, pero ha sido utilizada por otros autores y permite tener una idea de la conveniencia en la aplicación de los modelos [11, 22].
RESULTADOS Y DISCUSIÓN
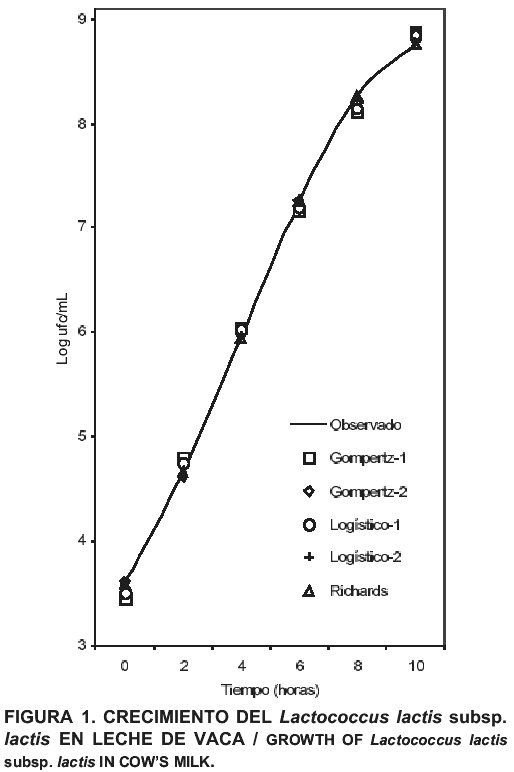
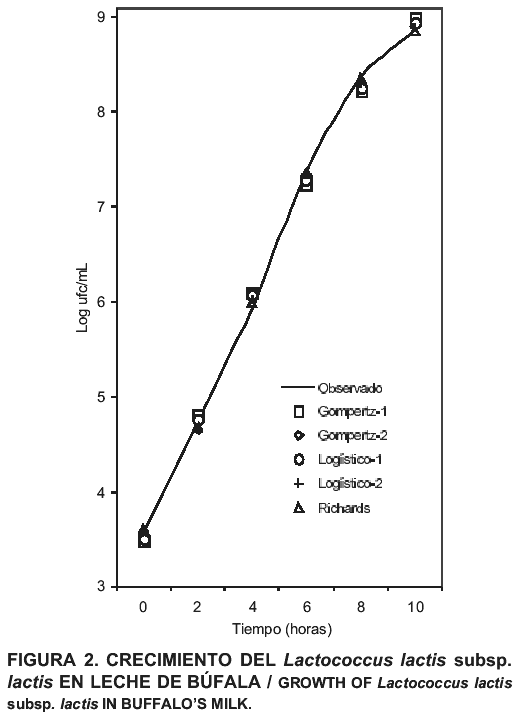
Las TABLA II y III, muestran los valores correspondientes de los diferentes parámetros estimados para cada uno de los modelos aplicados al crecimiento del L. lactis subsp. lactis en leche de vaca y búfala, respectivamente. Con los mismos se pueden construir las diferentes ecuaciones de predicción. En dichas TABLAS se puede observar que en algunos modelos, los valores para ciertos parámetros coinciden, lo cual da una idea de la similitud en el ajuste entre ellos.
TABLA II. PARÁMETROS ESTIMADOS POR LOS DIFERENTES MODELOS APLICADOS AL CRECIMIENTO DE Lactococcus lactis subsp. lactis EN LECHE DE VACA / PARAMETERS ESTIMATED BY DIFFERENT MODELS APPLIED TO THE GROWTH OF Lactococcus lactis subsp. lactis IN COWS MILK.
| Modelo | Parámetros | |||||||
| A | B | D | M | p | k | v | t | |
| Gompertz-1 | 11,4015 | 0,1673 | - | 0,1555 | - | - | - | - |
| Gompertz-2 | 3,1926 | 0,2963 | 6,4535 | 3,3829 | - | - | - | - |
| Logístico-1 | 10,1837 | 0,6407 | - | 0,2533 | - | - | - | - |
| Logístico-2 | 2,3701 | 0,3988 | 6,9584 | 3,8239 | - | - | - | - |
| Stannard | 9,0455 | -0,9170 | - | - | 0,2226 | 0,1335 | - | - |
| Richards | 9,0455 | - | - | - | - | 0,5998 | 4,4915 | 4,3629 |
TABLA III. PARÁMETROS ESTIMADOS POR LOS DIFERENTES MODELOS APLICADOS AL CRECIMIENTO DE Lactococcus lactis subsp. lactis EN LECHE DE BÚFALA / PARAMETERS ESTIMATED BY DIFFERENT MODELS APPLIED TO THE GROWTH OF Lactococcus lactis subsp. lactis IN BUFFALOS MILK
| Modelo | Parámetros | |||||||
| A | B | D | M | p | k | v | t | |
| Gompertz-1 | 11,4382 | 0,1740 | - | 0,1596 | - | - | - | - |
| Gompertz-2 | 3,0634 | 0,2848 | 6,7836 | 3,2946 | - | - | - | - |
| Logístico-1 | 10,2523 | 0,6567 | - | 0,2587 | - | - | - | - |
| Logístico-2 | 2,2350 | 0,3899 | 7,2408 | 3,7547 | - | - | - | - |
| Stannard | 9,1214 | -0,9274 | - | - | 0,2178 | 0,1353 | - | - |
| Richards | 9,1215 | - | - | - | - | 0,6214 | 4,5923 | 4,4007 |
En las TABLAS IV y V se presentan los SCR, cuadrado medio de las residuales (CMR) y los R2 de los diferentes modelos aplicados al crecimiento del Lactococcus en leche de vaca y búfala, respectivamente. Se observa que los R2 están por encima de 0,99 lo cual significa que todos los modelos ajustaron bien a los datos experimentales, tanto para leche de vaca como para leche de búfala. La similitud en el ajuste de los diferentes modelos puede ser apreciado por los valores obtenidos en el CMR y la SCR.
| TABLA IV. DIFICULTAD PARA EL AJUSTE DE LOS DIFERENTES MODELOS APLICADOS AL CRECIMIENTO DE Lactococcus lactis subsp. lactis EN LECHE DE VACA / DIFFICULTY FOR THE FIT OF DIFFERENT MODELS APPLIED TO THE GROWTH OF Lactococcus lactis subsp. lactis IN COWS MILK. | |||||
| Modelo | Tiempo | Nº Iteraciones | SCR | CMR | R2 |
| Gompertz-1 | 0,12 | 18 | 3,9368 | 0,1192 | 0,9975 |
| Gompertz-2 | 0,10 | 8 | 3,4947 | 0,1092 | 0,9978 |
| Logístico-1 | 0,09 | 8 | 3,7116 | 0,1125 | 0,9977 |
| Logístico-2 | 0,07 | 7 | 3,4574 | 0,1080 | 0,9978 |
| Stannard | 0,12 | 25 | 3,4377 | 0,1074 | 0,9978 |
| Richards | 0,12 | 15 | 3,4377 | 0,1074 | 0,9978 |
| SCR: suma de cuadrado de residuales; CMR: cuadrado medio de residuales; R2:coeficientes de determinación. | |||||
TABLA V. DIFICULTAD PARA EL AJUSTE DE LOS DIFERENTES MODELOS APLICADOS AL CRECIMIENTO DE Lactococcus lactis subsp. lactis EN LECHE DE BÚFALA / DIFFICULTY FOR THE FIT OF DIFFERENT MODELS APPLIED TO THE GROWTH OF Lactococcus lactis subsp. lactis IN BUFFALO´S MILK.
| Modelo | Tiempo | Nº Iteraciones | SCR | CMR | R2 |
| Gompertz-1 | 0,20 | 18 | 4,2812 | 0,1297 | 0,9974 |
| Gompertz-2 | 0,18 | 8 | 3,9169 | 0,1224 | 0,9976 |
| Logístico-1 | 0,14 | 9 | 4,0561 | 0,1229 | 0,9976 |
| Logístico-2 | 0,10 | 8 | 3,8438 | 0,1201 | 0,9977 |
| Stannard | 0,17 | 38 | 3,7772 | 0,1180 | 0,9977 |
| Richards | 0,17 | 17 | 3,7772 | 0,1180 | 0,9977 |
| SCR: suma de cuadrado de residuales; CMR: cuadrado medio de residuales; R2:coeficientes de determinación. | |||||
Las TABLAS IV y V también presentan los valores para el tiempo y número de iteraciones necesarias para alcanzar el criterio de convergencia, lo cual es utilizado para la comparación entre los modelos [9]. Se puede observar que el tiempo tomado por los diferentes modelos para encontrar el ajuste no es significativo (menos de 1 segundo), lo cual se debe a la poca cantidad de datos experimentales obtenidos (36 para cada tipo de leche).
Los modelos Stannard y Richards fueron los que tomaron mayor tiempo y además presentaron un mayor número de iteraciones, comparado con los demás, siendo el de Stannard el que presentó mayor número de iteraciones. La dificultad mayor se observó al ajustar los datos para leche de búfala. Lo anterior sugiere que estos modelos, a pesar de tener igual número de parámetros que el de Gompertz-2 y Logístico-2, presentan mayor dificultad en el ajuste, lo cual contradice uno de los principios antes expuestos y que se requiere deban cumplir los modelos predictivos, como lo es la parsimonia. Estos además, presentan parámetros de ajuste que no tienen significado biológico y por lo tanto, ninguna interpretación.
El modelo que según el tiempo y el número de iteraciones resultó con mayor facilidad, fue el modelo Logístico-2, tanto en leche de vaca como en la de búfala. Esta observación concuerda con lo reportado por López y col., quien al comparar los modelos de Richards, Brody y Von Bertalanffy, encontró que el primero fue el que presentó mayor dificultad en alcanzar el ajuste de los datos experimentales [9].
La comparación de los modelos por medio de los valores del R2 solo es válido entre aquellos con igual número de parámetros, ya que a medida que el número de éstos aumentan, el R2 se acerca a la unidad por disminución del error [22]. Tomando en cuenta esto, al comparar los modelos de tres parámetros, Gompertz-1 y Logístico-1 entre ellos, y los de cuatro parámetros Gompertz-2, Logístico-2, Stannard y Richard, por simple observación de los R2, se concluye que no hay diferencia en el ajuste realizado por los mismos.
La prueba de falta de ajuste utilizada para comparar los modelos con diferentes números de parámetros mostró diferencia significativa (P<0,05) entre el modelo Richards (tomado como referencia) y el modelo Gompertz-1. Mientras que en la comparación con el modelo Logístico-1, no se encontró diferencia significativa, aunque el error resultó mayor en este modelo que en los de cuatro parámetros. Esta diferencia fue encontrada para ambos tipos de leche evaluadas.
Las FIGS. 1 y 2 presentan las curvas obtenidas con los datos observados y ajustados por los diferentes modelos para leche de vaca y búfala, respectivamente. En ellas se pueden apreciar también la similitud en el ajuste de los diferentes modelos a los datos observados experimentalmente, además de la similitud del ajuste entre los diferentes modelos. Debido a que los modelos Stannard y Richards resultaron ser iguales en el ajuste, por lo cual los datos estimados son idénticos, se omitió la curva para el modelo de Stannard.
Zwietering y col. [22], compararon los modelos de Gompertz (tres parámetros), Logístico (tres parámetros), Stannard y Richards, encontrando que todos los modelos ajustaron bien a la data experimental, aunque concluyeron que el modelo de Gompertz fue suficiente para describir la curva de crecimiento bacteriano, debido en parte a su facilidad de ajuste. Este resultado contrasta con el obtenido en el presente estudio, donde el modelo que presentó mayor error fue el de Gompertz de tres parámetros.
Quirino y col. [14], utilizaron cuatro modelos sigmoidales (Brody, Logístico, Gompertz y Richards) para caracterizar la circunferencia escrotal en toros Nellore en crecimiento, utilizando la metodología de López para comparar los diferentes modelos, encontrando mayor error, menor R2 y mayor dificultad (numero de iteraciones) para el modelo de Richard y el mejor modelo en este estudio resultó el modelo Logístico.
Por su parte Cayré y col. [2], compararon el modelo de Gompertz y Logístico de cuatro parámetros ambos, en el crecimiento de bacterias lácticas en emulsiones cárnicas para predecir vida útil, concluyendo en que no existen diferencias entre los modelos ajustados.
CONCLUSIONES
Todos los modelos presentaron un ajuste aceptable a los datos experimentales obtenidos, tanto para leche de vaca como para leche de búfala.
No se observaron diferencias entre los modelos con igual número de parámetros.
El modelo de Gompertz de tres parámetros fue de todos, el que presentó mayor error en el ajuste.
Los modelos de Stannard y Richards fueron los que presentaron mayor dificultad para alcanzar el criterio de convergencia.
Se considera que los modelos Logístico (Logístico-2) y de Gompertz (Gompertz-2) modificados por Gibson, son los mejores para modelar el crecimiento del Lactococcus lactis subsp. lactis en leche estéril.
RECOMENDACIONES
Se recomienda hacer la comparación de los modelos con una mayor cantidad de datos y bajo diferentes condiciones de crecimiento, a fin de determinar como se comportan bajo diferentes tratamientos.
Es ideal también realizar estas comparaciones con otros microorganismos para determinar si se encuentran los mismos resultados.
Al momento de realizar estudios de esta naturaleza, se requiere de la integración de profesionales de las diferentes áreas involucradas (ingeniería, estadística, microbiología) en la microbiología predictiva, para facilitar la interpretación y discusión de los resultados obtenidos.
AGRADECIMIENTO
Los autores agradecen a la División de Investigación de la Facultad de Ciencias Veterinarias de la Universidad del Zulia, por haber colaborado con el financiamiento necesario para la realización del estudio.
REFERENCIAS BIBLIOGRÁFICAS
1. BIRCH, C. A New logistic sigmoid growth equation compared with the Richards growth equation. Ann Bot-London. 83: 713-723. 1999. [ Links ]
2. CAYRE, M.; VIGNOLO, G.; GARRO, O. Validación y comparación de modelos de crecimiento microbiano. Universidad Nacional Del Nordeste, Comunicaciones Científicas y Tecnológicas. Argentina. 4 pp. 2001. [ Links ]
3. COMISIÓN VENEZOLANA DE NORMAS INDUSTRIALES (COVENIN) Leche fluida. Determinación de acidez titulable. 3a Rev. No 658-1997. 5 pp.1997. [ Links ]
4. COMISIÓN VENEZOLANA DE NORMAS INDUSTRIALES (COVENIN). Alimentos. Método para recuento de colonias de bacterias aerobias en placas de Petri. No 902-87. 6 pp. 1987. [ Links ]
5. GARCÍA, R.; HERVÁS, C.; RODRÍGUEZ, R.; ZURERA, G. Modelling the growth of Leuconostoc mesenteroides by artificial neural networks. Int J Food Microbiol. 105(3): 317-332. 2005. [ Links ]
6. GEERAERD, A.; HERREMANS, C.; HERREMANS, M.; CENES, C.; VAN IMPE, J. Application of artificial previous neural networks as a non linear technique to describe bacterial growth in chilled food products. Int J Food Microbiol. 44: 49-68. 1998. [ Links ]
7. HAJMEER, M.; BASHEER, I. Comparison of logistic regression and neural network-based classifiers for bacterial growth. Food Microbiol. 20(1): 43-55. 2003. [ Links ]
8. LATRILLE, E.; CORRIEU, G.; THIBAULT, J. Neural network models for final process time determination in fermented milk production. Comp Chem. 18(11-12): 1171-1181. 1994. [ Links ]
9. LÓPEZ DE T., G.; CANDOTTI, J. ; REVERTER, A.; BELLIDO, M.; VASCO, P.; GARCÍA, L.; BRINKS, J. Effects of growth curve parameters on cow efficiency. J Anim Sci. 70: 2668-2672. 1992. [ Links ]
10. McKELLAR, R.; LU, X. Primary Models. In: McKellar, R., Lu, X. (Eds.). Modeling microbial responses in foods. CRC Press, Boca Raton, USA, 21-62 pp. 2004. [ Links ]
11. MCMEEKIN, T.; OLLEY, J.; ROSS, T.; RATKOWSKY, D. Basic concepts and methods. In: Predictive Microbiology: theory and application. Research Studies Press LTD, Taunton, Englan. 11-84 pp. 1993. [ Links ]
12. MCMEEKIN, T.; ROSS, T. Modeling applications. J Food Protect. 59(Suppl): 37-42. 1996. [ Links ]
13. PANAGOU, E.; KODOGIANNIS, V.; NYCHAS, G. Modelling fungal growth using radial basis function neural networks: The case of the ascomycetous fungus Monascus ruber van Tieghem. Int J Food Microbiol. 117(3): 276-286. 2007. [ Links ]
14. QUIRINO, C.; BERGMANN, J.; VALE, V.; ANDRADE, V.; PEREIRA, J. Evaluation of four mathematical functions to describe scrotal circumference maduration in Nellore bulls. Theriogenol. 52(1): 25-34. 1999. [ Links ]
15. SCHEPERS, A.; THIBAULTB, J.; LACROIXA, C. Comparison of simple neural networks and nonlinear regression models for descriptive modeling of Lactobacillus helveticus growth in pH-controlled batch cultures. Enz Microbiol Tech. 26: 431-445. 2000. [ Links ]
16. SCHILLINGER, U.; GEISEN, R.; HOLZAPFEL, W.H. Potential of antagonistic microorganisms and bacteriocins for the biological preservation of food. Trends Food Sci Tech. 7: 158-164. 1997. [ Links ]
17. SPELHAUG, S.; HARLANDER, S. Inhibition of foodborne bacterial pathogens by bacteriocins from Lactococcus lactis y Pedococcus pentosaceous. J Food Protect. 52(12): 856-862. 1989. [ Links ]
18. STATISTICAL ANALYSIS SYSTEMS INSTITUTE (SAS). Users guide statistic. SAS Institute, Cary, North Carolina. 646 pp. Version 8,1. 2000. [ Links ]
19. VALBUENA, E.; BARREIRO, J.; SÁNCHEZ, E.; CASTRO, G.; BRÍÑEZ, W.; TOVAR, A. Modelos cinéticos aplicados al crecimiento de Lactococcus lactis subsp. lactis en leche. Rev Científ FCV-LUZ. XV(5): 464-475. 2005. [ Links ]
20. VALERO, A.; HERVÁS, C.; GARCÍA-GIMENO, R.; ZURERA, G. Product unit neural network models for predicting the growth limits of Listeria monocytogenes. Food Microbiol. 24(5): 452-464. 2007. [ Links ]
21. WHITING, R.; BUCHANAN, R. Microbial modeling: Scientific status summary. Food Technol. 48(6): 113-120. 1994. [ Links ]
22. ZWIETERING, M.; JONGENBURGER, I.; ROMBOUTS, F.; VANT RIET, K. Modeling of the bacterial growth curve. Appl Environ Microbiol. 56(6): 1875-1881. 1990. [ Links ]












 uBio
uBio