Revista Científica
versión impresa ISSN 0798-2259
Rev. Cient. (Maracaibo) v.19 n.2 Maracaibo mar. 2009
IDENTIFICACIÓN DE ESPECIES EN PRODUCTOS DE ORIGEN ANIMAL MEDIANTE PCR.
José Aranguren-Méndez1, María Portillo1, Jorge Ruiz1, Yenen Villasmil-Ontiveros1, Luis Yáñez1, Lisbeth Borjas2
y William Zabala2
1Facultad de Ciencias Veterinarias. 2Laboratorio de Genética Molecular. Universidad del Zulia, Apdo. 15252. Maracaibo, Venezuela. E-mail: atilioaranguren@gmail.com
RESUMEN
La identificación de la especie en productos de origen animal (carne, leche o sus derivados) se hace necesaria y de exigencia por los consumidores modernos, entre otras razones: i) para evitar fraude económico, ya sea por sustitución o adulteración del mismo, ii) por motivos de salud humana, tales como alergias alimentarías, iii) por implicaciones culturales; de allí que se debe contar con herramientas analíticas y sensibles para dicha identificación tales como el análisis de fragmentos de ADN en especial de origen mitocondrial (gen 12S ARNr) dada su particularidad de ser especifica de especies. A tal fin se estableció una metodología de identificación mediante la amplificación de fragmentos específicos de ADN mitocondrial (ADNm) a partir de muestras biológicas de las principales especies animales implicadas en la producción de carnes o alimentos (bovina, porcina, ovina, caprina, equina, asnos, felina y canina), específicamente de una fracción parcial del gen 12s ARNr de una región conservada usando unos cebadores comunes para dichas especies, un reverse especifico de especie y análisis posterior mediante geles de agarosa al 1,5% y amplificación de fragmentos que oscilaron entre 150 y 364 pb. Los resultados indican que se puede identificar la especie a la que pertenece la muestra analizada en el 100% de los casos, ofreciendo una herramienta especifica para determinar la especie en alimentos de origen animal.
Palabras clave: Especie, PCR, mitocondrial, ADN.
PCR-Species-Identification in Animal Products.
ABSTRACT
Nowadays identification of animal-origin products (meat, milk and dairy products) is of paramount importance to costumers and specially as for species identification for a number of reasons: i) to avoid economic fraud for substitution or alteration of the product ; ii) to avoid health issues such as food allergies; iii) for culturals reasons. There for the value of analytic and sensitive identification tools such as DNA fragments analysis, especially those of mitochondrial origin (12S rRNA gene) because of its species-specific character. An identification methodology through amplification of a species-conserved region of 12S rRNA gene (forward primer) and of a species-specific region of the same gene (reverse primer). Template DNA was extracted from biological samples of bovine, swine, ovine, caprine, equine and canine origin. After 1.5% agarose gel electrophoresis, fragments ranging from 150 to 364 bp were observed. Results show that species could be easily identified through PCR in all cases and that this methodology could be a specific tool for determining the origin of animal products.
Key word: Species, PCR, mitochondrial, DNA.
Recibido: 09 / 03 / 2008. Aceptado: 29 / 05 / 2008.
INTRODUCCIÓN
La calidad de los productos de origen animal, incluyendo su autenticidad es importante y una exigencia que cada día es más solicitada por parte de los consumidores modernos, los cuales ejercen presión sobre los entes gubernamentales a fin de que se le garantice la calidad de los mismos [17, 19].
La adulteración de materias primas como la carne o la leche de una especie por otra, sucede comúnmente, sobre todo cuando existen marcadas diferencias de precio o disponibilidad de los productos, sean de origen nacional o de importación y en especial, cuando se vende como una especie por otra de menor valor comercial [16].
La identificación clara de la especie de la cual provienen los alimentos es muy importante y existen al menos tres razones para su justificación:
i) Evitar el fraude económico por sustitución o adulteración del producto (ejemplo carne de perro (Canis familiaris) o gato (Felis catus) por carne de bovino (Bos taurus-indicus), en especial en alimentos ofrecidos en expendios sin el control sanitario correspondiente; en otros casos es el uso de leche bovina en la producción de quesos cuya fórmula original requiere leche de otras especie bien sea de búfala (Bubalus bubalus) (mozzarella) o cabra (Capra hircus) u oveja (Ovis aries).
ii) Requerimientos especiales, relacionados con aspectos de salud o de orden sanitarista, tales como problemas de alergias alimentarías en individuos susceptibles o que reaccionan ante ciertas proteínas de origen animal; así como aquellos individuos que no toleran la ingesta de ciertos componentes de la leche, tales como la lactosa (caso de infantes).
iii) Por razones culturales, tal es el caso del no consumo por parte de los musulmanes de productos de origen porcino [19].
Para identificar la especie de la que proceden determinados productos de origen animal, tradicionalmente se han venido estudiando proteínas, ya fuese mediante la electroforesis de proteínas solubles [1, 6], la separación mediante HPLC [3, 16], y/o métodos inmunoenzimáticos [2, 5, 7, 8]. Sin embargo, con los tratamientos culinarios de rutina empleados en la preparación de los alimentos, las proteínas se desnaturalizan, lo que no ocurre con la molécula de ADN, dado que es más estable a tales tratamientos (calor, salado, ahumado, etc.), ya que no es degradada en la cadena de producción de los alimentos y más termoestable que las proteínas [11].
En la actualidad, estas técnicas convencionales han sido desplazadas por el uso de metodologías mediante el estudio de ADN, la cual permite determinar de manera más exacta el origen y la identificación de todos los productos que llegan al consumidor aunque ya estén procesados. Debido a que dicha información genética se encuentra en todas las células nucleadas de cualquier tejido o fluido del animal (sangre, pelo o fibra, músculo, leche, entre otros) y lo más resaltante es que, la información del ADN permanece invariable a lo largo de la vida del animal y tras su sacrificio [10, 14, 15, 19].
El ADN mitocondrial ha sido utilizado ampliamente como un marcador de especies, en particular la región 12s ARNr [9, 19], dado que cumple una serie de requisitos para identificación. Esta región posee una longitud aceptable, suficiente diferenciación entre especie congenéricas o próximas, además que acumula puntos de mutación interesantes que originan cambios puntuales capaces de diferenciar géneros o especies, ideal para este tipo de estudios [12, 13, 18, 21].
Debido a la importancia del tema, el presente trabajo tuvo como objetivo desarrollar un protocolo de laboratorio que permita la identificación de la especie animal en diferentes muestras biológicas.
MATERIALES Y MÉTODOS
Muestras y extracción de ADN
El ADNmt se extrajo a partir de glóbulos blancos (leucocitos) de muestras sanguíneas obtenidas de cada una de las especies de interés y siguiendo para ello la metodología de Ausubel y col. [4]. El ADN de tejido fue aislado por extracción de una mezcla con proteinasa K-SDS [20].
La estandarización del protocolo para determinar con un cien por ciento de exactitud el origen de la muestra, se realizó a partir de muestras sericas y de tejido muscular de: bovino (Bos taurus-indicus), ovino (Ovis aries), caprino (Capra hircus), equino (Equus caballus), cerdo (Sus scrofa), gato (Felis catus), asno (Equus asinus) y perro (Canis familiaris), a las cuales se les diseñaron unos cebadores específicos para la especie de una región parcial del gen 12S (ARNr) perteneciente al ADN mitocondrial (ADNmt).
Diseño de los cebadores especie-específico
Se obtuvieron las secuencias de los genes (12S ARNr) de bovino, ovino, caprino, equino, cerdo, asno, felino y canino del GenBank (http://www.ncbi.nlm.nih.gov/genbank), para ello se diseñaron 2 cebadores forward universales, uno común para las especie bovina, ovina, caprina, cerdos, felino y canino y otro para las especies equinas (caballos y asnos) y fueron diseñados oligonucleótidos reversos específicos para cada una de las especies estudiadas, que permitían amplificar fragmentos de diferentes tamaños de acuerdo a la especie de interés (TABLA I).
Amplificación de ADN
Para la amplificación del fragmento de ADNmt se preparó una reacción final de 15 microlitros (µL), que contenía entre 150 a 200 ng de ADN, 200 µM de desoxirribonucleótidos (dNTP), 1,5 mM de MgCl2 y 0,5 U de ADN polimerasa, 5 pmol de cada oligo o cebador (TABLA I).
La PCR (Reacción en cadena de la polimerasa) fue llevada a cabo en un termociclador Eppendorf Mastercycler epgradient S®, para ello se diseñó un programa térmico, comportando una primera etapa a 94°C durante 1 min., seguido de 25 ciclos que comprendían, una primera fase de desnaturalización a 94°C durante 1 min, una fase de hibridación a 54°C durante 1 min y una fase de extensión de la cadena a 72°C durante 1 min. El programa térmico finalizó con una extensión a 72°C durante 5 min.
Los productos de la PCR fueron posteriormente detectados mediante geles electroforéticos de agarosa al 1,5%.
RESULTADOS Y DISCUSIÓN
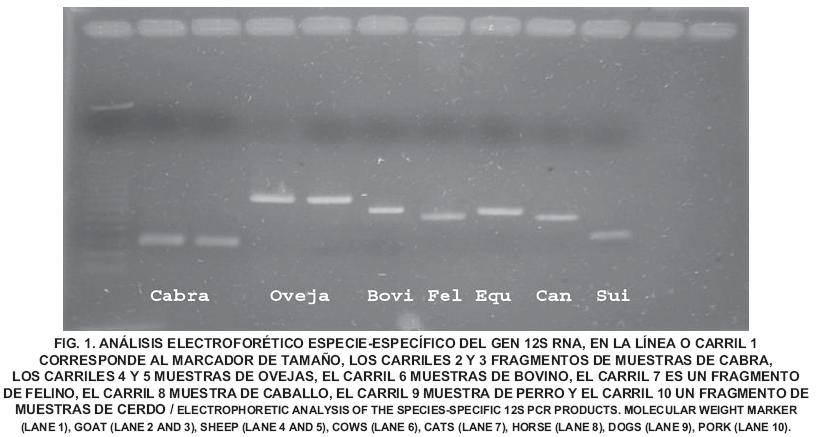
Los patrones de los productos amplificados por la PCR obtenidos de cada una de las especies, correspondieron exactamente a los tamaños esperados (FIG. 1). De las amplificaciones se obtuvieron fragmentos que oscilaron entre 150 y 364 pares de bases (pb), siendo dichos fragmentos específicos de 150 pb para las muestras de origen caprino, un fragmento de 160 pb para muestras de cerdos, fragmento de 221 pb perteneciente a muestras de bovino, fragmento de 260 pb correspondiente a muestras de perro, un fragmento de 264 pb para muestras de gato, fragmentos de 292 pb para los caballos y asnos, y un fragmento de 364 pb a muestras de ovino,
Cabe destacar, que cada juego de oligos o cebadores fueron construidos para que amplificaran única y exclusivamente la especie de interés, es decir que en caso de mezclas entre productos, se debería amplificar las muestras, ya sea de forma individual para las especies sospechosa o bien realizar una mezcla en una sola corrida de PCR de dos o más juegos de primers, en cuyo caso se podría apreciar dos o más bandas en el producto amplificado.
Reportes previos [12, 18, 21] ya indicaban la ventaja de trabajar con mtDNA y en especial con el gen 12S en vertebrados, dado que el material biológico es bastante estable, y se requiere tan sólo de una pequeña cantidad de ADN molde, además que puede detectarse el origen del producto a partir de muestras frescas o cocidas y además con el ADNmt, la integridad que se presenta es de mayor grado en el, que en el ADN nuclear.
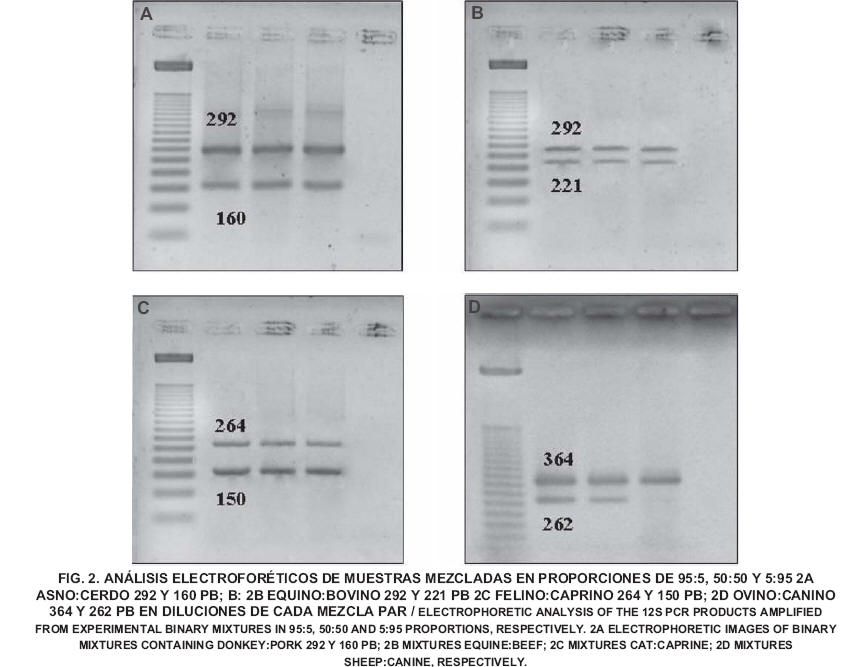
Con el objeto de validar el método bajo ensayo, se realizaron además mezclas de ADN de las distintas especies estudiadas por parejas, tratando de buscar la mejor combinación que permitiera el análisis simultáneo de más de un tejido o mezclas de las mismas. En la FIG. 2, se puede apreciar varias combinaciones de muestras que lograron amplificarse en parejas y en diferentes proporciones que oscilaron entre 95:5; 50:50 y 5:95% para cada par de muestras, respectivamente. Cabe destacar, que de las distintas combinaciones realizadas, aquellas que presentaron o mostraron los mejores comportamientos, es decir que ambos fragmentos fueron fácilmente detectados y apreciados bajo la luz ultravioleta resultaron ser las de asno:cerdo, equino:bovino, gato:caprino y ovino:perro. Las demás combinaciones, o bien no amplificaron claramente u obligan a realizar análisis independientes para cada especie, ya que no se logró de forma conjunta diferenciar las especies amplificadas con facilidad.
Dichas combinaciones de productos de ADN arrojaron los fragmentos esperados y que de forma individual ya habían sido comprobados; de esta manera se obtuvieron fragmentos amplificados de 292 y 160; 292 y 221; 264 y 150 y 364 y 262 pares de bases, respectivamente, de las especies citadas previamente. Uno de los fraudes que se presenta es la mezcla de productos de alto valor con otros de menor precio comercial; no obstante, se puede indicar que con mezclas de hasta 5% (mínima probada) se podría a través de este protocolo detectar en las muestras y diagnosticar con 100% de exactitud, la o las especies involucradas en mezclas de los ADN extraidos. Resultados similares de la especificidad fueron obtenidos por otros autores en estudios comparativos a estos [13, 19].
CONCLUSIONES
Dada la necesidad de proteger, tanto a los consumidores como a los productores, al garantizar el origen de los productos de origen animal, se hace necesario contar con herramientas sensibles, exactas como la PCR. Todas las especies estudiadas pudieron ser confirmadas en el laboratorio mediante la PCR, lo que indica que con la ayuda del ADNmt se puede identificar claramente la especie a la que pertenece el producto con un 100% de certeza, al igual que en caso de mezclas entre productos de varias especies.
Así mismo, se puede indicar que, mediante esta técnica se dispone de una metodología útil para programas de inspección sanitaria e identificación animal.
AGRADECIMIENTO
Los autores desean agradecer al Consejo de Desarrollo Científico y Humanístico de La Universidad del Zulia (CONDES) por el financiamiento de la presente investigación (CC-0928-06) y a la Policlínica Veterinaria de la Universidad del Zulia, quien gentilmente cedió algunas de las muestras analizadas.
REFERENCIAS BIBLIOGRÁFICAS
1.Addeo, F.; MOJO, L.; Chianese, L.; Nota, G. Evaluation of bovine and water buffalo milk in mixteures of liquid milk and mozzarella cheese by gel isoelectric focusing. Ital. J. Food Sci. 1: 71-80. 1989. [ Links ]
2.Addeo, F.; Nicloai, M.A.; CHIANESE, L.; Mojo, L.; Spagna-Musso, S.; Bocca, A.; Del Giovine, L. A control method to detect bovine milk in ewe and water buffalo chesse using immunoblotting. Milchwissenschaft. 50: 83-85. 1995. [ Links ]
3.Ashoot, S.H.; Monte, W.C.; Stiles, P.G. Liquid chromatographic identification of meats. J. Assoc. Off. Anal. Chem. 71:397-403. 1988. [ Links ]
4.Ausubel, F.M.; Brent, R.; Kingston, R.; Moore, D.; Seidman, G.; Smith, J.; Struhl, K. Current protocols in molecular biology. Green Publishing Associates and Wiley-Interscience, New. York. 243 pp. 1987. [ Links ]
5.Berger, R.; Mageau, R.P.; Schwab, B.; Johnston, R.W. Detection of poultry in cooked and canned meat foods by enzymelinked immunosorbent assays. J. Assoc. Off. Anal. Chem. 71:406-409. 1988. [ Links ]
6.Cartoni, G.P.; Coccioli, F.; Jasionowka, R.; Masci, M. Determination of cow milk in buffalo milk and mozzarella cheese by capillary electrophoresis of the whey protein fractions. Ital. J. Food Sci. 2: 127-131. 1998. [ Links ]
7.Chen, F.C.; Hsieh, H. Detection of pork in heat-procesed meat products by monoclonal antibody-based ELISA. J. AOAC Int. 83:79-85. 2000. [ Links ]
8.Cordal de B., M; Petracca, A; Moro, A. Preprations of reactants for the detection of proteins from different animal species by immunodiffusion. Rev. Arg. Microbiol. 17:209-216. 1985. [ Links ]
9.Feligini, M.; Bonnizi, I.; CUBRIC, V.; Parma, P.; Greppi, G.; Enne G. Detection of Adulteration in Italian Mozzarella Cheese using Mitochondrial DNA Templates as biomarkers. Food Technol. Biotechmol. 43:91-95. 2005. [ Links ]
10.Feligini, M.; Alim, N.; BONIZZI, I,; Enne, G.; Aleandri, R. Detection of cow milk water buffalo cheese by SYBR green real-time PCR: sensitivy test on governing liquid samples. Pakistan J. of Nutr. 6 :94-98. 2007. [ Links ]
11.Hofmann, K. Proof of proper heating of meat and bone meals. Fleischwirtschaft. 10:1037-1039. 1996. [ Links ]
12.Hurng-Yi, W.; Mung-PEI, T.; Ming-Chung, T.; Sin-Che, L. Universal primers for amplification of the complete mitocondrial 12S rRNA gene in vertebretes. Zool. Stud. 39: 61-66. 2000. [ Links ]
13.Martín, I.; García, T; Fajardo, V; ROJAS, M; Hernández, P.E; González, I; Martín, R. Detection of cat, dog, and rat or mouse tissues in food and animal feed using species-specific polymerase chain reaction. J. Anim. Sci. 85:2734-2739. 2007 [ Links ]
14.Matsunaga, T.; Chikuni, K.; Tanabe, R. A quick and simple method for the identification of meat species and meat products by PCR assay. Meat Sci. 51:143-148. 1999. [ Links ]
15.Parejo, J.C.; Padilla, J.A.; Tabasco, A.; Sansinforiano, M; Martinez-Trancon, M. Populations structure in the endangered blanca cacereña bovine breed demonstrated by RAPD analyses. Gen. Genet. Syst. 77:51-58. 2002 [ Links ]
16.Pellegrino, L.; De Noni, I.; Tirelli, A.; Resmini, P. Detection of cow milk in non-bovine cheese by HPLC of whey proteins. Sci. Tecn. Latt. Cas. 42: 87-94. 1991. [ Links ]
17.Pfeiffer, I.; Burger, J.; Brenig, B. Diagnostic polymorphisms in the mitochondrial cytochrome b gene allow discrimination between cattle, sheep, goat, roe buck and deer by PCR.RFLP. BMC Genet.. 5: 30-36. 2004. [ Links ]
18.Rastogi, G.; Dharne, M.; Bharde, A.; Pandav, V.S.; Ghumatkar, S.V.; Krishnamurthy, R.; Patole, M.S; Shouce, Y. Species determination and authentification of meat samples by mitocondrial 12S rRNA gene sequence analysis and conformation-sensitive gel electrophoresis. Curr. Sci. 87: 1278-1280. 2004. [ Links ]
19.Rodriguez, M.; García, T.; González, I.; Asensio, L.; Hernández, P.; Martín, R. PCR Identification of beef, sheep, goat and pork in raw and heat-treated meat mixtures. J. Food Protect. 67:172-177. 2004. [ Links ]
20.Sambrook, J.; Fritsh, E.F.; Maniatis, T. Commonly used techiniques in Molecular Cloning. Molecular Clonning; A laboratory manual, 2nd Ed., Cold Spring Harbor, New York. 89-112. pp. 1989. [ Links ]
21.Unseld, M.; BEYERMANN, B.; Brandt, P.; Hiesel, R. Identification of the species origin of highly processed meat products by mitochondrial DNA sequences. PCR Meth. Appl. 4: 241-243. 1995. [ Links ]












 uBio
uBio