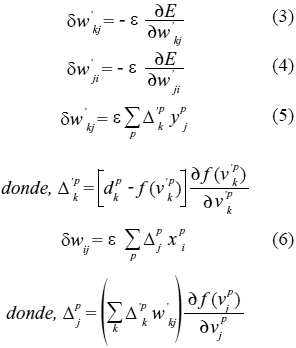Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de la Facultad de Ingeniería Universidad Central de Venezuela
Print version ISSN 0798-4065
Rev. Fac. Ing. UCV vol.26 no.3 Caracas Sept. 2011
Detección de microcalcificaciones en imágenes mamográficas usando redes neuronales
Aníbal Guerra, Joel Rivas
Facultad de Ciencias y Tecnología, Universidad de Carabobo, Venezuela.
ajguerra@uc.edu.ve, jrivas@uc.edu.ve
RESUMEN
El cáncer de mama es una de las principales causas de muerte entre las mujeres a nivel mundial, según la Asociación Americana del Cáncer. El factor clave para reducir el impacto de esta enfermedad es su detección temprana. El presente documento, describe el desarrollo de un software que tiene como objetivo principal constituirse como base de un
mecanismo de segunda opinión en el proceso de detección de microcalcificaciones, a través del estudio de imágenes mamográficas. El software trabaja a partir de una mamografía digitalizada, la cual es procesada para ingresarla como dato de entrada a una Red Neuronal Artificial (RNA) del tipo perceptrón multicapas; ésta se encarga de detectar si la imagen presenta microcalcificaciones. La RNA se implementó en lenguaje C++, con una arquitectura de una capa oculta y el algoritmo de aprendizaje Backpropagation, en combinación con técnicas basadas en el análisis estadístico sobre la textura de imágenes. La data base para el desarrollo de la aplicación proviene de la base de datos de la Sociedad de Análisis de Imágenes Mamográficas (MIAS, en sus siglas en inglés). La efectividad alcanzada en la evaluación del software fue de 94.4% de aciertos en su predicción, mostrando así el potencial de la aplicación de ambas técnicas para el abordaje del problema planteado.Keywords
: Mamografía, Redes neuronales artificiales, Detección asistida por computador, Microcalcificaciones.Micro-calcification detection in mammographic images using neural networks
ABSTRACT
According to the American Cancer Society, breast cancer is one of the leading causes of death in women worldwide. The key factor to reduce the impact of this disease is an early diagnosis. The software described in this document aims to be a
mechanism for second opinion in detection of micro-calcifications in mammographic images. In this software, digitalized mammographies are processed and inserted as entry data to a multi-layer perceptron, which is able to detect presence of micro-calcifications in the provided images. The neural network was implemented in C++ language; its architecture has one hidden layer and uses a back-propagation learning algorithm in combination with techniques of statistical analysis over the image texture. The data used in this research was extracted from the MIAS database. The software assessment reported 94.4% of sensitivity in prediction tasks, showing the potential of both techniques in the resolution of the problem.Palabras clave
: Mammography, Artificial neural network, Computer aided detection, Micro-calcification.Recibido: julio de 2010 Recibido en forma final revisado: julio de 2011
INTRODUCCIÓN
Para el año 2007, el cáncer de mama se había constituido el tipo de cáncer con mayor número de muertes entre las mujeres a nivel mundial, además de ser el primero en número de nuevos casos al año (American Cancer Society, 2007). Esta enfermedad posee un alto potencial mortal, pero de ser detectada a tiempo puede tratarse evitándose el fallecimiento de la paciente en un alto porcentaje de casos. El proceso de detección consiste en identificar la clase de lesión que se encuentre en la mama, la cual puede ser una masa o una calcificación, mientras que el proceso de diagnóstico consiste en identificar en qué zona de la mama se encuentra la lesión e identificar si ésta es benigna o maligna.
Las microcalcificaciones son pequeños depósitos de calcio en el tejido mamario, indicadores de la presencia de cáncer de mama en el 60% de los casos; por ello, resulta muy importante reconocerlas cuando se desea detectar esta enfermedad tempranamente. Actualmente, la principal herramienta que existe para la detección de una microcalcificación, en etapas cuando ésta aún no es palpable, es la mamografía (Salvador, 2006). La lectura adecuada de la mamografía es determinante en este proceso de detección temprana, pero no resulta una tarea sencilla dada su alta dependencia de la experticia del especialista y la influencia de variables de índole humano que afectan al proceso de interpretación (cansancio, fatiga, entre otros).
A fin de incrementar la eficiencia y eficacia con la que se lleva a cabo el proceso de detección de la enfermedad, surge la propuesta de desarrollar un sistema de detección asistida por computador (CAD) cuyo objetivo principal es brindar soporte a los médicos especialistas en la detección de microcalcificaciones en mamografías.
En este documento se describe el desarrollo de la aplicación (denominada SARA) que constituye la base para la construcción de un sistema computarizado de detección de microcalcificaciones en mamografías, partiendo de la premisa de requerir la inversión más baja posible para su implementación, proveer un alto nivel de usabilidad y requerir únicamente la imagen mamográfica digitalizada como insumo para la realización de la detección. Se presentan a continuación los aspectos relativos a las técnicas y procedimientos que condujeron el desarrollo propuesto, los resultados obtenidos de su implementación y las conclusiones producto de la investigación.
TÉCNICAS EXPERIMENTALES
La metodología utilizada en la investigación se encuentra inspirada por lo planteado en Aguilar & Rivas (2001), quienes integran enfoques, técnicas y otras metodologías provenientes de áreas vinculadas con sistemas basados en conocimiento e Ingeniería de Software.
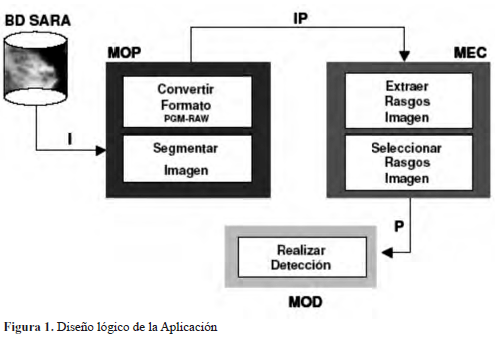
A fin de organizar y coordinar los requerimientos y las labores inherentes al desarrollo de la aplicación, se planteó un modelo lógico en el cual se separaban las tareas que debían realizarse en torno a 4 unidades conceptuales, cada una correspondiente a una tarea que debía ser resuelta. De esta forma podían darse soluciones independientes a cada sub-problema, las cuales se integrarían posteriormente resolviendo así el asunto original. En la Figura 1 se presenta el modelo lógico de la aplicación.
El primer paso consistió en la conformación de una base de datos de imágenes mamográficas digitalizadas que incluyese información sobre el diagnóstico asociado con cada una de ellas, de esta base de datos se extraerían las imágenes y éstas se ingresarían a un módulo de Preprocesamiento (MOP), en este módulo convierten a un formato estándar, se les elimina parte del ruido presente en la imagen y se les extrae la zona de interés. Esta Imagen Pre-procesada (IP) pasaría al módulo de extracción de características (MEC), en el cual se convierte en un conjunto de parámetros (P) que serán datos de entrada para el módulo de detección donde se ubicará la red neuronal artificial, la cual realizará la predicción asociada con la presencia o no de microcalcificaciones en esa mamografía.
1.- Conformación de la Base de datos SARA
Las imágenes mamográficas para este proyecto se tomaron de la base de datos: The Mammographic Image Analysis Society (MIAS) (Suckling, 2003). MIAS es creada a partir de la base de datos Digital Database for Screening Mammography (DDSM), de la Universidad del Sur de Florida. Digital Database for Screening Mamography (Kopans et al. 2006). Esta base de datos está conformada por 322 imágenes mamográficas correspondientes a la proyección medio lateral oblicua (MLO); de estos 322 casos, sólo 25 contienen microcalcificaciones y más de 160 son casos de mamas sanas. Las imágenes se encuentran en formato PGM de 1024 x 1024 píxeles digitalizadas a 8 bits y su tamaño es de 1.0 Mb.
La base de datos SARA se conformó con las 25 imágenes de mamas que presentan microcalcificaciones; de estos 25 casos, 10 son mamas densas glandular (D), 6 son fatty (F) y 9 son fatty glandular (G). Además se incluyeron 67 casos correspondientes a mamas sanas, de los cuales 27 (D), 16 (F) y 24 (G).
2.- Módulo Pre-procesamiento (MOP)
La primera tarea radicó en la conversión de formato de la imagen de pgm al formato raw, a través de un algoritmo implementado para tal fin. Luego, debía reducirse el ruido de la imagen que había sido producido por la digitalización de la mamografía original. Dicho algoritmo se construyó tomando como base el algoritmo de reducción de ruido desarrollado para las imágenes mamográficas originales de la base de datos de la Universidad del Sur del Florida (Kopans et al. 2006), el cual se basa en el análisis de las diferencias de niveles de gris entre las diversas zonas de la mamografía.
A continuación, se procedió a determinar la región de interés en la mamografía. En conjunto con los especialistas, luego de realizar múltiples observaciones y distintas pruebas de segmentación basadas en el estudio de la geometría mamaria, se definió una región de interés general enmarcada en un rectángulo que incluye la imagen del tejido del seno de la mujer. Se implementó un programa para automatizar la extracción de esta zona de interés, basado en la identificación de una región en la mamografía en forma de rectángulo, la cual comprende la zona del tejido mamario entre un límite izquierdo (LI) y un límite derecho (LD). Ambos límites se calculan, correlacionando la media y la desviación estándar de los píxeles en cada columna de la imagen, en función de un umbral (Barrio et al. 1999).
3.- Módulo Extracción de Características (MEC)
El objetivo en este módulo es extraer un conjunto de características de la región de interés que permita diferenciar entre una mama sana y una mama microcalcificada. Este proceso incluye dos fases: el cálculo de un conjunto amplio de rasgos de la imagen y el determinar cuáles de esos rasgos aportan información relevante para la detección.
Cálculo de rasgos de la imagen
Se realizó un exhaustivo estudio de las características relevantes indicadas por los mastólogos asesores del proyecto, así como lo planteado por otros autores en el área. Se seleccionó una técnica de extracción de características que se centra en el estudio de la textura asociada con los niveles de gris existentes en la imagen basándose en la matriz de co-ocurrencia de niveles de gris (Stelios et al. 1998), dado que es considerado el método de extracción de características de mayor robustez desde el punto de vista estadístico y matemático (Singh & Singh, 2003). Luego del proceso de análisis se encontró que los atributos producto de la evaluación realizada por los médicos asesores resultaron coherentes con los atributos estadísticos planteados para el estudio sobre la matriz de co-ocurrencia de niveles de gris.
La matriz de co-ocurrencia de niveles de gris,
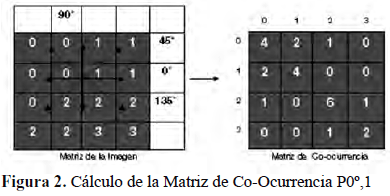
Gray Level Coocurrence Matrix (GLCM) o Spatial Grey Level Dependence Method (SGLDM) (Conners & Harlow, 1980), es el método de mayor alcance de todos los métodos considerados en esta investigación, entre ellos: Grey Level Run Length Method (GLRLM), Grey Level DifferenceMethod (GLDM) y Power Spectral Method (PSM); adicionalmente se considera uno de los más importantes en cuanto a descripción de la textura, especialmente para análisis de imágenes médicas (Svolos & Todd-Pokropek, 2008). La matriz de co-ocurrencia refleja las ocurrencias dadas de un valor de nivel de gris entre los vecinos de un píxel determinado. En esta investigación, se consideran las vecindades de grado: 0°, 45°, 90° y 135°, con distancia uno (1), tal como se ilustra en la Figura 2.De acuerdo a lo observado en investigaciones consultadas (Bovis & Singh, 2001; Burns, 1997), se planteó el cálculo de 11 valores estadísticos sobre la GLCM: energía, entropía, correlación, momento inverso de la diferencia, inercia, forma del racimo, prominencia del racimo, media y desviación estándar de la GLCM; además se incluyeron la media y desviación estándar de la matriz de la imagen mamográfica, según lo planteado en Kook & Wook (1999). Para cada imagen, se calculan los valores asociados con cada estadístico en la matriz de co-ocurrencia de acuerdo a cada grado (0°, 45°, 90° y 135°), el promedio entre ellos es el valor final del estadístico calculado para la imagen dada.
Determinación de los rasgos relevantes
A fin de identificar los rasgos determinantes para la
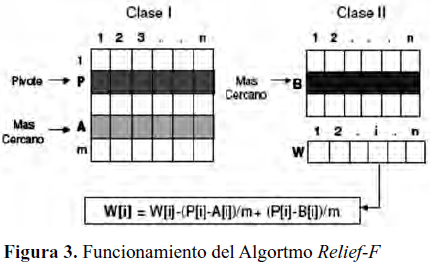
diferenciación entre el grupo de mamas sanas y el grupo de mamas con microcalcificaciones, se utilizó el método de selección de rasgos Relief-F (Kononenko, 1994). Para cada atributo a evaluar, Relief-F toma cada patrón y lo compara contra los demás patrones de su misma clase y los demás patrones de la otra clase involucrada en el problema; finalmente arroja un valor que representa la medida en la que ese atributo caracteriza a los patrones de su misma clase y lo diferencia de los patrones pertenecientes a la otra clase.Algoritmo
Relief-FPara todos los atributos: W[A]:=0
Inicio
Para i:=1 Hasta m Hacer
Seleccionar aleatoriamente un ejemplo de la clase R
Encontrar el ejemplo más cercano de la misma clase
H y el ejemplo más cercano de la otra clase M
Para A:=1 Hasta Num_Atributos Hacer
W[A] := W[A] – ( Diff(A,R,H) + Diff(A,R,M) )/m
Fin
Donde:
Diff(Atr,Instancia1,Instancia2) calcula la diferencia euclidiana entre los valores del atributo Atr de ambas clases involucradas, normalizada en el intervalo [0,1]. El peso final de cada atributo (W[A]) se encuentra en el intervalo [0,1], y refleja la medida en que el atributo permite establecer diferencias con respecto a los elementos que conforman la otra clase y la medida en que él establece semejanzas con los elementos de su propia clase. A medida de que este valor se acerque a uno (1), aportará información importante en la discriminación de distintas clases. La Figura 3 representa el principio de funcionamiento del Algoritmo Relief-F:4.- Módulo de Detección (MOD)
En el módulo de detección se concentran los procesos directamente inherentes a la detección de las microcalcificaciones; esto corresponde al diseño e implementación de la red neuronal artificial (RNA), la cual dada su capacidad de abstracción, clasificación y predicción, permitirá alcanzar los objetivos propuestos.
Las RNA son sistemas de cómputo compuestos por un gran número de elementos básicos (neuronas artificiales) agrupados en capas y que se encuentran altamente interconectados (sinapsis). Esta estructura posee varias entradas y salidas, las cuales serán entrenadas para reaccionar, de una manera deseada, a los estímulos de entrada, emulando el funcionamiento del cerebro humano. Requieren aprender a comportarse en situaciones no conocidas previamente (interrogatorio) y alguien debe encargarse de enseñarles o entrenarles (entrenamiento), sobre la base de un conocimiento previo del entorno del problema. Para construir la RNA se deben considerar los procesos que abarcan: la generación de los insumos para la red neuronal artificial (construcción del vector de entrada de la red, seleccionar el conjunto de patrones de entrenamiento, pre-interrogatorio e interrogatorio) y el diseño e implementación de la arquitectura de la RNA.
Construcción del vector característico
A partir de los rasgos seleccionados mediante el algoritmo Relief-F, se construye el vector de entrada a la RNA, adicionalmente, se anexa la salida deseada para cada patrón, ya que ésta debe incluirse durante la fase de entrenamiento. Los parámetros se normalizan en la escala [0,1] y la salida de la red se define como cero (0) en el caso de dar negativo, o uno (1) en caso de dar positivo, respecto al test de presencia de microcalficaciones.
Definición de los Conjuntos de Entrenamiento/Interrogatorio RNA
A partir de las 92 imágenes contenidas en la BD SARA, se realizó la construcción aleatoria de los conjuntos de patrones correspondientes a cada actividad de la red (entrenamiento, pre-interrogatorio e interrogatorio), empleando la técnica de validación simple y la técnica de validación cruzada, adaptadas para aplicaciones de inteligencia artificial según Galindo (1999). Dada la poca cantidad de patrones microcalcificados que existen en la base de datos MIAS (25 casos), se decidió seleccionar una cantidad equivalente de mamas sanas para estructurar los conjuntos, por ende, al conformar los conjuntos de prueba se escogieron aleatoriamente 25 mamas sanas de la totalidad de 67 disponibles. Se realizaron seis (6) experimentos, cada uno utilizando un grupo de datos distinto, tanto para la etapa de pruebas I (Entrenamiento, Pre-Interrogatorio e Interrogatorio) como para la etapa de pruebas II (Entrenamiento e Interrogatorio). Como la etapa de pruebas I se plantea con el propósito de entonar la arquitectura de la red, en este documento se presentan específicamente los resultados asociados con la fase de pruebas II, específicamente respecto a la fase de interrogatorio pues es la que permite evaluar la calidad de la predicción realizada por la red.
Diseño de la Red Neuronal Artificial
Se seleccionó como modelo de RNA el Perceptrón multicapa generalizado, con el algoritmo de aprendizaje de Retropropagación del error, por su convergencia a una solución aceptable con una arquitectura de solo 1 capa oculta. El esquema de desarrollo de la RNA se basó en modelo general del perceptrón multicapa (Aguilar & Rivas, 2001). El modelo de arquitectura que se va a definir corresponde al presentado en la Figura 4.
La RNA está compuesta de solo una capa oculta y el número
de neuronas en esta capa se determina por un proceso de experimentación al que se somete la red (etapa de prueba I). Dado que la capa de salida posee sólo una neurona, cuya salida se encuentra definida en el intervalo discreto [0,1], se seleccionó como función de transferencia a función sigmoide. La inicialización de los pesos sinápticos se hace aleatoriamente con números entre -0.3 y 0.3. La principal medida que permite estudiar al desempeño de la red es el Error Cuadrático Medio, el cual se modifica a través de las distintas iteraciones según la variación que cada uno de los pesos wij (intensidad de la conexión entre la i-ésima señal de entrada del patrón y la unidad de procesamiento o neurona j-ésima). Siendo cada patrón de entrada de la forma (xk;dk), correspondiente a los atributos característicos del patrón p y su salida deseada asociada, la medida del error se calcula: ![]()
Corresponde a la sumatoria de la diferencia del error existente entre la salida deseada y la salida calculada por la red para ese patrón, esta última según la ecuación:
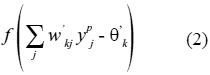
donde: los
yj corresponden a cada una de la salidas de las unidades de procesamiento de la última capa oculta y f es la función de activación de las neuronas de la capa de salida. Si estas salidas son muy diferentes de las salidas deseadas, la desviación cuadrática entre ambas resultará en un valor alto y debe minimizarse.Sobre esta función de coste global se aplica un procedimiento de minimización. En este caso, se hace mediante un descenso en la dirección contraria del gradiente de
E( wij, qj, wkj, q k). Las expresiones que resultan aplicando la regla de la cadena son las siguientes:siendo:
yj las salidas de la capa oculta y e la tasa de aprendizaje.El aprendizaje por
backpropagation comprende los siguientes procesos:1. Inicializar los pesos sinápticos y los umbrales de cada neurona, entre -0.3 y 0.3.
2. Para cada patrón del conjunto de los datos de entrenamiento.
a) Obtener la respuesta de la red ante ese patrón. Esta parte
se consigue propagando la entrada hacia adelante, ya que este tipo de red es feedforward (Unidireccional). Las salidas de una capa sirven como entrada a las neuronas de la capa siguiente, procesándolas de acuerdo a la regla de propagación y la función de activación correspondientes.b) Calcular los errores asociados según las ecuaciones
correspondientes con los términos delta, usando las ecuaciones (3),(4),(5) y (6).c) Calcular los incrementos parciales (sumandos de las
sumatorias). Estos incrementos dependen de los errores calculados en 2.b.3. Calcular el incremento total, para todos los patrones,
de los pesos y los umbrales según las expresiones en la ecuación de los términos delta, (3),(4),(5) y (6).4. Actualizar pesos y umbrales.
5. Calcular el error cuadrático medio actual y volver al paso 2 si no es satisfactorio.
Determinación de la Arquitectura de la RNA
El número de neuronas de la capa oculta que optimizó el desempeño de la red, se obtuvo siguiendo un procedimiento sistemático, evaluando el desempeño de la RNA desde 2 neuronas, incrementando este número en uno hasta llegar a 20 neuronas.
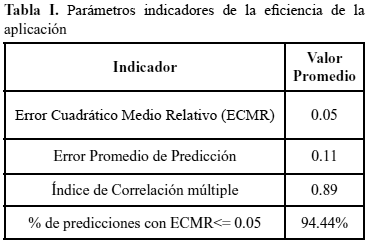
Para establecer los valores finales de los parámetros de la red: constante de aprendizaje (ETA), razón de momento (ALFA) y máximo número de iteraciones de la RNA, se realizaron más de 230 experimentos (correspondientes a la etapa de pruebas I) evaluándose: ETA en el rango de 0.1 a 0.175, ALFA de 0.95 a 0.975 (ambas, con una variación de 0.01) y la cantidad de iteraciones desde 500.000 hasta 50.000.000. A fin de determinar los valores de cada parámetro que optimicen el desempeño de la red, se calculan los siguientes estadísticos en cada ejecución de la RNA: Error promedio y máximo error total del sistema, máximo error individual por patrón, error cuadrático medio e índice de correlación múltiple.
RESULTADOS
Utilizando el algoritmo Relief-F y analizando los 11 valores estadísticos calculados a cada imagen, se encontró que aquellos que realmente aportan información relevante a la predicción a realizarse son: Desviación estándar de la imagen mamográfica, desviación estándar de la GLCM, entropía, inercia, Momento inverso de la diferencia, media de la mamografía digital, media de la matriz de coocurrencia y la media Mu_i, calculada sobre la suma de las GLCM. Estos constituyen los elementos finales del vector de entrada o vector característico de la RNA.
Una vez realizadas las pruebas correspondientes a la etapa de pruebas I, que buscaba determinar el resto de la arquitectura y el valor adecuado para cada uno de los parámetros de la red se obtuvo los siguientes resultados: el mejor desempeño de la red se alcanzó con una capa oculta conformada por 10 neuronas y con la siguiente combinación de parámetros: razón de momento ALFA = 0.175, constante de aprendizaje ETA = 0,965 y número de iteraciones ITER = 7.500.000. La función de transferencia utilizada fue la función sigmoide. Así se completó la configuración final de la red junto con los 8 componentes en la capa de entrada de acuerdo a los rasgos de textura relevantes de la imagen ya especificados y un nodo correspondiente a la capa de salida.
Definida la arquitectura de la red, se tomaron los conjuntos correspondientes a la fase de pre-interrogatorio y se incorporaron equitativamente a los grupos de patrones para entrenamiento e interrogatorio, de este modo, se obtiene 60% de los 50 patrones originales para la fase de entrenamiento y 40% para la fase de interrogatorio. El mecanismo empleado fue el de validación cruzada.
Se inició la etapa de pruebas II, con el objetivo único de medir la calidad de la predicción de la red diseñada. En cada uno de los seis (6) experimentos correspondientes, uno por cada conjunto de prueba, durante la fase de entrenamiento de la red se alcanzó una sensibilidad del 100%. En la fase de interrogatorio, se alcanzó un 94.4% de aciertos en el estudio de los seis (6) grupos de dieciocho (18) imágenes cada uno, la tasa de falsos positivos fue inferior al 6% de las evaluaciones. Los indicadores de desempeño de esta etapa de pruebas se presentan en la Tabla I.
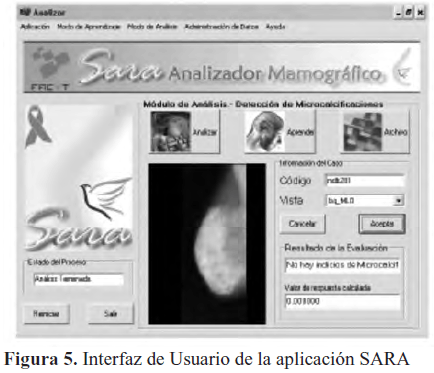
Finalmente, se diseñó una interfaz grafica de usuario (Figura 5) que permitiera utilizar a las funcionales provistas por cada uno de los módulos de la aplicación diseñada, de manera sencilla y amigable.
CONCLUSIONES
Se pudo verificar la validez de los atributos seleccionados en el desarrollo de SARA a través de la aplicación de Relief-F comparando los resultados con los obtenidos en el estudio de Rikxoort, 2005, en el que se determinan las características de textura más relevantes al hacer análisis de imágenes, utilizando inteligencia artificial o técnicas similares. En dicho estudio, de los 9 atributos destacados por el autor como altamente pertinentes, 8 de ellos coincidieron con los utilizados en la presente investigación.
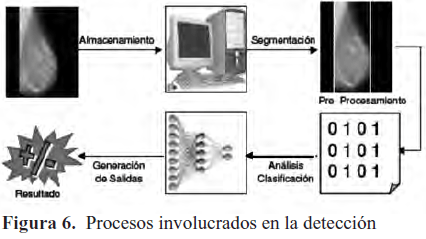
La experiencia obtenida, permitió constatar la gran utilidad de las redes neuronales artificiales en el desarrollo de sistemas de detección asistida por computador, constituyéndose en una alternativa eficaz para la producción de programas robustos y de bajo costo que ofrezcan un mecanismo de segunda opinión a los especialistas. Particularmente, se pudo comprobar la utilidad de esta técnica en la detección de microcalcificaciones en mamografías digitales al combinarse con técnicas de procesamiento de imágenes, lo cual podría ayudar a optimizar la labor realizada por los especialistas del área. Al respecto, se pudo verificar la utilidad del análisis textural en el estudio imágenes mamográficas, específicamente en la caracterización a través de atributos, ofreciendo un balance adecuado entre la calidad de la caracterización y la complejidad computacional de los cálculos asociados. Ambos aspectos permiten satisfacer el objetivo inicialmente planteado, la Figura 6 refleja los procesos involucrados en la detección que realiza la aplicación.
En la Tabla II, se contrastan los resultados obtenidos en esta investigación con otras aplicaciones de objetivo similar, que aplican otras técnicas sobre el mismo dominio de datos.
Se evidenció la alta complejidad que envuelve el proceso de detección de microcalficaciones, específicamente respecto a los falsos positivos. Estos casos fueron llevados ante especialistas en mastología, quienes concluyeron que estos falsos positivos se dan por la presencia de notables cortes transversales de vasos sanguíneos que, por su similitud, son comúnmente confundidos con microcalcificaciones. Acotaron que esto sólo puede evitarse con la experticia producida por los años de ejercicio de la profesión.
Se recomienda profundizar el estudio de las técnicas planteadas con un conjunto de datos de mayor dimensión y contrastarlo con la aplicación de otros modelos de máquinas inteligentes como las máquinas de soporte vectorial, máquinas de vectores relevantes, y otras.
AGRADECIMIENTOS
Se agradece a los especialistas mastólogos Dr. Luis Torres
S. y Dr. Humberto López por su invalorable contribución al desarrollo de la presente investigación.REFERENCIAS
1. Aguilar, J. & Rivas, F.
(2001). Introducción a las Técnicas de Computación Inteligente. Merida: José Aguilar Castro, Francklin Rivas Echeverría Editores. p 164-169. [ Links ]2. American Cancer Society
. (2007). Global Cancer Facts & Figures 2007. Recuperado el 08 de octubre de 2007, de http://www.cancer.org/downloads/STT/Global_Facts_and_Figures_2007_rev2.pdf [ Links ]3. Barrio, D., Manrique, D., Ríos, J., Vilarrasa, A.
(1999). Detección Automática de Regiones de Interés en Mamografías Digitalizadas para el Diagnóstico Asistido por Ordenador. Facultad de Informática (U.P.M). Campus de Montegancedo, Madrid. Recuperado el 20 de enero de 2006 de http://www.seis.es/i_s/i_s20/is20c.htm [ Links ]4. Bovis, K. & Singh, S
. (2001). Classification of Mammographic Breast Density Using a Combined Classiffier Paradigm. PANN Research, Department of Computer Science. University of Exeter, Exeter, UK. Recuperado el 20 de Enero de 2008, de http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.19.1806 & rep=rep1 & type=pdf [ Links ]5. Burns, I.
(1997). Grey Level Cooccurence Matrix. Recuperado el día 18 de Agosto de 2008, de http://www.cssip.uq.edu.au/meastex/www/for_code.html [ Links ]6. Conners, R. & Harlow, C. A
. (1980). A Theoretical Comparison of Texture Algorithms. IEEE Transactions on Pattern Analysis and Machine Intelligence, Vol. PAMI-2, p. 204-222. [ Links ]7. Galindo, P
. (1999). Técnicas de Validación de Sistemas Inteligentes de Computación SIC. Universidad de Cádiz, España. Recuperado el día 26 de octubre de 2006, de http://www.uca.es/dept/leng_sist_informaticos/preal/23041/transpas/F-Validacion/ppframe.htm [ Links ]8. Kononenko, I
. (1994). Estimating Attributes: Analysis and Extensions of Relief. Lecture Notes in Computer Science, Springer Berlin / Heidelberg, Volume 784/1994, p 171-182. [ Links ]9. Kook, J. & Wook, H
. (1999). Statistical Textural Features for Detection of Microcalcifications in Digitized Mammograms. IEEE Transactions on Medical Imaging. Vol 18, No 2, p 231-238. [ Links ]10. Kopans, D., Moore, R., Bowyer, K., Kegelmeyer, P.
(2006). Digital Database for Screening Mamography DDSM. University of South Florida. Recuperado el 15 de agosto de 2007 de: http://marathon.csee.usf.edu/Mammography/Database.html [ Links ]11. Oporto, S.
(2005). Detection of Microcalcification Clusters in Mammograms Using a Difference of Optimized Gaussian Filtres. Lecture Notes in Computer Science, Springer Berlin / Heidelberg. Volume 3656, 2005, p. 998-1005. [ Links ]12. Rikxoort, E
. (2005). Content-Based Image Retrieval: Utilizing color, texture, and shape. The features describing texture, (Magister Degree Thesis). Radboud University Nijmegen Faculty of Social Sciences Nijmegen, The Netherlands. [ Links ]13. Salvador, R
. (2006). Mamografía Digital. Recuperado el día 5 de noviembre de 2007, de: www.imaginebarcelona.com/divulgacion/mamografia.htm [ Links ]14. Singh, M. & Singh, S
. (2003). Texture Algorithms: Performance Variability Across Data Sets. Cybernetics and Systems, Volume 34, Issue 1 January 2003, p 1-17. [ Links ]15. Stelios, H., Mantas, J., Taxiarchis, B
. (1998). Computer Aided Detection of Clustered Microcalcifications in Digital Mammograms. University of Athens Faculty of Nursing. Recuperado el 20 de Enero de 2007, de http://fmi.uni-sofia.bg/courses/biomed/morphgeom/papers/Halkiotis.pdf [ Links ]16. Suckling, J.
(2003). The Mini-MIAS Database of Mamograms (MIAS). Recuperado el 01 de Octubre de 2007, de http://peipa.essex.ac.uk/info/mias.html [ Links ]17. Svolos, A. & Todd-Pokropek, A
. (2008). Time and Space Results of Dynamic Texture Feature Extraction in MR and CT Image Analysis. IEEE Trans Inf Technol Biomed, Jun;2(2), p 48- 54. [ Links ]