Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Zootecnia Tropical
versión impresa ISSN 0798-7269
Zootecnia Trop. v.27 n.4 Maracay dic. 2009
Variabilidad genética del ADN mitocondrial de poblaciones de abejas Apis mellifera (Hymenoptera: Apidae) en Colombia
Guillermo Salamanca Grosso *
Grupo de Investigaciones Mellitopalinológicas y Propiedades Fisicoquímicas de Alimentos. Facultad de Ciencias Departamento de Química. Universidad del Tolima, B. Santa Helena. A.A. 546. Ibagué, Tolima. Colombia. *Correo electrónico: salamancagrosso@gmail.com
RESUMEN
El ADN mitocondrial de Apis mellifera fue estudiado en 105 colmenas colectadas de 7 zonas geográficas colombianas asociadas a 14 departamentos. Se amplificó mediante PCR un fragmento de la región intergénica ARNtleu y de la citocromo oxidasa II (COII), usando posteriormente la endonucleasa DraI. Tras la digestión con la enzima DraI, se identificaron 13 haplotipos, 8 asociados al linaje africano A (con 90,5% del total para A1, A2, A4, A5, A6, A8, A9 y A13), 4 al linaje europeo M (7,6%, M2, M4, M5 y M7) y 1 al C de Europa del Este (C1 con 1,9%). La presencia de los 8 haplotipos A diferentes para las abejas de Colombia, sugiere que ha habido más de un episodio de hibridación, introgresión y expansión del fenómeno de africanización en el territorio colombiano, aspecto que supone existan otros haplotipos en poblaciones no incluidas en el estudio. El gradiente de distribución del linaje africano, esta representado principalmente por el haplotipo A1, desde la Costa Caribe colombiana al centro del país, donde se distinguen además los haplotipos A4 y A6. Los linajes europeos no se ven representados de manera importante en la población estudiada, por ello se demuestra que la actividad apícola colombiana muestra una marcada dependencia de colonias africanizadas.
Palabras clave: Apis mellifera, ADN mitocondrial, genética, Colombia, polimorfismos, haplotipos.
Genetic variability in mitochondrial DNA of honeybee populations of Apis mellifera (Hymenoptera: Apidae) in Colombia
ABSTRACT
The mitochondrial DNA of Apis mellifera, sampled in 105 beehives and 7 geographical zones associate to 14 localities in Colombia have been studied. A fragment of the intergenic region between the RNAtleu and the subunit II of the cytochrome oxidase gene was amplified by PCR and digested with the endonuclease DraI. Thirteen haplotypes were identified: eight belonging to the African lineage A (90,5% for A1, A2, A4, A5, A6, A8, A9 and A13), 4 to European lineage M (7,6% M2, M4, M5 and M7) and 1 to C (C1, 1,9%), of the east European lineage. The presence of eight different A haplotipes, suggests the occurrence of more than one episode of hybridization, introgression and expansion of the africanized phenoma which suggests the presence of other haplotypes in other populations not included in the study. The gradient of distribution of African lineage is represented by the A1 haplotype, to the center of the country from the Caribbean coast with A4 and A6 haplotypes. The european lineages, are not represented significantly on the population studied. Colombia beekeeping activities depend on the africanized honeybee.
Keywords: Apis mellifera, mitochondrial DNA, genetic, Colombia, polymorphism, haplotypes.
Recibido: 06/05/08 Aceptado: 14/10/09
INTRODUCCION
En Colombia se distinguen diferentes razas de abejas que fueron introducidas desde Europa, su aparición es concomitante con el descubrimiento de América y la actividad en el período de la colonización con presencia de A. mellifera en Norte América 1622, Cuba 1763, Brasil y Chile 1839, Salamanca, (2005). La comunidad salesiana a comienzos de los años 1900, marcó para el país el inicio de la apicultura técnica. En ese período se importaron abejas Italianas, Caucasianas y Carniolas; en los años 30`s las explotaciones eran rústicas y fue fortalecida la importación de reinas como política nacional, en procura de incentivar el sector. Esto se logró entre 1940 a 1960, con la participación de la comunidad la sallista y jesuita, que propiciaron las condiciones para la expansión del conocimiento entorno al manejo de las abejas a nivel rural. Desde el punto de vista productivo las razas de abejas de mayor interés a nivel mundial, se han centrado en A. mellifera mellífera L. 1758, A. mellifera Ligústica Spinola, 1806, A. mellifera Cárnica Pollmann, 1879 y A. mellifera Caucásica Gorb, 1758. Ruttner (1988); Roubik, (1991); Heras (1994); Sheppard et al. (1997).
La geografía colombiana está enmarcada sobre un área de 1141.748 km2, que comprende tierras emergidas y continentales. La circulación atmosférica es dependiente del sistema de confluencia intertropical (ZCIT), Osorio, et al. (2002). Las condiciones climáticas predominantes y la topografía del terreno, hacen que la actividad apícola sea considerada bajo el criterio de cotas altitudinales, más que zonificaciones políticas. Los pisos térmicos de interés para el sistema apícola corresponden a las zonas fría, (2.000 m.s.n.m.), templada (1.000 m.s.n.m.) y caliente (0 a 900 m.s.n.m.), donde actualmente se encuentran instaladas colmenas para el beneficio y cosecha de miel y polen, Kent (1976); Franky, (2008); Salamanca et al. (2008). A finales de los 70´s, Colombia recibió el impacto del proceso de africanización, generado en Brasil 20 años atrás, después de la expansión de A. mellifera scutellata (antes adansonii), con los trabajos de Kerr en 1956. Fallas en la manipulación de reinas africanas importadas desencadenaron la fuga de 26 enjambres en 1957, que sin control colonizaron Venezuela, Colombia y Panamá, Fisher (1985); a su paso, A. mellifera scutellata se apoderó de los hábitats, ocupados hasta entonces, por las abejas europeas, Kerr, (1970; 1991); Kerr et al. (1974); Núñez (2000), cruzándose con ellas o eliminándolas, Castaño, et al. (1979). El fortalecimiento de la actividad apícola en Colombia, antes del proceso de africanización obedeció a las políticas del Estado y la Federación de Cafeteros, Cornejo (1978, 1993).
En el mundo, los estudios sobre dispersión geográfica de las razas de abejas, se han fundamentado en valoraciones morfométricas, permitiendo su clasificación e identificación, Ruttner (1988); Daly y Balling (1978); Molina (1979); Díaz (1981); Malaspina (1982), Dinitz- Filho y Malaspina (1995) y Dinitz- Filho (1995). Existen cerca de 50 caracteres que permiten la diferenciación de razas, según su grado de complejidad. Los trabajos sobre africanización en Colombia, han sido realizados por Amaya y Roldan (1983); Morales (1995); Mantilla (1997); Salamanca, et al. (1998), quienes usaron criterios morfométricos y técnicas de análisis discriminante en la clasificación y diferenciación de abejas hibridas europeas y africanizadas. Con el advenimiento y desarrollo de la Biología Molecular, el análisis de restricción de fragmentos (RFLP´s) y microsatélites, en las que se usa la amplificación de ADN mediante reacciones en cadena de la polimerasa (PCR), facilitando los estudios sobre filogenia y evolución que han sido aplicados a diferentes especies y particularmente útiles en la dispersión de las abejas e identificación de razas, De la Rúa et al. (1998); Cánovas et al. (2008).
La caracterización molecular basada en ADN mitocondrial (ADNm), se ha convertido en una técnica ampliamente utilizada para el estudio de la diferenciación de subespecies y razas de abejas de los géneros A.s mellifera L. y Melliponinae entre otros, Smith (1991); Garnery et al. (1992; 1993; 1995); De la Rúa et al. (1999, 2002); Cánovas, et al. (2002; 2008); Del Lama, et al. (2004); Prada et al. (2005); Collet, et al. (2006). El ADNm de Apis mellifera, es de estructura circular, de herencia materna y ha sido usada en la diferenciación y caracterización de reinas de distintas colonias y razas; al evaluar la estructura molecular del ADNm de la progenie, se logra establecer un marcador genético de referencia a través del cual se puede indagar sobre el origen de una colonia, De la Rúa et al. (2001). Actualmente se han identificado para el caso de las abejas A. mellifera 5 linajes evolutivos, 1 de ellos (A) que incluye las subespecies africanas adansonii, unicolor, capensis, montícola y scutellata, entre otras; el (M), constituido por razas de Europa Occidental, incorporando A. mellifera mellifera, ibérica, sahariensis, major e intermissa; el (C), conformado por abejas de Europa del Este, entre ellas A. m. ligústica, cárnica, macedónica, cecropia y sícula; el linaje (O), que comprende las subespecies de Oriente Próximo, con A. mellifera meda, anatoliaca, syriaca, cipria y adami; el linaje (Y) que incluye a la subespecie A. m. yemenitica de Etiopía, Franck et al. (2000; 2001); Hernández (2002).
Cada uno de lo grupos descritos anteriormente, presentan estructura característica en la secuencia de diferentes regiones de su ADNm; entre los genes del ARNtleu y de la citocromo oxidasa II (COII), sección para la cual se ha observado en el linaje M una diana de la endonucleasa HincII que no se encuentra en el linaje A, Hall y Smith (1991), Franck et al. (2001). Los estudios genéticos en poblaciones de abejas, han demostrado la dominancia de los genes africanos en las abejas hibridas establecidas en áreas tropicales y subtropicales del Centro, Norte y Sur América; Crozier et al. (1989); Hall y Muralidharan (1989); Del Lama, et al. (1990); Roubick, (1991); Rinderer et al. (1993); Burgett et al. (1995); Sheppard et al. (1999); Lobo (2000); Quezada-Eúan, (2000); DeGrandi- Hoffman, (2001); Scott et al. (2004); Quezada-Eúan et al. (2003), evidenciaron la presencia de zonas comunes con incidencia de genes africanizados. La dominancia de los híbridos de abejas, son el resultado de una superioridad competitiva frente al hábitat de trópico. El presente estudio, tiene como objetivo caracterizar poblaciones de abejas de Apis mellifera, distribuidas en las principales regiones naturales colombianas usando técnicas moleculares de análisis para identificar la variabilidad del ADNm.
MATERIALES Y MÉTODOS
Muestras
Se colectaron entre 20 y 30 abejas operarias de los cuadros interiores de 105 colmenas de abejas A. mellifera de instalaciones productivas correspondientes a las zonas biogeográficas de los departamentos de Antioquia: (Fredonia); Bolívar: (Carmen de Bolívar); Boyacá: (Cerinza, Nobsa, Paz del Río, Socha, Tutasá, y Tibasosa); Caldas: (La Dorada); Cauca (Cajibio, Caloto, Inzá, Piendamó); Cesar: (Valledupar, Aguas blancas), Cundinamarca (La Calera); Huila: (Pitalito, Timaná); Nariño: (Buesaco); Santander del Sur: (Oiba); Tolima: (Anzoátegui, Cajamarca, Chaparral, Cunday, Ibagué- Salado, La Herrera, Mariquita y Venadillo) y Valle del Cauca: (Caicedonia). Cada muestra fue preservada usando etanoles absolutos y almacenados a -20°C hasta su procesado y análisis, en el laboratorio del Departamento de Zoología y Antropología Física de la Universidad de Murcia. La relación de muestras y su origen se recoge en el Cuadro 1.
Cuadro 1. Tamaño y composición de los productos amplificados por PCR y fragmentos de la región intergénica RNAtleu-COII en poblaciones de abejas colombianas.
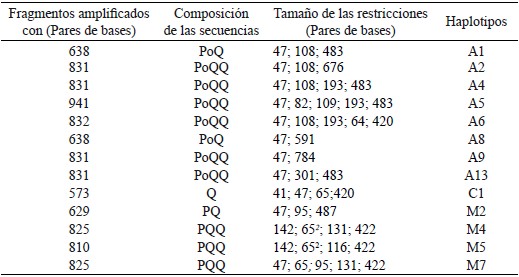
Los exponentes indican el número de fragmentos de restricción de igual tamaño.
Extracción de ADN
El ADN se obtuvo siguiendo la metodología de extracción con Chelex; se diseccionaron los músculos torácicos de las obreras y se dejaron secar por media hora en una estufa a 37ºC. La extracción se realizó según protocolo descrito por Walsh et al. (1991), con ligeras modificaciones. De la solución final del protocolo se tomo 1ml de ADN para la reacción de amplificación por PCR.
Amplificación por PCR y análisis de restricción
El análisis de ADN se llevó a cabo en un termociclador Perkin Elmer Cetus 480, según el método descrito por Garnery et al. (1993) para amplificar la región intergénica ARNtleu-COII, utilizando los cebadores: E2 (5-GGC AGA ATA AGT GCA TTG-3) y H2 (5-CAA TAT CAT TGA TGA CC-3). Se utilizaron PCR beads de Pharmacia a un volumen final de 25 ml con una concentración 0,16 mM de cada cebador. Los programas de amplificación consistieron en: desnaturalización a 96ºC durante 5 min, 35 ciclos de 1 min con una temperatura de anillado de 53ºC para la región intergénica ARNtleu-COII, con extensión a 72ºC durante 1 min y desnaturalización de 45 seg a 96ºC y por último 10 min de extensión a 72ºC. Los productos amplificados se examinaron en gel de agarosa (Nusive, al 5% en tampón TBE 1X). Una alícuota de 10 ml del producto de amplificación por PCR, fue digestado con 5 unidades de la enzima DraI a 37ºC y por 4-12 horas, teñidas con bromuro de etidio y visualizados con luz ultravioleta. Se empleó la enzima de restricción con capacidad de diagnóstico: DraI, especifica para la región intergénica ARNtleu-COII, Garnery et al. (1993).
Análisis estadístico
Para establecer las diferencias de la distribución de haplotipos y linajes predominantes en la población de abejas estudiadas, se realizó una prueba de ANOVA, usando el paquete estadístico Statgraphics centurión XV.
RESULTADOS Y DISCUSION
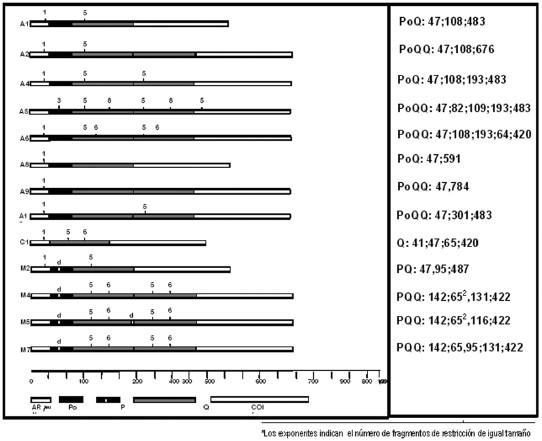
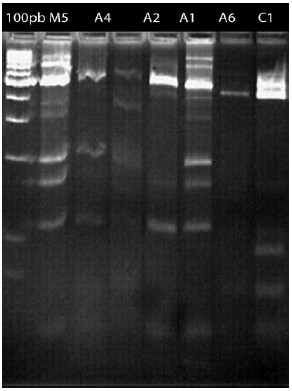
La estructura genética del ADNm, secuenciada por Crozier y Crozier (1993), presenta un tamaño aproximado de 16.343 pb, se ha considerado como marcador filogenético. La región intergénica sobre la cual fue realizado el estudio, está comprendida entre ARNtleu y COII; la digestión y amplificación del ADN con DraI (Figura 1), luego de la electroforesis en gel de agarosa, revela secuencias P, Po y P1 (51 a 68pb) y Q (192 a 196 pb) descritos por Cornuet et al. (1991); Garnery, et al. (1993); Collet, et al. (2006), en consecuencia, la región intergénica se caracteriza por la variabilidad en tamaño y depende del número de copias de la secuencia Q, además de la selección completa o parcial de las secuencias tipo P, Hernández, (2002), que para el caso de la población estudiada de abejas colombianas, se menciona en el Cuadro 1, cuyo patrón es análogo a las descritas por Lobo (2000) y consideradas en los trabajos de De la Rúa et al. (2003). En la Figura 2 se ilustran los patrones de restricción de algunos haplotipos A1, A2, A4, A6, M4, M5 y C1.
Figura 1. Mapas de restricción (izquierda) y Longitud de los fragmentos de restricción en pares de bases (derecha) de los haplotipos de abejas colombianas (según Garnery, et al., 1993; Collet, et al., 2006). d, indica delección en secuencia Po y Q. Los números sobre las barras indican los puntos de corte de la endonucleasa DraI.
Figura 2. Patrones de restricción de la región Intergénica ARNtleu-COII del ADNm de muestras de abejas colombianas, en gel de Agarosa 5%, teñido con bromuro de etidio. Se observan los haplotipos A1, A2, A4 y A6 (canaleta 4, 3, 2 y 5), M4 y M5 y C1. A la izquierda, se relaciona el marcador de tamaño molecular de 100 pb.
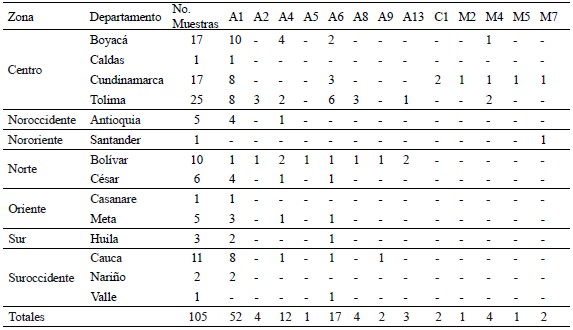
En el Cuadro 2, se relaciona la distribución de haplotipos de ADNm de las poblaciones de abejas de las localidades consideradas en el estudio, obtenidos mediante test con DraI, Garnery et al. (1993). El haplotipo con mayor predominancia es el A1 (50,0%), presente en el centro del país (Boyacá, Caldas, Cundinamarca y Tolima), seguido de A6 y A4, con 16,2 y 11,0% respectivamente. Los haplotipos M y C, están débilmente representados; las abejas colombianas de la Costa Norte (Bolívar y Cesar) y las otras localidades incluidas en el estudio, indican que estas poblaciones presentan en su estructura genética los haplotipos ya definidos por Estoup, et al. (1995).
Cuadro 2. Distribución de haplotipos de ADN identificados en muestras de abejas colombianas mediante el test de DraI. Garnery et al. (1993). Zona
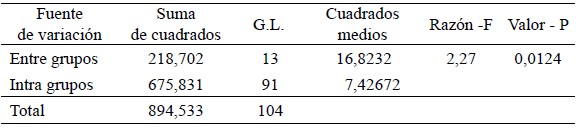
El análisis de varianza para la distribución de haplotipos observados de los departamentos considerados en el estudio, revela diferencias estadísticamente significativas (Pv: 0,0124, Cuadro 3). Entre tanto en la comparación de haplotipos y linajes mitocondriales, no se observan diferencias significativas (Pv: 0,2293, datos no mostrados), indicando alta incidencia de linajes africanos.
Cuadro 3. Resultados del análisis de varianza para la distribución de haplotipos de abejas colombianas por departamento.
La frecuencia de haplotipos observada en la costa Caribe colombiana corresponde a los haplotipos A1, A4, A6 y A13, (75%), con un 25% distribuido entre A2, A5, A8 y A9. Este mismo comportamiento, se presenta para el Noroccidente con A1 (80%) y A4 (20%). Es importante indicar que entre estas 2 zonas, dadas las condiciones climáticas y de oferta floral, existe una alta actividad trashumante, que contribuye a la dispersión de los haplotipos A1 y A4. La zona de los Llanos (Casanare y Meta), afectada de manera importante por el proceso de introgresión de las abejas africanizadas en la década de los 80´s, revela dominancia de genes de los haplotipos A1 (66%), A4 y A6 (34% en total). La mayor variabilidad y grado de entrecruzamiento se ha encontrado en la estructura apícola de la zona Centro (Caldas, Cundinamarca, Boyacá y Tolima). El patrón dominante es A1 (45%), A6 (18%) y A4 (10%), además de abejas con haplotipos A2, A13 y A8 (11,6%), que alternan con abejas de linajes C y M (15%); la estructura genética en abejas del sur y suroccidente del país, igualmente presenta linaje predominantes africanos A1 (70,6%), A6 (17,6%), A4 y A9 (11,8%). Esta variabilidad se puede explicar si se tienen en cuenta las prácticas comerciales e intercambio de material genético entre los apicultores, quienes desarrollan su sistema productivo importando abejas reinas de otras regiones o capturando enjambres silvestres. La dispersión y grado de entrecruzamiento de la población analizada, presenta patrones similares a los estudiados por Collet, et al. (2006), para poblaciones de abejas de Brasil y Uruguay con mayor frecuencia para A1 al norte y sur de Brasil. Los resultados observados explican el comportamiento en la distribución altitudinal reportado por Salamanca et al. (2005), en su estudio morfométrico de abejas colombianas.
CONCLUSIONES
En el estudio del ADN mitocondrial de abejas de Apis mellifera colombianas, se ha encontrado una dominancia de haplotipos Africanos A (A1, A2, A4, A5, A6, A8, A9 y A13). De los 13 linajes identificados, 8 pertenecen a (A), 4 al Europeo M (M2, M4, M5 y M7) y 1 al Europeo del Este C (C1), que en términos porcentuales correspondió al 90,5; 7,6 y 1,9%, respectivamente. La dominancia del haplotipo Africano (A), sugiere más de un episodio de hibridación, introgresión y expansión del fenómeno de africanización en el territorio colombiano. El gradiente de distribución del linaje africano, fue representado principalmente por el haplotipo A1, (Costa Caribe Colombiana). Al Centro se evidencian los haplotipos A4 y A6, que enmarcaron a grupos de abejas con alguna dominancia. Los linajes europeos, no representan dominancia y no se vieron representados respecto de la población estudiada. Es necesario estimar el efecto de la composición genética de las poblaciones de abejas, ya, que la actividad apícola en Colombia esta por desarrollarse y demanda el estudio e implementación de procedimientos de selección de material biológico para el fortalecimiento de la producción y el aprovechamiento de los recursos naturales por parte de los apicultores conforme a las políticas del Estado y de las cadenas productivas.
AGRADECIMIENTOS
El autor desea expresar su gratitud a los apicultores colombianos de las zonas de estudio incluidas en el trabajo de campo por facilitar las condiciones para el muestreo. A la Agencia Española de Cooperación (AECI), por facilitar los recursos económicos para el desarrollo del trabajo en el Laboratorio de Departamento de Zoología y Antropología Física de la Facultad de Veterinaria de la Universidad de Murcia, A los Doctores Pilar de La Rúa, José Galián y Fernando Cánovas, por sus sugerencias en el proceso de valoración genética. A Mónica Patricia Osorio, investigador asociado de la Universidad del Tolima, por su colaboración. A los evaluadores del artículo y colaboradores permanentes de la revista Zootecnia Tropical, por las observaciones en el proceso de arbitraje. Al Grupo de Investigaciones Mellitopalinológicas y Propiedades Fisicoquímicas de Alimentos de la Universidad del Tolima.
LITERATURA CITADA
1. Amaya F. y E. Roldan. 1983. La abeja africanizada en la región de Lenguapá. Trabajo de grado. Facultad Medicina Veterinaria y Zootecnia. Universidad Nacional. Santafé de Bogotá. 123p. [ Links ]
2. Burgett M., S. Shorney, J. Cordara, G. Gardiol and W. S. Sheppard. 1995. The present status of Africanized honey bees in Uruguay. Am. Bee J., (135): 328-330. [ Links ]
3. Cánovas F., P. De la Rúa, J. Serrano y J. Galián. 2002. Variabilidad del ADN mitocondrial en poblaciones de Apis mellifera ibérica de Galicia (NW España). Arch. Zootec., (51): 441-448. [ Links ]
4. Cánovas F., P. De la Rúa, J. Serrano and J. Galián. 2008. Geographical patterns of mitochondrial DNA variation in Apis mellifera iberinensis (Hymenoptera: Apidae). Journal of Zoological Systematics and Evolutionary Research., 46(1): 24-30. [ Links ]
5. Castaño M., A. Morales y O. Moreno. 1979. Informe de reconocimiento de la situación apícola en la región de la intendencia de Arauca. ICA. Bogotá. Pp. 4-11. [ Links ]
6. Collet T., K. M. Ferreira, M. C. Arias, A. E. Soares and M. A. Del Lama. 2006. Genetic structure of Africanized honeybee populations (Apis mellifera L.) from Brazil and Uruguay viewed through mitochondrial DNA COI–COII patterns. Heredity (97): 329–335. [ Links ]
7. Cornejo L. G. 1978. Estúdio sobre mercadeo para mieles de abeja em Colombia. UNCTAD/GAT. Ministério de Agricultura. Colombia. Pp. 147- 168. [ Links ]
8. Cornejo L. G. 1993. Apicultura prática en America latina. Boletín 105. Servicios Agrícolas. FAO. Roma. Pp. 167. [ Links ]
9. Cornuet J. M., L. Garnery and M. Solignac 1991. Putative function of the intergenic region between COI and COII of Apis mellifera L. mitochondrial DNA. Genetics, (128): 393–403. [ Links ]
10. Crozier R. H., Y. Crozier and A. Mackinlay. 1989. The CO-I and CO-II region of honeybee mitochondrial DNA: evidence for variation in insect mitochondrial evolutionary rates. Molecular Biology and Evolution, (6): 699-411. [ Links ]
11. Crozier R. H. and Y. Crozier. 1993. The mitochondrial genome of the honeybee Apis mellifera: complete sequence and genome organization. Genetics, (133): 97-177. [ Links ]
12. Daly H. V. and S. Balling. 1978. Identification of the Africanized honeybee in the wester hemisphere by discrimination analysis. J. Kansas Entomol. Soc. 51 (4): 657-669. [ Links ]
13. DeGrandi-Hoffman G. 2001. Update on Africanized honey bee research. Proceedings of the American Bee Research Conference. Am. Bee J., (12):886. [ Links ]
14. Del Lama M.A., J.A. Lobo E., E. Soares A., S.N. Del Lama. 1990. Genetic differentiation estimated by isozymic analysis of Africanized honeybee populations from Brazil and from Central America. Apidologie., (21): 271-280. [ Links ]
15. Del Lama M. A., R. O. Souza A. A. Duran X. and E. E. Soares A. 2004. Clinal variation and selection on MDH allozymes in honeybees in Chile. Hereditas, Lund, Sweden., 140(2): 149-153. [ Links ]
16. De la Rúa P., J. Serrano, J. Galián. 1998. Mitochondrial DNA variability in the Canary Islands honeybees (Apis mellifera L.). Molecular Ecology., (7): 1543-1547.
17. De la Rúa P., J. Serrano, J. Galián. 1999. Variabilidad mitocondrial en poblaciones de abejas de la miel (Apis mellifera L.) de la región de Murcia. Invest. Agr. Prod. Sanid. Anim., (14): 41-50.
19. De la Rúa P., J. Serrano and J. Galián. 2002. Biodiversity of Apis mellifera populations from Tenerife (Canary Islands) and hybridization with East European races. Biodiversity and conservation., (11): 59-67. [ Links ]
20. De la Rúa P., J. Galián, J. Serrano, R. Moritz. 2003. Genetic estructure of Balearic honeybee populations based on microsatellite polymorphism. Genet. Sel. Evol., (35): 339-350.
21. Díaz M. 1981. Estudio morfométrico de la abeja Apis mellifica en la antigua provincia de las villas. Agrotecnia de Cuba., 13(2): 25-32.
22. Diniz Filho J., A.F., O. Malaspina. 1995. Abelhas africanizadas nos anos 90. Ciência Hoje. 18(106): 73-76.
23. Estoup A.L., M. Garnery, M. Solignac, J.M. Cornuet. 1995. Microsatellite variation in honey bee (Apis mellifera L.) populations: Hierarchical genetic structure and test of the infinite allele and stepwise mutation models. Genetics. (140): 679-695.
24. Franck P., L. Garnery, M. Solignac, J.M. Cornuet. 2000. Molecular confirmation of a fourth lineage in honeybees from the Near East. Apidologie, (31): 167-180.
26. Franky A. 2008. Producir polen es la mejor alternativa en las zonas de alta montaña tropical. In: Memorias IX Congreso Iberoamericano de Apicultura. Calidad y sanidad, desafíos para el desarrollo de una apicultura sustentable. Concepción Chile. Pp. 42. [ Links ]
27. Fisher A. 1985. Las reinas africanas. Boletín técnico. El CIID informa. 14 (3-4): 4-6. [ Links ]
28. Hall H. G. and K. Muralidharan. 1989. Evidence from mitocohondrial DNA that African honey bees spread as continuous maternal lineages. Nature. (339): 211-213. [ Links ]
29. Hall H.G. and D. R. Smith. 1991. Distinguinshing African and European honeybee matrilines using amplified mitochondrial DNA. Proc. Natl. Acad. Sci. USA. (88): 4548-4552. [ Links ]
30. Heras F. 1994. Estudios biométricos y enzimáticos de la abeja de la abeja de la miel (Apis mellifera Linnaeus, 1758. Hymenoptera, Apoidae. Universidad de Salamanca. Departamento de Biología Animal, Ecología, Parasitología, Edafología y Química Agrícola. Pp. 178. [ Links ]
31. Hernández G. R. 2002. Trashumancia y variabilidad genética de las poblaciones de Apis mellifera ibérica (Hymenoptera, Apidae) de la región de Murcia. Tesina Facultad de Veterinaria. Facultad de Zoología y Antropología Física. Universidad de Murcia. España. Pp: 50. [ Links ]
32. Garnery L., J. M. Cornuet and M. Solignac. 1992. Evolutionary history of the honey bee Apis mellifera inferred from mitochondrial DNA analysis. Mol. Ecol. (1): 145-154. [ Links ]
33. Garnery L., M. Solicnac, G. Celebrano and J. M. Cornuet. 1993. A simple test using restricted PCR-amplified mitochondrial DNA to study the genetic structure of Apis mellifera L. Experiencia. (49): 1016-1021. [ Links ]
34. Garnery L., E. H. Mosshine, B.P. Oldroyd y J. M. Cornuet. 1995. Mitochondrial DNA variation in Mooccan and Spanish honey bee populations. Molecular Ecology. (4): 465-471. [ Links ]
35. Kent R. 1976. Beekeeping regions and the beekeeping industry in Colombia. Bee world. 57(4): 151-158. [ Links ]
36. Kerr W. 1970. Reproduçao da abelha Africana, Italiana e suas híbridas. I Congresso Brasileiro de Apicultura. Florianópolis-SC. Pp: 130-135. [ Links ]
37. Kerr W. E., M. S. Blum, J. F. Pisani y A. C. Stort. 1974. Correlation between amounts of 2-heptanone and iso-amyl acetate in honey bees and their agressive behaviour. J. Apic. Res. (13): 173-176. [ Links ]
38. Kerr W. E. 1991. Abelhas africanas: Sua introdução e expansão nas Américas, suas subespecies e ecotipos africanos. In: XXXXII Congresso Nacional de Genética. Caxambú, MG, Brasil. Pp. 123-127. [ Links ]
39. Lobo S. J. A. 2000. Highly polymorphic DNA markers in Africanized honeybee populations in Costa Rica. Genetics and Molecular Biology. 23(2): 317-322. [ Links ]
40. Malaspina O. 1982. Análise do comportamento de coleta de alimento e morfometria em abelhas africanizadas, caucasianas e em descendentes dos seus cruzamentos. Doutorado em Ciências Biológicas (Zoologia). Universidade Estadual Paulista Júlio de Mesquita Filho, UNESP, Brasil. Pp. 125. [ Links ]
41. Mantilla C. 1997. Principios de apicultura africanizada. Universidad Nacional de Colombia. Facultad de Ciencias. Departamento de Biología. Ed. Universidad Nacional de Colombia. Pp. 214. [ Links ]
42. Molina A. 1979. La abeja africanizada: algunos aspectos sobre su origen, biología y manejo. In: Memorias VI Congreso Nacional de la Sociedad Colombiana de Entomología. Cali Colombia. Pp. 56-60. [ Links ]
43. Morales S.G. 1995. Estacionalidad Reproductiva de las colonias de abeja africanizada en distintas zonas de vida. In: III Seminario Internacional de apicultura. Santa Fe de Bogotá, D.C, Colombia. Pp. 71-77. [ Links ]
44. Núñez J.A. 2000. Foraging Efficiency and Survival of African Honeybees in the Tropics. Anais do IV Encontro sobre Abelhas, Ribeirão Preto. SP. Brasil. Pp. 9-16. [ Links ]
45. Osorio T. M.P., T.E. Osorio y G.G. Salamanca. 2002. Aspectos climáticos relacionados con la actividad apícola tropical. In: IX Congreso Internacional de Actualización Apícola. Zacatecas México. Pp. 66-68. [ Links ]
46. Prada C., M. Del Lama y G. G. Salamanca. 2005. Distribución y Perfil Altitudinal de poblaciones de abejas africanizadas en Colombia. In: I Congreso Internacional de Apicultores de los Andes. III. Convención de Apicultores de Venezuela. Universidad Nacional Experimental del Táchira. Pp. 126. [ Links ]
47. Quezada-Euán J. 2000. Hybridization between European and Africanized honeybees in tropical Yucatan, Mexico. II. Morphometric, allozymic and mitochondrial DNA variability in feral colonies. Apidologie 31 Pp. 443–454. [ Links ]
48. Quezada-Euán J., E Pérez-Castro and W. May- Itzá. 2003. Hybridization between European and African-derived honeybee populations (Apis mellifera) at different altitudes in Perú. Apidologie. (34): 217–225. [ Links ]
49. Rinderer T. E., B.P. Oldroyd and W. S. Sheppard. 1993. Africanized bees in the U.S. Sci. Am. 269: 52-58. [ Links ]
50. Roubik D. W. 1991 .Aspects of Africanized honeybee ecology in tropical America. In: The Africanized honey bee. Ed Spivak, M., Fletcher, D.J. and Bress M.C. San Francisco, CA. Westview Press. Pp. 259-281. [ Links ]
51. Ruttner F. 1988. Biogeography and taxonomy of honeybee. Springer-Verlag. Berlin. Alemania. Pp. 284. [ Links ]
52. Salamanca G. G., F. Londoño, P. F. Rivera y O. M. Zapata. 1998. Análisis morfométrico y grado de africanización de la abeja Apis mellifera en algunos municipios del Tolima. In: XXXIII Congreso Nacional de Ciencias Biológicas. Pp. 166 [ Links ]
53. Salamanca G. G. 2005. Estudio analítico comparativo de las propiedades fisicoquímicas de mieles de Apis mellifera, en algunas zonas apícolas de los departamentos de Boyacá y Tolima. Vol. I. Anales de las Tesis Doctorales Fruto de la Cooperación Interuniversitaria con Colombia. Universidad Politécnica de Valencia. España. Pp.143-158. [ Links ]
54. Salamanca G. G., T. M. Osorio, E. F. Vargas, J. Yate y R. García R. 2008. El sistema de ecorregiones biogeográficas colombianas y su estado apícola productivo. Memorias IX Congreso Iberoamericano de Apicultura. Calidad y sanidad, desafíos para el desarrollo de una apicultura sustentable. Concepción. Chile. Pp. 34 [ Links ]
55. Sheppard W.S., M. C. Arias, A. Greech and M. D. Meixner.1997. Apis mellifera ruttneri, a new honey bee subspecies from Malta. Apidologie (28): 287-293. [ Links ]
56. Sheppard W. S., T. E. Rinderer, L. Garnery, and H. Shimanuki. 1999. Analysis of Africanized honey bee mitochondrial DNA reveals further diversity of origin. Genet. Mol. Biol., 22: 73-75. [ Links ]
57. Scott S., D. DeGrandi-Hoffman and D. S. Roan. 2004. The african honey bee: Factors Contributing to a Successful Biological Invasion. Annu. Rev. Entomol., (49): 351–76. [ Links ]
58. Smith D.R. 1991. Mitochondrial DNA and honeybee biogeography, in: Smith D.R. (Ed.), Diversity in the Genus Apis. Westview Press and IBH Publ., Oxford. Pp: 131-176. [ Links ]
59. Walsh P. S., D. A. Metzqer and R. Higuchi R. 1991. Chelex 100 as a médium for simple extraction of DNA for PCR- based typing from forensic material. Biotechniques (10): 506-512. [ Links ]