Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Opción
versión impresa ISSN 1012-1587
Opcion v.24 n.56 Maracaibo ago. 2008
Alelos del complejo principal de histocompatibilidad clase II DRB1*/DQB1* de la población wayúu de la Guajira venezolana
Miriam Echeverría, Sergio Rivera, Manzur Hassanhi, Alba Fuenmayor, Georgina Márquez, Maczi González y William Zabala
Instituto Hematológico de Occidente (Banco de Sangre del estado Zulia). Venezuela E-mail: mcecheverria60@hotmail.com
Resumen
Con el propósito de establecer las frecuencias alélicas y haplotípicas de los antígenos HLA de clase II de la población wayúu de la Guajira venezolana, se estudiaron 53 individuos pertenecientes a ese grupo étnico, adultos de ambos sexos, no relacionados y ubicados en diferentes comunidades del municipio Páez del estado Zulia, Venezuela. El ADN de sangre periférica se amplificó utilizando la técnica de Reacción en Cadena de la polimerasa (PCR-SSP) y posterior hibridación con sondas secuencia específica (PCR-SSO), para identificar los loci DQB1* y DRB1.* Las frecuencias alélicas (fa) se calcularon con la fórmula fa = 2 x homocigotos + heterocigotos/2 x N; para el desequilibrio de enlace se aplicó D=Ö d/N - Ö b+d/N x c+d/N y X2. Las distancias genéticas fueron determinadas sobre la base de los datos previamente reportados de las poblaciones indígenas wayúu colombiana; barí, yucpa y warao de Venezuela, la caucásica de Madrid y la negra de Sao Paulo Brasil, según la fórmula Cos qn = åÖPi x Pj, d = Ö1 – cos qn. El alelo más frecuentemente observado en los wayúu venezolanos fue DRB1* 0411, con 19,54%, seguido del DRB1* 0403, con 18,75% y el DRB1* 1602, con 10,94%. La frecuencia más alta para el locus DQB1* fue el 0302, con 53,58%, seguido por el DQB1* 0301, con 27,14% y el DQB1* 0402, con 7,86%. Los haplotipos DRB1*0411-DQB1*0302 y DRB1* 1602-DQB1*0301 se mostraron en desequilibrio de ligamiento en la población wayúu venezolana. El dendograma muestra a la población wayúu venezolana con identidad propia, muy cercana a la población wayúu colombiana, seguida del par yucpa-barí del mismo estado Zulia, seguido, a su vez, de la población warao (ubicada al este de Venezuela). El bloque de las poblaciones indígenas se observa bien separado de los caucásicos españoles y de los negros de Sao Paulo, Brasil.
Palabras claves: HLA clase II, wayuú venezolanos, frecuencias alélicas, desequilibrio de enlace, distancia genética.
Mayor histocompatibility complex class II, DRB1*/DQB1* alleles of amerindian wayúu from Venezuela
Abstract
In order to establish the alelic and haplotypic frequencies of HLA of class II systems in Wayúu population of the Venezuelan Guajira 53 individuals of the Wayúu ethnic group studied of the Venezuelan Guajira, adult of both sexes, not related located in different communities from the Páez municipality of the state Zulia ,Venezuela. The DNA of peripheral blood was extracted , was amplified using the technique of Chain Reaction of Polimerasa (PCR) and later hibridation with sequences specific oligonucleotides (PCR-SSO), for the tipificación of loci DQB1* and DRB1*. The genetic distances were determined on the basis of the data previously reported of the indigenous populations Colombian Wayúu, Barí, Yucpa, Warao of Venezuela, the caucasian of Madrid and the black of Sao Paulo, Brazil, by Cos qn = åÖPi x Pj, d = Ö1 – cos qn. The allelic frequency (af) was determined by af = 2 x homozygous + heterozygous /2 x N. I stupefy more frequently observed in Venezuelan the Wayúu population was DRB1* 0411 with 19.54%, followed of DRB1* 0403 with 18.75% and DRB1* 1602 with 10.94%. The high frequency but for locus DQB1* was the 0302 with 53.58% followed by DQB1* 0301 with 27.14% and DQB1* 0402 with 7.86%. Haplotipos DRB1*0411-DQB1*0302 and DRB1* 1602-DQB1*0301 were in linkage disequilibria (D= Öd/N - Öb+d/N x c+d/N and X2 test) in Venezuelan the Wayúu population. The dendrograma shows Venezuelan the Wayúu population with own identity, very near colombian the Wayúu population, followed of the pair Yucpa - Barí of he himself Zulia state followed of the Warao population (this of Venezuela). The block of the indigenous populations is observed well separated of the Spanish and black caucasians of Sao Paulo, Brasil.
Key words: HLA class II, Venezuelan Wayúu, allelic frenquencies, linkage disequilibria, genetic distances.
Recibido: 17 de octubre de 2007 Aceptado: 25 de mayo de 2008
INTRODUCCIÓN
Venezuela es uno de los países de América con el mayor número de comunidades indígenas, después de Brasil, Colombia y México (Fernández-Viña et ál., 1991; Gorodesky et ál., 2001; Layrisse y Wilbert, 1966; Yunis et ál., 2001). Según el Censo Nacional 2001, la población indígena venezolana alcanza 534.816 habitantes, lo cual representa solo el 2,2% de la población total del país. De esos indígenas, 138.729 constituyen la población wayúu (57,5%), que mayoritariamente habita en el estado Zulia (Allais, 2004).
La población wayúu venezolana (constituida por los guajiros, palabra que traduce hombres de tierra) habita principalmente en las áridas regiones de la península de la Guajira, la cual se extiende hacia el mar Caribe a 11º N y 13º O. Una gran parte de este territorio pertenece políticamente a Colombia y la otra, a Venezuela, especialmente la franja sureste. Los guajiros, que hablan la lengua arawakan, tuvieron su asiento central en la cuenca amazónica, en el actual Brasil, y debido a fricciones interétnicas (por problemas internos) unidas al deseo de expansión se fragmentaron en varios grupos, uno de los cuales es el guajiro. Este emigró a la península Guajira, pasó a lo largo de la costa venezolana y por el lago de Maracaibo y estableció contactos con otros grupos dispersos en todas las regiones (Allais, 2004; Ariends, 1992; Layrisse y Wilbert, 1966; Semprún, 1993).
Son muchas las publicaciones que en Latinoamérica y en el mundo hablan sobre la tipificación de los antígenos de histocompatibilidad de clase I y II de las poblaciones indígenas (Gorodesky et ál., 1985; Bostulini et ál., 1992; Cerna et ál., 1993; Fernández-Viña et ál., 1991; García Ortiz et ál., 2006; Layrisse et ál., 1995; Mack and Erlich, 1998; Tokunaga et ál., 2001). En Venezuela se han estudiado las frecuencias antigénicas de las poblaciones indígenas barí, warao, yucpa, yanomami, makiritare, yekuana y otros (Guédez et ál., 1994; Layrisse, 1992; Layrisse et ál., 1994; Layrisse et ál., 1997; Olivo et ál., 1996).
Los indios wayúu de Colombia (también conocidos como guajiros), con una población estimada entre 40.000 y 80.000 individuos, están localizados en la península de la Guajira. Los estudios realizados por Yunis et ál. (1994) mostraron una alta frecuencia de los alelos DRB1*04, seguidos por los DRB1*1602,1402/1406,1501 y 0802. Las frecuencias más alta para los alelos DQB1* fueron 0301,0402 y DQB1* en los wayúu colombianos.
2. FUNDAMENTACIÓN TEÓRICA
Tradicionalmente los antígenos HLA clase I y II han sido detectados por métodos serológicos. Sin embargo, con el desarrollo de los métodos fundamentados en la reacción en cadena de la polimerasa (PCR), para la tipificación de los genes HLA clase II, la habilidad para detectar la variabilidad genética dentro de CMH se ha incrementado considerablemente.
Es conocido que la América fue poblada por olas de migración que comenzaron hace 20.000 o 40.000 años y que la población amerindia se estableció definitivamente en Suramérica hace 10.000 o 12.000 años. Varias hipótesis han sido desarrolladas para explicar el origen de la población indígena en Suramérica. La ruta de entrada terrestre sugiere una migración a través de la jungla de Darién, entre Panamá y Colombia. Una segunda posibilidad es la ruta marítima desde Florida hasta Suramérica, a través de las islas del Caribe. Una tercera posibilidad es la existencia de una ruta a través del Pacífico, que llega hasta las costas peruanas. Adicionalmente, una segunda ola de migraciones hacia Suramérica ha sido postulada. Esta incluye la clásica ruta a lo largo de las costas del Pacífico y los Andes y olas de migración originadas en el este del Amazonas y directamente a través de Venezuela, Colombia y Guyana (Schmitz, 1983; Yunis et ál., 1994; Crawford, 1998).
La identificación de las moléculas HLA clase II en las poblaciones amerindias ha sido utilizada para estudios antropológicos. Los resultados obtenidos han permitido conocer características propias y exclusivas de los antígenos HLA- DRB1*/DQB1*, algunos de los cuales se encuentran presentes con las mismas características en varias poblaciones indígenas, muy distantes entre sí, de Norteamérica y Suramérica, como los warao de Venezuela y los mapuches de Argentina. Gracias a estas características comunes se han podido determinar posibles rutas migratorias entre las mencionadas poblaciones (Layrisse, 1992; Statsny et ál., 1991).
Nuevas variantes alélicas HLA clases I y II han sido identificadas en indios Suramericanos, lo cual ha aportado información relevante para la genética de poblaciones, asociación HLA y enfermedades y estudios de trasplante (Belich et ál., 1992).
El propósito de este trabajo es determinar la frecuencia de los alelos y haplotipos HLA clase II de la población wayúu de la Guajira venezolana, por diversos métodos de Biología molecular, y compararla con otras poblaciones, tales como amerindias, indígenas venezolanas (Guédez et ál., 1994; Layrisse et ál., 1997; Olivo et ál., 1996), wayúu colombiana (Yunis et ál., 1994), negra de Sao Paulo, Brasil (Louzada-Junior et ál., 2001) y caucasoide de España (Vicario et ál., 1996) con el objeto de evaluar su propia identidad y los estudios antropológicos tendientes a dilucidar las hipótesis de las migraciones de las poblaciones indígenas hacia el continente Suramericano.
3. METODOLOGÍA
Se procesaron 53 muestras de sangre periférica de individuos pertenecientes a la etnia wayúu de la Guajira venezolana, adultos de uno u otro sexo, no relacionados que habitan en diferentes comunidades del municipio Páez, como Paraguaipoa, Cojoro, Pochoure, Polomar y Castillete, todas del estado Zulia, Venezuela. Un cuestionario previo permitió realizar el análisis antropológico y determinar el grado de relación filogenética de la población evaluada.
La extracción del ADN se realizó por la técnica de Salting-out, previamente descrita por Millar et ál. (1987). La amplificación del ADN mediante la Reacción en Cadena de la polimerasa (PCR) se hizo con iniciadores específicos, seguido por la hibridación con sondas oligonucleotídicas de secuencia específica marcadas, y revelado enzimático denominada técnica PCR-SSOP (Lindel y col., 1997).
Se emplearon los reactivos recomendados en el IV Taller Latinoamericano de Histocompatibilidad (Sao Paulo, 1994), donde se trabajó con la técnica Eldot (Enzyme-Linked DNA Oligotyping), descrita por Lázaro et ál. (1993). Los primers usados para las amplificaciones por locus o grupos específicos por este método fueron: El DR genérico (Drama, PSP49 con biotina), DR1 alelo específico ( PSP 43 y PSP 49 con biotina), DR2 DRb1 (PSP 68, PSP 49 con biotina), DR2 DR b5 (PSP67, PSP49 con biotina), el DR4 alelo específico (PSP 25, PSP 49 con biotina), DRw52 DR b1 (PSP 26, PSP 49 con biotina), DRw52 DR b3 (PSP 37, PSP49 con biotina) DQ b1 (PQP 25, PQP 27) (Lázaro et ál., 1993). El producto de amplificación con cebadores biotinilados se inmovilizó en pozos de microplacas fondo plano contentivas de Avidina. Las sondas sintetizadas con una cola Poly A fueron hibridizadas en cada uno de los pozos para luego ser reveladas con un procedimiento enzimático cuyo procedimiento fue descrito por Statsny et ál. (1991). Se utilizaron 17 sondas para el DRB1* genérico, 18 para el DQB1*, 26 para el DR52-DRB1*, 9 para DR4-DRB1* y 6 para DR2-DRB1*, cuyas especificidades fueron previamente publicadas por el mismo autor.
Las pruebas comerciales Elpha (Enzyme Linked Probe Hybridization Assay), Biotest, Cantype (Dot-Blot) de la Cruz Roja (Cantype 1996), DRB-SSOP typing kit Dot-Blot (Biotest 1997) e iniciadores secuencia específica de alta resolución (séquense specif primers kit higth resolution PCR-SSP C-six), se utilizaron para completar o confirmar la tipificación de algunos de los subtipos DRB1*, DRB3*, DRB4* y DQB1*.
4. ANÁLISIS ESTADÍSTICO
Las frecuencias alélicas (fa) se calcularon con la formula fa = 2 x homocigotos + heterocigotos/2 x N; para el desequilibrio de enlace se aplicó D=Öd/N - Öb+d/N x c+d/N y X2. (Mattiuz et ál., 1970). Las distancias genéticas fueron determinadas sobre la base de los datos previamente reportados de las poblaciones indígenas wayúu colombiana, barí, yucpa y warao de Venezuela, la caucásica de Madrid y la negra de Sao Paulo, Brasil, según la fórmula Cos qn = åÖPi x Pj, d = Ö1 – cos qn. (Cavalli-Sforza & Bodmer, 1971).
Las pruebas estadísticas utilizadas incluyen técnicas de distribución unidimensional, cálculo de proporciones, media aritmética y tabla de contingencia. Además se utilizó estadística no paramétrica para establecer las diferencias existentes entre los antígenos dentro de la misma población y en comparación con las poblaciones relacionadas (Sydney, 1972). La corrección de p se realizó utilizando la fórmula siguiente: Pc= 1-2 (1-p) n. En esta, n es el número de casos recogidos, independientemente usados en una prueba estadística (Goodman, 1969).
4.1. Distancia genética
La distancia genética y los árboles filogenéticos son parámetros que miden el grado de diferenciación génica existente entre poblaciones, así como también el tiempo de separación entre ellas. En este sentido, se construyó un árbol filogenético con el método de pares de grupos no ponderados a través de promedios aritméticos (Upgma- Unweighted Pair-Group Method using Arithmetic Averages), para visualizar las relaciones evolutivas entre las poblaciones consideradas para este estudio, utilizando la matriz de distancias genéticas DA, con los valores de frecuencia de DRB1* y DQB1* obtenidos de la población wayúu venezolana y los reportados en la literatura para las poblaciones wayúu colombiana (Yunis et ál., 1994), barí (Guédez et ál., 1994), warao (Olivo y col., 1993), yucpa (Layrisse et ál., 2001) y caucásicos españoles (Vicario y col., 1996) y negros de Sao Paulo (Louzada-Junior et ál., 2001). Tanto la matriz de distancias genéticas DA como el árbol filogenético se calcularon con el programa estadístico computarizado Dispan (Nei & Roychoudthurg, 1974), utilizando solo la frecuencia de aquellos alelos presentes en las poblaciones estudiadas.
5. RESULTADOS
El análisis molecular de la población wayúu revela que el alelo con más frecuencia es el DRB1*0411, con 19,54%, seguido del DRB1* 0403, con 18,75% y el DRB1*1602, con 10,94%. En la tabla 1: la frecuencia más alta para el locus DQB1* fue el 0302, con 53,58%, seguido por el DQB1* 0301, con 27,14% y el DQB1*0402, con 7,86%.
Tabla 1 Frecuencias alélicas DRB1*DQB1* de la población wayúu de la Guajira y otras poblaciones
| DQB1* | Wayúu venezolana | Wayúu colombiana Yunis et ál., 1994 | Yucpa Layrisse et ál., 2001 | Barí Layrisse et ál., 1995 | Warao Olivo, 1996 | Madrid Vicario, 1996 | Negros Sao Paulo Louzada et ál.. 2001 |
| 1303 | 0 | 0 | 0 | 0 | 0 | 0,014 | 0,011 |
| 1305 | 0 | 0 | 0 | 0 | 0 | 0,0059 | 0 |
| 1401 | 0 | 0,0178 | 0 | 0 | 0 | 0,0253 | 0,032 |
| 1402/1406 | 0,0156 | 0,08 | 0,0455 | 0,018 | 0,2955 | 0,0005 | 0 |
| 0801 | 0 | 0 | 0 | 0 | 0 | 0,0258 | 0,021 |
| 0806 | 0 | 0 | 0 | 0 | 0 | 0,0022 | 0 |
| 0802 | 0,0782 | 0,0535 | 0 | 0,009 | 0,1364 | 0,0005 | 0 |
| 0804 | 0 | 0,0089 | 0 | 0 | 0 | 0 | 0,045 |
| 0803 | 0 | 0 | 0 | 0 | 0 | 0,0016 | 0 |
| 0810 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| 0807 | 0,0078 | 0 | 0,125 | 0 | 0 | 0 | 0 |
| 0811 | 0,0078 | 0 | 0 | 0 | 0 | 0 | 0 |
| 0401 | 0,0234 | 0,0089 | 0 | 0 | 0 | 0,0215 | 0 |
| 0402 | 0 | 0,0089 | 0 | 0 | 0 | 0,0215 | 0,011 |
| 0403 | 0,1875 | 0,117 | 0,1136 | 0,036 | 0 | 0,0156 | 0,011 |
| 0404 | 0,0234 | 0,0359 | 0 | 0 | 0 | 0,0307 | 0,011 |
| 0405 | 0 | 0 | 0 | 0 | 0 | 0,0242 | 0 |
| 0407 | 0,0859 | 0,1072 | 0,0228 | 0,273 | 0 | 0,0091 | 0 |
| 0408 | 0 | 0 | 0 | 0 | 0 | 0,0022 | 0 |
| 0411 | 0,1954 | 0,1339 | 0,6136 | 0,255 | 0,1591 | 0 | 0,021 |
| 0417 | 0,0078 | 0 | 0 | 0 | 0 | 0 | 0 |
| 0409 | 0 | 0 | 0 | 0 | 0 | 0,0005 | 0 |
| 0701 | 0,0156 | 0 | 0 | 0 | 0 | 0,1566 | 0 |
| 0901 | 0 | 0 | 0 | 0 | 0 | 0,0102 | 0,021 |
| 1001 | 0 | 0 | 0 | 0 | 0 | 0,0168 | 0,043 |
| 0302 | 0 | 0 | 0 | 0 | 0 | 0 | 0,064 |
| 03 | 0,0156 | 0,0267 | 0 | 0 | 0 | 0 | 0 |
| 0301 | 0 | 0 | 0 | 0 | 0 | 0 | 0,074 |
| 04 | 0,1328 | 0,0357 | 0 | 0 | 0 | 0 | 0 |
| 06 | 0 | 0,0178 | 0 | 0 | 0 | 0 | 0 |
| 0406 | 0 | 0 | 0 | 0 | 0 | 0,0022 | 0 |
| 08041 | 0 | 0 | 0 | 0 | 0 | 0,0032 | 0 |
| 7 | 0 | 0,0267 | 0 | 0 | 0 | 0 | 0,119 |
| DQB1* | Wayúu venezolana | Wayúu colombiana Yunis et ál., 1994 | Yucpa Layrisse et ál., 2001 | Barí Layrisse et ál., 1995 | Warao Olivo, 1996 | Madrid Vicario, 1996 | Negros Sao Paulo Louzada et ál.. 2001 |
| 0501 | 0,0071 | 0 | 0 | 0 | 0,134 | 0,191 | |
| 0502 | 0,0071 | 0 | 0 | 0 | 0 | 0,0237 | 0,049 |
| 0503 | 0 | 0,0295 | 0 | 0 | 0 | 0 | 0,027 |
| 0602 | 0,0357 | 0,0889 | 0 | 0 | 0 | 0,0802 | 0,138 |
| 0603 | 0,0143 | 0,0087 | 0 | 0 | 0 | 0,0689 | 0,066 |
| 0604 | 0,0143 | 0,0538 | 0 | 0 | 0 | 0,0339 | 0,018 |
| 0609 | 0 | 0 | 0 | 0 | 0 | 0 | 0,018 |
| 0201 | 0,0357 | 0,035 | 0 | 0 | 0 | 0,2788 | 0,1065 |
| 0202 | 0 | 0 | 0 | 0 | 0 | 0 | 0,1065 |
| 0302 | 0,5358 | 0,421 | 0,7386 | 0,579 | 0,1591 | 0,0936 | 0,045 |
| 0301 | 0,2714 | 0,2667 | 0,1364 | 0,421 | 0,7045 | 0,1749 | 0,128 |
| 0304 | 0 | 0 | 0 | 0 | 0 | 0,0032 | 0 |
| 03032 | 0 | 0 | 0 | 0 | 0 | 0,0307 | 0 |
| 0401 | 0 | 0 | 0 | 0 | 0 | 0,0005 | 0,048 |
| 0402 | 0,0786 | 0,0964 | 0,125 | 0 | 0,1364 | 0,034 | 0,048 |
| 0605 | 0 | 0 | 0 | 0 | 0 | 0,0075 | 0 |
| 0601 | 0 | 0 | 0 | 0 | 0 | 0,0124 | 0,011 |
| 05031 | 0 | 0 | 0 | 0 | 0 | 0,0221 | 0 |
| 0305 | 0 | 0 | 0 | 0 | 0 | 0,0016 | 0 |
| 64 | 114 | 13 | 57 | 22 | 929 | 47 |
En la misma tabla 1 se comparan estadísticamente con los datos publicados de los alelos HLA clase II de los 114 wayúu colombianos (Yunis et ál, 1994), 57 indígenas barí (Guédez et ál., 1994), 22 indígenas warao (Olivo et ál., 1993), 13 indígenas yucpa (Layrisse et ál., 2001), 929 caucásicos españoles (Vicario y col., 1996) y 47 negros de Sao Paulo (Louzada-Jounior et ál., 2001), las frecuencias de los 64 individuos no relacionados wayúu venezolanos, y se observaron varias diferencias significativas que se mantuvieron luego de aplicarles la fórmula de la p corregida (Goodman, 1969).
En la tabla 2 se observan los haplotipos en desequilibrio de ligamiento. El haplotipo más frecuente en la población wayúu fue el DRB1* 0411/DQB1*0302, con (18,24%), seguido por el haplotipo DRB1* 1602/DQB1*0301, con (6,59%) y el DRB1* 0802/DQB1* (3,69%). El haplotipo que más caracteriza a la población wayúu venezolana es el 0411/0302, seguido del 1602/0301.
Tabla 2 Haplotipos en desequilibrio de ligamiento de la población wayúu de la Guajira venezolana
| Haplotipos | ||||
| DRB1* | *DQB1 | D | Frecuencia de haplotipos | P |
| 03 | 0502 | 0.005 | 0.004 | .005 |
| 03 | 0201 | 0.23 | 0.24 | .005 |
| 03 | 0603 | 0.24 | 0.24 | .005 |
| 0411 | 0302 | 5.11 | 18.24 | .005 |
| 0802 | 0402 | 2.3 | 3.27 | .005 |
| 0802 | 0502 | 0.44 | 0.54 | .005 |
| 0802 | 0301 | 1.41 | 0.90 | .005 |
| 0807 | 0402 | 0.45 | 0.55 | .005 |
| 0811 | 0402 | 0.45 | 0.55 | .005 |
| 1105 | 0301 | 0.823 | 1.29 | .005 |
| 1402 | 0604 | 0.468 | -0.49 | .005 |
| 1406 | 0604 | 0.047 | -0.49 | .005 |
| 1501 | 0602 | 1.39 | 1.523 | .005 |
| 1602 | 0301 | 3.580 | 6.59 | .005 |
P<0.05
En la tabla 3 se observa la comparación de haplotipos más frecuentes en wayúu con los publicados de otras poblaciones indígenas de Venezuela.
Tabla 3 Comparación haplotipos más frecuentes en wayúu con otras poblaciones indígenas venezolanas
| Haplotipos | WV | WC | YU | BA | WA | |
| DRB1* | *DQB1 | % | % | % | % | % |
| 0411 | 0302 | 18.24 | 13.11 | 60.23 | 25 | 12.5 |
| 1602 | 0301 | 6.59 | 21.31 | 7.95 | 39.7 | 36 |
| 0411 | 0402 | 0 | 8.19 | 0 | 0 | 0 |
| 1501 | 0602 | 1.52 | 0 | 0 | 0 | 0 |
| 1105 | 0502 | 1.29 | 0 | 0 | 0 | 0 |
| 0403 | 0302 | 0 | 13.11 | 12.5 | 3.5 | 0 |
| 0407 | 0302 | 0 | 11.47 | 2.27 | 30.2 | 0 |
| 0807 | 0402 | 0.55 | 0 | 12.5 | 0 | 0 |
| 1402 | 0301 | 0 | 1.63 | 4.55 | 1.7 | 27.8 |
| 0802 | 0402 | 3.27 | 8.19 | 0 | 0.9 | 22.99 |
WV = wayúu venezolano
WC = wayúu colombiano
YU = yucpa
BA = barí
WA = warao
El grado de homocigosis fue del 40,74%, y las combinaciones haplotípicas homocigotas más frecuentes son la DRB1*0403/ DQB1*0302, seguido por la DRB1* 0411/DQB1* 0302.
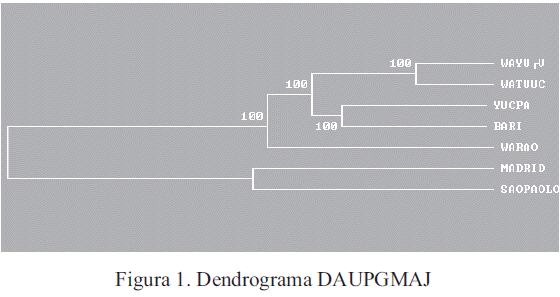
La distancia genéticas de los wayúu de la Guajira venezolana respecto de los wayúu colombianos, barí y warao, así como también con las poblaciones caucásicas de España y negros de Sao Paulo, Brasil, se muestran en la tabla 4. El árbol DAUPMGAJ agrupa a los indígenas separadamente de españoles y negros. Los wayúu de Colombia y Venezuela siempre están más cerca entre sí, pero no tanto de los otros indígenas del Zulia, como se esperaría por la poca distancia geográfica que los separa. El par yucpa-barí se separa del par wayúu venezolano-wayúu colombiano a una distancia menor que la observada en relación con los indígenas warao del Oriente del país. Finalmente, el bloque de las poblaciones indígenas se retira de manera notoria y significativa del par Madrid-Sao Paulo. El dendograma DAUPGMAJ correspondiente se muestra en la figura 1.
Tabla 4 Distancia genética obtenida usando frecuencias alélicas HLA – DRB1* – DQB1* en las poblaciones wayúu venezolana (1), wayúu colombiana (2), yucpa (3), barí (4), warao (5), Madrid (6), Sao Paulo (7)
|
| 1 | 2 | 3 | 4 | 5 | 6 |
| 2 | 0.0903 | |||||
| 3 | 0.1960 | 0.2626 | ||||
| 4 | 0.1958 | 0.2258 | 0.1796 | |||
| 5 | 0.3041 | 0.2844 | 0.3040 | 0.2167 | ||
| 6 | 0.4712 | 0.4136 | 0.7208 | 0.6920 | 0.7027 | |
| 7 | 0.5608 | 0.4190 | 0.7032 | 0.7055 | 0.6881 | 0.3008 |
6. DISCUSIÓN
El análisis de los alelos HLA de clase I y II de la población wayúu de la Guajira venezolana proporciona una información de gran valor para la conformación genética, porque asienta las bases para esclarecer el origen y posibles rutas migratorias de esta población.
Los alelos HLA clase II más frecuentes de la población wayúu de la Guajira venezolana son el DRB*0411 y el DQB1*0302; ambos son igualmente frecuentes en la misma población indígena del lado colombiano, con la cual no se observaron diferencias significativas. Sin embargo, cabe destacar que en la población wayúu venezolana se observaron en muy bajas proporciones los alelos DRB1* 0417, 0807, 0811 y el 1105, ausentes en los wayúu colombianos, barí y warao (Guédez et ál., 1994; Vicario y col., 1996; Yunis et ál., 1994). Estos alelos podrían marcar la diferencia entre estas poblaciones indígenas con respecto a los wayúu.
En relación con las otras poblaciones con las cuales se compararon las frecuencias alélicas de los wayúu venezolanos, se observa de manera interesante que los alelos DRB1* 0401 y DRB1*0404 solamente son compartidos por las poblaciones wayúu (venezolanos y colombianos) y caucásicos españoles, y están ausentes en negros de Sao Paulo. Por ello se puede inferir que estos alelos son de origen caucásico, producto de la mezcla de los españoles con los indígenas wayúu suscitada durante la época de la conquista de América y favorecida por la situación geográfica de la península de la Guajira, donde se asientan estas poblaciones indígenas. El DRB1* 03 y el DRB1*1301/1302 son alelos presentes en las poblaciones wayúu, y son igualmente compartidos con los caucásico españoles y negros. El DRB1*1501 es un alelo que se observa también y de manera exclusiva en Madrid, wayúu y negros. Por otra parte, los alelos DRB1*0401, *0404 y *1501 de las poblaciones wayúu colombiana y venezolana son compartidos con la población caucásica de España, pero se encuentran ausentes en los negros de Sao Paulo. El conjunto de estos datos evidencian la posible mezcla de los wayúu venezolanos y colombianos con esas dos poblaciones, lo cual los diferencia del resto de las poblaciones indígenas venezolanas.
El DRB1* 0411 y el DRB1*1602 son alelos que caracterizan la población indígena nacionalmente y no están presentes en la población caucásica de España ni en los negros de Sao Paulo. Las poblaciones indígenas del estado Zulia (wayúu, barí y yucpa), en Venezuela, comparten los alelos DRB1*0403 y *0407, los cuales tampoco se han reportado en la población indígena warao del Oriente del país. Estos alelos aparecen pobremente representados en las poblaciones caucásica española y en la negra de Sao Paulo; ambas diferencias son altamente significativas, pues marcan la distancia entre los indígenas del Oriente y del Occidente del país, respectivamente, y con el resto de las poblaciones caucásica y negra.
El alelo 1402/1406 es un antígeno diferencial con los wayúu colombianos, con una frecuencia del 8,0%, estadísticamente diferente de la observada en los wayúu venezolanos (1,88%). En los indígenas barí, su frecuencia es igualmente baja (1,8%); se muestra relativamente bajo en yucpa (4,55%), pero sumamente elevado en los indígenas warao (29,55%), (Vicario, 1996). Esto pudiera reflejar algún contacto de la población wayúu colombiana con los warao, en comparación con los wayúu venezolanos, los cuales recibieron aparentemente mayor influencia de las poblaciones caucásica y negra (Rivera y col., 1998). Este alelo 1402/1406 está prácticamente ausente en los caucásicos españoles y en los negros.
El alelo DRB1* 0417 está presente solamente en la población wayúu venezolana, pero su bajo porcentaje no identifica a esta población; de la misma manera, los alelos DRB1*0811, DRB1* 1105 están presentes en proporciones muy bajas. El alelo DRB1*0417 fue descrito por Shaohua et ál. en 1993 como un nuevo alelo en las tribus toba, mataco y toba pilaga, de la región Gran Chaco, en el noreste de Argentina. En estas mismas poblaciones se observó, además, el alelo DRB1*0411 con la más alta frecuencia, al igual que ocurre en los indígenas wayúu venezolanos. El alelo DRB1*0417, originario de las tribus indígenas de Sudamérica, pudiera ser una variante del *0411 (Cerna et ál., 1993).
El alelo DRB1*0807 fue descubierto recientemente en la población yucpa, ubicada también en la línea fronteriza entre Colombia y Venezuela (Layrisse et ál., 2001); igualmente fue descrito en la población indígena ticuna de Brazil (Mack & Erlich, 1998). Este alelo y el DRB1*0811 fueron descritos en las poblaciones indígenas amerindias navajo y tlingit (Williams et ál., 1994). El DRB1*0807 se generó como consecuencia de la entrada de los humanos en Suramérica. La generación en las Américas de los nuevos alelos DRB1*08, tales como el 0807 y 0811, sugiere que la ruta de evolución del DRB1* en la población nativa americana es rápida, lo que indica probablemente que fuerzas similares a las responsables de la rápida tasa de evolución observada para los alelos de CPH clase I, especialmente el locus B, en otras poblaciones nativas suramericanas (Belich et ál., 1992), pudieran actuar de la misma manera sobre el locus DRB1*. El DRB1*0802 es el más frecuente DRB1*08 de todas las poblaciones indígenas zulianas, y es el más frecuente también en todas las poblaciones indígenas que habitan el norte y el sur de los países americanos (Cerna et ál., 1993; Layrisse et ál., 1997; Mack & Erlich, 1998).
Es evidente que la población wayuú de la Guajira venezolana exhibe como alelos HLA DRB1* más frecuentes los DRB1*1602, 0403 y 0411, seguidos de los DRB1*0802 y 0407, propios de la mayoría las poblaciones amerindias previamente evaluadas. Los alelos DRB1* 1501 y 0701 son, evidentemente, producto de la mezcla de la población wayuú venezolana inicial con la población caucásica colonizadora.
Cerna et ál. muestran las frecuencias génicas HLA DRB1* de cinco poblaciones, cuatro de ellas indígenas suramericanas, una indígena norteamericana y caucásicos norteamericanos, en las cuales se pueden definir dos grupos indígenas suramericanos cuya distribución de frecuencias alélicas es francamente diferente: los xavantes del centro de Brasil y las poblaciones indígenas del área del Gran Chaco, al noroeste de la Argentina (toba del este, toba pilaga del oeste, y mataco-wichi). El alelo DRB1*1602 se encuentra altamente frecuente solo en los xavantes y no en el resto de las poblaciones mencionadas, mientras que todas comparten los DRB1* 1402 y 0802. Estos tres alelos se encuentran presentes, altamente frecuentes, en la población indígena norteamericana. Los autores reportan varias razones para estas diferencias y similitudes. Ambas pudieran relatar el origen de las tribus suramericanas y las diferentes vías de migración: unas transitaron a lo largo de las costas del Pacífico y los Andes, y posiblemente también a través del Pacífico, provenientes de Oceanía; otra estaría dibujada por el este, a través del alto y medio Amazonas.
En la población indígena wayuú venezolana, la presencia de los alelos HLA DRB1* 1602, 0407 y 0802 refuerza la hipótesis de la existencia de una ruta migratoria terrestre vía Panamá y Colombia, proveniente de las tribus de Norteamérica, donde estos alelos aparecen con una alta frecuencia génica. Igualmente, la existencia de dichos alelos en los indígenas xavantes de Brasil orienta su procedencia desde esta misma ruta migratoria. Por otra parte, el alelo HLA DRB1* 0411, con muy importante frecuencia génica en la población wayuú venezolana, está ausente en los indígenas xavantes y norteamericanos, pero se encuentra reportado en las tres tribus indígenas argentinas antes mencionadas. Esto revela posiblemente un aporte genético de alelos provenientes de la ruta Pacífica transoceánica, de aborígenes australianos. La presencia del alelo HLA DRB1*0417, aun cuando aparece con baja frecuencia génica en la población wayuú venezolana, refuerza esta última teoría. A la luz de estos resultados, la población wayúu venezolana constituiría una posible encrucijada para el resto de las poblaciones amerindias suramericanas, con una primera oleada migratoria proveniente del norte y otra segunda originaria del Pacífico.
El alelo DQB1* más frecuente en wayúu fue el DQB1*0302, seguido del 0301 y 0402. Se pudo observar que los alelos DQB1*0201, DQB1*0501, DQB1*0502 y DQB1*0602 están presentes solamente en las poblaciones wayúu, caucásica española y negros de Sao Paulo. El DQB1*0301 está alto en todas las poblaciones indígenas. El DQB1*0302 está presente en porcentajes altos en todas las poblaciones indígenas, y bajos en negros y caucásicos. El DQB1* 0402 está ausente únicamente en la población barí; el DQB1* está presente en wayúu y caucásica española, y su más alto porcentaje se observa en negros de Sao Paulo.
Ciertos genes que caracterizan a las poblaciones amerindias de Norteamérica y Suramérica, entre ellos a los tarahumara de México (García-Ortiz y col., 2006), han permitido establecer enlaces que confirman el origen ancestral de los wayúu venezolanos, quienes comparten cuatro de los principales alelos que forman el grupo de genes indígenas de esta población, como son el DRB1*0411, DRB1*1402, DQB1* 0402 y DQB1*0302.
En las poblaciones wayúu colombiana y venezolana se encuentran presentes la mayoría de los alelos DQB1* reportados para las poblaciones caucásicas y negras. El alelo DQB1*0302 es el más frecuente en wayúu y en todas las poblaciones amerindias de Venezuela. Sin embargo, llama la atención que los indígenas barí solo reportan los alelos DQB1*0301 y 0302 (Guedez et ál., 1994). En yucpa y warao se reporta, además, el DQB1*0402. Esta es otra razón más para indicar que el grado de mezcla de esta población wayúu con caucásicos y negros es mayor que la del resto de las poblaciones indígenas de Venezuela.
Los haplotipos DRB1*0411-DQB1*0302 y DRB1*1602-DQB1*0301, en desequilibrio de ligamiento en la población wayúu venezolana, fueron compartidos con todas las poblaciones indígenas de Venezuela. Sin embargo, la combinación DRB1*1501-DQB1*0602 y DRB1*1105-DQB1*0502 se observó exclusivamente en wayúu venezolanos y no así en el resto de las poblaciones indígenas evaluadas. El haplotipo DRB1*0411-DQB1*0402 se reportó únicamente en wayúu colombianos. El haplotipo DRB1*1402-DQB1*0301 se encuentra reportado en todas las poblaciones indígenas, con excepción de los wayúu venezolanos. Este resultado refuerza la individualidad propia de la población wayúu venezolana.
El porcentaje de homocigocis de la población wayúu venezolana fue calculado en 40,74%, propio de poblaciones indígenas, mucho más elevado que en la población mestiza zuliana de Venezuela, el cual es de 6,88% (resultados no publicados). Este factor podría dar cuenta de un efecto fundador o de entrecruzamiento en la población indígena wayúu, extensivo a otras poblaciones indígenas de Venezuela y al resto del continente americano, y sería responsable de un polimorfismo restringido de los alelos HLA.
De la misma manera como lo expresa la distribución de los alelos y haplotipos antes mencionados, el dendograma elaborado a partir de los datos de las frecuencias alélicas estudiadas y reportadas de las regiones DRB1* y DQB1*, nos indica que las poblaciones indígenas wayúu venezolana y colombiana aparecen muy cerca entre sí y separadas del resto de las poblaciones indígenas zulianas barí y yucpa. Los indígenas warao, del Oriente del país, se separan aún más indicando mayores diferencias. El par Madrid- Sao Paulo se separa considerablemente del resto de las poblaciones indígenas.
El estudio de la distribución de las frecuencias alélicas en la población indígena wayúu de la Guajira venezolana, permitió identificar alelos propios de esta población, los cuales la diferencian del resto de las poblaciones amerindias de Venezuela y del continente americano.
7. CONCLUSIONES
1 La población venezolana y colombiana comparten la mayoría de sus alelos y haplotipos, y no existen diferencias significativas entre las dos poblaciones indígenas. Sin embargo, algunos alelos se observan muy aumentados en una o muy disminuidos en la otra, lo cual les confiere características propias a cada una de las poblaciones indígenas wayúu.
2. Se observaron diferencias y semejanzas con otras poblaciones indígenas de Venezuela: barí, warao y yucpa, lo que sugiere posibles orígenes comunes.
3. Se observó que en la población wayúu están presentes alelos pertenecientes a las poblaciones caucásica y negra, lo que es indicativo de varios grados de mezcla.
4. La presencia de alelos HLA clase II comunes en las poblaciones indígenas norteamericanas, mexicanas, brasileras y argentinas en la población wayúu venezolana, y la distribución selectiva de sus alelos hacia una u otra de las poblaciones indígenas de Suramérica, sitúan a los wayúu en una encrucijada entre las rutas migratorias del norte de América y transoceánicas del Pacífico.
Referencias documentales
1. ALLAIS, M.L. 2004. La población indígena de Venezuela según los censos nacionales. Cambio demográfico y desigualdad social en Venezuela al inicio del tercer milenio. 22-13. [Recuperado el 27-6-2006]. Http:// www.ucab.edu.ve/eventos/iiencuentropoblacional/ponencias/Allais.pdf [ Links ]
2. ARENDS, T. 1992. Estructura genética de la población indígena de Venezuela. Universidad de las Naciones Unidas. Caracas, Venezuela. Trabajo de incorporación a la Academia Nacional de Medicina. [ Links ]
3. BELICH, M.P.; MADRIGAL, J.A.; HILDEBRAND, W.H. (1992). Unusual HLA-B alleles in two tribes of Brazilian indians. Nature, 357: 326-329. [ Links ]
4. BOSTULINI, M.C., WEINWE, T.A., FRANCO, M.H.L.P., SALAZANO, F.M., LAYRISSE, Z., SCHNEIDER, H. and SCHNEIDER, M.P.C. (1992). Genetic studies in three South American black populations. Gene Geog, 6: 1-16. [ Links ]
5. CAVALLI-SFORZA, L.L. & BODMER, W.F. (1971). The genetic of human population. Freeman. San Francisco, USA. [ Links ]
6. CERNA, M., FALCO, M., FRIEDMAN, H., RAÍMONDI, E., MACCAGNO, A., FERNÁNDEZ-VIÑA, M. and STASTSNY, P. 1993. Differences en HLA class II alleles of isoblated South American indian populations from Brazil and Argentina. Hum Immunol, 37: 213. [ Links ]
7. CRAWFORD, M. 1998. The origins of native America, in Evidence from anthropological genetics. Cambridge University Press, New York (USA). [ Links ]
8. FERNÁNDEZ-VIÑA, M., MORAES, J.R., MORAES, M.E., MILLAR, S., STASTNY, P. 1991. HLA class II haplotypes in Amerindians and in black North and South Americans. Tissue Antigens, 38: 235. [ Links ]
9. FERNÁNDEZ-VIÑA, M., XIAOJIANG, G., MORAES, E., MORAES, R. et ál. 1991. Alleles at four class II loci determined by oligonucleotide hybridatization and their associations in five ethnic groups. Immunogenetics, 34: 299-312. [ Links ]
10. GARCÍA ORTIZ, J., SANDOVAL RAMÍREZ, L., RANGEL VILLALOBOS, H. 2006. High- resolution molecular characterization of the HLA class I and II class II in the Tarahumara Amerindian population. Tissue Antigens, 67(3): 135-146. [ Links ]
11. GORODESKY, C., CASTRO, E.L.E., ESCOBAR, G.A. 1985. The HLA system in the prevalent Mexican indian group: the Nahuas. Tissue Antigens, 25 (1): 38-46. [ Links ]
12. GORODESKY C., ALAEZ, C., VÁSQUEZ-GARCÍA M., DE LA ROSA G., INFANTE, E. et ál. 2001. The genetic structure of different locations: tracking back their orings through MHC genes, blood group systems and microsatellites. Humman Immunology, 62: 979-991. [ Links ]
13. GOODMAN L. 1969. On partitioning chi-square and detecting partial association in three-way contingency tables. Y.R. Stat Soc., 831: 486. [ Links ]
14. GUÉDEZ, Y., LAYRISSE, Z., DOMÍNGUEZ, E., RODRIGUEZ-LARRALDE, J., SCORZA. 1994. Molecular analysis of MHC class II alleles and haplotypes (DRB1, DQA1 and DQB1) in the Bari Amerindians. Tissue Antigens, 44: 125-128. [ Links ]
15. LAYRISSE, Z., GUÉDEZ, Y., DOMÍNGUEZ, E., RODRIGUEZ-LARRALDE, J., SCORZA. 1995. Extended HLA haplotype among the Barí Amerindians of the Perija range relationship to other tribes based on four-loci haplotype Frequencies. Humman Immunology, 44: 228-235. [ Links ]
16. LAYRISSE M., W. 1966. Indian societies of Venezuela: Their bood group types. Editorial Sucre. Caracas, Venezuela. [ Links ]
17. LAYRISSE, Z., GUÉDEZ, Y., DOMÍNGUEZ, E., PAZ, N. et ál. 2001. Extended HLA haplotype in a Carib Amerindian population: the Yucpa of the Perija range. Humman Immunology, 63: 992-999. [ Links ]
18. LAYRISSE, Z., MATOS, M., MONTAGNANI, S., HERRERA, F. et ál. 1997. The novel HLA-DRB1*0807 allele is present in a Carib Amerindian population: the Yucpa. Tissue Antigens, 49(4): 427-428. [ Links ]
19. LAYRISSE, Z. 1992. Impacto de la Biología molecular en el estudio del polimorfismo del complejo principal de histocompatibilidad (CPH) en poblaciones humanas. Revista de la Real Academia de Ciencias Exactas, Físicas y Naturales: 71-74. [ Links ]
20. LÁZARO, A.M., FERNANDEZ-VIÑA, M., LIU, Z., STASTNY, P. 1993. Enzyme-linked DNA oligotyping a practical method for clinical HLA-DNA typing. Human Immunology, 36: 243-248. [ Links ]
21. LINDEL, N., ACHINO, B., PANDO, M., SATZ, L. 1997. Manual de técnicas. Curso teórico-práctico. Recientes avances en histocompatibilidad e inmunogenética. Facultad de Medicina, Universidad de Buenos Aires. Buenos Aires, Argentina. [ Links ]
22. LOUZADA-JUNIOR, P., SMITH, A.G., HANSEN, J.A., DONADI, E.A. 2001. HLA-DRB1 and DQB1 alleles in the Brazilian population of the northeastern region of the state of Sao Paulo. Brief Com. Tissue Antigens, 57: 158-162. [ Links ]
23. MACK, S.J., ERLICH, H.A. 1998. HLA class II polymorphin in the Ticuna of Brazil: evolutionary implications of the DRB1* 0807 allele. Tissue Antigens, 51: 41-50. [ Links ]
24. MATTIUZ, P.L., IHDE, D., PIAZZA, A., CEPELLINI, R., BODMER, W. F. 1970. New approaches to the population genetic segregation analysis of the HLA system, en Terasaki, P.I. (ed.). Histocompatibility Testing. Munksgaard, Copenhagen (Denmark). [ Links ]
25. MILLER, S.A., DYKES, D.D. and Polesky, F.H. 1988. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Reserch, 16 (3): 1215. [ Links ]
26. NEI, M., ROYCHOUDTHURG, A.K. 1974. Sampling variances of hetero zygosity and genetic distance. Genetic: 76-379. [ Links ]
27. OLIVO, A., DEBAZ, H., DE LA ROSA, G., ALAEZ, C., MORENO, M., JUÁREZ, V., GORODESKY, C., GUÉDEZ, Y., DOMÍNGUEZ, E. et ál. 1996. Alelos y haplotipos del complejo principal de histocompatibilidad en tres poblaciones indígenas americanas: Revista de la Real Academia de Ciencias Exactas, Físicas y Naturales, 197-210. [ Links ]
28. RIVERA, S., WEIR, J., ECHEVERRÍA, M., MÁRQUEZ, G., FUENMAYOR, A., HASSANHI, M., GONZÁLEZ, M. et ál. 1998: HLA en la población zuliana de Venezuela. Inmunología, 17: 137-145. [ Links ]
29. SEMPRÚN, C.E. 1993. La cultura guajira se extingue. Consejo Nacional de la Cultura, Dirección General Sectorial de Desarrollo Regional. Caracas, Venezuela. [ Links ]
30. SCHMITZ, P.I. 1983. Mecanismos das migracoes paleoindigenas na América tropical. 1. O territorio nacional, menos os trés estados sulinos. Comun XXXV Reuniáu An Soc Bras Progr Ciéncia, Belén. PA, 1-10. [ Links ]
31. STATSNY, P., GAO, X., MORAES, M., CERNA, M. et ál. 1991. Class II haplotype defined by oligonucleotide typing in II differents ethnic groups, in Tsuji, K.; Aizawa, M. and Gasazuki, T. (eds.). HLA, 2: 233-234. [ Links ]
32. SIDNEY, S. 1972. Estadística no paramétrica. Editorial Trillas. México, México. [ Links ]
33. SHAOHUA, A.; FÉRNANDEZ-VIÑA, M.; FALCO, M.; CERNÁ, M.; RAIMONDI, P.S. 1993. A novel HLA-DRB1 allele (DRB1* 0417) in South American indians. Inmunogenetics: 38-463. [ Links ]
34. TOKUNAGA, K.; OHASHI, J.; BANNAI, M.; JUJI, T. 2001. Genetic link between Asians and native Americans. Evidence from HLA genes and haplotypes. Hum Immunol, Sep 62(9): 1001-8. [ Links ]
35. VICARIO, J.L.; BALAS, A.; TROMPETA, E.; BARRAGÁN, R.; MEZQUILLA G.; GARCÍA, P.; MUNICIO, A. 1996. Frecuencias de antígenos y haplotipos HLA en españoles: comparación con distintas poblaciones. Revista de la Real Academia de Ciencias Exactas, Físicas y Naturales: 248-268. [ Links ]
36. WILLIAMS, T.; WU, J.; FOUTZ, T.; MCAULEY, J.D.; TROUP, G.M. 1994. A new DRB1 allele (DRB1*0811) identified in native Americans. Immunogenetics, 40: 314. [ Links ]
37. YUNIS, J.J.; OSSA, H.; SALAZAR, M.; DELGADO, M.B.; DEULOFENT, R.; DE LA HOZ, A.; BING, D.H.; RAMOS, O.; YUNIS, E.J.; YUNIS, E.J. 1994. Major histocompatibilitis complex class II alleles an haplotypes and blood groups of four Amerindian triles of northern Colombia. Human Immunology, 41: 248-258. [ Links ]
38. YUNIS, J.J.; YUNIS, E.J.; YUNIS, E. 2001. Genetic relationship of the Guambino, Páez and Ingano Amerindians of souhwest Colombia using major histocompatibility complex class II haplotypes and blood groups. Human Immunology, 62: 970-978. [ Links ]