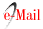Revista de la Sociedad Venezolana de Microbiología
versão impressa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.28 n.1 Caracas jun. 2008
Estafilococos oxacilino resistentes en queso blanco fabricado en el estado Anzoátegui, Venezuela
Druvic Lemus Espinoza*, María Teresa Maniscalchi Badaoui, Mazn Hassoun, Heycarid Vizcaya
Dpto. Microbiología y Parasitología. Escuela de Ciencias de la Salud. Universidad de Oriente (UDO) Núcleo de Anzoátegui. Venezuela.
* Correspondencia: E-mail: lemusd@yahoo.com
Resumen:
El objetivo de esta investigación fue detectar especies estafilocócicas oxacilino resistentes en quesos blanco duro y blando, fabricados artesanalmente en diversos municipios de la zona norte del estado Anzoátegui. Las cepas de Staphylococcus fueron aisladas e identificadas empleando métodos convencionales y se confirmaron las especies coagulasa negativas mediante galerías API 32 Staph (BioMérieux); la susceptibilidad a oxacilina y otros antibióticos por el método de difusión en disco y la presencia del gen mecA, por prueba de látex MRSA Slidex (BioMérieux) que pone de manifiesto la proteína PBP2a. De 130 quesos evaluados, se aislaron 171 cepas estafilocócicas, 26 (15,2%) resistentes a oxacilina (61,5% con presencia del gen), distribuidas como sigue: 14 cepas de S. epidermidis (8,2%), 6 de S. capitis (3,4%), 2 de S. aureus (1,2%), 2 de S. hominis (1,2%), 1 de S. warneri (0,6%) y 1 de S. saprophyticus (0,6%). La presencia del gen que codifica para la proteína PBP2a se detectó tanto en cepas con homorresistencia (76,9%) como con heterorresistencia (23,1%). S. haemolitycus fue 100% sensible a oxacilina. La presencia de cepas estafilocócicas oxacilino resistentes en el queso blanco, puede representar un riesgo para salud pública ya que podría servir como fuente de diseminación de éstas y acrecentar el problema de la resistencia.
Palabras clave: Staphylococcus spp. oxacilino resistente, proteína PBP2a, queso blanco
Oxacyllin resistant Staphylococcus in white cheese manufactured at the Anzoategui State, Venezuela
Abstract:
The purpose of this study was to detect oxacyllin resistant Staphylococcus species in hard and soft white cheese manufactured by artisans at various municipalities of the northern area of Anzoátegui State. The Staphylococcus strains were isolated and identified using conventional methods and coagulase negative species were confirmed through API 32 Staph (BioMérieux) galleries; susceptibility to oxacyllin and other antibiotics was determined by the disk diffusion method and presence of the mecA gene with the MRSA Slidex (BioMérieux) latex test that identifies PBP2a protein. From 130 cheeses evaluated, we isolated 171 Staphylococcus strains, 26 of which (15,2%) were oxacyllin-resistant (61,5% with presence of the gene), distributed as follows: 14, S. epidermidis strains (8,2%); 6, S. capitis (3,4%); 2, S. aureus (1,2%); 2, S. hominis (1,2%); 1, S. warneri (0.6%); and 1, S. saprophyticus (0.6%). Presence of the protein PBP2a codifying gene was detected in both homoresistant (76.9%) and heteroresistant (23.1%) strains. S. haemolitycus was 100% oxacyllin sensitive. Presence of oxacyllin resistant strains in white cheese can represent a public health risk since it could serve as a dissemination source, increasing the resistance problem.
Key words: Staphylococcus spp, oxacyllin resistant, PBP2a protein, white cheese
Recibido 03 de septiembre de 2007; aceptado 13 de noviembre de 2007
Introducción
Aunque anualmente se dispone en el mercado de nuevos antimicrobianos para enfrentar el problema de la resistencia microbiana, las bacterias son capaces de desarrollar defensas más efectivas contra estos nuevos y poderosos antibióticos. En este sentido, el valor terapéutico de estos antimicrobianos ha estado en evolución a través de los años. Las características mismas de los microorganismos hacen que la lucha contra ellos se haya convertido en una carrera donde ha habido la necesidad de emplear todo tipo de estrategias, desde la búsqueda de compuestos nuevos
con mayor actividad biológica, hasta el diseño de moléculas nuevas mediante procedimientos de biotecnología, pasando por combinaciones de antimicrobianos [1,2].
La resistencia bacteriana es un tema muy importante en el estudio de los antibióticos, porque su comprobación implica el fracaso de la terapéutica. La terapia antibiótica ha conducido a una prolongación en la expectativa y calidad de vida, como parte de los avances en la medicina moderna que han reducido la morbimortalidad de numerosos padecimientos, en especial de las enfermedades infecciosas; en contraste, el aumento del uso de estos medicamentos, a veces de forma indiscriminada, desde la década de 1950 se ha acompañado del alza creciente en la resistencia, cuya principal causa es la selección de cepas bacterianas que fortalecen sus genes de resistencia a la mayoría de los antibióticos y la bacteria responsable de la infección logra evadir su acción [1,2].
La producción de ß-lactamasas es el medio más importante de resistencia a los antibióticos ß-lactámicos y, en la actualidad, hay varias clases de esta enzima de origen bacteriano [1,2]. Se ha señalado, que dentro de los antibió-ticos a los cuales diversas bacterias han desarrollado resistencia, está la meticilina, primera penicilina eficaz ante la acción de las b-lactamasas, que actúa inhibiendo la síntesis de la pared bacteriana al unirse a las proteínas fijadoras de penicilina (PBP), enzimas encargadas de llevar a cabo la transpeptidación que permite el entrecruzamiento del peptidoglicano de la pared celular bacteriana. La resistencia a meticilina, viene mediada principalmente por la adquisición del gen mecA, el cual codifica para la producción de la PBP2a, una transpeptidasa con baja afinidad por los antibióticos b-lactámicos, pero que conserva su función en la síntesis de la pared celular [3].
El género Staphylococcus es uno de los grupos bacterianos al que más se le ha estudiado su patrón de susceptibilidad a los antibióticos; se hallan en la piel y mucosas y pueden llegar a los alimentos a través de manipuladores de alimentos con infecciones piógenas agudas o por portadores asintomáticos, que los albergan en fosas nasales y faringe [4-8] asociándose, entonces, su presencia en alimentos a una inadecuada manipulación o al empleo de materia prima contaminada constituyéndose, a su vez, en fuente de diseminación a nuevos hospedadores susceptibles de adquirir la infección.
En Staphylococcus aureus (S. aureus), se han descrito, además, otros mecanismos, que pueden ser responsables de la resistencia a meticilina y que median la llamada resistencia borderline, con valores de concentración mínima inhibitoria (CMI) cercanos o justo en el punto de corte (4-8 mg/ml para la oxacilina), entre los que se encuentra la hiperproducción de b-lactamasa y la presencia de PBP modificadas (PBP 1, 2 ó 4) diferentes a la PBP2a, los cuales a diferencia de la resistencia mediada por el gen mecA, no comparten resistencia con el resto de los antibióticos b-lactámicos, sin embargo, su frecuencia e implicación terapéutica no está bien definida [3,9,10].
El queso blanco constituye uno de los principales alimentos dentro de la pirámide nutricional, ya que es una buena fuente de proteínas, vitaminas y minerales en especial de calcio [11]. Debido al tipo de proceso que conlleva su manufacturación, este alimento también es un caldo de cultivo de microorganismos, y por su alta salinidad, favorece a las especies de Staphylococcus. Aunado a ello, a veces no se siguen las normas de higiene adecuadas, lo que trae como consecuencia que el producto obtenido resulte contaminado o que esta contaminación se genere después de elaborado, posiblemente como consecuencia de un mal almacenamiento o a la exposición ambiental durante la comercialización y/o por parte de los manipuladores de alimentos y en los hogares de los consumidores. Por lo anteriormente expuesto se debe considerar todos los segmentos de la cadena alimentaría, en su integridad desde la producción primaria hasta el consumo, donde cada elemento tiene potencial de influir sobre la inocuidad del producto, convirtiendo a ésta en una vía potencial de transmisión de microorganismos resistentes desde los animales y el ambiente al hombre.
Hoy día, la función de los laboratorios de microbiología ha sido ampliada incluyendo dentro de sus responsabilidades la vigilancia de la resistencia bacteriana a los antibióticos. Como objetivo de esta investigación, se propuso detectar especies estafilocócicas oxacilino resistentes en queso blanco duro y blando, fabricado artesanalmente en diversos municipios de la zona norte del estado Anzoátegui, Venezuela.
Materiales y métodos
Se procesaron 130 muestras de queso blanco blando y duro de fabricación artesanal de los municipios Urbaneja, Sotillo, Peñalver, Simón Rodríguez, Bolívar, Mesa de Guanipa, Freites, Aragua de Barcelona y Anaco, obtenidas de forma aleatoria en diversos establecimientos, durante el período Junio 2005 – Enero 2006. Las muestras fueron colocadas por los mismos vendedores en bolsas transparentes, estériles, con cierre hermético y posteriormente fueron transportadas en cavas a 18ºC hasta el Laboratorio de Microbiología y Parasitología de la Escuela de Ciencias de la Salud de la Universidad de Oriente, Núcleo de Anzoátegui. El aislamiento bacteriano se realizó por el método convencional descrito por la Norma COVENIN 1292-89 [12] y según recomendaciones del manual analítico de la Food and Drug Administration (FDA) de los Estados Unidos [13]. Las cepas de Staphylococcus aisladas fueron identificadas empleando pruebas específicas para ello (crecimiento en medios selectivos, detección de coagulasa en tubo, catalasa) [14] y se confirmó la identificación de las especies coagulasa negativas mediante el uso de galerías API 32 Staph (BioMérieux), empleándose las cepas control de ensayos S. aureus ATCC 25923 y S. epidermidis ATCC 14990. La susceptibilidad a oxacilina y otros antibióticos (vancomicina, clindamicina, gentamicina, eritromicina, tetraciclina y ciprofloxacina) se determinó por el método de difusión en disco [15] y se detectó la presencia del gen mecA, por prueba de látex MRSA Slidex (BioMérieux) que pone de manifiesto la proteína PBP2a. El análisis descriptivo de los resultados se realizó mediante
el programa Statistical Product and Service Solutions (SPSS) versión 9.0 para Windows y para determinar las relaciones estadísticas se aplicó Chi cuadrado (X2) con un nivel de significancia del 95% (p < 0,05).
Resultados
Se aislaron un total de 171 cepas de Staphylococcus spp., de 130 muestras de queso blanco (blando y duro) procesadas. Predominó el aislamiento de Staphylococcus coagulasa negativa (67,7 %) sobre los aislamientos de Staphylococcus coagulasa positiva (32,20%). Las especies detectadas por orden de frecuencia fueron (Tabla 1): en quesos blandos: S. epidermidis (28,1%), S. aureus (11,7%), S. capitis (4,1%), S. haemolitycus (1,7%), S. saprophyticus (1,7%), S. hominis (1,2 %) y S. warneri (0,6%); en las muestras de queso duro la distribución de las especies fue: S. epidermidis (21,6 %), S. aureus (20,5%), S. capitis (4,1%), S. haemolitycus (1,7%), S. saprophyticus (1,7%) y S. hominis (1,2 %). La presencia de S. aureus en las muestras de queso duro, obtuvo un valor estadísticamente significativo, así como cuando se relacionó la presencia de S. epidermidis en queso blando.
Tabla 1. Estafilococos aislados de muestras de queso blanco (blando y duro) fabricado artesanalmente en el Edo. Anzoátegui.
| Especie | Tipo de queso | Total | ||||
| Blando | Duro | |||||
| Nº Aislados | % | Nº Aislados | % | Nº Aislados | % | |
| S. aureus | 20 | 11,7 | 35 | 20,5 * | 55 | 32,2 |
| S. epidermidis | 48 | 28,1 * | 37 | 21,6 | 85 | 49,7 |
| S. capitis | 7 | 4,1 | 7 | 4,1 | 14 | 8,2 |
| S. haemolitycus | 3 | 1,7 | 3 | 1,7 | 6 | 3,5 |
| S. saprophyticus | 3 | 1,7 | 3 | 1,7 | 6 | 3,5 |
| S. hominis | 2 | 1,2 | 2 | 1,2 | 4 | 2,3 |
| S. warneri | 1 | 0,6 | 0 | 0 | 1 | 0,6 |
| TOTAL | 84 | 49,1 | 87 | 50,9 | 171 | 100 |
*p < 0,05
La resistencia asociada a los antibióticos evaluados fue la siguiente: eritromicina (67,7%), gentamicina (64,7%), ciprofloxacina (57%), clindamicina (49,2%) y tetraciclina (46,2%). Todas las cepas de Staphylococcus aisladas fueron sensibles a la vancomicina y 26 (15,2%) resistentes a oxacilina (Tabla 2). Todos los aislados de S. haemolyticus (100%) fueron sensibles a oxacilina. En una de las cepas oxacilino-resitente de S. aureus no se detectó la presencia de la proteína PBP2a (Figura 1), no obstante, en este estudio se observó una significancia estadística (p < 0,05) cuando se relacionó a las especies S. aureus, S. capitis, S. hominis y S. warneri, que fueron resistentes a oxacilina y en ellas se evidenció la presencia de la proteína PBP2a
Tabla 2. Susceptibilidad a oxacilina de especies estafilocócicas aisladas de muestras de queso blanco (blando y duro) fabricado artesanalmente en el Edo. Anzoátegui.
| Especie | Oxacilina | Total | ||||
| Sensible | Resistente | |||||
| Nº aislados | % | Nº aislados | % | Nº aislados | % | |
| S. aureus | 53 | 31 | 2 | 1,2* | 55 | 32,2 |
| S. epidermidis | 71 | 41,5 | 14 | 8,2 | 85 | 49,7 |
| S. capitis | 8 | 4,8 | 6 | 3,4* | 14 | 8,2 |
| S. haemolitycus | 6 | 3,4 | 0 | 0 | 6 | 3,4 |
| S. saprophyticus | 5 | 2,8 | 1 | 0,6 | 6 | 3,4 |
| S. hominis | 2 | 1,2 | 2 | 1,2* | 4 | 2,4 |
| S. warneri | 0 | 0 | 1 | 0,6* | 1 | 0,6 |
| TOTAL | 145 | 84,75 | 26 | 15,2 | 171 | 100 |
* p < 0,05
La presencia del gen que codifica para la proteína PBP2a se detectó tanto en cepas con homorresistencia como en cepas con heterorresistencia (Tabla 3); de las cepas que mostraron resistencia absoluta u homorresistencia se evidenció la presencia del gen mecA en el 30,8%, mientras que solo en 2 casos (7,7%) de aquellas que presentaron heterorresistencia a oxacilina.
Tabla 3. Distribución porcentual de la presencia del gen mecA de acuerdo al tipo de resistencia observada en cepas aisladas de queso blanco (duro y blando) fabricado artesanalmente.
| Tipo de Resistencia | Gen mecA | Total | ||||
| Ausente | Presente | |||||
| Nº Aislados | % | Nº Aislados | % | Nº Aislados | % | |
| Homorresistente | 12 | 46,2 | 8 | 30,8 | 20 | 76,9 |
| Heterorresistente | 4 | 15,4 | 2 | 7,7 | 6 | 23,1 |
| Total | 16 | 61,6 | 10 | 38,5 | 26 | 100 |
Discusión
Tomando en consideración que el género Staphylococcus está integrado por especies halófilas, y existen factores fisicoquímicos y de almacenaje del queso que permiten el óptimo desarrollo de las mismas (rangos de temperatura de 35ºC a 36ºC, pH entre 4.0 y 9.8, atmósfera aeróbica y una actividad del agua, Aw, 0.98, entre otros) que crean un hábitat idóneo para la proliferación de estos mesófilos [16], se puede justificar su presencia en quesos blancos elaborados artesanalmente.
Sin embargo, al comparar los valores de frecuencia obtenidos para S. aureus y S. epidermidis en queso duro y blando respectivamente, se observó una diferencia estadísticamente significativa que puede ser explicada si consideramos los siguientes parámetros: 1.- La variación en la concentración de sal en la elaboración de los diferentes tipos de quesos (en duros, aproximadamente 4% de NaCl y en semi-duros o blandos cerca de 3% de NaCl [17], 2.- Una capacidad de adaptabilidad del S. aureus a concentraciones de NaCl superior que el S. epidermidis, debido probablemente, a diferencias en los constituyentes lipídicos de la célula (principalmente por la diferencia en las cantidades de cardiolipina que son mayores en S. aureus [18-22] y la protección que pueden conferirle al S. aureus los iones Ca++ del queso [23]. Cabe destacar, que la contaminación bacteriana del queso de producción artesanal, es facilitada por la exposición de las manos de las personas que procesan el alimento, al suelo, al agua, a objetos contaminados, el contacto con animales reservorios [24-26], así como también por la diseminación microbiana desde las mucosas que tapizan fosas nasales y garganta [4-6] de los manipuladores del alimento como ha sido demostrado por varios autores en la evaluación microbiológica del personal que procesa alimentos y su relación con el grado de contaminación del producto alimenticio [6,27].
Estudios sobre la calidad microbiológica de los quesos blancos frescos venezolanos de elaboración artesanal, indican que en más del 90% de las muestras analizadas, las poblaciones de especies de Staphylococcus están por encima de los límites aceptables [28-30]. En la elaboración de los quesos blancos frescos se emplea leche cruda y en muchos casos hay deficiencias en las buenas prácticas de manufactura (pobres condiciones higiénicas del ordeño, la no refrigeración de la leche después de éste, el ordeño de vacas con mastitis, entre otros), lo que explica que estos productos sean riesgosos para la salud de los consumidores [29,30].
Todas las cepas de S. haemolyticus aislados de los quesos fueron sensibles a oxacilina, a diferencia de los hallazgos de Louie et al. [31], quienes evaluaron la susceptibilidad a oxacilina de cepas provenientes de muestras clínicas encontrando que 11/15 aislados de S. haemolyticus eran resistentes. Este resultado es interesante y significativo, ya que pudiera deberse a que éstas habrían estado en contacto con cepas o variantes de bacterias con desarrollo de mecanismos de resistencia antibiótica, en concordancia con lo demostrado por Teuber et al. [32] quienes señalaron la posibilidad de la transferencia genética de información de resistencia bacteriana, entre cepas de la misma o diferentes especies provenientes de animales tratados con antibióticos y cepas de alimentos, como el queso.
La frecuencia de los aislados de muestras de quesos, resistentes a oxacilina, fueron menores a los reportados por la mayoría de los investigadores para el grupo de Staphylococcus provenientes de muestras de pacientes hospitalizados [31,33], pero similares a los obtenidos por Normanno et al. [34], quienes igualmente estudiaron la presencia de especies oxacilino-resistentes en quesos italianos (mozarella y pecorino). Aún cuando es obvio que es más factible el hallazgo de cepas de origen nosocomial u hospitalarias resistentes a cualquier antibiótico (incluso en portadores asintomáticos que acuden o trabajan en estos centros de salud), en donde este ambiente termina siendo fuente de diseminación para dichos microorganismos, se justifica el estudio de la detección de cepas de origen comunitario provenientes de alimentos, debido a que implica, epidemiológicamente, la existencia de cepas de circulación ambiental que ya poseen la capacidad de resistir los efectos de la oxacilina y por lo tanto a todos los β-lactámicos y a las combinaciones de β-lactámicos con inhibidores de β-lactamasas. Este tipo de investigación ha sido poco implementado tanto en el ámbito internacional como en el nacional.
No se detectó la proteína PBP2 expresada por el gen mecA, en todas las cepas oxacilino resitentes aisladas, lo que implica que se puede atribuir la resistencia a la presencia de otras PBP modificadas o a la hiperproducción de b-lactamasa [3,9,10]. Sin embargo, en este estudio se observó una correlación estadísticamente significativa cuando se relacionó la presencia de la proteína PBP2a y las especies aisladas de Staphylococcus aureus, S. capitis, S. hominis y S. warneri, lo que amerita más estudios para determinar si las cepas locales de estas especies han desarrollado mayor oportunidad y/o capacidad de expresar este mecanismo de evasión a los antibioticos, en comparación con otras especies o es simplemente un hallazgo estadístico fortuito. La presencia del gen que codifica para la proteína PBP2a se observó tanto en cepas con homorresistencia como en cepas con heterorresistencia, probablemente debido a que este tipo de cepas oxacilino resistentes poseen una variabilidad extrema en la expresión de rasgos de resistencia [3].
Además del S. aureus, los Staphylococcus coagulasa negativos son reconocidos como importante causa de infecciones nosocomiales; el origen de la contaminación con este microorganismo puede ser de endógeno o exógeno, comúnmente transferido a través de las manos del personal que está involucrado [35-37]. Otro factor crítico de la transmisión, es la capacidad que poseen estos gérmenes para sobrevivir en diferentes ambientes, así como también la capacidad de adherencia en biopelículas, formación implicada como factor de virulencia [36,38]. La tasa de resistencia a oxacilina de este grupo de microorganismo tiene una tendencia al aumento en varios países, como por ejemplo Estados Unidos de América [37], e inclusive en Venezuela [39], de aquí la importancia de la identificación de las especies y su comportamiento de susceptibilidad individual para el mejor monitoreo epidemiológico de estas bacterias.
Son muchos los trabajos que evalúan la calidad microbiológica de alimentos de diversos orígenes, como lácteos o cárnicos, no obstante, en muy pocos se determina la posibilidad del desarrollo de resistencia a los antibióticos y su posible papel como fuente de infección desde los alimentos al hombre o animales [25,32,34,40-42].
A pesar de que los hallazgos porcentuales de Staphylococcus oxacilino resistentes descritos en este estudio son bajos, conforman un modelo factible como fuente de diseminación de los mismos que deberían ser considerados desde un enfoque sanitario, como un problema de salud pública donde se incrementan los factores de riesgo de adquirir en la comunidad, una infección por un Staphylococcus oxacilino resistente. Esto requerirá medidas educativas y preventivas dirigidas a los operadores de alimentos y a los productores agropecuarios, orientadas a la modificación de las condiciones que favorecen el desarrollo de estas cepas a fin de minimizar el riesgo en la comunidad.
Conclusiones
· Se observó el predominio de cepas Staphylococcus coagulasa negativas en ambos tipos de queso blanco: blando y duro.
· La resistencia de los Staphylococcus spp. a la oxacilina mediada por el gen mecA está presente en cepas ambientales de fácil intercambio entre individuos.
· No se observó relación estadísticamente significativa entre el tipo de resistencia a oxacilina y la presencia de la proteína PBP2a.
· La resistencia de algunas cepas de Staphylococcus spp. a la oxacilina se asoció con multirresistencia (resistencia simultánea a eritromicina, gentamicina, ciprofloxacina, clindamicina y tetraciclina).
· La presencia de cepas de estafilococo oxacilino resistentes en un alimento de consumo masivo, como son los quesos de fabricación artesanal, puede representar un problema de salud pública por el aumento del riesgo de diseminación de estas cepas en la comunidad.
Agradecimiento
Expresamos nuestro agradecimiento al Centro Venezolano de Colecciones de Microorganismos (CVCM), Instituto de Biología Experimental (IBE), por la certificación de las cepas Staphylococcus coagulasa negativas.
Referencias
1. Martin G. Resistencia Bacteriana a ß-lactámicos. Evolución y Mecanismos. AVFT 2002; 21: 107-16. [ Links ]
2. Cavalieri S, Hardbeck R, McCarter Y, Ortez J, Rankin I, Sautter R y col. Manual de pruebas de susceptibilidad antimicrobiana. Editor Marie Coyle. Departments of Laboratory Medicine and Microbiology University of Washington. OPS/ASM. (2005). Disponible en: http://www.paho.org/-Spanish/AD/THS/EV/labs_susep_antimicro.htm. [ Links ]
3. Chambers H. Methicillin resistance in Staphylococci: molecular and biochemical basis and clinical implications. Clin Microbiol Rev 1997; 10:781-91. [ Links ]
4. Boyce J. Are the epidemiology and microbiology of methicillin-resistant Staphylococcus aureus changing? JAMA 1998; 279:623-4. [ Links ]
5. Rao G. Risk factors for the spread of antibiotic-resistant bacteria. Drugs 1998; 55:323-30. [ Links ]
6. Hatakka M, Bjorkroth KJ, Asplund K, Maki-Petays N, Korkeala HJ. Genotypes and enterotoxicity of Staphylococcus aureus isolated from the hands and nasal cavities of flight-catering employees. J Food Prot 2000; 63:1487-91. [ Links ]
7. Camargo LF, Strabelli TM, Ribeiro FG, Iwahashi ER, Ebaid M, Filho HH, y col. Epidemiological investigation of an outbreak of coagulase negative Staphylococcus primary bacterium in a newborn intensive care unit. Infect Control Hosp Epidemiol. 1995; 16: 595-6. [ Links ]
8. Al-Shemari H, Abou-Hamad W, Libman M, Desrosiers M. Bacteriology of the sinus cavities of asymptomatic individuals after endoscopic sinus surgery. J Otolaryngol 2007; 36:43-8. [ Links ]
9. Brakstad O, MacLand J. Mecanism of methicilin resistente in Staphylococci. APMIS 1997; 105:264-76. [ Links ]
10. Calvo A, Rodríguez C, Andrade O, Bertuglia F, Márquez N. Staphylococcus aureus meticilino resistente: detección de PBP2 mediante la técnica de látex y evaluación de la resistencia asociada. Bol Venez Infectol 2004; 15: 18-22. [ Links ]
11. Torres E., Pacheco de D. E. Evaluación nutricional, física y sensorial de panes de trigo, yuca y queso llanero. Rev Chil Nutr 2007; 34: 133-41. [ Links ]
12. Comisión Venezolana de Normas Industriales (COVENIN). Norma 1292-89. Alimentos. Aislamiento y Recuento de Staphylococcus aureus. FONDONORMA. 1989. [ Links ]
13. Food and Drug Administration USA (FDA). Bacteriological Analitycal Manual (BAM). Chapters 12. 8º ed. Washington. 1998. [ Links ]
14. Forbes B., Sahm D., Weissfeld A. Bailey & Scott. Diagnóstico microbiológico. 11ª edición. Argentina: Editorial Médica Panamericana. 2004. [ Links ]
15. NCCLS. National Comittee for Clinical Laboratory Standards. Perfomance Standards for Antimicrobial Susceptibility Testing; 15th Informational Supplement. M2-A8. 2005. Vol 25-1. [ Links ]
16. Adams M. Microbiología de los Alimentos. Zaragoza- España: Acribia., S.A. 1997. [ Links ]
17. Comisión Venezolana de Normas Industriales (COVENIN). Norma 3821:2003. Queso Blanco. Categoría B. ICS 2003:67.100.30. FONDONORMA. [ Links ]
18. Short S., White, D. Metabolism of phosphatidylglycerol, lysylphosphatidylglycerol and cardiolipin of Staphylococcus aureus. J Bacteriol 1971; 108: 219-26. [ Links ]
19. Komaratat P, Kates M. The lipid composition of a halotolerant species of Staphylococcus epidermidis. Biochem Biophys Acta 1975; 398:464-84. [ Links ]
20. Kanemasa Y, Takatsu T, Sasai K, Kojima I, Hayashi H. The salt-resistance mechanism of Staphylococcus aureus examined by salt-sensitive mutants. Acta Med Okayama 1976; 30: 271-6. [ Links ]
21. Hurst A, Ofori E, El-Banna AA, Harwig J. Adaptational changes in Staphylococcus aureus MF31 grown above its maximum temperature when protected by NaCl: physiological studies. Can J Microbiol 1984; 30:1105-11. [ Links ]
22. Hurst A, Ofori E, Vishnubhatla I, Kates M. Adaptational changes in Staphylococcus aureus MF31 grown above its maximum growth temperature when protected by sodium chloride: lipid studies. Can J Microbiol 1984; 30:1424-7. [ Links ]
23. Ochiai T. Staphylococcus aureus requires increased level of Ca2+ or Mn2+ to grow normally in a high-NaCl/low-Mg2+ medium. Microbiol Inmunol 2001; 45: 769-76. [ Links ]
24. Van Duijkeren E, Wolfhagen M, Box A, Heck M, Wannet W. Human-to-dog transmission of methicillin-resistant Staphylococcus aureus. Emerg Infect Dis 2004; 10: 2235-7. [ Links ]
25. Vautor E, Abadie G, Guibert JM, Chevalier N, Pépin M. Nasal carriage of Staphylococcus aureus in dairy sheep. Vet Microbiol 2005; 106:235-9. [ Links ]
26. Bagcigil FA, Moodley A, Baptiste KE, Jensen VF, Guardabassi L. Occurrence, species distribution, antimicrobial resistance and clonality of methicillin- and erythromycin-resistant staphylococci in the nasal cavity of domestic animals. Vet Microbiol 2007;121:307-15. [ Links ]
27. Fueyo JM, Mendoza MC, Martín MC. Enterotoxins and toxic shock syndrome toxin in Staphylococcus aureus recovered from human nasal carriers and manually handled foods: epidemiological and genetic findings. Microbes Infect 2005; 7:187-94. [ Links ]
28. Mendoza C, Oyón R. Estudio comparativo de dos coberturas para queso llanero madurado. Rev Fac. Agron 2002; 28: 1-11. [ Links ]
29. Cava R, Sangronis E, Lucci E, Woyzechowsky L. Efecto de adición de nisina en queso fresco "telita" sobre la supervivencia de Staphylococcus aureus. An Venez Nutr. 2006; 19: 69-73. [ Links ]
30. Miró A, Ríos de Selgrad M. Calidad microbiológica de los quesos blancos venezolanos, analizados en el Instituto Nacional de Higiene "Rafael Rangel". Período: Enero 1988 a Junio 1998. Rev Inst Nac Hig "Rafael Rangel" 1999; 30: 14-20. [ Links ]
31. Louie A, Goodfellow J, Louie M, Simor A. Evaluation of a latex agglutination test (mrsa-screen) from detection of oxacillin resistance in coagulase-negative Staphylococci. Rev Clin Microbiol 2001; 39:4149-51. [ Links ]
32. Teuber M, Meile L, Schwarz F. Acquired antibiotic resistance in lactic acid bacteria from food. Antonie van Leeuwenhoek 1999; 76:115-37. [ Links ]
33. Tenover F, Jones R, Swenson J, Zimmer B, McAllister S, Jorgens J. Methods for improved detection of oxacillin resístance in coagulase-negative Staphylococci: results of a multicenter study. J Clin Microbiol 1999; 37:4051-8. [ Links ]
34. Normanno G, Corrente M, La Salandra G, Dambrosio A, Quaglia NC, Parisi A, y col. Methicillin-resistant Staphylococcus aureus (MRSA) in foods of animal origin product in Italy. Int J Food Microbiol 2007; 117:219-22. [ Links ]
35. Sandri AM, Dalarosa MG, Ruschel de Alcantara L, da Silva EL, Zavascki AP. Reduction in incidence of nosocomial methicillin-resistant Staphylococcus aureus (MRSA) infection in an intensive care unit: role of treatment with mupirocin ointment and chlorhexidine baths for nasal carriers of MRSA. Infect Control Hosp Epidemiol. 2006; 27:185-7. [ Links ]
36. Satorres S, Alcaráz L. Prevalence of icaA and icaD genes in Staphylococcus aureus and S. epidermidis strains isolated from patients and hospital staff. Cent Eur J Public Health 2007; 15:87-90. [ Links ]
37. Edmond M, Wallace D, Mc Chish M, Pfaller R, Wenzel P. Nosocomial Infections in United States hospitals a three-year analysis. Clin Infect Dis 1999; 29:239-44. [ Links ]
38. Alcaráz L, Satorres S, Lucero R, Puig de Centorbi O. Species identification, slime productin and oxacillin susceptibility in coagulase-negative Staphylococcus isolated from nosocomial specimens. Brazilian J Microbiol 2003; 34:45-51. [ Links ]
39. De La Parte-Perez Ma, Brito A, Hurtado P, Landaeta JM, Guzmán M, Carmona O. Cambios en la resistencia de Staphylococcus aureus a los antimicrobianos en centros clínicos del área metropolitana de Caracas, Venezuela. Período 1995-2002. Rev Soc Ven Microbiol 2003; 23: 190-5. [ Links ]
40. De Buyser M, Dufour B, Maire M, Lafarge V. Implication of milk and milk products in food-borne diseases in France and in different industrialized countries. Int J Food Microbiol 2001; 67:1-17. [ Links ]
41. Hwa Lee J. Methicillin (oxacillin)-resistant Staphylococcus aureus strains isolated from major food animals and their potential transmission to humans. Appl Environ Microbiol 2003; 69: 6489–94. [ Links ]
42. Jørgensen HJ, Mørk T, Rørvik LM. The occurrence of Staphylococcus aureus on a farm with small-scale production of raw milk cheese. J Dairy Sci 2005; 88:3810-7. [ Links ]












 uBio
uBio