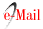Revista de la Sociedad Venezolana de Microbiología
versão impressa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.28 n.2 Caracas dez. 2008
Identificación de micobacterias no tuberculosas: comparación de métodos bioquímicos y moleculares
María José Godoya Loren Orozcoa Cohinta Hernándezb Omaira DaMatac Jacobus De Waardc Susana González Ricob,*
aEscuela de Bioanálisis, Facultad de Medicina
bSección de Bacteriología, Instituto de Medicina Tropical, Universidad Central de Venezuela. Caracas, Venezuela.
cLaboratorio de Tuberculosis. Instituto de Biomedicina Caracas - Venezuela.
* Correspondencia: E-mail: susana.gonzalez@ucv.ve
Resumen: Las infecciones causadas por micobacterias no tuberculosas (MNT) o atípicas constituyen en la actualidad un grave problema de salud, especialmente en pacientes inmunocomprometidos. Estas micobacterias presentan patrones de susceptibilidad a antibióticos particulares y distintos a M. tuberculosis, por lo que la administración del tratamiento adecuado requiere de un método rápido, sencillo y sensible de identificación. La técnica de PRA (Análisis de Restricción de Productos de PCR), basada en la digestión enzimática del producto de amplificación del gen hsp65, ha mostrado ser un método adecuado de identificación de micobacterias. En el presente trabajo se comparó la técnica de PRA con el estándar de identificación de micobacterias representado por las pruebas bioquímicas en 30 aislados provenientes del Laboratorio de Tuberculosis del Instituto de Biomedicina. La técnica de PRA permitió identificar 96% de las cepas analizadas, en comparación con 92.% de cepas identificadas por las técnicas bioquímicas. Los resultados obtenidos fueron idénticos en 18 de 22 cepas, correspondiendo al 82% de los resultados. Se concluye que el PRA es un método rápido, sencillo y económico que produce resultados concordantes con las técnicas tradicionales, con un menor grado de error. Basados en estos resultados se recomienda el uso del PRA en los laboratorios clínicos como método de identificación de rutina para micobacterias.
Palabras clave: micobacterias no tuberculosas, identificación molecular, PRA, pruebas bioquímicas
Identification of non-tuberculosis mycobacteria: comparison between biochemical and molecular methods
Abstract: Infections caused by atypical mycobacteria at present constitute a serious health problem, especially in immunocompromised patients. These mycobacteria present particular susceptibility patterns, different from M. tuberculosis, due to which the administration of an adequate treatment requires a fast, simple and sensitive identification method. The PRA technique (PCR Restriction), based on the enzymatic digestion of the amplification product of the hsp65 gene has shown to be an adequate method for the identification of mycobacteria. In this study we compared the PRA technique with the standard mycobacterial identification method, represented by biochemical tests, in 30 isolates from the Tuberculosis Laboratory of the Instituto de Biomedicina. The PRA technique allowed the identification of 96% of the strains analyzed, as compared with 92% of strains identified through biochemical methods. The results obtained were identical in 18 of 22 strains, corresponding to 82% of the results. It is concluded that the PRA technique is a fast, simple and economical method that produces results in concord with traditional techniques, with a lesser degree of error. Based in these results, the use of PRA as routine identification technique for mycobacteria is recommended for clinical laboratories.
Keywords: non-tuberculosis mycobacteria, molecular identification, PRA, biochemical tests
Recibido 10 de marzo de 2008; aceptado 08 de octubre de 2008
Introducción
Las micobacterias son un grupo complejo de microorganismos que incluye especies causantes de enfermedades graves en el humano, tales como la lepra y la tuberculosis. Las especies pertenecientes al género Mycobacterium son bacilos, no móviles, ácido resistentes (BAR), de crecimiento lento, y un genoma con alto contenido de G+C.
Las llamadas micobacterias no tuberculosas (MNT) o atípicas, es decir, todas aquellas que no pertenecen al Complejo M. tuberculosis o M. leprae, pueden ser saprofíticas, patógenas u oportunistas, así mismo son causantes de infecciones tanto en pacientes inmunocomprometidos, como en inmunocompetentes. Estas infecciones, denominadas micobacteriosis, se localizan generalmente en los pulmones, el sistema linfático, la piel o el tejido óseo, pudiendo ser diseminadas. Las mas comunes son las asociadas a procedimientos cosméticos [1-3] o quirúrgicos [4-7], catéteres, diseminación de enfermedades cutáneas, enfermedades del sistema nervioso central y pulmonar [8- 11], en algunos casos indistinguibles clínicamente de la tuberculosis, que afectan especialmente a pacientes infectados por el Virus de la Inmunodeficiencia Humana (VIH) [12,13]. Las principales especies que aparecen como causa de infección son el complejo M. avium, M. kansasii, M. abscessus, M. marinum y el complejo M. fortuitumchelonae.
La disminución de la prevalencia de la tuberculosis en el mundo desarrollado durante los últimos años se ha visto acompañada por un aumento en la frecuencia de infecciones causadas por micobacterias no tuberculosas. No está muy claro si esto corresponde con un aumento real en la prevalencia o se debe a que las micobacteriosis están siendo reconocidas con más frecuencia por los clínicos, aunado a una mayor capacidad de los microbiólogos para detectar e identificar las micobacterias más inusuales y de difícil aislamiento.
Los regímenes de tratamiento para las micobacteriosis aún no se encuentran claramente definidos en muchos casos, y el resultado terapéutico suele ser decepcionante, a pesar de la disponibilidad de nuevos antimicrobianos. La resistencia in vitro a la mayoría de los fármacos antituberculosos de primera línea es una de las características más llamativas de las MNT. El éxito del tratamiento se ve afectado negativamente por la larga duración del mismo, sus efectos secundarios, y las interacciones de las drogas, que en muchos casos llevan a los pacientes a no completarlo. La elección del tratamiento varía en función de tres factores fundamentales, la forma de presentación clínica, el estado del sistema inmunológico del paciente y la especie de micobacteria responsable. Por ello se requiere de una correcta identificación de la especie implicada en la infección, para aplicar el tratamiento adecuado que garantice la pronta mejoría del paciente.
La identificación tradicional de las MNT se basa en el análisis de ciertas características fenotípicas (biológicas y bioquímicas) tales como la velocidad de crecimiento, la producción de pigmento, la actividad de ciertas enzimas y/o la utilización de algunos sustratos. Muchas de estas técnicas requieren de varias semanas y no es posible en muchos casos, la diferenciación de miembros pertenecientes a un mismo complejo.
Recientemente se han venido utilizando técnicas de biología molecular para realizar la identificación en base a características genotípicas; estas pruebas no solo son más rápidas, sino también mas sensibles y precisas, ya que no son modificables por condiciones ambientales. Entre los métodos moleculares destacan los basados en la Reacción de Cadena de la Polimerasa (PCR por sus siglas en ingles Polymerase Chain Reaction), los cuales presentan una alta sensibilidad y especificidad. En el caso particular de la identificación de micobacterias, a principios de la década de los 90, Telenti et al. implementaron con éxito la técnica llamada Análisis de Restricción de productos de PCR o PRA (PRA por las siglas de su nombre en inglés: PCR Restriction Analysis), la cual se basa en la digestión enzimática con las enzimas de restricción BstEII y HaeIII del producto de amplificación del gen hsp65. Con esta metodología se genera un patrón de restricción específico de cada especie que permite la rápida identificación de las micobacterias no tuberculosas [14].
En un estudio publicado el año pasado donde se comparaban las pruebas bioquímicas y la técnica de PARA, esta última fue considerada una metodología rápida y sencilla de implementar en el laboratorio para la identificación de micobacterias [15]. Otros autores han llegado a la conclusión de que el PRA es un método rápido, sensible y económico por lo que debe ser considerado y aplicado en los laboratorios clínicos para la identificación de micobacterias [16-18]. Así mismo, el PRA ha sido probado directamente sobre muestras clínicas, aportando buenos resultados, lo que disminuiría substancialmente el tiempo de diagnóstico [19].
La técnica de PRA además de ser de gran utilidad en el diagnóstico de infecciones por micobacterias, también ha sido descrita en la realización de análisis filogenéticos [20].
Es por todo ello, que nos planteamos evaluar y comparar ambos métodos (bioquímicos y PRA), resaltando sus respectivas ventajas y desventajas, para de esta manera analizar la factibilidad de implementación de la técnica de PRA como rutina en el laboratorio.
Materiales y Métodos
En este trabajo se analizaron 30 cepas aisladas de pacientes provenientes del Laboratorio de Tuberculosis del Instituto de Biomedicina, previamente identificadas por métodos moleculares, enviadas bajo codificación a la Sección de Bacteriología del Instituto de Medicina Tropical (IMT) en cultivos en Lowenstein–Jensen (L-J). La información sobre la identidad de cada una de las cepas solo fue remitida al IMT al concluir los análisis, de manera de no influir en los resultados. En el IMT, estas cepas fueron identificadas mediante las siguientes metodologías:
Pruebas bioquímicas
Se emplearon los ensayos de catalasa, reducción de nitratos, aril sulfatasa, hidrólisis del Tween 80, reducción del telurito, NaCl, ureasa, captación de hierro, crecimiento en MacConkey, utilización citrato, manitol e inositol como sustratos y producción de pigmentos. Estas pruebas se realizaron de acuerdo con la metodología tradicional descrita en la bibliografía [21] y utilizada en la identificación rutinaria de micobacterias no tuberculosas en la Sección de Bacteriología del Instituto de Medicina Tropical, UCV.
Análisis de restricción de productos de PCR (PRA)
Se emplearon las especificaciones del protocolo utilizado en el Laboratorio de Tuberculosis del Instituto de Biomedicina [22].
Extracción de ADN
Dos a tres colonias de la cepa a identificar crecida en medio Lowenstein-Jensen (L-J) se resuspendieron en 100 μl de buffer TE (10mM tris/HCL, 1 mM EDTA, pH 8.0), y se incubaron en un baño de agua en ebullición durante 10 min. Luego de centrifugar a 7000 rpm por 2 min, se tomaron 5 μl del sobrenadante para utilizarse como sustrato para la PCR.
PCR
Se emplearon los iniciadores TB11 (5`-ACCAACGATGGTGTGTCCAT) y TB12 (5`-CTTGTCGAACCGCATACCCT) diseñados originalmente por Telenti y col. (14). Para cada aislado se preparó una mezcla de reacción que contenía 6% de glicerol, 100 μM de cada uno de los desoxinucleotidos trifosfato (dATP, dGTP, dCTP, dTTP), 2.5 mM de MgCl2 25 mM, buffer de PCR (50 mM Tris/HCL, 10 mM KCl, 0.1% tritón, pH 9.0), 0.5 μM de cada uno de los iniciadores (TB11 y TB12) y 1.25 U de Taq polimerasa, y 5 μl del ADN aislado de la cepa de interés en un volumen final de 50 μl. Para la amplificación se utilizó un ciclo inicial de desnaturalización de 94ºC por 3 minutos, 35 ciclos de amplificación de 94ºC por 30 segundos, 55ºC por 30 segundos y 72ºC por 30 segundos, finalizando con una extensión de 72ºC por 5 minutos.
Visualización del producto de PCR
La presencia de un amplicón de tamaño, integridad y concentración adecuada, se verificó mediante electroforesis horizontal en gel de agarosa 1% en buffer TAE (40 mM tris acetato, 2 mM EDTA, pH 8.0) conteniendo bromuro de etidio 0.5 μg/ml, durante 1 hora a 100 V. Se corrieron 5 μl del producto de la PCR con 2 μl de buffer de carga (glicerol 50% en buffer TE, 0.05% p/v azul de bromofenol) junto con 1 μg por carril de un marcador de peso molecular Lambda HindIII. El producto de amplificación debe tener un tamaño de 439 pb, correspondiente a las posiciones 398 y 836 del gen hsp65.
Digestión de amplicones
Una vez verificada la presencia de un producto de amplificación, se procedió a digerir el mismo con las enzimas de restricción BstEII y HaeIII. Un volumen total de 15 μl del producto de amplificación obtenido fue digerido con una mezcla de reacción conteniendo 100 μg/ml de albúmina sérica bovina, el buffer de reacción apropiado para cada enzima (para BstEII 100 mM NaCl, 50 mM Tris-HCl, 10 mM MgCl2, 1 mM DTT, pH 7.9; y para HaeIII 50 mM NaCl, 10 mM Tris-HCl, 10 mM MgCl2, 1 mM DTT, pH 7.9) y 5 U de la enzima de restricción correspondiente. La digestión enzimática se llevó a cabo durante una hora a 60ºC para el caso de BstEII, o a 37ºC para HaeIII.
Visualización y análisis de los patrones de digestión
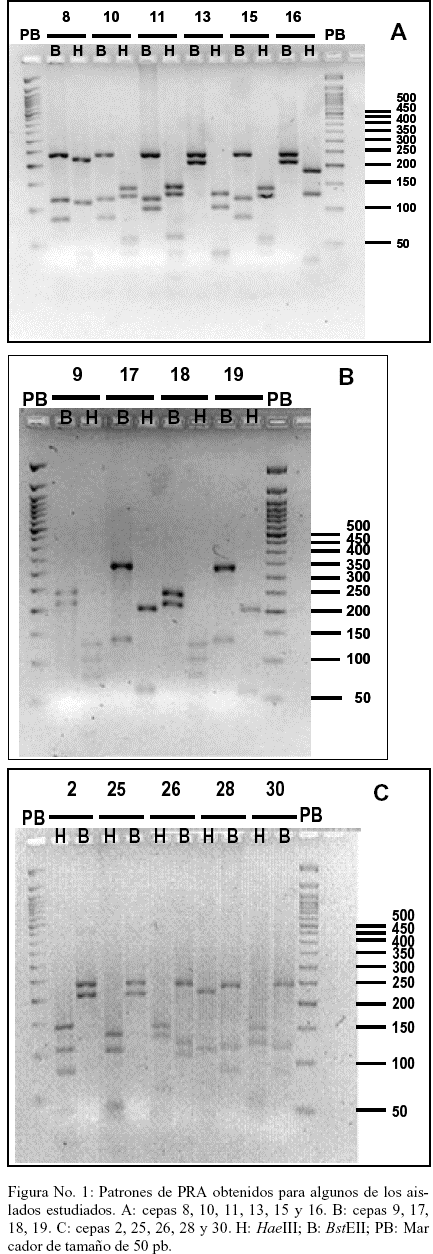
Una vez transcurrido el tiempo de digestión, se procedió a realizar la electroforesis horizontal de los productos en un gel de agarosa al 3% en TAE conteniendo bromuro de etidio, por una hora y media a 100 V. Para ello se colocaron en cada pozo 25 μl del producto de digestión con 2 μl de buffer de carga. En cada corrida se incluyeron uno o dos carriles con 1 μg de marcador de peso molecular de 50 pb. El patrón de bandas obtenido por digestión, se visualizó mediante exposición a luz UV, registrándose el resultado con una cámara fotográfica digital (Kodak DC100). La estimación del tamaño de cada una de las bandas que conforman el patrón de restricción, se realizó por comparación con la distancia de migración del marcador de 50 pb incluido. La identificación se realizó al comparar los patrones obtenidos en cada caso con los publicados en el banco de datos PRAsite (http://app.chuv.ch/prasite/index.htm) o con algoritmos descritos en la literatura.
Resultados
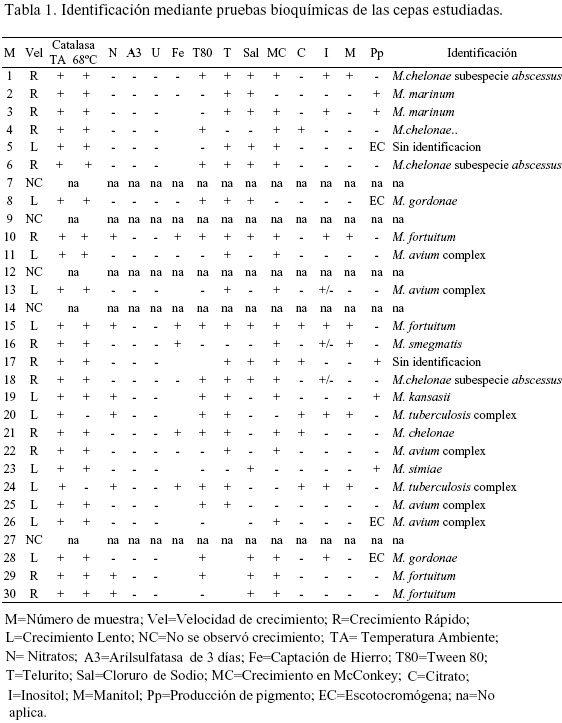
Identificación mediante pruebas bioquímicas
De las 30 cepas sub-cultivadas en medio L-J, se obtuvo crecimiento en 25 (83.3%), a las cuales se les realizaron las pruebas bioquímicas. Los resultados se resumen en la tabla 1. De los 25 aislados se identificaron 23, correspondiente a un 92%. Dos cepas (5 y 17) no pudieron ser identificadas por este método debido a que no se observó suficiente crecimiento en los medios o por que los resultados obtenidos en las distintas pruebas no eran concordantes con ninguna especie. La cepa No. 5, era una micobacteria de crecimiento lento y escotocromógena, mientras que la No. 17 era de crecimiento rápido y fotocromógena.
De acuerdo con la identificación bioquímica, las cepas más abundantes en nuestra muestra pertenecían al complejo M. avium (5 cepas, 21,7%), seguidas por M. fortuitum (4 cepas, 17,4%), M. chelonae subespecie abscessus (3 cepas, 13%), M. marinum (2 cepas, 8,7%), M. gordonae (2 cepas, 8,7%), el Complejo Tuberculosis (2 cepas, 8,7%), M. chelonae (2 cepas, 8,7%), M. smegmatis (1 cepa, 4,3 %), M. simiae (1 cepa, 4,3%), y M. kansasii (1 cepa, 4,3%). Es importante aclarar que las pruebas bioquímicas no permiten distinguir entre especies pertenecientes al mismo complejo, y tampoco permiten identificar especies recientemente descritas.
Identificación mediante PRA
Cuando se comparan los resultados de identificación obtenidos por ambos métodos, las pruebas bioquímicas y el PRA realizadas en el IMT hubo coincidencia en 18 de las 22 cepas que fueron identificadas por ambos métodos, lo cual constituye un 81% de coincidencia (Tablas 1 y 2).
Los aislados cuya identificación molecular no coincidió con la identificación bioquímica fueron el No. 16 identificado como M. smegmatis por pruebas bioquímicas y M. simiae por PRA, el No. 18 identificado como M. chelonae subespecie abscessus por pruebas bioquímicas M. kansasii por PRA, el No. 19 identificado como M. kansasii por pruebas bioquímicas y M. chelonae por PRA, y el No. 21 identificado como M. chelonae por pruebas bioquímicas y M. abscessus tipo 1 por PRA. (Figura 1).
Comparación de resultados de identificación obtenidos en el IMT con los reportados por el Laboratorio de Tuberculosis del Instituto de Biomedicina.
En la Tabla 3 se comparan los resultados de identificación obtenidos en el IMT, con los reportados por el laboratorio de Tuberculosis del Instituto de Biomedicina (LTIB) para las mismas cepas, los cuales fueron obtenidos previamente y de manera independiente mediante el análisis de PRA.
En cuanto a la comparación con los resultados obtenidos por las pruebas bioquímicas tradicionales, la técnica de PRA realizada en el LTIB coincidió en 18 de las 23 cepas (78,2%) que pudieron ser identificadas por métodos bioquímicos. Este valor es similar al que se obtuvo cuando se compararon los resultados del PRA y de las pruebas bioquímicas realizadas en el IMT.
En total, de las 22 cepas para las cuales se pudieron obtener resultados de identificación mediante los 2 métodos empleados en el IMT, 15 fueron identificadas de forma definitiva en este estudio, ya que se encontró concordancia entre los resultados obtenidos en el IMT y los correspondientes al LTIB, lo cual constituye un 68% de éxito en la identificación.
Las infecciones causadas por micobacterias no tuberculosas constituyen un problema de salud de importancia, como lo demuestra el creciente número de reportes en la literatura médica. En muchos casos el diagnóstico de estas infecciones se ve limitado por las características clínicas, que las hacen indiferenciables de otras infecciones incluidas las causadas por M. tuberculosis. La identificación del organismo causal es esencial para la instauración de un tratamiento adecuado, que permita una rápida recuperación del paciente.
Discusión
Convencionalmente la identificación de micobacterias no tuberculosas se lleva a cabo mediante pruebas que permiten detectar características bioquímicas y microbiológicas.
Estas pruebas son extensivamente utilizadas, pero presentan importantes limitaciones que contribuyen al problema de identificación y de manejo clínico de las micobacteriosis. Las pruebas bioquímicas son poco reproducibles, ya que la expresión de un fenotipo determinado depende de condiciones ambientales y por lo tanto presenta una marcada variabilidad. Así, la metodología puede presentar diferentes resultados al modificarse factores tales como la preparación de los medios y reactivos, la cantidad y tiempo de crecimiento de la cepa a identificar, e incluso el observador, ya que se requiere de un ojo entrenado y un cierto grado de subjetividad. Todo ello dificulta notablemente la interpretación de los resultados obtenidos. Otra importante limitante de los métodos bioquímicos es que, como se requiere de un cierto número de bacterias viables para producir un resultado detectable, el tiempo necesario para obtener un resultado puede ser de varias semanas, particularmente en especies de crecimiento lento. Además las bases de datos de las características fenotípicas se encuentran limitadas a las especies más comunes [23] y no son actualizadas con frecuencia, por lo que las especies recientemente descritas no pueden ser identificadas por estos métodos. Finalmente y no menos importante, la aplicación de estas técnicas involucra un alto riesgo de bioseguridad para el personal, pues se trabaja en todo momento con bacterias vivas. Las pruebas moleculares, basadas en la detección de características genotípicas, surgen como respuesta a muchas de estas limitaciones, y se ha reportado que las mismas permiten una identificación más rápida, confiable y sensible de las micobacterias no tuberculosas. Durante el desarrollo de este trabajo, se pudieron constatar en la práctica las ventajas de la técnica de PRA frente a las pruebas bioquímicas. Algunas de estas ventajas incluyen el corto periodo de tiempo requerido para la obtención de resultados, la relativa facilidad de realización e interpretación de la prueba, y la limitada manipulación de material infectante que se traduce en disminución de riesgo biológico.
En relación al PRA, la principal dificultad que encontramos tuvo que ver con la determinación del tamaño de las bandas presentes en los patrones de restricción, ya que como no contábamos con un software de análisis de imágenes, la misma se realizó de forma manual. En algunos casos el tamaño de las bandas no coincidía con los patrones publicados, dificultándose la identificación definitiva especialmente cuando se trata de patrones muy similares con pequeñas diferencias de tamaño (10-5 pb) entre bandas. Esta limitante se hacía particularmente importante en las bandas de bajo peso molecular, debido a la interferencia con el azul de bromofenol del buffer de carga, y la baja resolución del gel de agarosa en estas regiones. Este tipo de limitaciones ha sido descrita en algunos trabajos publicados, como es el caso de las investigaciones realizadas por Araya y col. [24] y Leao et al [25] en donde se expone que los errores o desventajas encontrados en el PRA están relacionados con la interpretación de los patrones, por lo que recomiendan la estandarización de las condiciones de la electroforesis (concentración del gel, tipo de agarosa utilizada y tiempos de corrida) y la utilización de software de medición de bandas para facilitar así la interpretación de los resultados. Así mismo, dicha dificultad a la hora de identificar bandas con pequeñas diferencias en el tamaño podría corregirse utilizando geles de poliacrilamida (PAGE) como se explica en la investigación realizada por Brunello et al [26].
Desde el punto de vista técnico, también observamos que la cantidad de biomasa utilizada para la extracción del ADN afecta la realización del PRA, ya que cuando se toma demasiada (o muy poca) cantidad de material no se logra amplificación. Esto puede deberse a que, al haber una gran cantidad las células, las mismas no pueden ser lisadas con facilidad durante el tiempo de incubación, o a un aumento de la cantidad de inhibidores de la reacción de amplificación. Es importante notar que este es el único paso de la técnica que es hasta cierto punto subjetivo, ya que es difícil establecer una cantidad óptima, medible y fija de material a tomar para el aislamiento de ADN. Además nuestra experiencia es que esta cantidad depende también del tipo de colonia (seca, incrustada, mucosa, etc.) que se esta estudiando.
En relación al grado de dificultad, si bien ambos métodos necesitan un adiestramiento previo, las pruebas bioquímicas son más laboriosas y requieren de una inversión de tiempo considerable porque deben ser observadas prácticamente de forma diaria por varias semanas, además de lo engorroso de trabajar con material contaminante por largos períodos de tiempo. La técnica de PRA requiere de 2-3 días de trabajo a lo sumo, y sólo se mantiene contacto con material infectante durante la extracción de ADN. La técnica utiliza menor número de soluciones y las mismas pueden ser preparadas con anterioridad en grandes volúmenes, y almacenadas por largos períodos de tiempo.
Las pruebas bioquímicas también resultan más costosas que el PRA, debido a que en éste la inversión inicial en los equipos necesarios para realizar la técnica es en donde radicaría el mayor gasto. Así mismo, las pruebas bioquímicas necesitan de la preparación de numerosos medios de cultivo y reactivos, y suelen requerir varias repeticiones de cada prueba, bien sea por contaminación o por problemas de crecimiento, así como su descontaminación y limpieza posterior. Esta sería otra de las muchas ventajas del PRA frente a las pruebas bioquímicas, las cuales ya se encuentran documentadas [27-29]. Se ha calculado que el PRA es 50% mas económico que las pruebas bioquímicas tomando en cuenta sólo los reactivos utilizados para cada una de las técnicas [28].
Nuestros resultados demuestran que el PRA permite obtener una identificación definitiva (a partir de colonias aisladas) en 2 días aproximadamente, mientras que las pruebas bioquímicas podrían demorar hasta 7 semanas. Esta importante ventaja del PRA con respecto a las pruebas bioquímicas, permite suministrar en un tiempo menor el tratamiento adecuado al paciente, esto ha sido ampliamente reportado [24,28-33], en donde coinciden que dicho método molecular es mucho más rápido comparado con el estándar de diagnóstico representado por las pruebas bioquímicas, lo que se traduce en el beneficio del paciente. Como se discutió previamente, el menor tiempo constituye una de las mas importantes ventajas operativas de la técnica de PRA.
En nuestro trabajo, la técnica de PRA permitió identificar un porcentaje mayor de cepas (96,2%) en comparación con las pruebas bioquímicas (92%), lo que demuestra una vez mas la ventaja de esta técnica como método de identificación, comparado con las pruebas bioquímicas tradicionales, ya que adicional a las ventajas en cuanto a tiempo de obtención de resultados , costo, seguridad, objetividad y facilidad de implementación que ya fueron discutidas, se pueden identificar un mayor número de aislados a nivel de especie.
En cuanto a la concordancia entre las pruebas bioquímicas y el PRA realizados en el IMT, se encontraron incongruencias en 4 de las 22 cepas analizadas por ambos métodos, resultando en un 82% de concordancia en los resultados. Estas cifras son similares a lo publicado recientemente donde el 74% de los resultados de identificación por PRA coincidieron con los resultados de las pruebas bioquímicas en un estudio de 434 aislados clínicos [33].
En relación a los aislados que presentaron resultados incongruentes entre las dos metodologías, en 2 de ellos (16 y 21) los resultados del PRA realizado en el IMT coincidieron con los obtenidos en el LTIB, mientras que en los otros 2 (18 y 19), fueron los resultados de las pruebas bioquímicas los que coincidieron con el PRA realizado en el LTIB. En el caso de la cepa 16, la misma fue identificada por pruebas bioquímicas como de crecimiento rápido (M. smegmatis ) no cromogénica, mientras que M. simiae es de crecimiento lento y fotocromógena (ver Tabla No. 1). La cepa 21 era de crecimiento rápido y no cromogénica (M. chelonae ) , al igual que M. abscessus, pero a diferencia de esta especie, su crecimiento fue negativo en NaCl, positivo en citrato y negativo en inositol y manitol. Es poco probable que exista confusión en los patrones de PRA de estas especies, ya que los mismos son bastante diferentes (ver tabla No. 2): para M. smegmatis BstEII 235/130/85 y HaeIII 145/125/60 contra M. simiae BstEII 235/210 y HaeIII 185/130; para M. chelonae BstEII 320/130 y HaeIII 200/60/55/50 contra M. abscessus tipo 1 BstEII 235/210 y HaeIII 145/70/60/55. Es por ello que es posible pensar, que en el caso de las cepas 16 y 21, las pruebas bioquímicas dieron una identificación errónea o equivocada, ya que, tal como se discutió previamente, estas pruebas son susceptibles de error, bien sea por la variación intrínseca de las mismas, fallas de los reactivos, errores de interpretación, etc. Por ejemplo, la tasa de crecimiento podría verse influenciada por la cantidad de inóculo inicial presente en la muestra, el medio de cultivo y la edad del aislamiento original.
En cuanto a la comparación de los resultados obtenidos por PRA y pruebas bioquímicas con los reportados por el laboratorio de Tuberculosis del Instituto de Biomedicina (LTIB) se obtuvo una concordancia de 16 de las 22 cepas analizadas por ambos métodos en el IMT. Los aislados 13 y 25, que fueron identificados como M. interjectum en el LTIB, mostraron un patrón de PRA, así como una identificación bioquímica correspondiente a M. avium en nuestro trabajo. La concordancia de las técnicas fenotípicas y genotípicas, nos llevó a revisar una vez mas los patrones de PRA, concluyendo que el patrón obtenido en nuestro caso se asemeja más al patrón de M. avium (BstEII 235/210 y HaeIII 130/105) que al de M. interjectum (BstEII 235/210 y HaeIII 145/130).
Los aislados 18 (M. abscessus) y 19 (M. kansasii) son un caso particular, puesto que, si bien se obtuvo un patrón de PRA no coincidente, las pruebas bioquímicas coinciden con los resultados reportados por el LTIB. En cuanto a la cepa 9 (M. chelonae), las características bacteriológicas no permiten aclarar el panorama, puesto que no se obtuvo crecimiento en el subcultivo de esta cepa. El aislado 17 (M. kansasii) presentó crecimiento rápido, como M. chelonae, pero también era fotocromógena como M. kansasii.
Llama la atención que la mayor parte de las diferencias, entre los 2 laboratorios, se refieren a la identificación de M. chelonae y M. kansasii. Estas dos especies micobacterianas son fácilmente distinguibles por sus características bacteriológicas, porque M. chelonae es una micobacteria de crecimiento rápido y no pigmentada, mientras que M. kansasii es de crecimiento rápido y fotocromógena. Desde el punto de vista genotípico, los patrones de PRA de ambas especies son bastante distintos también, siendo para M. chelonae BstEII 320/130 y HaeIII 200/60/55/50 y para M. kansasii BstEII 235/210 y HaeIII 130/105/80. Sin embargo, la complejidad de los resultados obtenidos en este trabajo comprueba la dificultad de la identificación de micobacterias en los aislados clínicos.
Finalmente, y si bien no contábamos con un número estadísticamente significativo de muestras para hacer un cálculo de la sensibilidad y especificidad, la mayoría de los resultados coincidieron por las técnicas tradicionales y moleculares, y con los resultados previos de identificación suministrados por el LTIB. Esto nos confirma una vez mas que el PRA es un método confiable, sensible y preciso, que comparado con el estándar de diagnóstico como son las pruebas bioquímicas, produce mejores resultados en un menor tiempo.
Nuestros resultados nos permiten sugerir, que las pruebas bioquímicas podrían continuar siendo de utilidad como método confirmatorio, secundario al PRA, sobre todo en aquellos casos donde el patrón de restricción obtenido no se encuentra publicado en la base de datos, o es muy similar a otro, o cuando existen dudas por incongruencias con otras características del aislado.
Finalmente podemos concluir que el método de PRA posee múltiples ventajas en comparación con las pruebas bioquímicas convencionales, y que por ello se perfila como un método de elección para la identificación de micobacterias no tuberculosas, que proporciona resultados rápidos y confiables. Los resultados de esta investigación podrían contribuir a mejorar el diagnóstico de las micobacteriosis en Venezuela, permitiendo así una pronta implementación del tratamiento adecuado para los pacientes afectados.
Referencias
1. Sañudo A, Vallejo F, Sierra M, Hoyos JG, Yepes S, Wolff JC et al. Nontuberculous mycobacteria infection after mesotherapy: preliminary report of 15 cases. Int J Dermatol. 2007; 46:649-53. [ Links ]
2. Rivera-Olivero IA, Guevara A, Escalona A, Oliver M, Pérez-Alfonzo R, Piquero J, y col. Infecciones en tejidos blandos por micobacterias no tuberculosas secundarias a mesoterapia. ¿Cuánto vale la belleza?. Enferm Infecc Microbiol Clin. 2006; 24:302-6. [ Links ]
3. Nagore E, Ramos P, Botella-Estrada R, Ramos-Níguez JA, Sanmartín O, Castejón P. Cutaneous infection with Mycobacterium fortuitum after localized microinjections (mesotherapy) treated successfully with a triple drug regimen. Acta Derm Venereol. 2001; 81:291-3. [ Links ]
4. Sampaio JL, Chimara E, Ferrazoli L, da Silva Telles MA, Del Guercio VM, Jericó ZV. Application of four molecular typing methods for analysis of Mycobacterium fortuitum group strains causing post-mammaplasty infections. Clin Microbiol Infect. 2006; 12:142-9. [ Links ]
5. Thami GP, Kaur S, Chander J, Attri AK. Post surgical atypical mycobacterial infection due to Mycobacterium fortuitum. J Infect. 2002; 45:210-11. [ Links ]
6. Moshirfar M, Meyer JJ, Espandar L. Fourth-generation fluoroquinolone-resistant mycobacterial keratitis after laser in situ keratomileusis. J Cataract Refract Surg. 2007; 33:1978-81. [ Links ]
7. Alvarenga L, Freitas D, Hofling-Lima AL, Belfort R Jr, Sampaio J, Sousa L, et al. Infectious post-LASIK crystalline keratopathy caused by nontuberculous mycobacteria. Cornea. 2002; 21:426-9. [ Links ]
8. Le Bourgeois M, Sermet-Gaudelus I, Catherinot E, Gaillard JL. Nontuberculous mycobacteria in cystic fibrosis. Arch Pediatr. 2005; 12 Suppl 2:S117-21. [ Links ]
9. Jordan PW, Stanley T, Donnelly FM, Elborn JS, McClurg RB, Millar BC, et al. Atypical mycobacterial infection in patients with cystic fibrosis: update on clinical microbiology methods. Lett Appl Microbiol. 2007; 44:459-66. [ Links ]
10. Girón RM, Domingo D, Buendía B, Antón E, Ruiz-Velasco LM, Ancochea J. Nontuberculous mycobacteria in patients with cystic fibrosis. Arch Bronconeumol. 2005; 41(10):560- 65. [ Links ]
11. Griffith DE. Emergence of nontuberculous mycobacteria as pathogens in cystic fibrosis. Am J Respir Crit Care Med. 2003; 167:810-2. [ Links ]
12. Miguez-Burbano MJ, Flores M, Ashkin D, Rodriguez A, Granada AM, Quintero N, et al. Non-tuberculous mycobacteria disease as a cause of hospitalization in HIV-infected subjects. Int J Infect Dis. 2006; 10:47-55. [ Links ]
13. Raszka WV Jr, Skillman LP, McEvoy PL, Robb ML. Isolation of nontuberculous, non-avium mycobacteria from patients infected with human immunodeficiency virus. Clin Infect Dis. 1995; 20:73-6. [ Links ]
14. Telenti A, Marchesi F, Balz M, Bally F, Böttger EC, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J Clin Microbiol. 1993; 31:175-8. [ Links ]
15. Martin A, Uwizeye C, Fissette K, De Rijk P, Palomino JC, Leao S, et al. Application of the hsp65 PRA method for the rapid identification of mycobacteria isolated from clinical samples in Belgium. J Microbiol Methods. 2007; 71:39-43. [ Links ]
16. Kim H, Kim SH, Shim TS, Kim MN, Bai GH, Park YG, et al. PCR restriction fragment length polymorphism analysis (PRA)-algorithm targeting 644 bp Heat Shock Protein 65 (hsp65) gene for differentiation of Mycobacterium spp. J Microbiol Methods. 2005; 62:199-209. [ Links ]
17. Bahrmand AR, Bakayeva TG, Bakayev VV. Use of restriction enzyme analysis of amplified DNA coding for the hsp65 gene and polymerase chain reaction with universal primer for rapid differentiation of mycobacterium species in the clinical laboratory. Scand J Infect Dis. 1998; 30:477-80. [ Links ]
18. Devallois A, Goh KS, Rastogi N. Rapid identification of mycobacteria to species level by PCR-restriction fragment length polymorphism analysis of the hsp65 gene and proposition of an algorithm to differentiate 34 mycobacterial species. J Clin Microbiol. 1997; 35:2969-73. [ Links ]
19. de Magalhães VD, de Melo Azevedo Fda P, Pasternak J, Valle Martino MD. Reliability of hsp65-RFLP analysis for identification of Mycobacterium species in cultured strains and clinical specimens. J Microbiol Methods. 2002; 49:295- 300. [ Links ]
20. Kim H, Kim SH, Shim TS, Kim MN, Bai GH, Park YG, et al. Differentiation of Mycobacterium species by analysis of the heat-shock protein 65 gene (hsp65). Int J Syst Evol Microbiol. 2005; 55:1649-56. [ Links ]
21. Isenberg H.D. (editor). Clinical Microbiology Procedures Handbook. Washington DC: American Society for Microbiology, 1992; pp 3.0.1-3.16.3. [ Links ]
22. De Waard J, Da Mata O. PRA Manuel de Identificación de Micobacterias Atípicas. Instituto de Biomedicina, Laboratorio de Tuberculosis. [ Links ]
23. Springer B, Stockman L, Teschner K, Roberts GD, Böttger EC. Two-laboratory collaborative study on identification of mycobacteria: molecular versus phenotypic methods. J Clin Microbiol. 1996;34:296-303. [ Links ]
24. Araya P, Velazco M, Fernandez J. Identificación rápida de micobacterias no tuberculosas mediante análisis de patrones de restricción. Rev. Méd. Chile. 2006; 134: 868-73. [ Links ]
25. Leao SC, Bernardelli A, Cataldi A, Bernardelli A, Cataldi A, Zumarraga M, Robledo J, Realpe T, et al. Multicenter evaluation of mycobacteria identification by PCR restriction enzyme analysis in laboratories from Latin America and the Caribbean. J Microbiol Methods. 2005; 61:193-9. [ Links ]
26. Brunello F, Ligozzi M, Cristelli E, Bonora S, Tortoli E, Fontana R. Identification of 54 mycobacterial species by PCR-restriction fragment lengthpolymorphism analysis of the hsp65 gene. J Clin Microbiol. 2001;39:2799-806. [ Links ]
27. Da Silva C, Ueki S, Geiger D, Leão S. Hsp65 PCRrestriction enzyme analysis (PRA) for identification of mycobacteria in the clinical laboratory. Rev. Inst. Med. Trop. S. Paulo. 2001; 43: 25–8. [ Links ]
28. Prammananan T, Cheunoy W, Na-Ubol P, Tingtoy N, Srimuang S, Chaiprasert A. Evaluation of polymerase chain reaction and restriction enzyme analysis for routine identification of mycobacteria: accuracy, rapidity, and cost analysis. Southeast Asian J Trop Med Public Health. 2005; 36: 1252-60. [ Links ]
29. Castro CM, Puerto G, García LM, Orjuela DL, Polo CL, Garzón MC, y col. Identificación molecular de micobacterias no tuberculosas. Biomedica. 2007; 27:439-46. [ Links ]
30. Cheunoy W, Prammananan T, Chaiprasert A, Foongladda S. Comparative evaluation of polymerase chain reaction and restriction enzyme analysis: two amplified targets, hsp65 and rpoB, for identification of cultured mycobacteria. Diagn Microbiol Infect Dis. 2005; 51: 165-71. [ Links ]
31. Cormican MG, Glennon M, Ni Riain U, Flynn J. Evaluation of a PCR based method for identification of mycobacterial isolates. Ir J Med Sci. 1995; 164: 20-3. [ Links ]
32. Wang SX, Tay L, Sng LH. Rapid identification of pathogenic rapidly growing mycobacteria by PCR-restriction endonuclease analysis. Ann Acad Med Singapore. 2005; 34: 137-40. [ Links ]
33. Chimara E, Ferrazoli L, Ueky SY, Martins MC, Durham AM, Arbeit RD, et al. Reliable identification of mycobacterial species by PCR-restriction enzyme analysis (PRA)- hsp65 in a reference laboratory and elaboration of a sequence- based extended algorithm of PRA-hsp65 patterns. BMC Microbiol. 2008; 20;8:48. [ Links ]












 uBio
uBio