Revista de la Sociedad Venezolana de Microbiología
versión impresa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. v.31 n.2 Caracas dic. 2011
Evaluación del tiempo de vida de bacterias potencialmente patógenas en sedimentos marinos
Adrián J. Acuña*, Cecilia F. Torres, Graciela N. Pucci, Oscar H. Pucci
Centro de Estudios e Investigación en Microbiología Aplicada (CEIMA). Facultad de Ciencias Naturales. Universidad Nacional de la Patagonia San Juan Bosco. Comodoro Rivadavia. Argentina.
* Correspondencia: E-mail: ajcuna@unpata.edu.ar
Resumen
El presente trabajo tuvo por objeto determinar el período de supervivencia en sedimentos marinos de bacterias con riesgo biológico o indicadoras. Se tomaron muestras de sedimento marino intermareal en dos playas de la ciudad de Comodoro Rivadavia, Argentina; una sometida a contaminación por efluentes domiciliarios, y otra sin pruebas de contaminación evidente. Sobre éstos se realizó una caracterización inicial por recuento de bacterias en diferentes medios de cultivo (TSA, VRB, VRBG, TCBS, agar bilis esculina y agar cetrimide) y por su identificación. También se realizó un estudio de supervivencia de los microorganismos de interés en microcosmos. Estos fueron monitoreados por conteo de bacterias y por identificación de los integrantes de interés. Los principales resultados indicaron que existe una marcada diferencia en la composición de la comunidad bacteriana entre ambas muestras. Los géneros bacterianos en la muestra contaminada fueron Aeromonas, Salmonella, Pseudomonas, Escherichia, Ochrobactum y Klebsiella, y en la playa no contaminada Bacillus, Paenibacillus, Micrococcus, Photobacterium y Vibrio. El ensayo en microcosmos demostró que, luego de 65 días, la comunidad bacteriana de la playa contaminada fue similar a la del sedimento sin contaminación, indicando que al retirar la fuente de contaminación, es probable la recuperación de la zona afectada.
Palabras clave: comunidad bacteriana, microcosmos, sedimento marino, Patagonia.
Evaluation of life-span of potentially pathogenic bacteria in marine sediments
Abstract
The present study had the purpose of determining the survival period of bacteria which represent a biological risk and are indicators of contamination. Inter-tide marine sediment samples were taken at two beaches of the Comodoro Rivadavia city in Argentine; one the beaches was submitted to contamination due to household effluents, and the other without proof of evident contamination. These samples were initially characterized by bacterial counts in different culture media (TSA, VRB, TCBS, bile esculin agar and cetrimide agar) and by their identification. A study of the survival of the target bacteria in microcosm was also done, monitored by bacterial counts and identification of these bacteria. The main results indicated that there was a marked difference in the composition of the bacterial community between the two samples. In the contaminated sample the bacterial genus were Aeromonas, Salmonella, Pseudomonas, Escherichia, Ochrobactum and Klebsiella, and in the non-contaminated beach Bacillus, Paenibacillus, Micrococcus, Photobacterium and Vibrio. The microcosm assay showed that after 65 days the bacterial community of the contaminated beach was similar to that of the non-contaminated beach sediment, indicating that by withdrawing the contamination source, the affected zone can probably be recovered.
Keywords: bacterial community, microcosm, marine sediment, Patagonia.
Recibido 26 de mayo de 2011; aceptado 12 de septiembre de 2011
Introducción
El mar es considerado como un gran vertedero en donde una serie de mecanismos físicos, químicos y biológicos pueden conseguir la depuración de los desechos que se arrojan en él. Sin embargo, en la actualidad la autodepuración del mar parece no ser efectiva por lo que la contaminación ocasiona un desequilibrio del ambiente marino [1].
Aunque el mar podría representar un entorno poco favorable para el desarrollo de agentes patógenos y bacterias que habitan el intestino humano, resulta un ambiente propicio para la supervivencia de diversos microorganismos, entre ellos, las enterobacterias y enterococos [2].
Por otra parte, al ingresar las enterobacterias al ambiente marino, se ven obligadas a interactuar con la comunidad bacteriana indígena, un hábitat muy diferente del cual provienen, especialmente por la agresividad de la salinidad contenida en el agua de mar [3].
Del número total de especies que componen a una comunidad bacteriana determinada, con frecuencia una pequeña porción es abundante o dominante (representado por un gran número de individuos, biomasa, productividad, etc.), y es posible decir que son estas especies las que con frecuencia caracterizan a dicha comunidad [4]. Es de esperar que al incorporar enterobacterias al medio, éstas interactúen generando disturbios en la dinámica normal de la comunidad original y podremos utilizar dichas especies para comparar las diferencias que surjan en la comunidad bacteriana antes, durante y luego de eliminar la fuente de contaminación [3,5].
La ciudad de Comodoro Rivadavia, provincia de Chubut, Argentina, vierte diversos efluentes en el cuerpo de agua más cercano, en este caso el mar. El grado de salud ambiental que presenta se puede dividir en dos estados, uno en el cual el hombre ha ejercido una escasa influencia y un segundo estado en donde los disturbios generados por la contaminación resultan evidentes [6]. En la serie de playas que se enumeran en las costas de la ciudad se puede encontrar los dos escenarios planteados anteriormente [7,8].
Teniendo en cuenta que se trata de una ciudad cuyas playas son de uso recreativo para la población, es de interés conocer el tiempo que resultaría necesario para que la presencia de bacterias patógenas o potencialmente patógenas para la salud humana sean eliminadas del ambiente.
El presente trabajo tiene por objeto determinar el período de supervivencia en sedimentos marinos de bacterias que impliquen riesgo biológico o bacterias indicadoras (Enterobacterias, E. coli, Pseudomonas, enterococos) mediante su estudio en microcosmos.
Materiales y métodos
Toma de muestras: Se seleccionaron dos puntos de muestreo de los cuales se extrajeron los primeros 10 cm de sedimento marino intermareal. El primero se encuentra situado en la Playa del 99 (S99) a 45°5259,89 de latitud sur y 67°3021,97 de longitud oeste en la ciudad de Comodoro Rivadavia, provincia de Chubut, Argentina, cuya muestra corresponde con un sedimento marino sometido a líquidos provenientes de efluentes cloacales. El segundo punto se encuentra situado en la Playa del Límite (SL) a 45°5940,09 de latitud sur y 67°3521,32 de longitud oeste, a unos 30 km al sur de Comodoro Rivadavia, cuya muestra corresponde con el sedimento no expuesto a efluentes cloacales. De cada punto de muestreo se tomaron tres muestras puntuales de cinco kilogramos con las que se realizó una muestra compuesta sobre la que se realizaron las experiencias.
Análisis microbiológico inicial:
Recuento de bacterias: Se realizó en ambas muestras por la técnica de recuento en placa. Para bacterias heterótrofas se utilizó en agar TSA (Britania®), para bacterias coliformes fecales agar VRB (Britania®), para bacterias coliformes totales agar VRBG (Britania®), para Vibrio agar TCBS (Britania®), para Enterococcus agar bilis esculina, BE, (Britania®) y para Pseudomonas aeruginosa agar cetrimide (Britania®). El tiempo de incubación fue de 24 h a 37 ºC.
Identificación de bacterias: A partir de los recuentos bacterianos realizados, se aislaron en medio TSA cepas autóctonas hasta cultivo puro, para su posterior identificación por ácidos grasos de membrana (FAMEs). La extracción se realizó sobre 40 mg de bacterias (peso húmedo) crecidas en la tercera estría en medio TSBA (tripteína 15 g, peptona de soya 5 g, cloruro de sodio 5 g, agar 15 g, glucosa 2,5 g, agua destilada 1000 ml, pH 7) cultivadas a 28 ºC durante 24 h, efectuando una saponificación con alcohol metílico-hidróxido de sodio-agua (150 mL:45 g:150 mL) seguida de una metilación con ácido clorhídrico 6N y alcohol metílico (325 :275 mL) y a continuación una extracción con n-hexano-metil térbutil éter (1:1) y lavado con hidróxido de sodio-agua (10,8 g-900 mL) de acuerdo con el procedimiento del sistema de identificación (MIDI Newark, Del., USA). Las muestras fueron almacenadas a -20 °C hasta su análisis [9].
Los ácidos grasos se determinaron como metil ésteres por cromatografía gaseosa usando una columna capilar Ultra 2 de 25 m de longitud, 0,2 mm de diámetro. El análisis se llevó a cabo con un cromatógrafo HP 6890 series II GC (inyección splitless; presión inicial 10 psi; programa de temperatura: 170-288 °C a 28 °C min-1, 288-310 °C a 60 °C min-1, 1,5 min de permanencia a 310 °C, detector por ionización de llama), la integración de los picos se efectuó mediante HP 10.01 Chem Station y los ácidos grasos fueron identificados utilizando Sherlock (versión 6.0) con el estándar Agilent Calibration standards kit for the microbial identification system. La composición en ácidos grasos fue calculada como porcentaje del área de pico [10].
Estudio de supervivencia de bacterias: Se confeccionaron cuatro microcosmos por triplicado utilizando 100 g de sedimento para cada uno. Tres correspondieron a la muestra SL, para lo que se realizó uno como sistema control (SL) conteniendo sólo el sedimento. Otro al que se le incorporó al sedimento marino un inóculo bacteriano (SLI) y un tercer sistema en el que se esterilizó el sedimento en autoclave durante 15 min a una atmósfera de presión, al que luego se le incorporó un inóculo bacteriano (SLIE). El inóculo bacteriano utilizado fue preparado a partir de cultivos de 24 h, conformado por cepas representativas de Escherichia coli, Pseudomonas aeruginosa, Enterococcus sp. y Vibrio meschnikevii, todas aisladas anteriormente a partir de la muestra S99. Para ello se realizaron suspensiones individuales de cada microorganismo en solución fisiológica hasta una turbidez correspondiente a 0,5 Mc Farland (108 UFC.mL-1), luego se colocaron 2 mL de cada suspensión bacteriana en un frasco estéril, se homogeneizó y se agregó a cada uno de los dos sistemas correspondientes. El cuarto microcosmo se realizó sólo con el sedimento de la playa sometida a los efluentes cloacales (S99). Los sistemas se incubaron durante 65 días a 10 °C en oscuridad. El seguimiento de los mismos se realizó por conteo en placa en agar TSA, VRB, VRBG, TCBS, BE y Cetrimide, y por aislamiento e identificación de bacterias por FAMEs. Con los datos de las identificaciones bacterianas se calculó el índice de diversidad de Shannon con el programa PAST.
Análisis estadístico: Para conocer como fue la variación de la comunidad bacteriana en función del tiempo, se realizaron análisis de agrupamiento y componentes principales con los microorganismos identificados en las muestras estudiadas a los diferentes tiempos de muestreo. Todos los análisis se realizaron utilizando el programa PAST.
Resultados
Análisis microbiológico inicial: El sedimento S99 mostró un mayor número de microorganismos en todos los medios de cultivo utilizados con respecto al sedimento SL (Tabla 1). La técnica de ácidos grasos permitió identificar en S99 los géneros bacterianos Aeromonas, Salmonella, Pseudomonas, Escherichia, Ochrobactrum y Klebsiella, y dentro de estos las especies Escherichia fergusonii, Ochrobactrum anthropi y Klebsiella pneumoniae. Por otro lado, en SL se observaron los géneros bacterianos Bacillus, Paenibacillus, Micrococcus, Photobacterium y Vibrio, y dentro de ellos las especies Photobacterium damselae y Vibrio alginolyticus.
Tabla 1. Población bacteriana (UFC.gr-1.102) inicial en los sedimentos intermareales. Comodoro Rivadavia, Argentina.
| Sedimento | TSA | VRB | VRBG | TCBS | BE | Cetrimide |
| SL | 13 ± 0,6 | 0 | 0 | 0 | 0 | 0 |
| S99 | 450 ± 24,7 | 6 | 22 ± 1,5 | 0 | 16 ± 1,1 | 0 |
SL: Playa del Límite, S99: Playa del 99, TSA: bacterias heterótrofas, VRB: coliformes fecales, VRBG: coliformes totales, TCBS: Vibrio, BE: enterococos, Cetrimide: Ps. aeruginosa.
Estudio de supervivencia de microorganismos: En el microcosmo SL, el número de bacterias heterótrofas se mantuvo en el orden de 103 UFC.g-1 durante todo el estudio, mientras que el resto de los grupos de microorganismos estudiados no presentaron desarrollo.
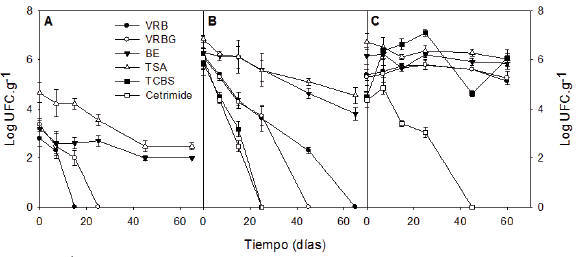
En el sistema S99 se observó una tendencia general a disminuir el número de colonias en todos los medios de cultivo utilizados (Figura 1.A). Los enterococos y las bacterias heterótrofas lo hicieron a una velocidad menor con respecto a los coliformes totales y fecales. La tasa de desaparición de microorganismos en el medio BE fue de un orden menor con respecto a los otros medios de cultivo.
Figura 1. Población bacteriana en los microcosmos. A corresponde al microcosmo S99, B al microcosmo SLI y C al microcosmo SLIE. El sistema SL no se incluyó en el gráfico ya que no hubo desarrollo en los medios de cultivo utilizados, a excepción de TSA. VRB: coliformes fecales, VRBG: coliformes totales, BE: Enterococos, TSA: bacterias heterótrofas, TCBS: Vibrio, Cetrimide: Ps. aeruginosa.
En el sistema SLI se observó, en los conteos bacterianos, una tendencia similar a lo mencionado para el microcosmo S99 (Figura 1.B). En este caso, nuevamente se encontró una mayor resistencia de los enterococos y bacterias totales, mientras que los otros grupos decaen a una mayor velocidad, de entre 1 y hasta 3 logaritmos de diferencia, con una rápida desaparición de microorganismos en los medios de cultivo Cetrimide y TCBS. Por otro lado, en el microcosmo SLIE el número de microorganismos se mantuvo en el tiempo (Figura 1.C), con excepción de Pseudomonas aeruginosa que decayó rápidamente, no encontrándose registros a partir del día 45. En este sistema, a diferencia de los anteriores, el grupo de los enterococos fue el que mostró una mayor tasa de muerte.
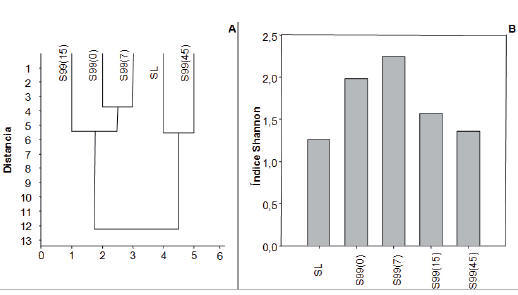
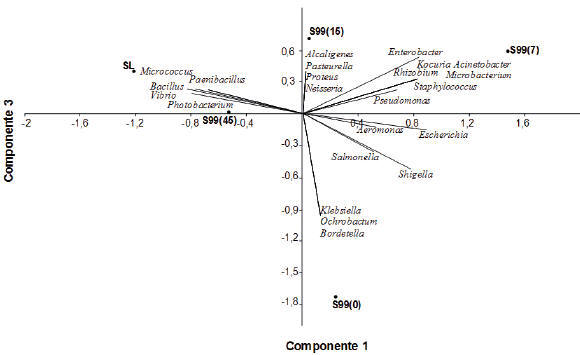
Se aislaron 129 cepas a partir de los microcosmos confeccionados con los sedimentos estudiados. Entre éstas se identificaron 26 géneros bacterianos diferentes y 18 especies. El análisis de agrupamiento realizado con los datos de los microcosmos SL y S99 (Figura 2.A) mostró que la comunidad bacteriana presente en el sedimento S99 en el día 45 es similar al del sistema SL. El índice de Shannon calculado en estos sistemas (Figura 2.B), se modificó durante la experiencia en S99, observándose que en el día 45 fue similar al índice calculado para SL. Por otro lado, en el análisis de componentes principales (Figura 3) se observó como, entre los días 0 y 7, la composición de la comunidad bacteriana de S99 estaba representada por grupos patógenos o considerados de riesgo para la salud humana. Hacia el día 15 los principales grupos cambiaron a géneros intermedios, y este reemplazo de géneros continuó hasta encontrar, en el día 45, una comunidad bacteriana muy similar a la observada en SL.
Figura 2. Análisis de agrupamiento (A) e Índice de Shannon (B) del microcosmo S99 a diferentes tiempos comparado con la situación inicial del sistema SL en función a los microorganismos identificados. Los números entre paréntesis indican el tiempo de incubación del microcosmo.
Figura 3. Análisis de componentes principales del microcosmo S99 a diferentes tiempos comparado con la situación inicial del sistema SL en función a las bacterias identificadas. Los números entre paréntesis indican el tiempo de incubación del microcosmo.
Discusión
Los efluentes cloacales descargados de forma continua y sin tratamiento en las costas marinas son una fuente permanente de microorganismos patógenos o potencialmente patógenos para la salud humana, volviendo éste un sitio con grandes posibilidades de ser un importante reservorio de genes de resistencia a antimicrobianos, los que pueden ejercer una acción negativa sobre la salud humana al utilizar estas playas con fines recreativos [2]. Pucci et al encontraron en la Playa del 99 una población para coliformes totales y coliformes fecales en el orden de 103 UFC.mL-1, demostrando que el vertido de estos efluentes de forma permanente hace que estos microorganismos se encuentren casi de forma constante [7]. Resultados similares fueron reportados por Ramos-Ortega et al en la Bahía de Santa Marta, Colombia, donde pudieron establecer la importancia del buen mantenimiento y diseño de los emisarios submarinos, en función de lograr una buena dilución de los efluentes domiciliarios vertidos en el cuerpo de agua [11]. Los sedimentos marinos son mejores para el mantenimiento de las bacterias entéricas, comparados con la columna de agua, porque protegen de la luz solar, de predadores, aumenta la biodisponibilidad de nutrientes y materia orgánica y provee de superficies colonizables por microorganismos [12-15].
En el sistema S99 no se observó desarrollo de los grupos bacterianos relacionados con Ps. aeruginosa y Vibrio. Esto pudo estar relacionado a lo observado en el sistema SLI, ya que en éste, estos grupos bacterianos fueron los primeros en desaparecer. Esto indicaría que en la Playa del 99, tanto Ps. aeruginosa como Vibrio, pueden ser incorporados por la contaminación antrópica, pero rápidamente pueden ser eliminados del medio ambiente [16]. La mortalidad de las bacterias estudiadas fue mayor en el sistema SLI que en S99. Esto se debió a que cuantificamos un número mayor de bacterias en SLI, debido a que el conteo inicial se ve incrementado por el inóculo incorporado. En S99 la población inicial de bacterias totales fue en el orden de 104 UFC.g-1, mientras que en SLI lo fue en el orden de 106 UFC.g-1. Las tasas de muerte mayores correspondieron a Pseudomonas, coliformes totales y en tercer lugar a Vibrio, mientras que los enterococos y coliformes fecales presentaron una mayor resistencia a la desaparición, exhibiendo mayor supervivencia.
Otros estudios, enfocados en variables abióticas (salinidad, pH, temperatura, etc.) que deben enfrentar las bacterias entéricas cuando son expuestas al ambiente marino concluyeron, que una vez liberadas al medio marino, la supervivencia de las mismas en éste depende directamente de su habilidad de superar la situación de estrés a las que se encuentran sometidas, demostrando con E. coli y Salmonella que la radiación UV es uno de los factores determinantes en su desaparición [3,17,18]. Numerosos géneros bacterianos han mostrado ser capaces de desarrollar dichas habilidades en sedimentos estériles, mientras que otras decaen rápidamente [19]. Con los resultados obtenidos en el sistema SLEI, en el cual los factores bióticos han sido suprimidos, especies como Ps. aeruginosa y V. meschnikevii parecen no desarrollar dicha capacidad de adaptación. Este sistema (SLIE) presentó un escenario diferente a SLI, ya que la desaparición correspondiente a enterococos resultó mayor que para coliformes fecales y totales. En este sistema, a diferencia de los anteriores, los microorganismos estudiados se encuentran compitiendo entre sí por los recursos pero no con la comunidad autóctona, con lo que podemos sugerir que dentro de los géneros relacionados con los efluentes cloacales, los enterococos poseen mecanismos de defensa para competir con los microorganismos autóctonos más eficientes que los coliformes fecales y totales.
Las bacterias entéricas decrecen más en aguas marinas naturales que en las estériles, debido a la interacción con los procesos biológicos naturales [20-22]. Las bacterias marinas autóctonas pueden utilizar la pared celular de bacterias entéricas como fuente de carbono [23]. En estudios previos se demostró que las bacterias entéricas son eliminadas del ambiente marino por competencia con los microorganismos autóctonos por los nutrientes o por la existencia de microorganismos que los parasitan o lisan [5,23].
La diversidad en S99 se modificó en función al tiempo, demostrando su mayor auge en el recuento del día 7. Debemos tener en cuenta que consideramos como contaminación antrópica al agregado de carga bacteriana a través de un efluente cloacal, por lo que en este caso, un aumento en la diversidad del ecosistema, nos está representando un escenario notablemente desfavorable. La tendencia, en el tiempo, según las mediciones del índice de Shannon en S99, fue disminuir hasta alcanzar una medida similar al índice calculado para SL, reduciéndose en una unidad desde su punto más alto. Esta disminución de la biodiversidad está relacionada con la desaparición de los microorganismos aportados por el efluente cloacal [16], logrando que la misma sea similar a la encontrada en el sedimento de la playa del límite.
De los estudios realizados, se puede inferir que al cortar la fuente de contaminación, las bacterias entéricas tienden a desaparecer del medio en períodos de tiempo considerablemente cortos. Las experiencias realizadas en microcosmos suelen actuar de forma similar pero no exactamente igual a lo que ocurre en los ecosistemas a gran escala, ya que la recuperación es rápida cuando se perturban áreas pequeñas [3].
Referencias
1. Atlas R, Bartha R. Ecología microbiana y microbiología ambiental, 4ta ed. Madrid: Pearson Addison-Wesley; 2002. [ Links ]
2. Al-Bahry SN, Mahmoud IY, Al-Belushi KI, Elshafie AE, Al-Harthy A, Bakheit CK. Coastal sewage discharge and its impact on fish with reference to antibiotic resistant enteric bacteria and enteric pathogens as bio-indicators of pollution. Chemosphere. 2009; 77:1534-9. [ Links ]
3. Rozen Y, Belkin S. Survival of enteric bacteria in seawater. FEMS Microbiol Rev. 2001; 25:513-29. [ Links ]
4. Süss J, B Engelen, H Cypionka, H Sass. Quantitative analysis of bacterial communities from Mediterranean sapropels based on cultivation depend methods. Microbiol Ecol. 2004; 51:109-21. [ Links ]
5. Jannasch HW. Competitive elimination of Enterobacteriaceae from seawater. Appl Microbiol. 1968; 16:1616-8. [ Links ]
6. Artiola JF, Pepper IL, Brusseau ML. Monitoring and characterization of the environment. In: Artiola JF, Pepper IL, Brusseau ML editors. Environmental monitoring and characterization. USA: Elsevier Academic Press; 2004. p. 1-9. [ Links ]
7. Pucci GN, Acuña AJ, Llanes ML, Tiedemann MC, Pucci OH. Identificación de bacterias cultivables de la ciudad costera Comodoro Rivadavia, Argentina. Rev Biol Mar Oceanogr. 2009; 44:49-58. [ Links ]
8. Pucci GN, Acuña AJ, Tonin N, Tiedemann MC, Pucci OH. Diversidad de bacterias cultivables con capacidad de degradar hidrocarburos de la playa de Caleta Córdova, Argentina. Rev Peru Biol. 2010; 17:237-44. [ Links ]
9. Härtig C, Loffhagen N, Harms H. Formation of trans fatty acids is not involved in growth-linked membrane adaptation of Pseudomonas putida. Appl Environ Microbiol. 2005; 71:1915-22. [ Links ]
10. Pucci GN, Pucci OH. Cambios en los ácidos grasos de membrana de Microbacterium esteraromaticum GNP-5 con diferentes temperaturas y osmolaridades. Acta Biol Colomb. 2006; 11:61-73. [ Links ]
11. Ramos-Ortega LM, Vidal LA, Vilardy S, Saavedra-Díaz L. Análisis de la contaminación microbiológica (coliformes totales y fecales) en la Bahía de Santa Marta, Caribe colombiano. Acta Biol Colomb. 2008; 13:87-98. [ Links ]
12. Sinton L, Finlay R, Lynch P. Sunlight inactivation of fecal bacteriophages and bacteria in sewage-polluted seawater. Appl Environ Microbiol. 1999; 65:3605-13. [ Links ]
13. Davies C, Bavor H. The fate of stormwater-associated bacteria in constructed wetland and water pollution control pond systems. J Appl Microbiol. 2000; 89:349-60. [ Links ]
14. Craig D, Fallowfield H, Cromar N. Use of macrocosms to determine persistence of Escherichia coli in recreational coastal water and sediment and validation with in situ measurements. J Appl Microbiol. 2004; 96:922-30. [ Links ]
15. Davies C, Long J, Donald M, Ashbolt N. Survival of fecal microorganisms in marine and freshwater sediment. Appl Environ Microbiol. 1995; 61:1888-96. [ Links ]
16. Darakas E, Koumoulidou T, Lazaridou D. Fecal indicator bacteria declines via a dilution of wastewater in seawater. Desalination. 2009; 248:1008-15. [ Links ]
17. Troussellier M, Bonnefont JL, Courties C, Derrien A, Dupray E, Gauthier M et al. Responses of enteric bacteria to environmental stresses in seawater. Oceanol Acta. 1998; 21:965-81. [ Links ]
18. Martin Y, Troussellier M, Bonnefont JL. Adaptative responses of E. coli to marine environmental stresses: a modelling approach based on viability and dormancy concepts. Oceanol Acta. 1998; 21:951-64. [ Links ]
19. Lee CM, Lin TY, Lin CC, Kohbodi GA, Bhatt A, Lee R et al. Persistence of fecal indicator bacteria in Santa Monica Bay beach sediments. Water Research. 2006; 40:2593-602. [ Links ]
20. Gauthier MJ, Munro PM, Mohajer S. Influence of salts and sodium chloride on the recovery of Escherichia coli from seawater. Curr Microbiol. 1987; 15:5-10. [ Links ]
21. Gonzalez JM, Iriberri J, Egea L, Barcina I. Characterization of culturability, protistan grazing, and death of enteric bacteria in aquatic ecosystems. Appl Environ Microbiol. 1992; 58:998-1004. [ Links ]
22. Le Guyader F, Pommepuy M, Cormier M. Implantation of Escherichia coli in pilot experiments and the influence of competition on the flora. Can J Microbiol. 1991; 37:116-21. [ Links ]
23. Mitchell R, Yankofsky S, Jannasch HW. Lysis of Escherichia coli by marine micro-organisms. Nature. 1967; 215:891-3. [ Links ]












 uBio
uBio