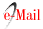Revista de la Sociedad Venezolana de Microbiología
versão impressa ISSN 1315-2556
Rev. Soc. Ven. Microbiol. vol.36 no.2 Caracas dez. 2016
Mutaciones K130M y V131I del gen X en aislados venezolanos del virus de hepatitis B
Idamelis Rodrígueza, Carmen Luisa Loureiroa, Roberto Leónb, Sharon Kay-Valeroc, Merita Seniord, Marisol Devesaa, Flor Helene Pujola,*
a Laboratorio de Virología Molecular, Centro de Microbiología y Biología Celular, Instituto Venezolano de Investigaciones Científicas, Venezuela. b Instituto Médico La Floresta, Caracas, Venezuela. c Instituto Clínico Médico Quirúrgico, Mérida, Venezuela. d Hospital de Clínicas Caracas, Caracas, Venezuela.
* Correspondencia: E-mail: fpujol@ivic.ve
Resumen: Aproximadamente el 50% de los carcinomas hepatocelulares (CHC) en el mundo están etiológicamente asociados con la infección por el virus de hepatitis B (VHB). Se han descrito 10 genotipos del VHB (A-J). En Venezuela y en varios países latinoamericanos predomina el genotipo F. Las mutaciones K130M y V131I presentes en la proteína HBx del VHB han sido asociadas al desarrollo del CHC. El objetivo de este trabajo fue estudiar la variabilidad genética de la proteína HBx del VHB circulante en pacientes venezolanos, con el fin de correlacionar estas mutaciones con los parámetros clínicos y virológicos de la enfermedad. Se analizó la secuencia del gen X del VHB, mediante amplificación por PCR de un fragmento de ese gen, en 45 pacientes infectados (35 crónicos y 10 agudos). Se observó una mayor frecuencia de las mutaciones K130M y V131I en pacientes de 25 o más años y con infección crónica. La presencia de estas mutaciones fue significativamente menor en el subgenotipo F3, comparado con el genotipo C. Estos resultados refuerzan la hipótesis de que el subgenotipo F3, predominante en Venezuela, podría estar asociado a una progresión menos severa de la enfermedad que la descrita para otros subgenotipos americanos, como F1b o F2.
Palabras clave: virus de hepatitis B, cáncer de hígado, mutaciones, genotipo, gen X.
K130M and V131L Mutations on the X gene of hepatitis B virus in Venezuelan isolates
Abstract: Approximately 50% of the hepatocellular carcinomas (HCC) in the world are etiologically associated to hepatitis B virus (HBV) infection. Ten HBV genotypes (A-J) have been described in Venezuela and in other Latin American countries where the F genotype predominates. The K130M and V131I mutations present in the HBx protein of HBV have been associated with the development of HCC. The aim of this work was to study the genetic variability of HBx protein from HBV circulating in Venezuelan patients, in order to correlate these mutations with clinical and virus factors involved in the disease. The X HBV gene sequence was analyzed by PCR amplification of that gene in 45 infected patients (35 with chronic and 10 with acute stages of hepatitis). A higher frequency K130M and V131I mutations was observed in subjects 25 years of age and older with chronic infection. The presence of these mutations was significantly lower in the F3 subgenotype compared with genotype C. These results support the hypothesis that the F3 subgenotype, predominant in Venezuela, could be associated with a less severe progression of the disease than that described for other American subgenotypes, such as F1b or F2.
Keywords: hepatitis B virus, liver cancer, mutations, genotype, X gene.
Recibido 29 de mayo de 2016; aceptado 29 de agosto de 2016
Introducción
El virus de la hepatitis B (VHB) es el agente etiológico responsable de 500.000 a 600.000 muertes al año a nivel mundial [1,2]. Cerca de un tercio de la población ha estado en contacto con este virus y 248 millones de personas son portadores crónicos del antígeno de superficie AgSHB [3], de las cuales entre el 15 y el 25% pueden desarrollar una enfermedad progresiva [4,5] con propensión a padecer carcinoma hepatocelular (CHC) [6]. Se estima que 33% de los casos de cirrosis y un 53% de los casos de CHC son atribuibles a este agente viral.
El VHB es miembro de la familia Hepadnaviridae, con un ADN parcialmente de doble cadena que contiene cuatro marcos de lectura abiertos, los cuales codifican las proteínas de envoltura (S), la cápside (preC/C), la polimerasa (P) y la proteína X (HBx) [7]. Con base en la variación de la secuencia nucleotídica, el VHB actualmente se clasifica en 10 genotipos, designados de la A a la J y dentro de cada genotipo se describen subgenotipos con números arábicos [8], siendo el genotipo F altamente prevalente en varios países suramericanos y el F3, en particular, en Venezuela [9,10].
El gen X (nucleótidos 1374-1838) codifica para la proteína HBx, indispensable para la infección y replicación eficiente del VHB. Esta proteína de 154 aminoácidos juega un papel importante en el control de la proliferación celular, la viabilidad y la transformación [11]. HBx no se encuentra incorporada al virión y se comporta como un elemento transactivador de la expresión genética celular y viral [12]. Se ha sugerido que HBx induce la carcinogénesis por diversos mecanismos, como por ejemplo la desregulación del ciclo celular. Sin embargo, el papel de las mutaciones específicas en el gen X no ha sido bien caracterizado, aún cuando se ha descrito que las deleciones en este gen se detectan con mucha frecuencia en pacientes con CHC [12,13]. HBx se solapa con el gen C que contiene el promotor de la cápside (PC).
El riesgo de desarrollar CHC se correlaciona con el nivel de replicación del VHB, el genotipo circulante y las mutaciones [14]. El genotipo C del VHB se ha asociado con un riesgo elevado de desarrollar CHC [15]. Se ha reportado que los nativos de Alaska infectados con el subgenotipo F1 poseen un riesgo mayor de desarrollar cáncer que con los genotipos A-D [16]. De hecho, la edad media de los pacientes infectados con el VHB genotipos A,C,D que presentaron CHC fue de 60 años, mientras que para el genotipo F fue sólo de 22,5 años. Sin embargo, los investigadores han indicado que este hecho puede estar relacionado con factores ambientales, como las altas concentraciones de aflatoxinas encontradas en los pacientes. La presencia de mutaciones en el genoma del VHB asociadas a severidad de la enfermedad, también se ha visto diferencialmente presente entre genotipos y subgenotipos del VHB [17]. En un trabajo previo encontramos una mayor frecuencia de mutaciones en distintas regiones del genoma del VHB en pacientes infectados con el subgenotipo F2, con respecto al subgenotipo F3 [18].
El objetivo de este trabajo fue estudiar la variabilidad genética de la proteína HBx con el fin de correlacionar estas mutaciones con parámetros clínicos y virológicos de la enfermedad por el VHB.
Materiales y métodos
Población de estudio: Se estudiaron un total de 77 muestras de sangre de pacientes positivos para VHB (18 con hepatitis aguda y 59 con hepatitis crónica), previamente diagnosticados y caracterizados con el desarrollo de diferentes patologías hepáticas, por gastroenterólogos del Hospital Dr. Domingo Luciani de Caracas y previo consentimiento escrito. Este estudio fue aprobado por el Comité de Bioética del IVIC.
La obtención del suero se realizó centrifugando cada muestra de sangre a 500g durante 5 min a temperatura ambiente, luego de dejar la muestra de sangre reposar por 10 min a temperatura ambiente. Los sueros se preservaron a -80 ºC hasta el momento de su uso.
El aislamiento del ADN viral se realizó siguiendo las indicaciones del protocolo de extracción de la casa comercial QIAampDNA Mini Kit (QIAGEN, Alemania); el ADN aislado fue conservado a -80 ºC hasta el momento de su uso.
Amplificación y secuenciación del gen X del VHB: El gen X del VHB fue amplificado mediante dos reacciones de amplificación consecutivas (Nested-PCR) usando dos juegos de cebadores externos 1182P: 5-ctcgccaacttacaaggccttt-3 y 1910N: 5-cratgtccatgyccyaaagc-3 ó 1101P: 5-ctcgccaacttacaaggccttt-3 y 1906N: 5-tccaaattcttttatagggg-3, para maximizar el número de muestras amplificadas. El proceso de amplificación consistió en una predesnaturalización a 94 ºC durante 3 min seguido por 42 ciclos a 94 ºC durante 1 min, 50 ºC por 1 min y 72 ºC por 1 min, con una extensión final de 1 min a 72 ºC. En la segunda ronda de PCR, se emplearon los cebadores 1601P: 5-gtttgctgacgcaaccccc-3 y 1860N: 5-cagcttggaggcttgaacagt-3 o 1263P: 5-gatccatactgcggaactcc-3 y 1860N: 5-cagcttggaggcttgaacagt-3 o 1450P: 5-tacgtcccgtcggctgaatc-3 y 1860N: 5-cagcttggaggcttgaacagt-3. Las condiciones de amplificación para la segunda PCR fueron: una predesnaturalización a 94 ºC durante 3 min seguido por 40 ciclos a 94 ºC durante 40 seg, 55 ºC por 40 seg y 72 ºC por 1 min y 40 seg, con una extensión final de 4 min a 72 ºC, esperándose un tamaño de 259 pb; 597 pb ó 410 pb, respectivamente.
Análisis de secuencia: Las muestras positivas fueron secuenciadas usando los cebadores internos sentido y antisentido, empleando el servicio Macrogen (Seúl, Corea). El análisis filogenético se realizó por el método estadístico Neighbor Joining o vecino más cercano, con distancia genética estimada con corrección de Kimura 2 parámetros, utilizando el programa DNAMAN 5.2.2 (LynnonBioSoft, Canada). Las secuencias nucleotídicas fueron traducidas a secuencias proteícas mediante el programa DNAMAN versión 5.2.2. (LynnonBioSoft, Canadá) para identificar la presencia de mutaciones en las posiciones M130K y V131I del ORF X del VHB.
Determinación del marcador serológico antígeno e (HBe) /anticuerpo anti-HBe: La presencia del HBe o de su anticuerpo fue evaluada mediante ensayo inmunoenzimático (ELISA) directo, tipo sándwich, utilizando el estuche de la casa comercial DiaSorin (Immunolab, Italia).
Análisis estadístico: Las diferencias estadísticas fueron evaluadas empleado la prueba de Chi-Cuadrado con corrección de Yates o mediante la prueba exacta de Fisher, cuando el valor de una celda era menor de 5. Para ello se empleó el software EpiInfo versión 7.1.1.14. La significancia de las diferencias en medias se evaluó mediante el test t de Student. Se consideró significativo cuando p fue menor o igual a 0,05.
Resultados
De los 77 pacientes analizados, se logró amplificar el fragmento del gen X en 45 aislados que correspondieron a 10 pacientes con infección aguda y 35 pacientes crónicos. En la tabla 1 se muestran las características relevantes de los 45 aislados. Se pudo observar que los valores de aminotransferasas tanto ALT como AST fueron significativamente más elevadas en los pacientes que cursaban una infección aguda.
Tabla 1. Características demográficas, clínicas y del VHB infectante en los pacientes estudiados.
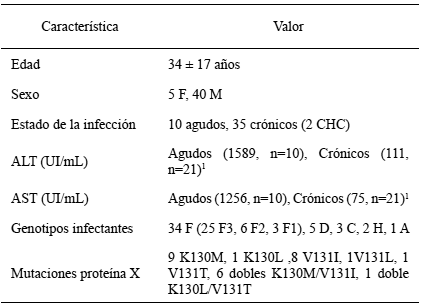
1Aminotransferasas ALT y AST, promedios con diferencia estadísticamente significativos (t Student 0,01 para ALT y 0,001 para AST).
El análisis de la secuencia del fragmento del gen X para los 45 aislados permitió la asignación de los genotipos y subgenotipos circulantes, observándose que un 75% de los aislados se agruparon dentro del genotipo F. Así mismo, se logró la identificación de las mutaciones en las posiciones K130M y V131I de la proteína X, de las cuales 9 aislados presentaron un cambio en la posición K130M, 8 en la posición V131I y en 6 aislados se logró identificar la doble mutación K130M/V131I. Un aislado de paciente infectado con VHB genotipo H presentó simultáneamente un inusual cambio en las posiciones K130L/V131T y mutaciones en el PBC específicamente en A1761C y T1765C.
De los dos pacientes que presentaban CHC, el análisis de secuencia del virus reveló la presencia de la doble mutación K130M/V131I en uno de los aislados (Tabla 2); dicha muestra correspondió al genotipo infectante C.
Tabla 2. Mutaciones en el gen X y parámetros clínicos y demográficos.
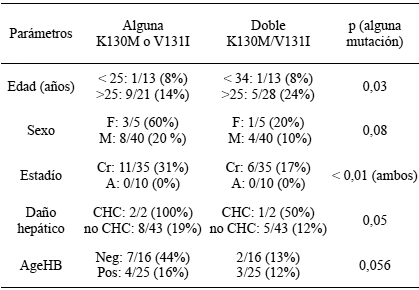
Cr: crónico. A: agudo.
Ninguno de los virus de pacientes en fase aguda presentó alguna de las mutaciones (K130M y/oV131I), mostrándose una prevalencia significativamente mayor de mutaciones en pacientes en fase crónica (Tabla 2). Se observó igualmente una mayor prevalencia de mutaciones en las muestras de pacientes de 25 o más años de edad. Por otra parte, se observó una tendencia a una mayor presencia de mutaciones en las muestras que no expresaron el antígeno e (AgeHB), aunque sin significancia estadística.
Se observó un alto grado de variación en la frecuencia de las mutaciones estudiadas con respecto al genotipo infectante (Figura 1). La frecuencia de las mutaciones fue alta para los genotipos C y D y menor en los genotipos F3. La prevalencia de las mutaciones simples y de la doble mutación fue significativamente menor en el genotipo F3 al compararlo con el genotipo C (p<0,01).
Figura 1: Frecuencia de mutaciones en el gen X en función del genotipo viral. Se muestran los números de aislados con mutaciones con respecto al número total de aisaldos del genotipo correspondiente. Se muestran las diferencias significativasentre el genotipo C y el F3 para la presencia de al menos una mutación (*) o doble mutación (**).
Discusión
Diferentes evidencias muestran que la infección crónica con el VHB está asociada con el progreso a cirrosis (C) y CHC. Se conoce que en el progreso de la enfermedad intervienen características clínicas, genéticas, virales y demográficas. Estos factores, sumados a la variabilidad de VHB, están asociados al incremento en la patogenicidad y al desarrollo de un daño hepático severo [19,20]. De esta forma, la frecuencia y tasa de progresión neoplásica está determinada por varios factores, siendo los más determinantes el genotipo, la edad, el sexo, el consumo de alcohol y de aflatoxinas, así como a factores genéticos del hospedero, como la hemocromatosis hereditaria [16,21-23]. En particular, existen múltiples evidencias de que la doble mutación K130M/V131I está asociada a una mayor frecuencia de CHC [21]. Este estudio muestra la correlación de distintos parámetros clínicos y virales, tales como la edad, la fase clínica de la enfermedad y el genotipo del virus infectante, con la aparición de mutaciones en el gen X. Se observó una mayor frecuencia de las mutaciones K130M y V131I, en los virus de pacientes en fase crónica de la enfermedad y en los pacientes con cáncer de hígado, como era esperado [20]. Por otra parte, las mutaciones K130L/V131T y A1761C y T1765C en el PBC encontradas en un aislado del VHB genotipo H no han sido descritas; se desconoce si están asociadas al desarrollo de CHC.
El AgeHB es una variante secretada de la proteína de la cápside, altamente inmunogénica, que actúa probablemente como inmunomodulador de la respuesta inmunitaria del hospedador. Una reducción en la expresión de este antígeno podría estar involucrada en la persistencia viral [24]. Diferentes autores reportan que la presencia de A1762T/G1764A reduce la síntesis de AgeHB por la supresión de la transcripción del ARNmpreC de un 50 a 70% [12,13]. El solapamiento de las mutaciones K130M/V131I, presentes en el gen X, con la doble mutación en PBCA1762T/G1764A, podría favorecer la desaparición del AgeHB durante la historia natural de la infección y por ende es compatible con una menor presencia del AgeHB en pacientes con virus que exhiben estas mutaciones, aunque en este estudio solo se observó una tendencia cercana a alcanzar significancia estadística (p=0,056), entre la presencia del alguna de estas mutaciones y la ausencia de este marcador serológico.
La variedad de genotipos del VHB en un país está muy relacionada con su composición étnica y es posible, a través del estudio de la distribución de genotipos virales, inferir las inmigraciones humanas que han ocurrido en un país [9,25,26]. En Venezuela, si bien circulan varios genotipos del VHB, es altamente predominante el genotipo F, siendo mayoritario el subgenotipo F3, seguido por el F2 [27]. El genotipo F ha sido asociado a una progresión a formas más severas de la enfermedad en otras latitudes [28,29]. El primer estudio, proveniente de España, encontró una mayor frecuencia de muertes asociadas al genotipo F del VHB en comparación con otros genotipos [29]. Sin embargo, en ese estudio el número de casos de pacientes infectados con el genotipo F fue bajo. El estudio más completo del genotipo F del VHB proviene de Alaska, donde los autores encontraron una mayor frecuencia de aparición de CHC en pacientes infectados con el genotipo F del VHB, a una edad menor [28].
Es importante destacar que el subgenotipo del VHB genotipo F que circula en Alaska es el F1b, que circula también en Argentina [10]. La frecuencia de aparición de mutaciones asociadas a severidad de infección parece también estar asociada no sólo al genotipo sino al subgenotipo infectante. En un estudio reciente en Argentina, los autores hallaron una mayor frecuencia de mutaciones en el PBC (que corresponden a las mutaciones K130M/V131I) en el VHB subgenotipo F1b, comparado con los genotipos A2, D y F4 [30]. En nuestro estudio, la frecuencia de mutaciones observada en el subgenotipo F3 fue significativamente menor que la observada en los aislados genotipos C, para el cual ha sido descrita una alta frecuencia de estas mutaciones [23]. Existen evidencias que muestran que los pacientes con genotipo C presentan un mayor daño hepático que los pacientes con genotipo B, ocurriendo en el genotipo C una mayor frecuencia de la doble mutación K130M/V131I [20], en concordancia con lo obtenido en este trabajo. En un estudio previo venezolano, se encontró que la prevalencia de mutaciones en el gen de la cápside y PBC eran menores en el subgenotipo F3 comparado con el F2 [18]. Todas estas evidencias apuntan a que podrían existir diferencias en la severidad de infección en función no solo del genotipo viral, sino también, en el caso del genotipo F, del subgenotipo. El subgenotipo F1b podría estar asociado a una mayor frecuencia de aparición de mutaciones y a una mayor severidad de infección, mientras que en el subgenotipo más común en Venezuela, el F3, la aparición de estas mutaciones no sería tan rápida.
En conclusión, este estudio confirma la relación entre la aparición de las mutaciones en la proteína X (o en el PBC) y distintos parámetros como la edad, la fase de la enfermedad y el daño hepático, en Venezuela. Estos resultados, aunados a otro estudio previo realizado en el país, sugieren que el subgenotipo F3 podría estar asociado a una progresión menos severa de la enfermedad que otros subgenotipos como el F1b o F2. Estas evidencias resaltan la importancia del seguimiento clínico y virológico de los pacientes infectados con el VHB para un mejor control de esta enfermedad.
Referencias
1. El-Serag HB, Rudolph KL. Hepatocellular carcinoma: epidemiology and molecular carcinogenesis. Gastroenterology. 2007; 132:2557-76. [ Links ]
2. Venook AP, Papandreou C, Furuse J, de Guevara LL. The incidence and epidemiology of hepatocellular carcinoma: a global and regional perspective. Oncologist. 2010; 15:5-13. [ Links ]
3. Schweitzer A, Horn J, Mikolajczyk RT, Krause G, Ott JJ. Estimations of worldwide prevalence of chronic hepatitis B virus infection: a systematic review of data published between 1965 and 2013. Lancet. 2015; 386:1546-55. [ Links ]
4. Zuckerman AJ, Zuckerman JN. Molecular epidemiology of hepatitis B virus mutants. J Med Virol. 1999; 58:193-5. [ Links ]
5. Yang HI, Yeh SH, Chen PJ, Iloeje UH, Jen CL, Su J, Wang LY, Lu SN, You SL, Chen DS, Liaw YF, Chen CJ. Associations between hepatitis B virus genotype and mutants and the risk of hepatocellular carcinoma. J Natl Cancer Inst. 2008; 100:1134-43. [ Links ]
6. Lavanchy D. Worldwide epidemiology of HBV infection, disease burden, and vaccine prevention. J Clin Virol. 2005; 34 Suppl 1:S1-S3. [ Links ]
7. Schäedler S, Hildt E. HBV Life cycle: entry and morphogenesis. Viruses. 2009; 1:185-209. [ Links ]
8. Kramvis A. Genotypes and genetic variability of hepatitis B virus. Intervirology. 2014; 57:141-50. [ Links ]
9. Devesa M, Pujol FH. Hepatitis B vírus genetic diversity in Latin America. Virus Res. 2007; 127:177-84. [ Links ]
10. Alvarado-Mora MV, Pinho JR. Distribution of HBV genotypes in Latin America. Antiviral Ther. 2013; 18:459-65. [ Links ]
11. Gong DY, Chen EQ, Huang FJ, Leng XH, Cheng X, Tang H. Role and functional domain of hepatitis B virus X protein in regulating HBV transcription and replication in vitro and in vivo. Viruses. 2013; 5:1261- 71. [ Links ]
12. Lazarevic I. Clinical implications of hepatitis B virus mutations: recent advances. World J Gastroenterol. 2014; 20:7653-64. [ Links ]
13. Gao S, Duan ZP, Coffin CS. Clinical relevance of hepatitis B virus variants. World J Hepatol. 2015; 7:1086-96. [ Links ]
14. Di Bisceglie AM. Hepatitis B and hepatocellular carcinoma. Hepatology. 2009; 49 (Suppl 5): S56–S60. [ Links ]
15. Chan HL, Hui AY, Wong ML, Tse AM, Hung LC, Wong VW, Sung JJ. Genotype C hepatitis B virus infection is associated with an increased risk of hepatocellular carcinoma. Gut. 2004; 53:1494-98. [ Links ]
16. McMahon BJ. The influence of hepatitis B virus genotype and subgenotype on the natural history of chronic hepatitis B. Hepatol Internat. 2009; 3:334-42. [ Links ]
17. Tong S, Revill P. Overview of hepatitis B viral replication and genetic variability. J Hepatol. 2016; 64 Suppl 1:S4-S16. [ Links ]
18. Puche ML, Kay-Valero S, Michelli P, Oropeza MD, Loureiro CL, Devesa M, Dagher L, Pujol FH. Genetic diversity of hepatitis B virus and mutations associated to hepatocellular carcinoma in patients with different stages of liver disease from Venezuela. Inv Clin. 2016; 57: en prensa. [ Links ]
19. Tseng TC, Liu CJ, Yang HC, Chen CL, Yang WT, Tsai CS, Kuo SF, Verbree FC, Su TH, Wang CC, Liu CH, Chen PJ, Chen DS, Kao JH. Higher proportion of viral basal core promoter mutant increases the risk of liver cirrhosis in hepatitis B carriers. Gut. 2015; 64:292-302. [ Links ]
20. Fang ZL, Yang J, Ge X, Zhuang H, Gong J, Li R, Ling R, Harrison TJ. Core promoter mutations A(1762)T and G(1764)A and viral genotype in chronic hepatitis B and hepatocellular carcinoma in Guangxi, China. J Med Virol. 2002; 68:33-40. [ Links ]
21. Montella M, DArena G, Crispo A, Capunzo M, Nocerino F, Grimaldi M, Barbieri A, DUrsi AM, Tecce MF, Amore A, Galdiero M, Ciliberto G, Giudice A. Role of sex hormones in the development and progression of hepatitis B virus-associated hepatocellular carcinoma. Internat J Endocrinol. 2015; 2015:854530. doi: 10.1155/2015/854530. [ Links ]
22. Pujol FH, Roman S, Panduro A, Navas MC, Lampe E. Hepatocellular carcinoma in Latin America. In: Chemin I, editor. Hepatocellular carcinoma: A global challenge. New York: Nova Science Publishers Inc.; 2011. p. 55-68. [ Links ]
23. Kim HJ, Park JH, Jee Y, Lee SA, Kim H, Song BC, Yang S, Lee M, Yoon JH, Kim YJ, Lee HS, Hwang ES, Kook YH, Kim BJ. Hepatitis B virus X mutations occurring naturally associated with clinical severity of liver disease among Korean patients with chronic genotype C infection. J Med Virol. 2008; 80:1337-43. [ Links ]
24. Tong S, Li J, Wands JK, Wen Y. Hepatitis B virus genetic variants: biological properties and clinical implications. Emerg Microbes Infect. 2013; 2:e10. doi:10.1038/emi.2013.10. [ Links ]
25. Kramvis A, Paraskevis D. Subgenotype A1 of HBV tracing human migrations in and out of Africa. Antiv Ther. 2013; 18:513-21. [ Links ]
26. Loureiro CL, Aguilar JC, Aguiar J, Muzio V, Pentón E, Garcia D, Guillen G, Pujol FH. HBV genotypic variability in Cuba. PLoS One. 2015; 10:e0118959. doi: 10.1371/journal.pone.0118959. [ Links ]
27. Devesa M, Loureiro CL, Rivas Y, Monsalve F, Cardona N, Duarte MC, Poblete F, Gutierrez MF, Botto C, Pujol FH. Subgenotype diversity of hepatitis B virus American genotype F in Amerindians from Venezuela and the general population of Colombia. J Med Virol. 2008; 80:20-6. [ Links ]
28. Livingston SE, Simonetti JP, McMahon BJ, Bulkow LR, Hurlburt KJ, Homan CE, Snowball MM, Cagle HH, Williams JL, Chulanov VP. Hepatitis B virus genotypes in Alaska Native people with hepatocellular carcinoma: preponderance of genotype F. J Infect Dis. 2007; 195:5-11. [ Links ]
29. Sánchez-Tapias JM, Costa J, Mas A, Bruguera M, Rodés J. Influence of hepatitis B virus genotype on the long-term outcome of chronic hepatitis B in western patients. Gastroenterology. 2002; 123:1848-56. [ Links ]
30. González López Ledesma MM, Mojsiejczuk LN, Rodrigo B, Sevic I, Mammana L, Galdame O, Gadano A, Fainboim H, Campos R, Flichman D. Hepatitis B Virus Genotype distribution and genotypespecific BCP/preCos substitutions in acute and chronic infections in Argentina. PLoS ONE. 2015; 10:e0121436. doi:10.1371/journal.pone.0121436. [ Links ]












 uBio
uBio