Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Bioagro
versión impresa ISSN 1316-3361
Bioagro v.20 n.2 Barquisimeto ago. 2008
Evaluación de la estabilidad genética mediante marcadores rapd, en brotes de Billbergia rosea Hortus Ex Beer, conservados in vitro
Adriana Pardo1, Claret Michelangeli2, Catalina Ramis2, Norca Mogollón1 y Carmen Silva3
1 Unidad de Biotecnología Vegetal, Decanato de Agronomía, Universidad Centroccidental Lisandro Alvarado. Apdo. 400. Barquisimeto. Venezuela. e-mails: apardo@ucla.edu.ve; norcam@intercable.net.ve
2 Centro de Investigaciones en Biotecnología Agrícola, Facultad de Agronomía, Universidad Central de Venezuela. Maracay. Venezuela. e-mails: claremiche@gmail.com; cmcramis@yahoo.es
3 Instituto Nacional de Investigaciones Agrícolas (INIA), Estación Local Yaritagua. Yaritagua estado Yaracuy. Venezuela. e-mail: carmens186@hotmail.com
RESUMEN
Se evaluó mediante la técnica RAPD, la estabilidad genética de brotes de Billbergia rosea procedentes de ápices caulinares sometidos a cinco regímenes de conservación bajo condiciones de crecimiento mínimo. Para ello se procedió a la estandarización del protocolo para la obtención de bandas RAPD en esta especie, siguiendo los pasos de extracción de ADN, amplificación del ADN polimórfico y visualización de las bandas generadas. El dendrograma obtenido basado en los coeficientes de similitud de Jaccard, así como el análisis de componentes principales, estableció tres grupos. En el grupo I se ubicaron los brotes almacenados en ¼ MS con o sin sacarosa, presentando una similitud mínima con respecto a las plantas madres de 0,91. El segundo, incluyó los tratamientos con agua destilada y ½ MS con sacarosa, cuya disimilitud fue de 0,15 con respecto al grupo anterior. El grupo III estuvo conformado por los brotes conservados en ½ MS con una similitud de 0,58 con respecto a los grupos anteriores. El análisis reflejó que los brotes conservados en ¼ MS con sacarosa, mostraron un perfil electroforético similar al plantel madre, lo que indica su estabilidad genética. Estos resultados pueden ser utilizados como punto de partida para la planificación de estrategias de conservación a mediano plazo de Billbergia rosea.
Palabras clave adicionales: Bromeliaceae, variaciones genéticas, coeficientes de similitud de Jaccard
Evaluation of genetic stability using RAPD markers, in Billbergia rosea Hortus Ex Beer buds conserved in vitro
ABSTRACT
Genetic stability in Billbergia rosea buds, obtained from stem tips subjected to five conservation regimes under minimal growth, was evaluated using RAPD techniques. Protocol was standardized to obtain RAPD bands for this species. For this proposal, DNA extraction, polymorphic DNA amplification and printing of generated bands were made. Dendrogram based in Jaccards similarity index and the principal components analysis showed three groups. Group I, buds kept in ¼ MS with or without sucrose were pooled with minimal similarity (0.91) compared to mother plants. Group II, all treatments with distilled water and ½ MS with sucrose were included with dissimilarity of 0.15 compared to previous group. Group III consisted of buds kept in ½ MS with similarity of 0.58 to both groups. Analysis revealed that buds kept in ¼ MS with sucrose, showed a electrophoretic profile similar to mother plant, thus indicating genetic stability. The results can be used as base to plant medium-term conservation strategies for Billbergia rosea.
Additional key words: Bromeliaceae, genetics variations, Jaccards similarity index
Recibido: Octubre 3, 2007 Aceptado: Mayo 6, 2008
INTRODUCCIÓN
La familia Bromeliaceae, autóctona del continente americano está conformada en su mayoría por especies de importancia ornamental las cuales representan 99 % del total de géneros que comprende la familia. Se caracterizan por presentar inflorescencias y follajes de colores vistosos, además de una fácil adaptación a diferentes ambientes, ya que son poco exigentes en lo que respecta a iluminación y requerimientos hídricos. Estas características hacen de las bromeliáceas, plantas altamente cotizadas en el comercio internacional para fines decorativos (Koh y Davies, 1997).
Dentro de la conservación de recursos fitogenéticos, las técnicas de cultivo in vitro constituyen una herramienta particularmente útil.
Al respecto, se debe considerar que durante la conservación es preciso garantizar la estabilidad o integridad genética de los materiales (Engelmann, 1999; Hodgkin et al., 2001; Rao, 2004). Tejidos y órganos utilizados como explante durante el cultivo in vitro, entre ellos, meristemas, embriones somáticos y yemas introducen un mínimo riesgo de variación genética en las plantas regeneradas, en relación con el uso de callos, suspensiones celulares y protoplastos (Rani y Raina, 2000; Pontaroli y Camadro, 2005).
Por tal razón, las plantas conservadas in vitro deben ser evaluadas y/o monitoreadas para determinar sus niveles de integridad o estabilidad genética. Esto ha sido realizado considerando aspectos morfológicos, bioquímicos y moleculares (Pérez et al., 1997; Hodgkin et al., 2001). Dentro de éstos, los marcadores moleculares proveen una excelente vía para obtener una gran cantidad de datos sobre los procesos de conservación que pueden dar origen a patrones de variación, proporcionando información que resulta de gran utilidad para un eficiente manejo y explotación de la variabilidad presente en las colecciones o bancos de germoplasma (Rao, 2004). Toda la información generada por los diversos tipos de marcadores moleculares, entre los que figuran los RAPD, son altamente sensibles para el análisis genético de las plantas obtenidas por cultivo de tejidos.
La variación genética originada durante la conservación in vivo e in vitro ha sido detectada por marcadores RAPD en Musa sp. (Damasco et al., 1996); Allium sp. (Al-Zahim et al., 1999); Digitalis sp. (Nebauer et al., 1999); Mangifera sp. (Lowe et al., 2000) y Lilium sp. (Varshney, et al., 2001), entre otros. En Digitalis obscura la variación genética detectada mediante RAPD fue utilizada para proponer estrategias para su conservación in vitro (Nebauer et al., 1999).
Dentro de la familia Bromeliaceae se ha estudiado la conservación in vitro de Billbergia rosea mediante la aplicación de técnicas de cultivo de tejidos (Pardo et al., 2007). Por lo anteriormente señalado, en el presente estudio se planteó como objetivo caracterizar molecularmente, mediante la técnica RAPD, brotes de Billbergia rosea procedentes de ápices caulinares sometidos a cinco regímenes de conservación bajo condiciones de crecimiento mínimo.
MATERIALES Y MÉTODOS
La investigación se realizó en el laboratorio de Genética Molecular del Centro de Investigaciones en Biotecnología Agrícola (CIBA) de la Facultad de Agronomía, Universidad Central de Venezuela (UCV) en Maracay, Venezuela.
Para verificar la estabilidad genética de los brotes de B. rosea conservados in vitro bajo cinco condiciones de crecimiento mínimo se empleó la técnica de polimorfismo del ADN amplificado al azar (RAPD).
Se utilizaron brotes en etapa de multiplicación con una altura de 3 a 4 cm y 3 a 4 hojas por brote, provenientes del cultivo de ápices caulinares (Pardo et al., 2007) los cuales fueron conservados durante nueve meses, sin subcultivos, bajo condiciones de crecimiento mínimo de acuerdo al protocolo establecido por Zee y Munekata (1992). El medio Murashige y Skoog (MS) se preparó manteniendo la concentración total de los componentes: tiamina-HCl (30 mg·L-1), glicina (2 mg·L-1), ácido nicotínico (10 mg·L-1), piridoxina (1 mg·L-1) e inositol (100 mg·L-1); el pH se ajustó a 5,8 ± 0,1. Los tratamientos se conformaron por: C1= Agua destilada; C2= ½ MS; C3= ½ MS con 30 g·L-1 sacarosa; C4 = ¼ MS y C5= ¼ MS con 30 g·L-1 sacarosa, todos con 9 g·L-1 de agar.
Para los análisis moleculares se tomaron al azar ocho muestras o brotes de cada uno de los tratamientos de conservación in vitro y como testigos se utilizaron dos plantas adultas del plantel madre.
Con la finalidad de estandarizar el protocolo para la obtención de bandas RAPD en esta especie se siguieron los pasos de extracción de ADN, amplificación del ADN polimórfico y visualización de las bandas generadas.
Extracción de ADN
En los brotes conservados in vitro se logró la extracción de cantidades suficientes de ADN genómico, utilizando el método de purificación del CTAB (Doyle y Doyle, 1999). En el caso de las plantas adultas, con tejido foliar coriáceo y suculento, la aplicación del protocolo del CTAB no resultó eficiente y limitó la extracción, por tal motivo se probó un método más agresivo de extracción como el utilizado en especies arbóreas por Risterucci et al. (2000). Esta metodología utilizada en plantas adultas permitió obtener cantidades suficientes de ADN de buena calidad.
Amplificación del ADN polimórfico
A fin de estandarizar la amplificación de los fragmentos al azar y seleccionar los iniciadores de mejor resolución y reproducibilidad, se seleccionaron ocho muestras al azar de ADN genómico a los cuales se les aplicó el protocolo para RAPD descrito por Williams et al. (1990). Se probaron tres concentraciones de ADN (10, 20 y 50 ng), tres de dNTPs (2,5, 5,0 y 10 mM) y dos cantidades de los iniciadores (1 y 2 mL), preparados a 10 mM. Para probar la reproducibilidad se realizaron tres repeticiones. Las condiciones más apropiadas para la amplificación de los RAPD fueron: Tampon Promega 1X, 2,5 mM MgCl2, 0,53 mM dNTPs, 0,9 mg BSA, 0,66 mM iniciador, 2,5 U Taq Promega y 50 ng de ADN en un volumen final de 15 mL.
La PCR fue realizada en un termociclador MS Research PTC-200 programado para un primer ciclo térmico de 5 min a 94 ºC, seguido por 45 ciclos correspondientes a la desnaturalización (1 min a 94 ºC), alineación del iniciador (36 ºC por 30 segundos) y elongación (72 ºC por 2 minutos). Posteriormente, el producto se mantuvo constante a 72 ºC por 7 min y luego fue conservado a 4 ºC hasta la realización de la electroforesis.
Para la amplificación del ADN se emplearon 11 iniciadores de la serie Operon Technologies de acuerdo a lo reportado en piña (Ananas comosus) por Sripaoraya et al. (2001). La identificación y secuencia de los iniciadores se presentan en el Cuadro 1.
Cuadro 1. Iniciadores decaméricos de la serie Operon Technologies, utilizados para la amplificación RAPD en tejido foliar de Billbergia rosea
| Iniciador | Secuencia |
| OPA-02 | 5-TGCCGAGCTG-3 |
| OPA-07 | 5-GTCCCGACGA-3 |
| OPA-09 | 5-GGGTAACGCC-3 |
| OPC-01 | 5-TTCGAGCCAG-3 |
| OPC-02 | 5-GTGAGGCGTC-3 |
| OPC-05 | 5-GATGACCGCC-3 |
| OPC-07 | 5-GTCCCGACGA-3 |
| OPC-09 | 5-CTCACCGTCC-3 |
| OPC-10 | 5-TGTCTGGGTG-3 |
| OPC-11 | 5-AAAGCTGCGG-3 |
| OPC-12 | 5-AAAGCTGCGG-3 |
Con la finalidad de seleccionar los iniciadores que permitieran la identificación de variantes se utilizó una muestra de planta adulta proveniente del plantel madre más tres muestras de ADN seleccionadas al azar de cada uno de los tratamientos de conservación in vitro (16 muestras). Se realizaron dos repeticiones en cada caso para un total de 32 muestras. Se observó una alta reproducibilidad para todos los iniciadores. Sin embargo, los de mejor resolución fueron los iniciadores OPC-01, OPC-02 y OPC-05, por lo que fueron seleccionados para el análisis de la estabilidad genética.
Visualización de las bandas
La visualización de los fragmentos amplificados en geles de agarosa al 3 % evidenció la presencia de bandas entre 300 y 600 pb. Sin embargo, la resolución de las bandas no fue la más apropiada, por lo que se utilizaron geles de acrilamida al 6 % (0,4 mm de espesor y 42 cm de longitud) revelados en nitrato de plata, obteniéndose en este caso una óptima resolución.
Análisis de la estabilidad genética
El polimorfismo genético entre los genotipos fue evaluado utilizando tres iniciadores cuyos productos de amplificación fueron registrados en matrices binarias de datos, asignando (0) para ausencia y (1) presencia de bandas amplificadas. Se construyó una matriz de similitud generada mediante el modelo matemático o índice de Jaccard (J) y con ésta un análisis de agrupamiento UPGMA (Unweighted Pair-Group Method Average) con su respectivo dendrograma. Así mismo, se aplicó el análisis de componentes principales (Cuadras, 1991). Ambos tipos de análisis permitieron visualizar las relaciones genéticas entre los genotipos evaluados.
RESULTADOS Y DISCUSIÓN
Los brotes conservados bajo condiciones de crecimiento mínimo, mostraron variaciones en sus patrones electroforéticos cuando fueron comparados con las plantas madres. En el Cuadro 2 se muestra el número de brotes que presentaron idénticos patrones electroforéticos a las plantas madres para cada uno de los iniciadores RAPD seleccionados.
Cuadro 2. Número de brotes con igual número de bandas presentadas en las plantas madres de Billbergia rosea para cada uno de los iniciadores seleccionados durante la conservación bajo cinco condiciones de crecimiento mínimo
| Iniciador | Número de brotes | |||||
| C1 | C2 | C3 | C4 | C5 | Total | |
| OPC-01 | 0 | 8 | 0 | 8 | 8 | 24 |
| OPC-02 | 2 | 0 | 0 | 0 | 2 | 4 |
| OPC-05 | 0 | 8 | 0 | 3 | 8 | 19 |
C1= Agua destilada, C2= ½ MS; C3= ½ MS con 30 g·L-1 sacarosa; C4= ¼ MS; C5= ¼ MS con 30 g·L-1 sacarosa
Con los iniciadores OPC-01 y OPC-05 todos los brotes conservados en agua destilada con agar y en ½ MS con sacarosa revelaron distintos patrones electroforéticos a los observados en las plantas madres, mientras que con el iniciador OPC-02 sólo dos brotes conservados en agua destilada y en ¼ MS con sacarosa se mostraron idénticos a las plantas madres (Cuadro 2).
Con el iniciador OPC-01 los brotes conservados en agua destilada presentaron dos bandas polimórficas o distintas a las plantas madres; en siete de los brotes se presentó una banda de más y en ocho de éstos una banda de menos; mientras que en todos los brotes conservados en ½ MS con sacarosa se observaron dos bandas polimórficas, una de ellas presente y la otra ausente (Cuadro 3).
Cuadro 3. Número de bandas distintas a las presentadas en las plantas madres de Billbergia rosea para cada uno de los iniciadores seleccionados durante la conservación bajo cinco condiciones de crecimiento mínimo
| Iniciador | Número de bandas observadas | |||||
| C1 | C2 | C3 | C4 | C5 | Total | |
| OPC-01 | 2 | 0 | 2 | 0 | 0 | 4 |
| OPC-02 | 3 | 21 | 2 | 1 | 3 | 31 |
| OPC-05 | 2 | 0 | 2 | 2 | 0 | 6 |
C1= Agua destilada, C2= ½ MS; C3= ½ MS con 30 g·L-1 sacarosa; C4= ¼ MS; C5= ¼ MS con 30 g·L-1 sacarosa
El iniciador OPC-05 diferenció a todos los brotes almacenados en agua destilada y ½ MS con sacarosa con la presencia de dos bandas polimórficas. Una de las bandas estuvo ausente en ocho de los brotes, y la otra banda no observada en las madres, se reveló en seis de los brotes sometidos a ambos tratamientos de conservación. En los materiales conservados en ¼ MS se observaron dos bandas polimórficas a las adultas; una de ellas estuvo ausente en cinco brotes, y la otra banda se reveló como nueva en tres de los brotes conservados en este medio.
El iniciador OPC-02 detectó polimorfismo entre las plantas madres y todos los brotes almacenados en ½ MS con o sin sacarosa y ¼ MS y en dos de los brotes conservados en agua destilada y en ¼ MS con sacarosa. El mayor polimorfismo fue observado en los brotes cultivados en ½ MS con un total de 21 bandas polimórficas; en siete brotes se observaron 12 bandas que no estaban presentes en las plantas madres; mientras que nueve bandas, presentes en las madres, no se observaron en cinco de estos brotes. Los restantes tratamientos mostraron entre una a tres bandas polimórficas (Cuadro 3).
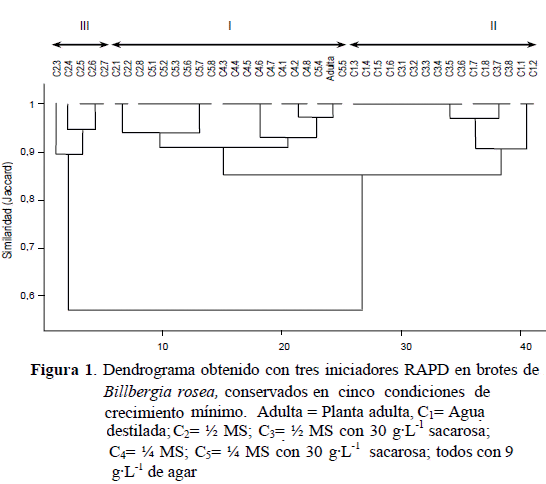
El dendrograma obtenido basado en los coeficientes de similitud de Jaccard, conformó tres grupos (Figura 1). En el grupo I se ubicaron todos los brotes almacenados en ¼ MS con o sin sacarosa (C5 y C4, respectivamente) y tres de los brotes conservados en ½ MS sin sacarosa (C2), con una similitud mínima con respecto a las plantas madres de 0,91. El grupo II incluyó a todos los brotes tratados con agua destilada (C1) y ½ MS con 30 g·L-1 sacarosa (C3) con una disimilitud de 0,15 con respecto al grupo anterior. El grupo III se conformó por cinco de los brotes conservados en ½ MS (C2) con una similitud de 0,58 con respecto a los grupos anteriores. Cabe destacar que sólo el iniciador OPC-02 detectó polimorfismo entre los brotes almacenados en ½ MS (Cuadro 2) con un total de 21 bandas distintas a las observadas en las plantas adultas (Cuadro 3). Por esto permitiría explicar que sólo cinco brotes con nueve bandas ausentes fueron incluidos en el grupo III y los restantes en el grupo I.
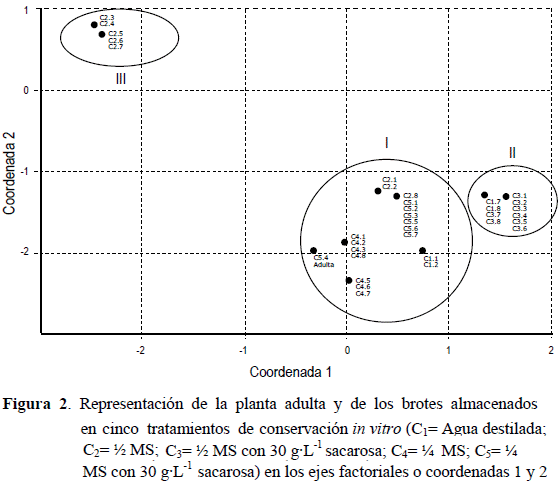
Los valores propios y la proporción de la variación observada entre los materiales conservados explicados por cada componente o coordenada principal se presentan en el Cuadro 4. La variación entre los cinco tratamientos de conservación in vitro pudo ser explicada en los dos primeros ejes o coordenadas con un 64,46 % de variabilidad, significativo para todos los tratamientos (P≤0,05).
Cuadro 4. Valores propios obtenidos del análisis de componentes principales
| Coordenada principal | Valor propio | Varianza (%) | Varianza Acumulada (%) |
| 1 | 1,4881700 | 43,30 | 43,30 |
| 2 | 0,7271060 | 21,16 | 64,46 |
| 3 | 0,4977190 | 14,48 | 78,95 |
| 4 | 0,2560980 | 7,45 | 86,40 |
| 5 | 0,1447730 | 4,21 | 90,61 |
| 6 | 0,1244470 | 3,62 | 94,23 |
| 7 | 0,0749877 | 2,18 | 96,41 |
| 8 | 0,0491181 | 1,43 | 97,84 |
| 9 | 0,0396427 | 1,15 | 99,00 |
La representación gráfica de las coordenadas principales se presenta en la Figura 2. Esta gráfica ubica en un plano cartesiano a la planta adulta y a los brotes conservados, utilizando los dos primeros factores como ejes de abscisas y ordenadas. De manera similar al dendrograma, este análisis diferenció tres grupos. En un primer grupo (I) se concentraron cercano a la planta adulta, todos los brotes conservados en ¼ MS con o sin sacarosa, y próximos a éstos, dos brotes conservados en agua destilada y tres de los brotes almacenados en ½ MS. En un segundo grupo (II), se ubicaron todos los brotes tratados con ½ MS con sacarosa; mientras que en un plano opuesto a ambos grupos, se agruparon cinco de los brotes conservados en ½ MS sin sacarosa (III).
Durante el estudio de conservación in vitro se utilizaron brotes regenerados a partir de ápices caulinares con el fin de minimizar el riesgo de variación genética, tal como fue propuesto por Rani y Raina (2000). Sin embargo, el análisis RAPD detectó polimorfismo entre las plantas madres y los diferentes tratamientos de conservación. Debido a que todos los brotes fueron almacenados por un período de nueve meses sin subcultivo, probablemente las causas de la variación están principalmente asociadas con el medio de cultivo empleado, tal como lo reporta Zee y Munekata (1992) durante el almacenamiento de A. comosus.
Cabe señalar que durante la conservación in vitro destacaron, en primer lugar, los brotes almacenados en ¼ MS con 30 g·L-1 de sacarosa con los mayores promedios de sobrevivencia (86 %) y vigor (3), así como para las variables altura de brotes, número de hojas y longitud de raíces (Pardo et al., 2007). Seguidamente los brotes conservados en ½ MS con 30 g·L-1 de sacarosa y en agua destilada alcanzaron promedios aceptables de sobrevivencia (79 y 74 %) y vigor (3 y 2,4), respectivamente; mientras que los brotes almacenados en ausencia de sacarosa (C2 y C4) presentaron los mínimos promedios para todas las variables evaluadas (Pardo et al., 2007).
El análisis RAPD ubicó en un mismo grupo a las plantas adultas y a todos los brotes almacenados en ¼ MS con o sin sacarosa (grupo I), reflejándose que a bajas concentraciones de sales en el medio los brotes mostraron un perfil electroforético similar al de las plantas adultas. Estos resultados pueden estar asociados a la probable susceptibilidad de B. rosea a la presencia de sales, ya que en su condición de planta epífita (CAM) puede absorber agua y nutrientes por las hojas, escapando de los sustratos salinos, y no estando forzadas a desarrollar mecanismos de tolerancia (Moradshahi et al., 1977; Luttge, 2004). En consecuencia, la susceptibilidad a la salinidad podría explicar que las plantas conservadas en medio nutritivo con bajo contenido de sales (C4 y C5) presentaran mayor similitud con las plantas adultas.
En segundo lugar, resalta el comportamiento de los brotes conservados en ½ MS con 30 g·L-1 de sacarosa y en agua destilada (grupo II) con una disimilitud de apenas 0,15 con respecto al grupo I. En el tratamiento ½ MS en presencia de sacarosa los brotes mantuvieron el balance carbohidratos-sales y lograron activar su mecanismo fisiológico, manteniendo además un lento crecimiento durante el transcurso del experimento (Pardo et al., 2007). En el caso del tratamiento con agua destilada, los brotes no fueron sometidos a condiciones de estrés salino logrando mantener el balance osmótico en ausencia de sales y azúcar en el medio, por lo cual no se deterioraron tan rápido como aquellos cultivados en medio ½ MS sin sacarosa.
Finalmente, en el grupo más disímil a las plantas adultas se ubicó la mayoría de los brotes almacenados en ½ MS sin sacarosa (C2-grupo III) los cuales se tornaron marrones, se secaron y mostraron un deterioro progresivo después de trascurridos los diez meses de la conservación in vitro, afectándose por ende la sobrevivencia y el vigor (Pardo et al., 2007). Probablemente el comportamiento observado en los brotes almacenados en ausencia de sacarosa y alto contenido de sales (C2) fue debido a que bajo condiciones de estrés, ocasionado por el desbalance de carbohidratos y sales en los tejidos, los brotes no lograron evitar la pérdida de agua y turgor en la célula, causando la deshidratación en los tejidos, tal como lo menciona Foyer (1988). Así mismo, Kaeppler y Phillips (1993) y Phillips et al. (1994) mencionan que el estrés celular puede afectar el control del ciclo celular o los sistemas de reparación del ADN con posibles daños o alteraciones a nivel celular. Lo anteriormente señalado posiblemente repercutió en el deterioro progresivo que se observó en estos brotes, lo cual pudo ser detectado como polimorfismos RAPD.
El análisis RAPD permitió discriminar por diferencias en sus patrones electroforéticos todos los brotes almacenados bajo cinco condiciones de crecimiento mínimo, permitiendo de esta manera detectar los tratamientos que presentaban mayor similitud con las plantas adultas. En tal sentido, los brotes de B. rosea conservados en medio ¼ MS con 30 g·L-1 de sacarosa (C5) presentaron estabilidad genética y buen comportamiento durante la conservación in vitro. Los resultados obtenidos por la técnica RAPD pueden ser utilizados como punto de partida para la planificación de estrategias de conservación a gran escala de B. rosea, tal como ha sido reportado por Zee y Munekata (1992) en otras especies como piña (Ananas comosus); por Nebauer et al. (1999) para digital negra (Digitalis obscura) y en mango (Mangifera indica) según Lowe et al. (2000).
CONCLUSIONES
El análisis RAPD determinó que los brotes almacenados en ¼ MS con sacarosa mostraron un perfil electroforético similar a las plantas adultas lo que indica su estabilidad genética. Esta información puede ser de utilidad para fitomejoradores o biólogos moleculares con el fin de planificar estrategias de conservación a mediano plazo de Billbergia rosea.
AGRADECIMIENTO
A Paúl Azuaje por su colaboración y apoyo a esta investigación. Al CDCH-UCV por el financiamiento. Al personal técnico y obrero de los laboratorios del CIBA-UCV.
LITERATURA CITADA
1. Al-Zahim, M., B. Ford-Lloyd y H. Newburry. 1999. Detection of somaclonal variation in garlic (Allium sativum L.) using RAPD and cytological analysis. Plant Cell Report 18: 473-477. [ Links ]
2. Cuadras, C. 1991. Métodos de análisis multivariante. Colección estadística y análisis de datos. Barcelona, España. [ Links ]
3. Damasco, O., G. Grahan, R. Henry, S. Adkins, M. Smith e I. Godwin. 1996. Random amplified polymorphic DNA (RAPD) detection of dwarf off-Types in micropropagated Cavendish (Musa spp. AAA) bananas. Plant Cell Reports 16: 118-123. [ Links ]
4. Doyle, J. y T. Doyle. 1999. Isolation of DNA from small amounts of plant tissues. Focus 12: 13-15. [ Links ]
5. Engelmann F. 1999. Management of field and in vitro germplasm collections. Proceedings of a Consultation Meeting (15-20 January 1996, CIAT). IPGRI, Roma. [ Links ]
6. Foyer, C. 1988. Feedback inhibition of photosynthesis through source-sink regulation in leaves. Plant Physiol. Biochem. 26: 483-492. [ Links ]
7. Hodgkin, T., R. Roviglioni, M. De Vicente y N. Dudnik. 2001. Molecular methods in the conservation and use of plant genetic resources. Acta Horticulturae 546: 107-118. [ Links ]
8. Kaeppler, S. y R. Phillips. 1993. DNA methylation and tissue culture induced variation in plants. In Vitro Cell Dev. Biol. 29: 125-130. [ Links ]
9. Koh, Y. y F. Davies. 1997. Micropropagation of Cryptanthus with leaf explants with attached meristems excised from greenhouse stock plants. Scientia Horticulturae 70(4): 301-307. [ Links ]
10. Lowe, A., A. Gillies, J. Wilson e I. Dawson. 2000. Conservation genetics of bush mango from central/west Africa: implications from random amplified DNA analysis. Molecular Ecology 9: 831-841. [ Links ]
11. Luttge, U. 2004. Ecophysiology of Crassulacean acid metabolism (CAM). Annals of Botany 93: 629-652. [ Links ]
12. Moradshahi, A., K. Vines y C. Black. 1977. CO2 exchange and acidity levels in detached pineapple, Ananas comosus (L.) Merr., leaves during the day at various temperatures, O2 and CO2 concentrations. Plant Physiol. 59: 274-278. [ Links ]
13. Nebauer, S. L. del Castillo-Agudo y J. Segura. 1999. RAPD variation within and among natural populations of outcrossing willow-leaved foxglove (Digitalis obscura L.). Theor. Appli. Genet. 98: 985-994. [ Links ]
14. Pardo, A., C. Michelangeli y N. Mogollón. 2007. Conservación in vitro de Billbergia rosea (Bromeliaceae). Boletín del Centro de Investigaciones Biológicas (LUZ) 41(4): 517-529. [ Links ]
15. Pérez, G., M. Isidrón, E. Arias, S. Pérez, J. González, y N. Nieves. 1997. Characterization phenotypic, biochemical and cytogenetic of pineapple plants obtained from somaclonal variation and mutagenesis. Acta Horticulturae 425: 221-232. [ Links ]
16. Phillips, R., S. Kaeppler y P. Olhoft. 1994. Genetic instability of plan tissue cultures: Breakdown of normal controls. Proc. Natl. Acad. Sci. 91: 5222-5226. [ Links ]
17. Pontaroli, A. y E. Camadro. 2005. Somaclonal variation in Asparagus officinales plants regenerated by organogenesis from long-term callus cultures. Genetic Molecular Biology 28(3): 423-430. [ Links ]
18. Rani, V. y S. Raina. 2000. Genetic fidelity of organized meristem derived micropropagated plants: a critical reappraisal. In Vitro Cell. 36: 319-326. [ Links ]
19. Rao, K. 2004. Plant genetic resources: Advancing conservation and use through biotechnology. African Journal of Biotechnology 3(2): 136-145. [ Links ]
20. Risterucci, M., L. Grivet, N. Goran, I. Pieretti, M. Flament y C. Lanaud. 2000. A high density linkage map of Theobroma cacao L. Theor. Appli. Genet. 101: 948-955. [ Links ]
21. Sripaoraya, S., W. Blackhall, R. Marchant, B. Power, C. Lowe y R. Davey. 2001. Relationship in pineapple by random amplified polymorphic DNA (RAPD) analysis. Plant Breeding 120: 265-267. [ Links ]
22. Varshney, A., M. Lakshmikumaran, P. Srivastava y V. Dhawan. 2001. Establishment of genetic fidelity of in vitro raised Lilium bulblets through RAPD markers. In Vitro Cellular and Development Biology 37(2): 227-231. [ Links ]
23. Willians, J., A. Kubelik, N. Livak, A. Rafalski y S. Tingey. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucl. Acids. Res. 18: 6531-6535. [ Links ]
24. Zee, F. y M. Munekata. 1992. In vitro storage of pineapple (Ananas spp.) germplasm. HortScience 27(1): 57-58. [ Links ]