Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Bioagro
versión impresa ISSN 1316-3361
Bioagro v.21 n.3 Barquisimeto dic. 2009
Huella genética de genotipos silvestres y comerciales de Passiflora spp. utilizando patrones RAPD
Iris Pérez-Almeida1, Sundry Vásquez García1, Delis Pérez1, Oscar De La Rosa1 y Efraín Salazar1
1 Unidad de Biotecnología Agrícola, Instituto Nacional de Investigaciones Agrícolas (INIA). Centro Nacional de Investigaciones Agropecuarias (CENIAP). Apdo. 4653. Maracay. Venezuela. email: iperez@inia.gob.ve; dperez@inia.gob.ve
RESUMEN
Una colección importante de especies cultivadas y silvestres de Passiflora se encuentra en el Banco de Germoplasma del INIA-CENIAP, en Maracay, Venezuela, y de las cuales es necesario conocer la identidad genética para evitar duplicaciones. Se utilizaron 60 iniciadores de la serie Operon para determinar la huella genética de 17 genotipos e identificar patrones de bandas para cada uno de ellos. Las bandas exhibidas por cada genotipo se numeraron en forma ascendente y secuencial creando así un patrón genético característico para cada genotipo en relación con cada iniciador utilizado así como la tipificación del iniciador en relación a cada genotipo estudiado. El análisis genético determinó que los RAPD permitieron generar para cada genotipo estudiado un conjunto de patrones de bandas que le son característicos y que se convierte en su huella genética. Se establecieron 19 iniciadores con un alto potencial discriminatorio, los cuales deberían ser la base de futuros estudios de diversidad genética en Passiflora. Estos iniciadores permitieron observar diferentes posiciones de banda similares entre los individuos sugiriendo la presencia de varios alelos para esas características. Los patrones RAPD permitieron separar los materiales cultivados de las especies silvestres, encontrándose además un alto grado de variabilidad entre los materiales cultivados.
Palabras clave adicionales: Banco de germoplasma, maracuyá, marcadores moleculares
Genetic fingerprint of wild and commercial genotypes of Passiflora spp. by using RAPD patterns
ABSTRACT
An important collection of cultivated and wild species of Passiflora is maintained at the Germplasm Bank of INIA-CENIAP in Maracay, Venezuela, and it is essential to know the genetic identity of such materials in order to avoid duplicates. Sixty primers from the Operon series were used to determine the genetic fingerprint of 17 genotypes and identify patterns of bands for each genotype. Displayed bands by each genotype were numbered in ascending and sequential order thus creating a distinctive genetic pattern for each genotype in relation to each primer used, as well as the primer characterization in relation to each genotype studied. The genetic analysis established that RAPDs allowed generating a set of specific band patterns characteristic for each genotype that becomes their genetic fingerprint. Nineteen primers with a high discriminatory potential were chosen and should be the base of further genetic studies of Passiflora genetic diversity. These primers permitted observing different positions of similar bands among the individuals suggesting the existence of several alleles for these characteristics. RAPD patterns allowed separating cultivated materials from wild types, finding a high degree of variability among the cultivated materials.
Additional key words: Germplasm bank, passion fruit, molecular markers
Recibido: Julio 1, 2008 Aceptado: Agosto 18, 2009
INTRODUCCIÓN
El género Passiflora (Passifloraceae) tiene una gran diversidad de especies, distribuidas en distintas zonas agroecológicas de las regiones tropicales. En Venezuela tiene representación en gran parte del territorio nacional, lo cual resalta la importancia florística del género (Delascio-Chitty, 2006; Mazzani et al., 1999; Pérez et al., 2001). De la misma manera, la importancia económica del género se acentúa con los distintos usos comerciales de algunas de las especies, principalmente Passiflora edulis f. flavicarpa, P. cuadrangularis y P. mollisima (Mazzani et al., 1999; Pérez et al., 2001).
Las expediciones realizadas en Venezuela han permitido colectar un amplio número de muestras de Passiflora reuniendo un número importante de materiales de este género, tanto cultivados como silvestres (Mazzani et al., 1999; Pérez et al., 2001). Estas accesiones se encuentran en el Banco de Germoplasma del INIA-CENIAP, en Maracay estado Aragua. Los materiales colectados han sido objeto de caracterizaciones morfológicas, fisiológicas y agronómicas. Sin embargo, para evitar duplicaciones en las colecciones, se hace imprescindible conocer con mayor precisión la identidad genética de las accesiones. En estos casos, el uso de técnicas moleculares basadas en el estudio directo del material genético permite el establecimiento de la huella genética a través de marcadores basados en la secuencia del ADN, estableciendo patrones específicos del individuo o planta bajo estudio (Henry, 1997). Una de las ventajas de usar las técnicas basadas en el ADN, es que la huella genética es independiente del efecto del ambiente y se mantiene constante en diversos tejidos y etapas de desarrollo del organismo. La semejanza entre huellas genéticas depende de la proximidad genética de los materiales probados (Jiménez, 1996).
Las huellas genéticas de plantas se han establecido mediante el uso de varias técnicas moleculares, entre las cuales destacan los patrones de bandas de amplificación al azar de ADN polimórfico (RAPD) (Fernández, 2004). Estos marcadores han ido creciendo en su utilidad en los últimos años por su alto nivel de resolución, facilidad de implementación y su confiabilidad (Hong, 2007).
Diversos autores han utilizado exitosamente la técnica RAPD para el estudio de P. edulis f. flavicarpa (Otoni et al., 1995; Carneiro et al., 2002; Viana et al., 2003) o de otros materiales de Passiflora (Fajardo et al., 1998; Crochemore et al., 2003). Asimismo, para este género se tiene la ventaja de contar con metodologías eficientes para la extracción de ADN de calidad para realizar análisis basados en PCR (Molinari y Crochemore, 2001).
En virtud de las experiencias en la caracterización molecular de Passiflora, el presente trabajo tuvo como objetivo determinar los mejores iniciadores e identificar mediante patrones de bandas RAPD los materiales de la colección de Passiflora del INIA-CENIAP, a fin de complementar la evaluación de la identidad genética de las plantas.
MATERIALES Y MÉTODOS
Se utilizaron 12 genotipos de Passiflora edulis f. flavicarpa cultivada y cinco genotipos de Passiflora silvestres (Cuadro 1), colectados en diversas regiones de Venezuela y conservados ex situ en el Banco de Germoplasma del INIA-CENIAP.
Cuadro 1. Materiales genéticos cultivados de Passiflora edulis f flavicarpa y silvestres del género Passiflora, y concentración de ADN genómico aislado
| Genotipo | Nº BG | Nombre científico | Nombre común | Procedencia | Concentración (ng·µL-1) |
| P1 | 138 | P. edulis f. flavicarpa | Parchita maracuyá | Est. Exp. Montalbán, UCV. Bejuma, Edo. Carabobo | 147,05 |
| P2 | 140 | Est. Exp.Montalbán, UCV. Bejuma, Edo. Carabobo | 251,65 | ||
| P3 | 142 | Est. Exp.Montalbán, UCV. Bejuma, Edo. Carabobo | 680,50 | ||
| P4 | 146 | Moralito, Santa Bárbara, Edo. Zulia | 496,90 | ||
| P5 | 148 | Tucani, Sur del Lago, Edo. Zulia | 108,15 | ||
| P6 | 152 | Semilla comercial. Agroisleña, Cagua, Edo. Aragua | 751,50 | ||
| P7 | 165 | La Pereza, Edo. Mérida | 910,50 | ||
| P8 | 167 | Sur del Lago, Edo. Zulia | 680,50 | ||
| P9 | 170 | Colectada en el Sistema de Riego Carambú, Edo. Trujillo | 752,00 | ||
| P10 | 171 | Donada. INIA-Monagas. Colectada en Aragua de Maturín | 553,00 | ||
| P11 | 172 | Donada. INIA-Monagas. Colectada en Caicara | 869,50 | ||
| P12 | 174 | Donada. INIA-Monagas. Colectada en Caripe | 501,00 | ||
| P13 | 122 | P. foetida | Parcha hedionda | Tucacas, Edo. Falcón | 1212,00 |
| P14 | 176 | P. maliformis | Granadilla de piedra | Sector La Trilla, Sta. Rosa del Sur, Edo. Carabobo. | 800,50 |
| P15 | 180 | P. subpeltata | Granada cimarrona | Monte Carmelo, Edo. Lara | 423,65 |
| P16 | 181 | P. cincinnata | Parchita andina | Colectada en la vía Barquisimeto-Duaca, Edo. Lara | 1828,00 |
| P17 | 182 | P. giberti | Burucuyá | Donada. UCLA, Edo. Lara | 628,00 |
Genotipo: Código asignado en este trabajo; Nº BG: Nº de accesión en el Banco de Germoplasma del INIA-CENIAP
Para el aislamiento de ADN se tomaron 0,2 g de tejido foliar y se maceraron con nitrógeno líquido. Para la extracción de ADN genómico se utilizó el método de CIAT (1999) modificado. Para la amplificación al azar de los ADN, se tomaron 15 µL por reacción de amplificación RAPD conteniendo 10 ng ADN, 1,5 µL solución tampon, 1,33 mM MgCl2, 0,22 mM de cada dNTP, 0,3 µM del iniciador, 1U Taq ADN polimerasa y 0,33 mM BSA. El proceso de amplificación se realizó en un termociclador PTC 100 MJ Research utilizando un programa con un paso inicial a 94 °C por 5 min, seguido por 45 ciclos a 93 °C por 30 s, 36 °C por 30 s y 72 °C por 1 min, más una extensión final a 72 °C por 7 min. Se utilizaron sesenta (60) iniciadores de anclaje al azar de las series OPA (01 al 20), OPB (01 al 10), OPF (01 al 20) y OPM (01 al 10) de Operon Technologies.
Los productos de amplificación se separaron en geles de agarosa al 1,5 % durante 1,5 h a 90 mA y 85 V, colocando como marcador de peso molecular ADN del plásmido pBR322 digerido con Bst 01. Los fragmentos RAPD fueron teñidos con bromuro de etidio al 0,0002 %, y visualizados en un digitalizador de imágenes Biorad Chemidoc, utilizando el programa Quantity One 4.2.
La identidad genética de los ADN obtenidos se basó en el conjunto de patrones de bandas RAPD amplificadas para cada uno de los genotipos con los 60 iniciadores estudiados. Cada patrón se comparó con el de todos los genotipos evaluados por iniciador, determinando así las diferencias y similitudes. Los patrones de bandas diferentes fueron numerados en secuencia ascendente, identificándose patrones iguales con el mismo número. Se evaluó el número de patrones diferentes obtenidos con cada iniciador, lo cual evidenció el nivel de polimorfismo visualizado y, por ende, la capacidad discriminatoria de genotipos por cada secuencia utilizada.
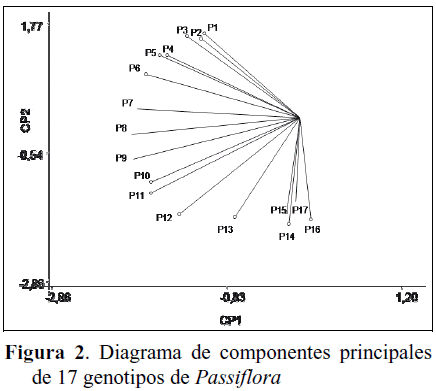
Con los datos obtenidos a través del procesamiento de imágenes de los geles de agarosa se construyó una matriz binaria que incluyó la presencia o ausencia de fragmentos amplificados en el gel. La distancia genética entre los materiales fue calculada usando el programa Popgene 1.31 (Yeh et al., 1999). Este programa establece matrices estandarizadas para la distancia genética (Nei, 1972) y matrices de distancia genética corregida para muestras pequeñas (Nei, 1978). Todos los agrupamientos se realizaron usando el método UPGMA y el agrupamiento resultante fue expresado como un dendrograma. Adicionalmente, se realizó un análisis de componentes principales usando el programa Infostat Profesional 1.1 para visualizar la distribución espacial de los 17 materiales como ejemplo de la diversidad genética encontrada.
RESULTADOS Y DISCUSIÓN
Los resultados del aislamiento de ADN se presentan en el Cuadro 1. El protocolo de extracción utilizado se considera que fue efectivo en el caso de muestras foliares de Passiflora, cuyas especies presentan altos contenidos de fenoles, taninos y otras sustancias en las hojas que dificultan la extracción. Las concentraciones obtenidas fueron muy superiores a las requeridas para realizar la amplificación al azar de los ADN (10 ng·µL-1).
En líneas generales se observó un mayor rendimiento de ADN en los materiales silvestres (980 ng·L-1 en promedio) que en los materiales cultivados (550 ng·µL-1 en promedio).
El ADN se separó electroforéticamente como una banda discreta, al mismo nivel que la banda correspondiente al ADN usado como patrón de concentración, lo cual indicó que las moléculas aisladas estaban intactas.
Con relación al polimorfismo mostrado por los 60 iniciadores para cada uno de los genotipos estudiados se obtuvo que 17 iniciadores presentaron un polimorfismo bajo (6-9 patrones de bandas), 24 iniciadores un polimorfismo intermedio (10-14) y un grupo de 19 iniciadores mostraron alto polimorfismo (15-17) considerados estos últimos como los de mayor potencial discriminante (Cuadro 2). Se destacaron los marcadores OPA02, OPA04 y OPA17, los cuales mostraron capacidad para discriminar claramente los 17 genotipos.
Cuadro 2. Patrones de bandas generados por cada iniciador con relación a los 17 genotipos de Passiflora estudiados
| Nº | Iniciador | Patrones de bandas generados | Nº | Iniciador | Patrones de bandas generados | Nº | Iniciador | Patrones de bandas generados | Genotipos no discriminados |
| 1 | OPA 06 | 6 | 21 | OPA 14 | 10 | 41 | OPM 07 | 14 |
|
| 2 | OPA 15 | 6 | 22 | OPA 20 | 10 | 42 | OPA 03 | 15 | P5-P6; P9-P10 |
| 3 | OPF 01 | 6 | 23 | OPF 03 | 10 | 43 | OPF 10 | 15 | P1-P2-P3, P8-P9 |
| 4 | OPF 05 | 7 | 24 | OPF 14 | 10 | 44 | OPA 05 | 15 | P5-P6; P10-P11 |
| 5 | OPF 06 | 7 | 25 | OPM 05 | 10 | 45 | OPA 09 | 15 | P2-P4,P8-P9 |
| 6 | OPF 13 | 7 | 26 | OPB 03 | 11 | 46 | OPA 11 | 15 | P11-P12, P4-P15 |
| 7 | OPB 06 | 8 | 27 | OPF 02 | 11 | 47 | OPA 19 | 15 | P5-P7,P8-P13 |
| 8 | OPA 12 | 8 | 28 | OPF 20 | 11 | 48 | OPB 08 | 15 | P3-P4; P7-P8 |
| 9 | OPM 08 | 8 | 29 | OPA 10 | 12 | 49 | OPB 09 | 15 | P12-P14, P15-P16 |
| 10 | OPF 07 | 9 | 30 | OPA 13 | 12 | 50 | OPM 03 | 15 | P5-P6; P9-P10 |
| 11 | OPF 09 | 9 | 31 | OPA 16 | 12 | 51 | OPM 10 | 15 | P3-P4, P8-P9 |
| 12 | OPF 04 | 9 | 32 | OPF 08 | 12 | 52 | OPA 18 | 16 | P11-P12 |
| 13 | OPF 18 | 9 | 33 | OPF 16 | 12 | 53 | OPB 02 | 16 | P7-P8 |
| 14 | OPF 19 | 9 | 34 | OPF 17 | 12 | 54 | OPB 05 | 16 | P1-P3 |
| 15 | OPM 01 | 9 | 35 | OPM 04 | 12 | 55 | OPB 07 | 16 | P7-P9 |
| 16 | OPM 02 | 9 | 36 | OPB 01 | 13 | 56 | OPB 10 | 16 | P14-P15 |
| 17 | OPM 09 | 9 | 37 | OPF 15 | 13 | 57 | OPF 12 | 16 | P7-P8 |
| 18 | OPA 01 | 10 | 38 | OPB 04 | 14 | 58 | OPA 02 | 17 | - |
| 19 | OPA 07 | 10 | 39 | OPF 11 | 14 | 59 | OPA 04 | 17 | - |
| 20 | OPA 08 | 10 | 40 | OPM 06 | 14 | 60 | OPA 17 | 17 | - |
Los iniciadores OPF1 y OPF14 no mostraron alto polimorfismo, lo cual contradice lo señalado por Bellon et al. (2007) quienes los reportaron como altamente polimórficos en Passiflora. Estas diferencias en la capacidad para detectar polimorfismo en este género hacen suponer que los diferentes genotipos juegan un papel importante en la efectividad discriminatoria de estos iniciadores.
Los patrones RAPD obtenidos permitieron establecer una huella genética para cada material estudiado. Se observó una alta variabilidad entre los genotipos de P. edulis f. flavicarpa (Figuras 1 y 2). Esta variabilidad podría explicarse por la naturaleza alógama de las Passiflora. Sin embargo a pesar de la variabilidad observada, los genotipos de P. edulis f. flavicarpa conformaron un grupo separado de las especies silvestres estudiadas. Las distancias genéticas obtenidas para los genotipos de la forma flavicarpa son comparables a las observadas entre las especies silvestres, corroborando la alta variabilidad genética en P. edulis (Figura 1).
Es importante destacar que los 19 iniciadores altamente polimórficos pueden ser la base de los futuros estudios de caracterización en genotipos del género Passiflora, ya que permitieron identificar los genotipos del género. Asimismo, permitieron observar diferentes posiciones de bandas similares entre los individuos sugiriendo la presencia de varios alelos para esas características. Esto reduciría significativamente los esfuerzos de caracterización molecular. Sin embargo a fin de afinar los detalles de la clasificación taxonómica, se hace necesario ampliar el estudio molecular de los genotipos con más marcadores RAPD e integrar otro tipo de marcadores.
En la Figura 2 se observa que las especies silvestres se orientan más hacia el eje con el componente principal 1, mostrando una correlación ligeramente positiva con éste. En el caso de las especies cultivadas se orientan más hacia el lado negativo del componente 1 y se distribuyen más ampliamente en relación al componente 2. El material 13 correspondiente a un genotipo de P. foetida es el que más cercano se encuentra a los materiales cultivados de P. edulis f. flavicarpa, dentro de los cuales se evidencia una mayor variabilidad genética.
CONCLUSIONES
Se generó para cada genotipo de Passiflora estudiado un conjunto de patrones de bandas que le son característicos y que se convierte en su huella genética.
Los patrones RAPD permitieron separar las especies silvestres de los materiales cultivados, encontrándose además, entre estos últimos, un alto grado de variabilidad.
Se establecieron 19 iniciadores con un alto potencial discriminatorio, los cuales deberían ser la base de futuros estudios de diversidad genética de este género.
AGRADECIMIENTO
Al Instituto Nacional de Investigaciones Agrícolas, por el financiamiento ID-ARA-05-01003.
LITERATURA CITADA
1. Bellon, G., F.G. Faleiro, K.P. Junqueira, N.T.V. Junqueira, E.C. Dos Santos, M.F. Braga y C.T. Guimarães. 2007. Genetic variability of wild and commercial passion fruit (Passiflora edulis Sims.) accessions using RAPD markers. Rev. Bras. Frutic. 29: 124-127. [ Links ]
2. Carneiro, M.S., L.E.A. Camargo, A.S.G. Coelho, R. Vencovsky, R.P.L. Júnior, N.M.C. Stenzel y M.L.C. Vieira. 2002. RAPD-based genetic linkage maps of yellow passion fruit (Passiflora edulis Sims. f. flavicarpa Deg.). Genome 45: 670-678. [ Links ]
3. CIAT. 1999. Taller integración de fitopatología, mejoramiento y biología molecular para el desarrollo de resistencia al añublo del arroz. Centro Internacional de Agricultura Tropical. Octubre 14-18, Cali, Colombia. [ Links ]
4. Crochemore, M., H.B. Correa y L.G. Esteves. 2003. Genetic diversity in Passion Fruit (Passiflora spp.) evaluated by RAPD Markers. Brazilian Archives of Biology and Technology 46: 521-527. [ Links ]
5. Delascio-Chitty, F. 2006. El género Passiflora L. en el Hato Piñero, Estado Cojedes, Venezuela. Acta Bot. Venez. 29: 27-38. [ Links ]
6. Fajardo, D., F. Angel, M. Grum, J. Tohme, M. Lobo, W. M. Roca e I. Sánchez. 1998. Genetic variation analysis of the genus Passiflora L. using RAPD markers. Euphytica 101: 341-347. [ Links ]
7. Fernández, H. 2004. Uso de marcadores moleculares en la caracterización de bancos de germoplasma. Ceniap Hoy N° 5. http://www.ceniap.gov.ve/pbd/RevistasTecnicas/ceniaphoy/articulos/n5/. Consulta del 05/05/2008. [ Links ]
8. Henry, R. 1997. Practical applications of plant molecular biology. Chapman & Hall. London. [ Links ]
9. Hong, J. 2007. DNA fingerprinting from botanicals. Vol. 6 Nº 2. http://www.Innovationmagazine.com/innovation/ (consulta 18/08/09). [ Links ]
10. Jiménez, A. 1996. Ingeniería genética. Aplicaciones e implicaciones. Puertas a la lectura. Universidad de Extremadura. Badajoz. España 2: 37-41. [ Links ]
11. Mazzani, E., D. Pérez y W. Pacheco. 1999. Distribución y uso de especies del género Passiflora (Passifloraceae) en las zonas altas de los estados Lara y Falcón, Venezuela. Plant Genetic Resources Newsletter 119: 24-32. [ Links ]
12. Molinari, H.B. y M.L. Crochemore. 2001. Genomic DNA extraction from Passiflora spp. for PCR-RAPD analyses. Rev. Bras. Frutic. 23: 447-450. [ Links ]
13. Nei, M. 1972. Genetic distance between populations. American Naturalist 106: 238-292. [ Links ]
14. Nei, M. 1978. Estimation of average heterozigosity and genetic distance from a small number of individuals. Genetics 89: 583-590. [ Links ]
15. Otoni, W. C., N. W. Blackhall, F. B. d'Utra Vaz, V.W. Casali, J.B. Power y M.R. Davey. 1995. Somatic hybridization of the Passiflora species, P. edulis f. flavicarpa Degener and P. incarnata L. J. Exp. Bot. 46: 777-785. [ Links ]
16. Pérez, D., E. Mazzani y W. Pacheco. 2001. Colecta de Passifloras silvestres y cultivadas en zonas altas de los estados Aragua y Miranda. Región centro-norte de Venezuela. Plant Genetic Resources Newsletter 125: 11-15. [ Links ]
17. Viana, A.P., T.N Santana, M. Gonzaga, M. Magalhães de Souza, J.F. Martinez y A. Teixeira. 2003. Diversidade genética entre genótipos comerciais de maracujazeiro-amarelo (Passiflora edulis f. flavicarpa) e entre espécies de passifloras nativas determinada por marcadores RAPD. Rev. Bras. Frutic. 25: 489-493. [ Links ]
18. Yeh, F., R. Yang y T. Boyle. 1999. Popgene: Microsoft Windows-based freeware for Population Genetic Analysis Version 1.32, University of Alberta. Edmonton. 29 p. [ Links ]