Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Bioagro
versión impresa ISSN 1316-3361
Bioagro vol.24 no.3 Barquisimeto dic. 2012
Variabilidad genética de Spongospora subterranea f. sp. subterranea en Colombia
Inés Osorio-Giraldo1, Marcela Orozco-Valencia1, Pablo Gutiérrez-Sánchez1, Paola González-Jaimes2 y Mauricio Marín-Montoya1
1 Escuela de Biociencias, Facultad de Ciencias, Universidad Nacional de Colombia. Medellín. e-mail: inesosorio@gmail.com, morozco@unal.edu.co, paguties@unal.edu.co, mamarinm@unal.edu.co
2 Facultad de Ciencias Agrarias, Politécnico Colombiano Jaime Isaza Cadavid, Medellín. e-mail: epgonzalez@elpoli.edu.co
RESUMEN
La sarna polvosa de la papa (Spongospora subterranea f. sp. subterranea - Sss) es una de las enfermedades re-emergentes más importantes de este cultivo en Colombia. El efecto deletéreo de Sss no sólo se debe al daño directo sobre el sistema radicular y la calidad de los tubérculos, sino también a que sus zoosporas transmiten el Potato mop-top virus (PMTV, Pomovirus). Sss genera quistosoros que actúan como estructuras de resistencia del patógeno en el suelo, pudiendo permanecer viables por varias décadas. Por esto, su manejo debe fundamentarse en el uso de variedades tolerantes a la enfermedad y en el empleo de tubérculo-semilla certificado. En esta investigación se evaluaron los niveles de variación de 127 aislamientos de Sss obtenidos en los departamentos de Antioquia, Boyacá, Cundinamarca y Nariño, con el fin de apoyar la selección de aislamientos de Sss a evaluar en programas de mejoramiento genético de papa y en el diseño de pruebas de detección asintomática del patógeno. Para esto se secuenciaron regiones ITS del ADN ribosomal y se condujeron análisis de agrupamiento, encontrándose la presencia de tres variantes principales de Sss, dos de las cuales (Tipos I y II) han sido reportadas en otros países del mundo, mientras que el Tipo III hasta ahora sólo se ha encontrado en Colombia. Con base en esta información se diseñó una prueba de PCR-RFLPs que permite identificar los tres ribotipos principales del patógeno.
Palabras clave adicionales: ADNr, ITS, PCR, sarna polvosa, Solanum tuberosum
Genetic variability of Spongospora subterranea f. sp. subterranea in Colombia
ABSTRACT
Powdery scab (Spongospora subterranea f. sp. subterranea – Sss) is one of the most important re-emerging diseases of potato crops in Colombia. Sss can cause severe root damage and compromise the quality of tubers. Moreover, Sss zoospores can also serve as vector of Potato mop-top virus (PMTV, Pomovirus). Sss can survive in the soil for decades due to the formation of resistance structures or cystosori. For this reason, the disease can only be efficiently controlled by using resistant potato varieties and certified seed tubers. In this work, the level of variation of 127 Sss isolates from the Colombian provinces of Antioquia, Boyacá, Cundinamarca and Nariño was evaluated. This information is of great value for genetic improvement programs of potato and can also be used in the design of asymptomatic detection tests. Cluster analysis of ITS rDNA sequences revealed the existence of three Sss variants. Types I and II have been reported in other countries while type III has only been found in Colombia. This information was used to design a PCR-RFLP test to discriminate the pathogen´s ribotypes.
Additional key words: rDNA, ITS, PCR, Powdery scab, Solanum tuberosum
Recibido: Marzo 5, 2012 Aceptado: Agosto 20, 2012
INTRODUCCIÓN
El cultivo de la papa (Solanum tuberosum L.) se ve afectado por diversos microorganismos fitopatógenos e insectos plagas, destacándose en los últimos años la re-emergencia de la enfermedad denominada sarna polvosa causada por el plasmodiofórido Spongospora subterranea (Wallroth) Lagerheim f. sp. subterranea Tomlinson (Sss). Su efecto no sólo se debe a la reducción en la producción, sino también al deterioro en la calidad y apariencia cosmética de los tubérculos que reduce drásticamente su valor en el mercado (Merz y Fallon, 2009). Sss es el vector natural del Potato mop top virus (PMTV), uno de los problemas virales de la papa que tiene carácter cuarentenario para diferentes países del mundo (Salazar, 2006; Gil et al., 2011).
Los síntomas de la sarna polvosa incluyen la presencia en los tubérculos de lesiones pustulosas corchosas con centros pulverulentos formados a partir de la agregación de los quistosoros del patógeno. Con el avance de estas lesiones es frecuente la ruptura del peridermo, aunque el nivel de penetración en los tubérculos es generalmente superficial (Carreño, 2009). Bajo condiciones óptimas para el desarrollo de la enfermedad, es frecuente la unión de lesiones que generan la deformación de los tejidos y la formación de cánceres o agallas (Harrison et al., 1997). En Colombia, la enfermedad además de causar los daños descritos, genera el deterioro del sistema radicular de las plantas, al inducir la formación de agallas múltiples en forma de cuentas que causan la disminución de la superficie de absorción de las raíces, induciendo marchitez y finalmente la muerte de las plantas (Gilchrist, 2009). La infección en las raíces, especialmente cuando se forman agallas, puede causar una reducción en la toma de agua y de nutrientes por parte de las plantas (Falloon et al., 2005).
En los departamentos de Cundinamarca, Boyacá, Nariño y Antioquia, Sss afecta severamente las variedades Parda Pastusa, Diacol Capiro y papa criolla (S. phureja). Los síntomas se observan en raíces y tubérculos, pero hay variación entre las zonas paperas de Colombia, en cuanto a la distribución de los síntomas en los diferentes órganos de la planta. En Cundinamarca, Boyacá y el Norte de Antioquia es más frecuente observar pústulas sobre los tubérculos de las variedades Parda Pastusa y Diacol Capiro, mientras que en el departamento de Nariño y el Oriente de Antioquia es común observar agallas en raíces. Hasta ahora no se conoce si estas diferencias se deben a variaciones en las condiciones ambientales de las regiones o a la ocurrencia de razas del patógeno (Jaramillo y Botero, 2007; Carreño, 2009).
El control de la sarna polvosa de la papa es difícil de lograr debido a la carencia de variedades comerciales resistentes y de tratamientos químicos o biológicos efectivos contra Sss. Por esto, su manejo debe estar enmarcado bajo un esquema integrado del cultivo, en el que se incluyan aspectos como la rotación, el manejo adecuado del riego y drenaje, la sanidad del tubérculo-semilla, la utilización de organismos antagonistas (ej. Trichoderma spp.), la utilización de cultivos trampa, la desinfestación de herramientas de trabajo y finalmente, una legislación que restrinja el movimiento de material contaminado y supervise más detalladamente los procesos de certificación de semilla. Desafortunadamente en el país pocas de éstas prácticas son utilizadas por los agricultores y por el contrario hay un desconocimiento generalizado del manejo de la enfermedad, llegándose incluso a confundir con problemas bacteriales o de Rhizoctoniasis (Carreño, 2009).
La problemática causada por esta enfermedad en las regiones cultivadoras de papa de Colombia, hace necesario que se intensifiquen los esfuerzos por desarrollar metodologías de diagnóstico de suelos y material vegetal, que eviten la diseminación generalizada del plasmodiofórido y de su virus asociado PMTV entre regiones infectadas y libres de enfermedad; siendo especialmente importante el desarrollo de procedimientos que garanticen la sanidad de los lotes dedicados a la producción de semilla. Entre las diversas metodologías desarrolladas en el mundo para el diagnóstico de este patógeno se destacan la técnica de ELISA con anticuerpos policlonales (Harrison et al., 1993; Walsh et al., 1996), PCR convencional usando cebadores específicos (Bulman y Marshall, 1998; Bell et al., 1999) y recientemente PCR en tiempo real basado en el uso de sondas TaqMan (Qu et al., 2011) y pruebas de flujo lateral (Bouchek-Mechiche et al., 2011). Sin embargo, estas técnicas son de aplicación limitada en nuestro medio al no estar diseñadas con base en los genotipos locales de los patógenos presentes en las regiones paperas del país. Con el fin de suplir dicha carencia de información, en esta investigación se evaluaron los niveles de variabilidad genética de aislamientos de Sss procedentes de las principales zonas cultivadoras de papa de Colombia, mediante el análisis de secuencias de las regiones ITS del ADN ribosomal (ADNr).
MATERIALES Y MÉTODOS
Colección y procesamiento de muestras. Para la colección de muestras de quistosoros de Sss proveniente de suelo, raíces y tubérculos, se seleccionaron lotes en fincas con antecedentes de infección según el registro histórico de los agricultores, siguiendo la metodología de Jaramillo y Botero (2007), en la cual se utiliza un juego de tamices de 100 y 25 µm para la obtención de los quistosoros. Por cada lote se tomaron tejidos de plantas o tubérculos sintomáticos y una muestra de suelo de 500 g. Los sitios de colección incluyeron las regiones cultivadoras de papa de los municipios de la Unión y Santa Rosa de Osos (Antioquia), Zipaquirá, Tabio, Subachoque y Villa Pinzón (Cundinamarca), Tunja, Siachoque, Soracá, Oicatá (Boyacá), y Pasto e Ipiales (Nariño). En la investigación se propuso obtener un mínimo de 30 muestras de Sss por departamento.
Los quistosoros obtenidos a partir de muestras de suelos fueron inoculados en plantas de Nicotiana benthamiana y S. phureja, utilizadas como plantas señuelo para incrementar el inóculo siguiendo la metodología de Vélez (2007), manteniéndose en casa de malla en el Centro Experimental Paysandú (corregimiento Santa Elena, municipio de Medellín, Colombia).
Extracción de ADN. Para la obtención del ADN se empleó una modificación de la metodología de Zolan y Pukkila (1986), a partir de quistosoros extraídos y de tejidos afectados de las plantas de papa y plantas señuelo. Cuando la presencia de inhibidores afectaba la eficiencia de la PCR, la extracción de ADN se realizó utilizando el kit DNeasy Plant (Qiagen, EEUU), siguiendo las instrucciones del fabricante. Para determinar la concentración del ADN extraído, se utilizó la lectura de absorbancia en las longitudes de onda de 260 y 280 nm mediante un Nanodrop 2000C (Thermo), obteniéndose un rango entre 10 y 112 ng·μL-1 de ADN con una relación 260:280 de 1,5 a 1,9. La integridad del ADN se determinó realizando electroforesis en gel de agarosa al 0,8% suplementado con bromuro de etidio (10 mg·mL-1). La visualización de las bandas se realizó utilizando el sistema digital de análisis Bio Doc Analyze (Biometra).
PCR y secuenciación de ITS del ADNr: Para la evaluación de los niveles de variación genética de Sss se realizaron PCR empleando los cebadores específicos Spo8 (5' CTG GGT GCG ATT GTC TGT TG 3') y Spo9 (5' CAC GCC AAT GGT TAG AGA CG 3') diseñados por Bulman y Marshall (1998), así como los cebadores SsF (5´ GTC GGT TCT ACC GGC AGA CC 3´) y SsR (5´ GCA CGC CAA TGG TTA GAG ACG 3´), reportados por Qu et al. (2006), que amplifican productos de 391 y 434 pb de la región ITS del ADNr, respectivamente. Las reacciones de PCR incluyeron 0,1 µM de cada cebador, 1 U de Taq ADN polimerasa recombinante (Fermentas, Lithuania), 0,2 mM de cada dNTP, 1X de buffer de enzima (100 mM Tris-HCl (NH4)2SO2 pH 8,8), 2 mM MgCl2, 1 µL de una dilución 50 ng·µL-1 de ADN y un volumen total de 25 µL. Las amplificaciones se realizaron en un termociclador T3 (Biometra) y consistieron de una desnaturalización inicial a 98 °C por 3 min, seguida por 35 ciclos de 94 °C por 30 s, 55 °C por 30 s, 72 °C por 1 min y un período final de extensión a 72 °C por 10 min. Luego de la amplificación, se tomaron 5 µL de los productos de reacción para su separación por electroforesis en gel de agarosa al 1,5 %.
Los amplicones obtenidos fueron purificados mediante los kits QIAquick PCR Purification y QIAquick Gel Extraction (Qiagen), para proceder a su secuenciación directa en ambas direcciones utilizando los cebadores empleados en el PCR y el kit Big Dye Terminator Cycle Sequencing Ready Reaction (PE Applied Biosystems). Su corrido y separación se realizó en un secuenciador ABI Prism 3730xl (PE Applied Biosystems) de la compañía Macrogen. Las secuencias obtenidas con cada cebador, fueron editadas mediante el software BioEdit 6.0.6 y Chromas 1.45, construyéndose consensos y confirmándose su validez por comparación con las bases de datos moleculares con el programa BLASTn (http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi). Paralelamente, se obtuvieron del GenBank secuencias de Sss de las regiones estudiadas para su alineación con las generadas en este trabajo mediante el software Clustal W. Además se utilizaron las secuencias de los plasmodiofóridos Plasmodiophora brassicae, Polymyxa graminis, Polymyxa betae, S. nasturtii y Sorosphaera viticola, como grupos externos de análisis (outgroups). Los alineamientos fueron utilizados en la construcción de una matriz Nexus y el análisis de agrupamiento se realizó mediante un algoritmo de Máxima Parsimonia utilizando el software PAUP 4.0b. Para evaluar el soporte estadístico de cada agrupación, se realizó un análisis de Bootstrap con 500 iteraciones. RFLPs. Con el fin de proponer una prueba de diagnóstico molecular de las variantes de Sss, se realizó un análisis in silico de RFLPs a partir de las secuencias de Sss generadas en esta investigación y otras depositadas en el GenBank. Para esto se utilizó el software Webcutter 2.0 (http://rna.lundberg.gu.se/cutter2/) con las enzimas Bsp143I, EcoRI, EcoRV, Hin6I, MspI, TaqI y Tru1I, seleccionadas por ser de fácil acceso para laboratorios de diagnóstico molecular. La validez de la prueba fue confirmada mediante PCR-RFLPs con las enzimas Bsp143I y Hin6I y aislamientos representativos de los tres grupos genéticos principales detectados en el trabajo. Las reacciones consistieron de 15 µL, conteniendo 1,5 µL de buffer 10X, 2 µL de enzima y 11,5 µL de producto de PCR. Las reacciones fueron incubadas al baño María a 37 °C durante 24 h. La separación se efectuó por electroforesis en gel de agarosa al 2,5 %.
RESULTADOS
Colección y procesamiento de muestras. Se obtuvieron 210 muestras de tubérculos y raíces de papa con síntomas de sarna polvosa y de suelos con historia de la presencia del patógeno en los departamentos de Antioquia, Boyacá, Cundinamarca y Nariño. Los análisis de variabilidad se realizaron con las secuencias que presentaron tamaños superiores a 390 pb, lográndose el objetivo de contar con al menos 30 secuencias por cada departamento bajo análisis (Cuadro 1).
Cuadro 1. Procedencia de muestras de raíz y tubérculos con síntomas de sarna polvosa y de suelos de lotes con registro histórico de la enfermedad en 4 departamentos cultivadores de papa de Colombia
| Departamento | Municipio | Procedencia | Nº de muestras |
| Antioquia (33 muestras) | La Unión | Tubérculo, S. tuberosum var. Capiro | 3 |
| La Unión | Suelo de lote con S. tuberosum | 7 | |
| La Unión | Raíz, S. tuberosum | 5 | |
| La Unión | Raíz, S. phureja | 6 | |
| La Unión | Tubérculo, S. tuberosum | 1 | |
| La Unión | Tubérculo, S. phureja var. Criolla Colombia | 1 | |
| La Unión | Raíz, S. phureja var. Criolla Colombia | 2 | |
| Santa Rosa | Raíz, S. tuberosum var. Capiro | 2 | |
| Santa Rosa | Tubérculo, S. tuberosum var. Nevada | 1 | |
| Santa Rosa | Tubérculo, S. phureja var. Criolla Colombia | 1 | |
| Santa Rosa | Tubérculo, S. tuberosum var. Capiro | 4 | |
|
|
|
| |
| Boyacá (30 muestras) | Tunja | Raíz, S. phureja var. Criolla Colombia | 2 |
| Tunja | Tubérculo, S. tuberosum | 9 | |
| Tunja | Raíz, S. tuberosum | 1 | |
| Tunja | Suelo con señuelo de N. benthamiana | 1 | |
| Soracá | Raíz, S. phureja var. Criolla Colombia | 2 | |
| Soracá | Suelo con señuelo de S. phureja var. Criolla Colombia | 4 | |
| Soracá | Suelo con señuelo de N. benthamiana | 3 | |
| Oicatá | Suelo con señuelo de N. benthamiana | 4 | |
| Oicatá | Suelo con señuelo de S. phureja var. Criolla Colombia | 1 | |
| Siachoque | Suelo con señuelo de N. benthamiana | 1 | |
| Siachoque | Tubérculo S. tuberosum var. Capiro | 2 | |
|
|
|
|
|
| Cundinamarca (33 muestras) | Zipaquirá | Raíz, S. tuberosum var. Parda Pastusa | 7 |
| Zipaquirá | Suelo con señuelo de N. benthamiana | 1 | |
| Zipaquirá | Raíz, S. tuberosum | 2 | |
| Zipaquirá | Raíz, S. phureja var. Criolla Colombia | 1 | |
| Villapinzón | Raíz, S. tuberosum var. Parda Pastusa | 3 | |
| Villapinzón | Raíz, S. tuberosum var. Suprema | 2 | |
| Villapinzón | Tubérculo, S. tuberosum var. Capiro | 2 | |
| Villapinzón | Raíz, S. tuberosum | 2 | |
| Villapinzón | Suelo con señuelo de S. phureja var. Criolla Colombia | 2 | |
| Villapinzón | Suelo con señuelo de N. benthamiana | 2 | |
| Tabio | Raíz, S. phureja var. Criolla Colombia | 2 | |
| Tabio | Raíz, S. tuberosum var. Parda Pastusa | 2 | |
| Tabio | Suelo con señuelo de S. phureja var. Criolla Colombia | 1 | |
| Tabio | Suelo con señuelo de N. benthamiana | 1 | |
| Subachoque | Raíz, S. tuberosum var. Parda Pastusa | 2 | |
| Desconocido | Tubérculo, S. tuberosum | 1 | |
|
|
|
| |
| Nariño (31 muestras) | Pasto | Suelo con señuelo de N. benthamiana | 11 |
| Pasto | Raíz, S. tuberosum var. Parda Pastusa | 2 | |
| Pasto | Suelo con señuelo de S. phureja var. Criolla Colombia | 10 | |
| Ipiales | Suelo con señuelo de N. benthamiana | 5 | |
| Ipiales | Suelo con señuelo de S. phureja var. Criolla Colombia | 3 |
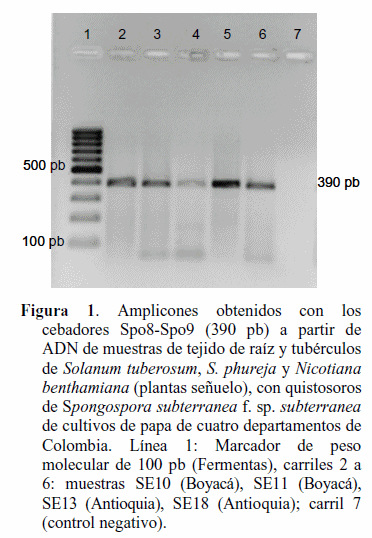
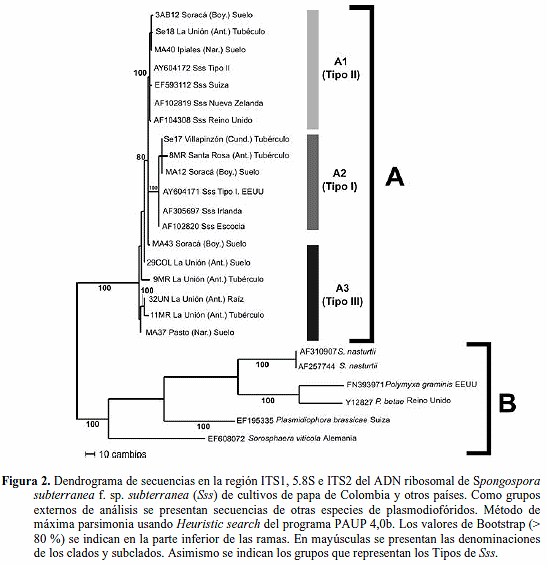
Análisis de variación de regiones ITS del ADNr. Las reacciones de PCR produjeron amplicones del tamaño esperado de ~390 pb con los cebadores Spo8-9 (Figura 1) y de ~430 pb cuando se usaron los cebadores SsF-SsR, en 79 y 48 muestras respectivamente. Para el análisis de agrupamiento sólo se incluyeron los consensos que resultaron de la secuenciación en ambos sentidos. De esta forma, se obtuvieron 33 secuencias de Antioquia, 30 secuencias de Boyacá, 33 secuencias de Cundinamarca y 31 secuencias de Nariño, para un total de 127 secuencias completas (Cuadro 1). El alineamiento final de las secuencias alcanzó 445 sitios, de los cuales 156 caracteres eran constantes, 55 variables pero no informativos y 236 informativos. La longitud del árbol con máxima parsimonia fue de 707, con índices de consistencia (IC) y retención (IR) de 0,78 y 0,88, respectivamente. Dado el alto número de muestras secuenciadas se presenta un dendrograma resultado del análisis de redundancia, mediante el programa Decrease Redundancy (http://web.expasy.org/decrease_redundancy) (Figura 2). Se presentaron dos grandes clados (A y B) soportados por valores de bootstrap de 100 %; el primer clado corresponde a los aislamientos de Sss, mientras que el otro grupo incluyó las demás especies de plasmodiofóridos (S. nasturtii, P. graminis, P. betae y Pl. brassicae), que presentaron un alto número de cambios entre ellos. El clado A se subdividió a su vez en tres grupos (A1, A2 y A3), dos de los cuales corresponden a los Tipos I (A2) y II (A1) definidos por Qu y Christ (2004). En términos porcentuales ambos Tipos representan el 15,7 % (Tipo I) y 18,9 % (Tipo II) de los aislamientos de Sss obtenidos en Colombia, además incluyeron secuencias del patógeno de Irlanda y Escocia para el Tipo I y de Suiza, Nueva Zelanda y Gran Bretaña para el Tipo II. El subgrupo que representa el Tipo I de Sss sólo presentó aislamientos obtenidos de tubérculos o de suelos, pero no de raíz; mientras que el subgrupo representando el Tipo II incluyó indistintamente aislamientos de raíz, tubérculos y suelos.
Por otra parte, el tercer subgrupo (A3) incluyó el mayor número de aislamientos Colombianos de Sss (65,4 %), con algunos de sus miembros presentando hasta 10 cambios entre ellos. En este subgrupo se encontraron indistintamente aislamientos de los cuatro departamentos, diferentes fuentes de muestreo (raíz, tubérculo o suelo) y hospedantes (diferentes variedades de S. tuberosum y S. phureja var. Criolla Colombia). Dadas las diferencias en secuencias de estos aislamientos, se propone nombrar a este grupo como el Tipo III de Sss.
El análisis de redundancia permitió también identificar 12 genotipos entre los 127 aislamientos de Sss de Colombia, cinco de los cuales estuvieron conformados por un sólo aislamiento, tres por dos aislamientos y cuatro presentaron más de diez miembros, siendo el genotipo 11 representante del Tipo I, el 12 del Tipo II y los genotipos 7 y 10 del Tipo III (Cuadro 2). Los demás genotipos al ser únicos o tener como máximo dos representantes, no fueron definidos a nivel de Tipos. Los niveles de identidad para la región ribosomal secuenciada entre estos genotipos fueron superiores al 95% en todos los casos, con una divergencia de 3,3% entre los Tipos I y II; del 5 % entre los Tipos I y III y del 2% entre el II y III. La divergencia entre secuencias de Sss y otras especies de plasmodiofóridos fue mayor al 44%, lo que demuestra la utilidad taxonómica de las regiones ITS para establecer relaciones filogenéticas en este grupo de protozoos (Cuadro 3).
Cuadro 2. Genotipos y posiciones de corte de siete enzimas de restricción en las regiones ITS1, 5.8S e ITS2 del ADN ribosomal de 127 aislamientos de Spongospora subterranea f. sp. subterranea de Colombia
| Genotipo | Rflps | Proporción (%) |
| 1 | 92/185/125,233/221,351/276,346/227/97,114 | 1,6 |
| 2 | 93/186/126,234/222,283,352/277,347/49,228/98,115 | 1,6 |
| 3 | 93/186/126,234/222,283,352/277,347/49,228/98,115 | 0,8 |
| 4 | 93/186/126,234/222,283,352/277,347/49,228/98,115 | 0,8 |
| 5 | 96,339/189/129,237/225,354/280,349/231/101,118 | 1,6 |
| 6 | 97,340/190/130,238/226,355/281,350/232/102,119 | 0,8 |
| 7 | 93/186/126,234/222,283,352/277,347/49,228/89,115 | 39,3 |
| 8 | 92,309/185/125,233/221,289,351/276,346/49,227/97,114 | 0,8 |
| 9 | 92/185/125,233/221,282,351/276,346/227/97,114 | 0,8 |
| 10 | 93/186/126,234/222,283,352/277,347/49,228/98,115 | 22 |
| 11 | 97,340/190/130,238/226,355/281,350/49,232/102,119 | 13,4 |
| 12 | 92/185/125,233/221,350/276,345/49,227/97,114 | 16,5 |
Los números indican las posiciones de los sitios de corte con las enzimas de restricción: Bsp143I/ EcoRI/ EcoRV/ Hind6I/ MspI/ TaqI/ Tru1I. Para las enzimas que presentan varios sitios de corte, se separan las posiciones por el signo coma (,)
Cuadro 3. Matriz de identidad basado en secuencias de las regiones ITS1, 5.8S e ITS2 del ADN ribosomal de aislamientos de Spongospora subterranea f. sp. subterranea de Colombia y otros países del mundo. Se incluyen a S. nasturtii y Plasmodiophora brassicae como especies externas de análisis
| Muestras* | SE17 | SE18 | MA37 | 29COL | AY604172 TipoII | AY604171 TipoI | AF310907 S. nasturtii | EF593112 | AF102820 | AF102819 | EF195335 P. brassicae | AF104308 |
| SE17 | ID | 0,962 | 0,957 | 0,952 | 0,964 | 0,992 | 0,564 | 0,959 | 0,992 | 0,964 | 0,586 | 0,959 |
| SE18 | 0,962 | ID | 0,979 | 0,974 | 0,992 | 0,959 | 0,574 | 0,987 | 0,959 | 0,992 | 0,584 | 0,987 |
| MA37 | 0,957 | 0,979 | ID | 0,984 | 0,982 | 0,954 | 0,579 | 0,977 | 0,954 | 0,982 | 0,598 | 0,977 |
| 29COL | 0,952 | 0,974 | 0,984 | ID | 0,982 | 0,959 | 0,579 | 0,977 | 0,959 | 0,982 | 0,6 | 0,977 |
| AY604172. TipoII | 0,964 | 0,992 | 0,982 | 0,982 | ID | 0,967 | 0,577 | 0,994 | 0,967 | 1 | 0,589 | 0,994 |
| AY604171. Tipo I | 0,992 | 0,959 | 0,954 | 0,959 | 0,967 | ID | 0,564 | 0,962 | 1 | 0,967 | 0,59 | 0,962 |
| AF310907. S. nasturtii | 0,564 | 0,574 | 0,579 | 0,579 | 0,577 | 0,564 | ID | 0,579 | 0,564 | 0,577 | 0,566 | 0,574 |
| EF593112 | 0,959 | 0,987 | 0,977 | 0,977 | 0,994 | 0,962 | 0,579 | ID | 0,962 | 0,994 | 0,589 | 0,989 |
| AF102820 | 0,992 | 0,959 | 0,954 | 0,959 | 0,967 | 1 | 0,564 | 0,962 | ID | 0,967 | 0,59 | 0,962 |
| AF102819 | 0,964 | 0,992 | 0,982 | 0,982 | 1 | 0,967 | 0,577 | 0,994 | 0,967 | ID | 0,589 | 0,994 |
| EF195335. P. brassicae | 0,586 | 0,584 | 0,598 | 0,6 | 0,589 | 0,59 | 0,566 | 0,589 | 0,59 | 0,589 | ID | 0,589 |
| AF104308 | 0,959 | 0,987 | 0,977 | 0,977 | 0,994 | 0,962 | 0,574 | 0,989 | 0,962 | 0,994 | 0,589 | ID |
* SE17 (Tipo I), SE18 (Tipo II), MA37 (Tipo III), 29COL (Tipo III): Aislamientos de Sss de Colombia representativos de los genotipos 11, 12, 7 y 10, respectivamente. AY604172: Sss del TipoII (EEUU), AY604171: Sss del Tipo I (EEUU), AF310907: S. nasturtii, EF593112: Sss (Suiza), AF102820: Sss (Escocia), AF102819: Sss Nueva Zelanda, EF195335: P. brassica, AF104308: Sss (Reino Unido).
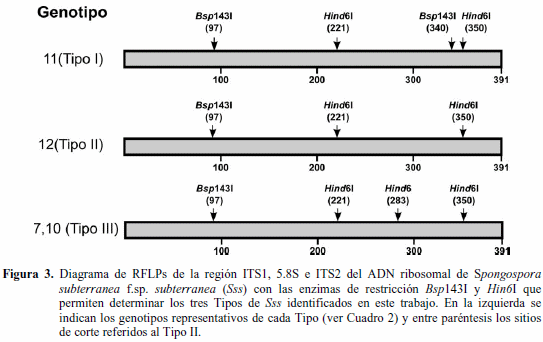
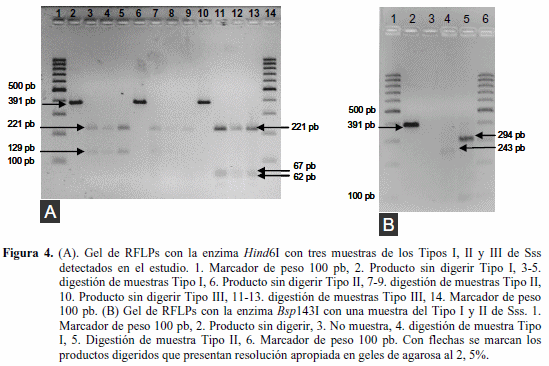
RFLPs. Los análisis de restricción indicaron la existencia de polimorfismos en los sitios de corte de algunas de las enzimas empleadas, entre los grupos principales señalados en el análisis de agrupamiento. Lo anterior puede ser utilizado con fines prácticos cuando se requiera evaluar el genotipo o la mezcla de genotipos asociados a un lote de cultivo, plantas sintomáticas o de tubérculo-semilla con síntomas de sarna polvosa. Por ejemplo, los aislamientos representativos del Tipo II de Sss presentan un sitio de corte para Bsp143I (posición [p.] 93), mientras que para los del Tipo I se presentan dos sitios (p. 97, 340). Los aislamientos del Tipo III tienen tres sitios de corte para la enzima Hin6I (p. 222, 283, 352) en contraste con dos sitios en los del Tipo I y II (p.125-130, 233-238) (Cuadro 2; Figura 3). Por esto se propone la realización de una prueba simple de PCR-RFLPs, a partir de la amplificación de las regiones ITS con cebadores específicos para Sss y su posterior digestión con las enzimas Bsp143I y Hin6I, con el fin de identificar los tres Tipos de Sss. Al evaluar la digestión de amplicones representantes de los tres Tipos de Sss detectados en el estudio, se validó la utilidad de la técnica en geles de agarosa al 2,5%, ya que los perfiles de restricción obtenidos con la enzima Hin6I permiten diferenciar a los aislamientos de los Tipos I y II (fragmentos visibles de 221 y 129 pb) del Tipo III (fragmentos visibles de 221, 67 y 62 pb) (Figura 4A); mientras que los representantes de los Tipos I y II se diferencian por la digestión con la enzima Bsp143I , al generar fragmentos visibles de 243 pb (Tipo I) y de 294 pb (Tipo II) (Figura 4B). Los fragmentos inferiores a 50 pb, no se tuvieron en cuenta en este análisis ya que salen del rango de resolución de la electroforesis en geles de agarosa.
DISCUSIÓN
La sarna polvosa de la papa causada por Sss se ha tornado en uno de los problemas fitosanitarios más limitantes de la producción de papa en Colombia durante los últimos años. Hasta el presente estudio se habían realizado dos estudios de variabilidad genética de Sss con base en análisis de las regiones ITS del ADNr. En el primero de ellos, Hincapié (2006), encontró altos niveles de identidad genética entre los aislamientos de Sss secuenciados y analizados mediante PCR-RFLP, lo que condujo al autor a plantear la posible ocurrencia de una estructura poblacional clonal para este patógeno en el país.
En un estudio más reciente, Carreño (2009) utilizando análisis de secuencias ITS y SSCP (Polimorfismo Conformacional de Banda Simple), que incluyó un alto número de aislamientos de diferentes regiones, estableció la presencia de subpoblaciones del patógeno relacionadas con el tipo de órgano afectado. Así, aislamientos con secuencias ITS del Tipo I, se asociaron exclusivamente con infecciones de tubérculo, mientras que aquellos del Tipo II lo hicieron con síntomas de agallas en raíz. En dicha investigación se descartó además la presencia de patotipos o ecotipos de Sss en Colombia, puesto que en ninguno de los casos se observó una asociación entre la variación genética y el cultivar del hospedero o la zona geográfica de origen de los aislamientos. Así mismo, se comprobó, que en el país existen variantes genéticas de Sss que difieren de las reportadas en otros países, registrándose la presencia de cinco haplotipos del patógeno, dos de los cuales corresponden a los Tipos I y II, mientras que los tres restantes correspondían a nuevas variantes no reportadas en otros países (Carreño, 2009); desafortunadamente las secuencias resultantes de dicho trabajo no fueron publicadas en GenBank y por tanto no pudieron ser incluidas en los análisis de agrupamiento aquí presentados.
El presente estudio amplía aun más la base de conocimiento de los niveles de variación de Sss en Colombia, al detectar 12 genotipos a partir de 127 secuencias de igual número de aislamientos del patógeno, obtenidos en cultivos de los cuatro principales departamentos productores de papa del país. El análisis basado en dichas secuencias, definió la presencia de un clado que representa a Sss, e incluye los dos Tipos mundialmente conocidos para este patógeno (Qu y Christ, 2004), además de un tercer subgrupo (III) que presenta un 5% y 2% de divergencia con respecto a los Tipo I y II, respectivamente. Este Tipo III contiene la mayor proporción de aislamientos del estudio e integró indistintamente cepas de diferentes orígenes geográficos, hospedantes y tejidos sintomáticos. De gran interés resultó el hecho que los aislamientos asociados con el Tipo I procedían de pústulas en tubérculos o de suelos, pero no de agallas de raíz, lo cual confirma la asociación realizada por Carreño (2009) para esta variante de Sss. Sin embargo, los aislamientos asociados al Tipo II provenían indistintamente de raíz, tubérculos o suelos. A partir de estos resultados es necesario evaluar el significado biológico de estas variantes de Sss bajo las condiciones agroecológicas de las regiones cultivadoras de papa del país. Así por ejemplo, es de interés estudiar las reacciones de las variedades colombianas de papa a dichos ribotipos, así como su capacidad de transmisión de PMTV. En este sentido, Ward et al. (2005), evaluando el efecto de dos ribotipos de Polymyxa graminis, un plasmodiofórido que afecta gramíneas en la zona templada, encontraron que los aislamientos del Tipo II fueron detectados principalmente en plantas de trigo y estaban asociados a la transmisión del Soil-borne cereal mosaic virus (SBCMV), mientras que aquellos del Tipo I afectaban cebada y su capacidad de transmisión de SBCMV era muy baja.
Los niveles de variación encontrados en esta investigación para el agente causal de la sarna de la papa en Colombia, condujeron a plantear un método de diagnóstico que permita identificar los principales genotipos del patógeno en el país y de esta forma apoyar los programas de mejoramiento genético, vigilancia cuarentenaria y de certificación de tubérculo-semilla. Para esto se realizó un análisis in silico de RFLPs de regiones ITS del ADNr con siete enzimas de restricción de uso frecuente en laboratorios de detección molecular de microorganismos. Los resultados demostraron polimorfismos en los sitios de corte de algunas de las enzimas utilizadas, dos de las cuales permiten distinguir los tres principales grupos definidos por el análisis de agrupamiento. De esta forma se confirmó que la digestión con la enzima Hin6I, diferenció los aislamientos de los Tipos I y II de aquellos del Tipo III, mientras que el corte con la enzima Bsp143I, separó a los miembros de los Tipos I y II. La prueba realizada en este estudio, puede ser implementada con variaciones como el uso de digestiones dobles y la utilización de geles de poliacrilamida que generen mayor resolución de los fragmentos de restricción de bajo peso molecular; sin embargo el esquema aquí presentado ofrece simplicidad y eficiencia en la separación de los tres Tipos, sin la necesidad de contemplar todos los productos de la digestión (>50 pb). Adicionalmente a esta prueba y con base en los resultados de las secuencias obtenidas en esta investigación y en aquella de Gil et al. (2011), nuestro grupo se encuentra diseñando y evaluando pruebas de PCR y RT-PCR en tiempo real para la detección asintomática de Sss y de su virus asociado PMTV.
Se espera que los resultados generados en estos trabajos, sean incorporados rápidamente en los programas de manejo integrado de la sarna polvosa en Colombia y otros países latinoamericanos afectados por esta enfermedad como Venezuela (Rodríguez et al., 2009) y Costa Rica (Montero-Astúa et al., 2008).
CONCLUSIONES
En este trabajo se encontró a partir de análisis de secuencias de las regiones ITS del ADNr, la presencia en Colombia de tres variantes principales (Tipos I, II y III) de S. subterranea f. sp. subterranea, agente causal de la sarna polvosa de la papa. Dichas variantes pueden ser identificadas a partir de una prueba simple de PCR-RFLPs utilizando cebadores específicos Spo8-9 y restricciones con las enzimas Hin6I y Bsp143I.
AGRADECIMIENTO
Esta investigación se realizó gracias al apoyo económico y técnico del Ministerio de Agricultura y Desarrollo Rural (proyecto 090-2007S4527-87-08), de la Universidad Nacional de Colombia, sede Medellín, el Politécnico Colombiano Jaime Isaza Cadavid, Fedepapa y Fritolay. Se agradece a la Prof. Luz Estela Lagos, de la Universidad de Nariño por su apoyo con la colección de muestras en el Departamento de Nariño.
Literatura Citada
1. Bell, K.S., J. Roberts, S. Verrall, D.W. Cullen, N.A. Williams, J.G. Harrison, I.K. Toth, D. Cooke, J.M. Duncan y J.R. Claxton. 1999. Detection and quantification of Spongospora subterranea f.sp. subterranea in soils and on tubers using specific PCR primers. European Journal of Plant Pathology 105: 905-915. [ Links ]
2. Bulman, S.R. y J.W. Marshall. 1998. Detection of Spongospora subterranea in potato tuber lesions using the polymerase chain reaction (PCR). Plant Pathology 47:759-766. [ Links ]
3. Bouchek-Mechiche, K., F. Montfort y U. Merz. 2011. Evaluation of the Sss AgriStrip rapid diagnostic test for the detection of Spongospora subterranea on potato tubers. European Journal of Plant Pathology 131: 277-287. [ Links ]
4. Carreño, A.J. 2009. Evaluación de la variabilidad genética de Spongospora subterranea f.sp. subterranea mediante la comparación de regiones ITS del ADN ribosomal de cepas procedentes de las regiones productoras de papa en Colombia. Tesis de Maestría en Ciencias Agrarias. Facultad de Agronomía. Universidad Nacional de Colombia, sede Bogotá. Colombia. 104 p. [ Links ]
5. Falloon, R.E., D. Curtin, R.A. Lister y R.C. Butler. 2005. Root function and growth of potato plants reduced by Spongospora subterranea infection. American Journal of Potato Research 82: 68. [ Links ]
6. Gil, J.F., P.A. Gutiérrez, J.M. Cotes, E.P. González y M. Marín. 2011. Caracterización genotípica de aislamientos colombianos del Potato mop-top virus (PMTV, Pomovirus). Actualidades Biológicas 33: 69-84. [ Links ]
7. Gilchrist, R.E. 2009. Alternativas para el manejo integrado de la sarna polvosa de la papa. (Spongospora subterranea f.sp. subterranea). Tesis de Doctorado en Ciencias Agrarias. Facultad de Ciencias Agropecuarias. Universidad Nacional de Colombia, sede Medellín. [ Links ]
8. Harrison, J.G., E.A. Rees, H. Barker y R. Lowe. 1993. Detection of spore balls of Spongospora subterranea on potato tubers by enzyme-linked immunosorbent assay. Plant Pathology 42: 181-186. [ Links ]
9. Harrison, J.G., R.J. Searle y N.A. Williams. 1997. Powdery scab disease of potato - A review. Plant Pathology 46:1-25. [ Links ]
10. Hincapié, L.A. 2006. Evaluación de la variabilidad molecular de Spongospora subterranea (Wallroth) Lagerheim f. sp. subterranea en las principales zonas paperas de Colombia. Tesis de Maestría en Biotecnología. Facultad de Ciencias. Universidad Nacional de Colombia, sede Medellín. Colombia. 65 p. [ Links ]
11. Jaramillo, S. y J.M. Botero. 2007. Respuesta de diferentes poblaciones de Spongospora subterranea f. sp. subterranea a la rotación entre dos variedades de papa (Solanum tuberosum ssp. andigena). Revista Facultad Nacional de Agronomía Medellín 60:3859-3876. [ Links ]
12. Merz, U. y R.E. Fallon. 2009. Powdery scab of potato-increased knowledge of pathogen biology and disease epidemiology for effective disease management. Potato Research 52:17-37. [ Links ]
13. Montero-Astúa, M., V. Vasquéz, W.W. Turechek, U. Merz y C. Rivera. 2008. Incidence, distribution, and association of Spongospora subterranea and Potato mop-top virus in Costa Rica. Plant Disease 92:1171-1176. [ Links ]
14. Qu, X.S. y B.J. Christ. 2004. Genetic variation and phylogeny of Spongospora subterranea f. sp. subterranea based on ribosomal DNA sequence analysis. American Journal of Potato Research 81:385-394. [ Links ]
15. Qu, X.S., J.A. Kavanagh, D. Egan y B.J. Christ. 2006. Detection and quantification of Spongospora subterranea f. sp. subterranea by PCR in host tissue and naturally infested soils. American Journal of Potato Research 83:21-30. [ Links ]
16. Qu, X.S., L.A. Wanner y B.J. Christ. 2011. Multiplex real-time PCR (TaqMan) assay for the simultaneous detection and discrimination of potato powdery and common scab diseases and pathogens. Journal of Applied Microbiology 110: 769-777. [ Links ]
17. Rodríguez, D., M. Ojeda, M. Pérez de Camacaro, M. Gallardo, R. Valera y F. Bittara. 2009. Producción, incidencia de la sarna polvorienta y calidad de clones avanzados de papa. Revista de la Facultad de Agronomía Universidad del Zulia 26: 508-531. [ Links ]
18. Salazar, L.F. 2006. Emerging and Re-emerging Potato Diseases in the Andes. Popato Research 49:43-47. [ Links ]
19. Vélez, P.B. 2007. Detección e identificación del Potato Mop - top virus (PMTV) en áreas de producción de papa donde se encuentra Spongospora subterranea en dos departamentos de Colombia. Tesis de Maestría en Ciencias Agrarias. Facultad de Agronomía, Universidad Nacional de Colombia Sede Bogotá. 115 p. [ Links ]
20. Walsh, J.A., U. Merz y J.G. Harrison. 1996. Serological detection of spore balls of Spongospora subterranea and quantification in soil. Plant Pathology 45: 884-895. [ Links ]
21. Ward, E., K. Kanyuka, J. Motteram, D. Kornyukhin y M.J. Adams. 2005. The use of conventional and quantitative real-time PCR assays for Polymyxa graminis to examine host plant resistance, inoculum levels and intraspecific variation. New Phytologist 165:875-885. [ Links ]
22. Zolan, M.E. y P.J. Pukkila. 1986. Inheritance of DNA methylation in Coprinus cinereus. Molecular and Cellular Biology 6: 195-200. [ Links ]