Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Bioagro
versión impresa ISSN 1316-3361
Bioagro vol.25 no.3 Barquisimeto dic. 2013
Caracterización molecular de doce aislamientos de Trichoderma spp. mediante rapd y rADN-ITS
Alexander Hernández1, María Jiménez1, Asdrúbal Arcia2, Dilcia Ulacio1 y Naileth Méndez1
1 Posgrado de Agronomía, Decanato de Agronomía, Universidad Centroccidental Lisandro Alvarado. Apdo. 400. Barquisimeto. Venezuela. e-mail: hernandez@ucla.edu.ve; mjimeneztamayo@gmail.com; dilciau@ucla.edu.ve, nailethmendez@ucla.edu.ve
2 Instituto de Agronomía, Universidad Central de Venezuela. Maracay. Venezuela. e-mail: aarcia.asdrubal@gmail.com
RESUMEN
La variabilidad genética entre aislamientos del biocontrolador Trichoderma asociados con los diferentes cultivos es un tema de importancia. Esta investigación estuvo dirigida a caracterizar, mediante marcadores RAPD y rADN-ITS, diez aislamientos de Trichoderma spp. y dos cepas comerciales asociados al cultivo del ajo. Los aislamientos fueron obtenidos de esclerocios, rizósfera, suelo y bulbos afectados por la pudrición blanca (Sclerotium cepivorum). Se analizó la variabilidad genética mediante técnicas de clasificación y ordenación a partir de los coeficientes de distancia (1-S de Jaccard). Los análisis realizados con RAPD coincidieron con los realizados con los marcadores ADNr-ITS. Se encontró que los aislamientos que pertenecen a las especies T. harzianum, T. koningiopsis, T. atroviride, T. pseudokoninngii y T. longibrachiatum se agruparon en relación con la diversidad genética sin una relación clara en cuanto al origen geográfico. El rango de distancia genética entre los distintos aislamientos estuvo entre 0,11 y 0,87 en el caso de RAPD, y 0,08 y 0,62 al utilizar los rADN-ITS. Fueron identificados los aislamientos Ar-01, Ar-02 y Ta-01 que por su clasificación y ordenación junto a las cepas comerciales pudieran ser buenos controladores e inductores de resistencia en plantas de ajo afectadas por la pudrición blanca.
Palabras clave adicionales: T. harzianum, T. koningiopsis, T. atroviride. T. pseudokoningii, T. longibrachiatum.
Molecular characterization of 12 isolates of Trichoderma spp. using RAPD and rDNA-ITS
ABSTRACT
Genetic variability among isolates of the bio-controller Trichoderma associated with different crops is an important topic of study. The purpose of this research was to characterize ten isolates of Trichoderma spp. and two commercial strains associated to garlic crop, using RAPD and rDNA-ITS. Isolates were obtained from sclerotia, rhizosphere, soil and bulbs affected by white rot (Sclerotium cepivorum). Genetic variability was analyzed by classification and management techniques from the Jaccard distance ratios. Results from RAPD analyses were similar to those conducted by rDNA-ITS markers. It was found that the isolates belonging to the species T. harzianum, T. koningiopsis, T. atroviride, T. pseudokoninngii and T. longibrachiatum were grouped in relation to genetic diversity without a clear relationship in terms of geographical origin. The range of genetic distance among the different isolates was 0.11-0.87 for RAPD, and 0.08-0.62 for rDNA-ITS. The isolates Ar-01, Ar-02 and Ta-01 were classified along with the commercial strains and could be good controllers and inducers of resistance in garlic plants affected by white rot.
Additional key words. T. harzianum, T. koningiopsis, T. atroviride, T. pseudokoningii, T. longibrachiatum
Recibido: Febrero 20, 2013 Aceptado: Septiembre 27, 2013
INTRODUCCIÓN
Debido a la importancia económica que posee el biocontrolador Trichoderma y a las múltiples aplicaciones biotecnológicas, resulta fundamental conocer su diversidad en Venezuela (Pavone, 2012). En el caso del cultivo del ajo, no se conocen estudios en los que se realice una determinación de la biodiversidad de las especies de Trichoderma asociadas con este cultivo.
El biocontrolador ha logrado altas reducciones de la pudrición blanca del ajo causada por el hongo Sclerotium cepivorum ya que compite y parasita tanto el micelio como los esclerocios del patógeno (Coventry et al., 2006; Jiménez et al., 2012).
La actividad de Trichoderma ha sido atribuida a un efecto antimicrobiano directo sobre los patógenos, especialmente mediante el micoparasitismo y la antibiosis, por competencia por espacio y nutrientes, aunque también puede establecer asociaciones con las raíces de las plantas (Harman et al., 2004). Dichas asociaciones hongo-raíz, estimulan las respuestas de defensa de las especies vegetales, mecanismo de acción, que está relacionado, con su capacidad para sintetizar e inducir enzimas. Esto último, proporciona un nuevo instrumento para entender la respuesta de dichas plantas y puede ser usado para producir organismos mucho más eficaces en el biocontrol. No obstante, estos estudios deben ser precedidos por una identificación precisa de los aislamientos.
La variabilidad genética entre aislamientos de Trichoderma asociados con plantas de ajo afectadas por S. cepivorum se desconoce; no obstante, se dispone de herramientas moleculares apropiadas, como son los marcadores moleculares RAPD y las secuencias espaciadoras transcritas internas 1 y 2 (ITS1 e ITS2) del ADN ribosomal (Pavone, 2012) que pueden ser usados para tal fin.
El objetivo de la presente investigación fue identificar y caracterizar mediante técnicas moleculares aislamientos de Trichoderma spp. y relacionarlos con cepas comerciales por su capacidad antagónica a S. cepivorum, inducción de defensa y origen geográfico, para seleccionar los más eficientes, capaces de promover múltiples efectos beneficiosos sobre la planta.
MATERIALES Y MÉTODOS
Se seleccionaron diez aislamientos de Trichoderma spp. obtenidos a partir de rizósfera, suelo, esclerocios de S. cepivorum y bulbos de ajo provenientes de huertos afectados por la pudrición blanca. Para ello se empleó el método de trampeo propuesto por Hurtado et al. (2007). Los esclerocios fueron desinfestados con hipoclorito de sodio al 10% secados y ubicados directamente sobre medio agar-agua. Los bulbos dientes se secaron y se ubicaron directamente sobre el mismo medio. Adicionalmente se emplearon como testigos comerciales Tricobiol (Agrobica) y Subiol (Insubiol) ambos identificados como T. harzianum. La procedencia, fuente e identificación de los aislamientos colectados en diferentes localidades productoras de ajo de los estados Lara, Trujillo, Aragua y Táchira se muestran en el Cuadro 1.
Cuadro 1. Procedencia e identificación de los aislamientos de Trichoderma spp. colectados en diferentes localidades productoras de ajo
| Aislamiento | Fuente | Localidad-Región | Identificación |
| Tr-01 | Rizósfera | Carache-Trujillo | T. harzianum |
| Tr-02 | Rizósfera | Carache-Trujillo | T. harzianum |
| Tr-03 | Esclerocios S. cepivorum | Carache-Trujillo | T. longibrachiatum |
| La-01 | Rizósfera | Agua Negra-Lara | T. pseudokoninngii |
| La-04 | Bulbos de ajo | Agua Negra-Lara | T. atroviride |
| La-06 | Esclerocios S. cepivorum | Agua Negra-Lara | T. koningiopsis |
| La-07 | Suelo | Agua Negra-Lara | T. koningiopsis |
| Ar-01 | Suelo | Colonia Tovar – Aragua | T. harzianum |
| Ar-02 | Suelo | Cagua – Aragua | T. harzianum |
| Ta-01 | Suelo | Táchira | T. harzianum |
| Ar-Th | Testigo comercial (Trichobiol) | - | T. harzianum |
| Po-Th | Testigo comercial (Subiol) | - | T. harzianum |
La identificación de las especies colectadas de Trichoderma fue realizada según morfología distintiva, de acuerdo con la Subcomisión Internacional para la Taxonomía del género Trichoderma (Samuels et al., 2011).
La extracción de ADN se realizó macerando 0,4 g de micelio seco en un tubo con 200 μL de buffer de extracción (CTAB 2 %) y luego se completó hasta 600 μL, se agregó 10 μL de mercaptoetanol agitando por inversión e incubando a 65 ºC por 30 minutos. La separación y limpieza se realizó con 800 μL cloroformo-isoamil-alcohol (24:1), se centrifugó a 12.000 rpm por 10 minutos y se transfirió el sobrenadante a otro tubo. La precipitación se realizó con 70 μL de NaCl 5M y 800 μL de isopropanol frío, incubando en hielo por 30 minutos. Se centrifugó en iguales condiciones y se descartó el sobrenadante. Por último, se secó el pellet por inversión y se resuspendió en 60 μL de buffer TE pH 8. La calidad del ADN se verificó mediante electroforesis en gel de agarosa al 1 % y se ajustó la concentración a 10 ng·µL-1 de ADN de cada muestra para su posterior utilización.
El análisis para la caracterización molecular se realizó en el laboratorio de Biología Molecular del Posgrado de Agronomía de la Universidad Centroccidental Lisandro Alvarado (UCLA).
En la reacción de amplificación tipo RAPD se observó una alta reproducibilidad para los iniciadores OPA01, OPA04, OPB01, OPC01, OPD06 y OPE01; sin embargo, los de mejor resolución fueron los iniciadores OPA01, OPB01, OPC01 y OPE01. Las reacciones de PCR fueron realizadas con 0,2 μM de dNTPs, 0,2 μM de cada cebador, Taq pol 2,5 U y 20 ng de ADN en un volumen final de 15 mL. La PCR fue ejecutada en la siguiente forma: 93 ºC desnaturalización por 60 s, seguida de 45 ciclos de amplificación con 92 ºC por 60 s, 36 ºC por 60 s, y 72 ºC por 60 s. La extensión final fue de 5 min a 72 ºC. La visualización de los segmentos amplificados se realizó por electroforesis en geles de agarosa 2 % y tinción con bromuro de etidio al 1 %.
La amplificación del fragmento 1 (ITS1) se realizó en presencia de los cebadores Its1 e Its2. Para el fragmento 2, formado por la región ITS1-5,8s-ITS2, se utilizaron los cebadores Its1 e Its4, siguiendo la metodología de White et al. (1990). Las reacciones se hicieron con 20 ng de ADN, buffer10X, 10mM de dNTP´s, 25mM de Mg+2, 1,5 mM de cada iniciador y 2,5 U Taq pol, para un volumen final de 30µL. La amplificación fue ejecutada con un primer ciclo de 95 ºC por 40 seg., seguida de 30 ciclos con 92 ºC por 40 seg., 59 ºC por 40 seg. y 72 ºC por 40 seg. La extensión final fue de 10 min a 72 ºC y se conservó a 4ºC.
Los productos de la amplificación de cada aislamiento fueron digeridos con las enzimas Hinf I, HaeIII, HpaII y MspI; en donde 4 µL del producto de PCR fue mezclado con 1,5 µL de tampón, 8,5 µL de agua destilada y 1 µL de cada enzima; se dejó incubar a 37 ºC por 2 horas. Posteriormente, la visualización del producto de la digestión se realizó mediante electroforesis en geles de poliacrilamida al 10 % y tinción con nitrato de plata al 0,2 %.
Para el análisis de los datos se registraron los productos de amplificación o digestión polimórficos, según el caso, en matrices binarias de datos, asignando (0) para ausencia y (1) para presencia de bandas. Se construyeron matrices de distancia (1-S de Jaccard) y con éstas, los análisis de agrupamiento UPGMA con su respectivo dendrograma. Así mismo, se aplicó el análisis de coordenadas principales (ACP) y el de procrustes generalizado (APG). Ambos tipos de análisis permitieron visualizar las relaciones genéticas entre los aislamientos evaluados. Todos los análisis fueron realizados usando el programa InfoGen (Balzarini et al., 2012).
RESULTADOS Y DISCUSIÓN
Los 4 iniciadores RAPD generaron 80 bandas amplificables y reproducibles (Cuadro 2). Los iniciadores OPC-01 y OPE-01 generaron el mayor número de bandas polimórficas, 23 y 26, respectivamente. El contenido de información polimórfica (PIC) de los iniciadores fue similar con valores entre 0,26 y 0,28.
Cuadro 2: Números y tipos de alelos detectados por los iniciadores de RAPD utilizados para la caracterización molecular de los 12 aislamientos de Trichoderma ssp.
| Iniciador | BP | BM | BT | PIC | SD |
| OPA01 | 15 | 0 | 15 | 0,28 | 0,02 |
| OPB01 | 16 | 0 | 16 | 0,26 | 0,02 |
| OPC01 | 23 | 0 | 23 | 0,28 | 0,02 |
| OPE01 | 26 | 0 | 26 | 0,26 | 0,02 |
| Total | 80 | 0 | 80 |
|
|
BP= Bandas polimórficas, BM= Bandas monomórficas, BT= Bandas totales, PIC= Contenido de información polimórfica y SD= Desviación estándar.
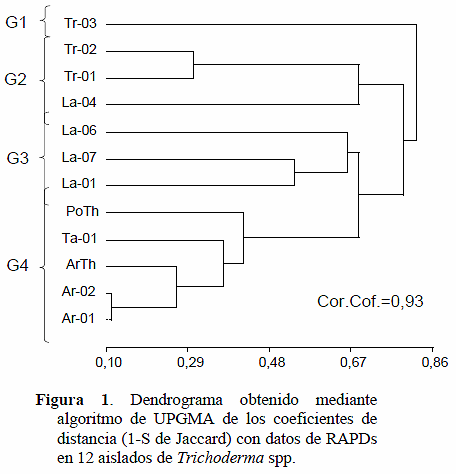
El análisis de agrupamiento con los datos de RAPD mostró variabilidad inter e intraespecífica entre los aislamientos examinados (Figura 1), formándose cuatro grupos. El primer grupo, incluye al aislamiento Tr-03 de la especie T. longibrachiatum, el cual se ubicó separadamente del resto de los aislamientos a una distancia de 0,83 indicando que sólo comparte un 17% del reservorio genético. Un segundo grupo incluyó a dos aislados de Trujillo (Tr-02 y Tr-01), identificados como T. harzianum y otro de Lara (La-04) de la especie T. atrovide. Este grupo estuvo ubicado 0,70 de distancia, compartiendo un 30 % de las características con el resto de la población. El tercer grupo conglomeró a las especies T. koningiopsis y T. pseudokonigii, todas provenientes de Lara. Este grupo se separó 0,65 del resto de los aislamientos. El cuarto grupo estuvo formado por aislamientos de T. harzianum de distintos orígenes geográficos. Es el grupo menos disperso que muestra un rango de distancia de 0,11 a 0,44 y agrupa a las cepas comerciales con dos cepas nativas de Aragua y otra de Táchira. Las cepas Ar-01y Ar-02 son las más similares y tienen en común un 89 % de los alelos explorados con los marcadores utilizados.
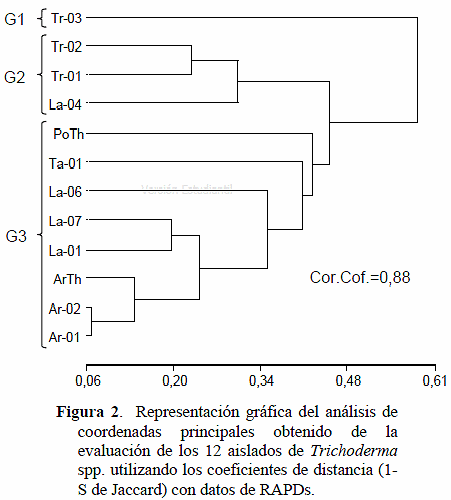
El análisis de coordenadas (Figura 2), permitió identificar dos ejes principales que explican el 53,1 % de la variación total. Ubicó en el espacio a las distintas especies de Trichoderma, permitiendo observar la diversidad genética intra e interespecífica. Este análisis se corresponde con el de clasificación mostrada en el dendrograma de la Figura 1. Los aislados de T. harzianum Ta-01, Ar-01 y Ar-02 se ubican con los dos biocontroladores comerciales en el cuadrante negativo del CP1 y son el grupo menos disperso. La especie T. longibrachiatum se ubicó alejada en el cuadrante positivo del CP1; mientras que T. atroviride se ubica en el cuadrante negativo del mismo eje, relacionándose con dos aislados de T. harzianum provenientes del estado Táchira. Al centro de los cuadrantes se ubicaron T. pseudokoninngii y T. koningiopsis, especies relacionadas entre sí e intermedias en distancia con el resto de las analizadas.
Al comparar la caracterización molecular de los aislamientos relacionados con S. cepivorum junto con los testigos comerciales utilizados como biocontroladores e inductores de resistencia a la pudrición blanca, se podría esperar que los aislamientos Ta-01, Ar-01 y Ar-02 tengan un comportamiento similar a los comerciales, por la poca distancia genética que los separa. En general, al utilizar los marcadores RAPD se encontró una relación entre el agrupamiento de los aislamientos con la procedencia de cada uno de ellos, y se demostró la variabilidad existente entre los mismos, observándose que es una técnica confiable para la caracterización de aislamientos de Trichoderma spp. Estos resultados concuerdan con los señalados por Latha y Mukherjee (2002), quienes señalaron que los marcadores RAPD revelaron gran variabilidad inter e intraespecífica entre las cepas de Trichoderma examinadas. De igual manera, Góes et al. (2002) señalaron que el uso de RAPD permitió demostrar variabilidad entre los aislamientos de Trichoderma.
El producto de amplificación del fragmento 1 (ITS1) fue de ~ 400 pb y el fragmento 2 (ITS1-5,8s-ITS2) ~ 500 pb. Los patrones de bandas de la restricción de estos productos generados por las enzimas HinfI, HaeIII, HpaII y MspI reflejaron polimorfismo para los aislamientos de Trichoderma spp, que permitieron el análisis de la diversidad genética.
Las digestiones de los productos ITS generaron un total de 84 bandas (Cuadro 3), de las cuales 8 fueron monomórficas. El fragmento 2 fue más variable y originó 55 bandas. La enzima HaeIII originó mayor cantidad de bandas, 11 en el fragmento 1 y 23 en el fragmento 2, para un total de 34. Por el contrario la HpaII apenas originó 15 bandas, 4 en el fragmento 1 y 11 en el 2.
Cuadro 3. Números y tipos de alelos detectados por los marcadores de rADN-ITS utilizados para la caracterización molecular de los 12 aislamientos de Trichoderma spp.
| Marcador | BP | BM | BT | PIC | D.E. |
| F1HinfI | 6 | 1 | 7 | 0,14 | 0,01 |
| F1HaeIII | 10 | 1 | 11 | 0,33 | 0,02 |
| F1HpaII | 4 | 0 | 4 | 0,26 | 0,02 |
| F1MpsI | 7 | 0 | 7 | 0,27 | 0,02 |
| Fragmento1 | 27 | 2 | 29 |
|
|
| F2Hinfl | 11 | 1 | 12 | 0,24 | 0,02 |
| F2HaeIII | 19 | 4 | 23 | 0,25 | 0,02 |
| F2HpaII | 11 | 0 | 11 | 0,23 | 0,01 |
| F2MspI | 8 | 1 | 9 | 0,28 | 0,01 |
| Fragmento 2 | 49 | 6 | 55 |
|
|
| Total | 76 | 8 | 84 |
|
|
BP= Bandas polimórficas, BM= Bandas monomórficas, BT= Bandas totales, PIC= Contenido de información polimórfica y D.E.= Desviación estándar
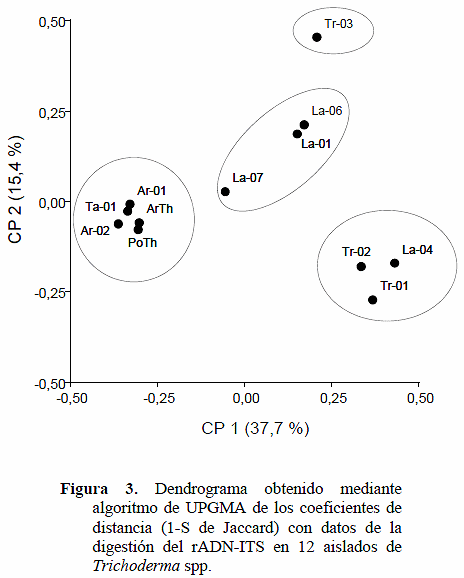
En la Figura 3 se aprecia el dendrograma que muestra la variabilidad genética intra e interespecífica entre los aislamientos de Trichoderma ssp. con datos de la digestión del rADN-ITS. Se pone de manifiesto la variación observada entre los aislamientos que van desde distancias de 0,11 a 0,62. En general esta clasificación coincidió con la realizada con los datos de RAPD al agrupar por especie y procedencia geográfica. Se originaron tres grupos principales claramente definidos. El primero incluyó un aislamiento de T. longibrachiatum proveniente del estado Trujillo (Tr-03) muy distante del resto de las especies (0,62) con las cuales comparte por lo menos un 38% del reservorio de alelos detectados con los marcadores rADN-ITS. El segundo grupo estuvo conformado por dos aislados de T. harzianum, provenientes del estado Táchira (Tr-02 y Tr-01) y uno de T. atroviride, (La-04), ubicado a 0,31 de distancia. El último grupo incluyó a los biocontroladores comerciales y tres aislamientos nativos (Ar-01, Ar-02 y Ta-01) de la especie T. harzianum. También están en este grupo las especies T. koningiopsis y T. pseudokoninguii, provenientes del estado Lara. Es el grupo más variable con aislados que están 0,08 a 0,40 de distancia. Basado en la poca distancia genética de los aislamientos de este grupo con los comerciales y porque comparten una alta proporción del reservorio de genes, se pudiera sugerir que también sean buenos biocontroladores.
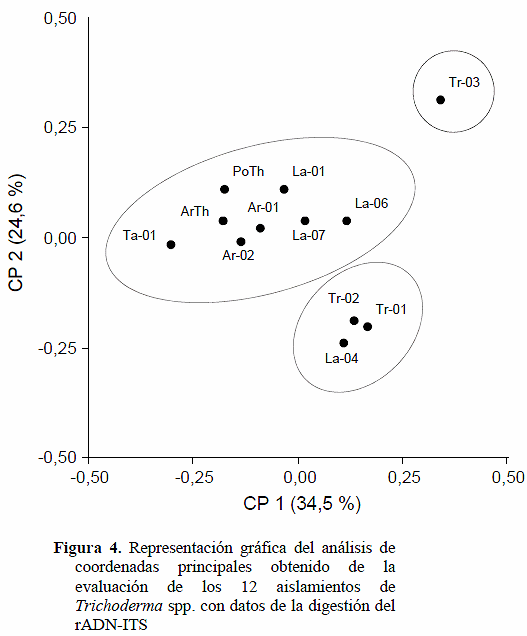
La Figura 4 corresponde a la representación gráfica del análisis de coordenadas principales de los datos de rADN-ITS, el cual explica el 59,1% de la variación. Se puede apreciar que este análisis de ordenación coincide con la clasificación presentada para este mismo marcador en el dendrograma.
La combinación de las dos coordenadas formadas permite ubicar espacialmente los grupos de aislamientos para evaluar el grado de dispersión y distanciamiento entre ellos según su afinidad genética detectada con este tipo de marcador.
El componente CP1 ubica en el cuadrante negativo a la mayoría de los aislados de T. harzianum, incluyendo los biocontroladores comerciales, junto con T. pseudokoninguii. En el cuadrante positivo a las especies T. atroviride, T. longibrachiatum y T. koningiopsis. También están ubicados en este cuadrante positivo de la CP1 dos aislados de Trujillo que fueron identificadas como T. harzianum (Tr-01 y Tr-02). El componente CP2, que sólo explica el 24,6 % de la variación, separa a la especie T. atroviride junto con los aislados Tr-01 y Tr-02 de T. harzianum del resto de los aislamientos que se ubican en el cuadrante positivo de este componente.
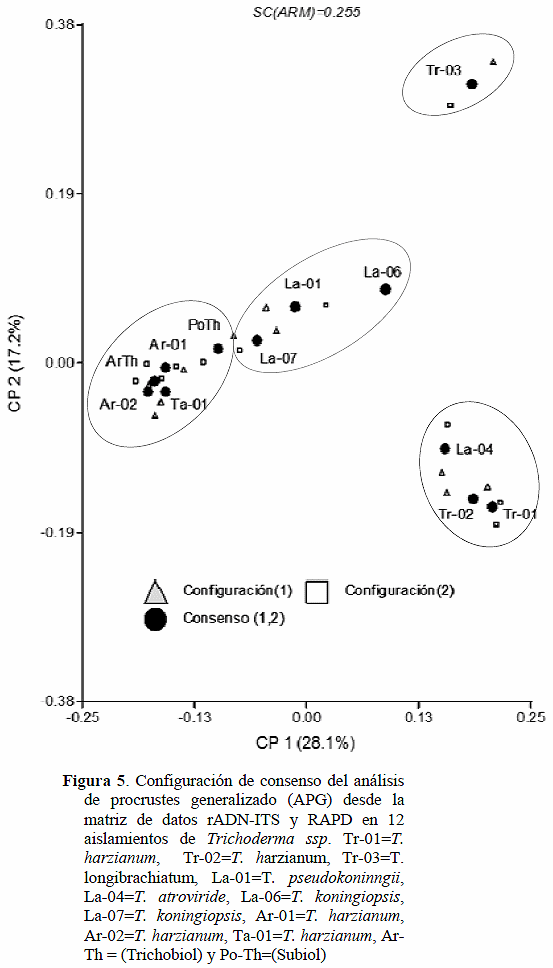
En el Cuadro 4 se muestran los resultados del análisis de procrustes generalizado (APG) y se confirma la posición de los aislamientos dentro de los grupos en los análisis de ordenación por rADN-ITS y RAPD. Se observan valores de proporción en relación al consenso por encima del 0,90. La suma de cuadrados por grupos de datos fue similar y al relacionarlos con el consenso se observan valores de proporción de 0,96, indicando que hay una discrepancia entre los dos análisis de 4 %, confirmando que estas técnicas son válidas para la caracterización molecular de aislamientos de Trichoderma.
Cuadro 4. Análisis de varianza del Procrustes Generalizado (APG) desde la matriz de datos rADN-ITS y RAPD en 12 aislamientos de Trichoderma ssp
| Sumas de cuadrados por caso | ||||
| Aislamiento | Consenso | Residuo | Total | Proporción de consenso |
| Tr-01 | 0,191 | 0,002 | 0,193 | 0,987 |
| Tr-02 | 0,174 | 0,005 | 0,179 | 0,974 |
| Tr-03 | 0,308 | 0,003 | 0,311 | 0,989 |
| La-01 | 0,156 | 0,012 | 0,168 | 0,929 |
| La-04 | 0,174 | 0,005 | 0,178 | 0,975 |
| La-06 | 0,181 | 0,004 | 0,185 | 0,979 |
| La-07 | 0,138 | 0,007 | 0,145 | 0,952 |
| Ar-01 | 0,090 | 0,008 | 0,099 | 0,915 |
| Ar-02 | 0,103 | 0,004 | 0,107 | 0,960 |
| ArTh | 0,115 | 0,001 | 0,117 | 0,988 |
| PoTh | 0,147 | 0,010 | 0,157 | 0,935 |
| Ta-01 | 0,153 | 0,007 | 0,161 | 0,955 |
| Total | 1,931 | 0,069 | 2,000 | 0,965 |
| Sumas de cuadrados por grupo | ||||
| ADNr-ITS | 0,960 | 0,034 | 1,000 | 0,960 |
| RAPD | 0,965 | 0,035 | 1,000 | 0,965 |
| Total | 1,931 | 0,069 | 2,000 | 0,965 |
En la Figura 5 se muestra la configuración de consenso del APG desde la matriz de datos rADN-ITS y RAPD. Este análisis explica en sus dos primeros componentes más del 45 % de la variación observada y confirma la ubicación de los aislamientos en los grupos ya analizados.
Los resultados indican que las especies de Trichoderma asociadas con esclerocios de S. cepivorum, rizósfera, suelo y bulbos de plantas de ajo afectadas por la pudrición blanca, son hongos antagonistas que se encuentran de forma natural en la microbiota de áreas cultivadas y se confirma el hecho de que los aislados de Trichoderma spp. tienen una amplia diversidad genética dentro y entre las regiones productoras de ajo del país.
La información permitió la caracterización molecular de los aislamientos relacionados con S. cepivorum y su posible uso como inductores de resistencia a la pudrición blanca. Se encontró una relación entre el agrupamiento de los aislamientos, según los datos moleculares, con las especies a la cual pertenecen cada uno de ellos, y se demostró la variabilidad existente entre los mismos, observándose que el uso de los marcadores RAPDs constituye una técnica confiable para la identificación de Trichoderma spp.
El análisis del patrón de bandas de los fragmentos rADN-ITS también mostró ser eficaz en la resolución del perfil taxonómico del género. Ambos marcadores revelaron gran variabilidad inter e intraespecífica entre las cepas de Trichoderma examinadas. No se observó una clara relación entre el polimorfismo y la procedencia geográfica. En el presente estudio no se comparó la variabilidad genética entre los aislamientos y la fuente de inóculo, pero se observó en el caso de RAPD que los del grupo 4 fueron aislados todos de suelo, y pertenecen a T. harzianum, mientras que los de los otros grupos fueron aislados a partir de rizósfera, bulbo y esclerocios.
La ubicación taxonómica de las cepas en estudio es importante ya que representa el punto de partida que permitirá un mejor diseño de experimentos y evaluar su actividad biológica bajo diferentes condiciones de crecimiento, determinar los mecanismos de acción que desarrollan y la interacción que establecen con la planta. Esta información en conjunto facilitará la identificación de los organismos que pueden seleccionarse como antagonistas patógenos e inductores de resistencia en las plantas.
Individuos pertenecientes a especies como T. atroviride y T. harzianum aparecen relacionados; a pesar que pertenecen a distintas especies fueron aislados de una misma región geográfica y bajo condiciones climáticas similares (La-04, Tr-01 y Tr-02). Dicha discrepancia puede representar un ejemplo, de cómo un aislado, puede desarrollar propiedades de otro grupo, quizás a través de la selección impuesta por el ambiente o condiciones ecológicas particulares (Lieckfeldt y Seifert, 2000).
El hecho de que aislamientos de distintas zonas geográficas como Aragua, Portuguesa, Táchira y Trujillo sean identificados como variantes de T. harzianum pone en evidencia el flujo de genes por la actividad agrícola al incorporar biocontroladores en las aéreas cultivadas, al traslado de bulbos de una zona a otra y la selección por las condiciones ambientales.
Dentro del género Trichoderma spp. existe una gran diversidad intraespecífica (Bissett, 1991) y ésta viene establecida por las diferencias entre las distintas circunstancias donde se desarrollaron los aislados, contrastes entre las áreas geográficas donde se encuentran y las fuentes de inóculo muestreadas, características propias de cada individuo y la forma cómo se reproducen (Kredics et al.,2003, 2006; Siddiquee et al., 2007).
En el presente estudio se evidenció, al integrar los atributos moleculares con los criterios morfológicos que se usaron para la identificación de especies, la existencia de grupos dentro de T. harzianum, que pueden estar relacionados con diferentes niveles de actividad biológica. Además, apoyan la existencia de especies con características morfológicas y moleculares similares.
El análisis molecular de Trichoderma spp, corroboró que los atributos genéticos, pueden ser utilizados para ubicar los aislados en grupos o relacionarlos por áreas geográficas similares pero si lo que se persigue es la selección como inductores de resistencia o agentes de control biológico, deben realizarse pruebas bioquímicas y fisiológicas para determinar si tal diversidad, está relacionada con mecanismos de acción y posteriormente compararlos con los respectivos patrones moleculares.
CONCLUSIONES
La información generada en este estudio permitió la caracterización molecular de los aislamientos de Trichoderma spp. relacionados con S. cepivorum y su posible uso como biocontroladores a la pudrición blanca del ajo. Se encontró relación entre el agrupamiento, según los datos moleculares, con las especies a la cual pertenecen cada uno de ellos, y se demostró la variabilidad genética existente entre los aislamientos. Se comprueba que los marcadores RAPDs y rADN-ITS son técnicas confiables para la caracterización e identificación de Trichoderma ssp.
Agradecimiento
Los autores agradecen al Consejo de Desarrollo Científico, Humanístico y Tecnológico (CDCHT) de la Universidad Centroccidental Lisandro Alvarado por el financiamiento de esta investigación bajo el código 020 AG-2008.
LITERATURA CITADA
1. Balzarini, M., J. Di Rienzo. InfoGen versión 2012. FCA, Universidad Nacional de Córdoba, Argentina. http://www.info-gen.com.ar. (consulta 12/6/2013). [ Links ]
2. Bissett, J. 1991. A revision of the genus Trichoderma. II. Infrageneric classification. Canadian Journal of Botany 60: 2357-2372. [ Links ]
3. Coventry, E., R. Noble, A. Mead, F. Martin., J. Perez. y J. Whipps. 2006. Allium white rot suppression with composts and Trichoderma viride in relation to Sclerotia viability. Phytopathology 96(10): 1009-1020. [ Links ]
4. Góes, L., A. Lima Da Costa, L. López De Carvalho Freire y N. Tinti De Oliveira. 2002. Randomly Amplified Polymorphic DNA of Trichoderma isolates and antagonism against Rhizoctonia solani. Brazilian Archives of Biology and Technology 45(2): 151-160. [ Links ]
5. Harman G., C. Howell, A. Viterbo, I. Chet y M. Lorito. 2004. Trichoderma species-opportunistic, avirulent plant Symbionts. Nature Review Microbiology 2: 43-56. [ Links ]
6. Hurtado, T., L. Bautista y A. Arcia. 2007. Captura de aislamientos de Trichoderma a través de prácticas o técnicas de enriquecimiento. Fitopatol. Venez. 20(2): 56. [ Links ]
7. Jiménez, M., A. Arcia, D. Ulacio y A. Hernández. 2012. Evaluación Trichoderma ssp. y Acibenzolar-S-Metil (Bion) como inductor de resistencia a la pudrición blanca Sclerotium cepivorum Berk. en ajo (Allium sativum) bajo condiciones de campo. J. Selva Andina Res. Soc. 1(1): 14-25. [ Links ]
8. . 2003. Trichoderma. Acta Microbiologica et Immunologica Hungarica 50: -. [ Links ]
2006 Trichoderma longibrachiatum. Acta Microbiologica et Immunologica Hungarica 51: - [ Links ]
10. Latha, J. y P. Mukherjee. 2002. PCR-fingerprinting of some Trichoderma isolates from two Indian type culture collections - a need for re-identification of these economically important fungi. Current Science 83(4): 372-375. [ Links ]
11. Lieckfeldt, E. y K. Seifert. 2000. An evaluation of the use of ITS sequences in the taxonomy of the Hypocreales. Studies in Mycology 45: 35-44. [ Links ]
12. Moreno, B. y R. Acevedo. 2002. Caracterización patogénica y estudio de los grupos de compatibilidad micelial en Sclerotium cepivorum Berk. Revista Iberoamericana de Micología 19: 115-119. [ Links ]
13. Pavone, D. 2012. Biocontrol de Rhizoctonia solani Kühn por Trichoderma spp. Tesis Doctoral en Ciencias Mención Biología Celular. Universidad Central de Venezuela. 192 p. [ Links ]
14. Samuels, G., P. Chaverri, D. Farr y E. McCray. 2011. Trichoderma Online, Systematic Mycology and Microbiology Lab. ARS, USDA. http://www.isth.info/tools/morphkey/index.php(consulta del 12/06/2013). [ Links ]
15. Siddiquee, S., F. Abdullah, T. Guan y E. Rohaza. 2007. Level in Allozyme variations of malaysian isolates of Trichoderma harzianum and its taxonomic implications. Research Journal of Microbiology 2(10): 717-726. [ Links ]
16. White, T., T. Bruns, S. Lee y J. Taylor. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: M. Innis, D. Gelfand, J. Sninsky y J. White (eds.). PCR Protocols: A Guide to Methods and Applications. Academic Press Inc. New York. pp. 315-322. [ Links ]