Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Bioagro
versión impresa ISSN 1316-3361
Bioagro vol.26 no.1 Barquisimeto abr. 2014
Diversidad de linajes y virulencia de una población venezolana del hongo Pyricularia oryzae, causante de la piricularia en arroz
Alex González1, Geraldine Guzmán, Alexander Hernández2, Erika Arnao3 y Juan Pineda2
1 Fundación para la Investigación Agrícola Danac, San Javier, estado Yaracuy. Venezuela. e-mail: alex.gonzalez@danac.org.ve
2 Posgrado de Agronomía Universidad Centroccidental Lisandro Alvarado. Apdo. 400. Barquisimeto. Venezuela.
3 Plant, Soil and Agriculture Systems. Southern Illinois University. Carbondale, Illinois, USA.
RESUMEN
El principal problema fitopatológico de la producción de arroz en Venezuela es ocasionado por el hongo Pyricularia oryzae (= P. grisea), agente causal de la piricularia en este cultivo. Las poblaciones del patógeno presentan una alta variabilidad genética, lo cual reduce la durabilidad de las variedades resistentes y disminuye la eficacia de los fungicidas específicos. Dado el nivel de importancia de esta enfermedad, el objetivo del trabajo fue determinar la diversidad de linajes y la virulencia en una población venezolana del hongo P. oryzae con la finalidad de usar cepas con amplio poder discriminatorio en el mejoramiento para resistencia a piricularia. Para ello, 81 aislados del hongo de la colección (2005 y 2006) y seis representantes de los linajes venezolanos, fueron evaluados con el marcador molecular Pot2 y 45 aislados de la colección 2006 fueron inoculados sobre 26 cultivares de arroz. Utilizando el coeficiente de Dice se lograron agrupar los aislados en 13 linajes de los cuales tres son linajes conocidos y 10 resultaron ser nuevos linajes. No se hallaron aislados para tres linajes previamente reportados. El análisis de varianza arrojó diferentes espectros de virulencia en el comportamiento de las cepas frente a los cultivares. El análisis de Procrustes determinó que las dos metodologías discriminan de igual manera (68 %), dando consenso (>70 %) entre los resultados arrojados por ambas, evidenciándose correspondencia entre haplotipos y patotipos. En la población de P. oryzae evaluada se identificaron nuevos linajes y cepas con amplio espectro de virulencia que pueden ser usados en estudios posteriores para fines de resistencia a piricularia.
Palabras clave adicionales: Pyricularia grisea, Oryza sativa, aislado, marcador molecular.
Lineage diversity and virulence of a Venezuelan population of Pyricularia oryzae, the cause of rice blast
ABSTRACT
The main phytopathological problem of rice production in Venezuela is caused by fungus Pyricularia oryzae (= P. grisea), causal agent of rice blast. The pathogen populations have high variability which reduces the durability of resistant varieties and decreases the efficacy of specific fungicides. Given the level of importance of this disease, the objective of this study was to determine the diversity of lineages and virulence of a Venezuelan population of the fungus P. oryzae in order to use strains with large discriminatory power in the breeding programs for resistance to rice blast. Thus, 81 isolates from the fungus collection (2005 and 2006) and representatives of the six Venezuelan lineages were evaluated using the Pot2 molecular marker; and 45 isolates of the 2006 collection were inoculated on 26 rice cultivars. By calculating the Dice coefficient, isolates were grouped in 13 patterns, from which three are known lineages, and ten came out to be new lineages. No isolates were found for three previously reported lineages. Analysis of variance showed different virulence spectra in the behavior of isolated strains to the cultivars. A Procrustes analysis determined that both methods discriminate similarly (68 %), giving more than 70 % consensus between them, and showed the existence of a relationship between haplotypes and patotypes. We identified new lineages and strains with a broad virulence spectrum that can be used in later studies for purposes of blast resistance in rice.
Additional key words: Pyricularia grisea, Oryza sativa, isolate, molecular marker
Recibido: Junio 25, 2013 Aceptado: Febrero 24, 2014
INTRODUCCIÓN
El arroz es el alimento básico para más de la mitad de la población mundial, y el continente asiático representa el mayor productor y consumidor de la región, la más poblada del mundo. En los últimos años, el arroz también se ha convertido en un importante alimento básico en toda África (FAO, 2013). En Venezuela, el consumo de este cereal forma parte de la dieta de la población, sobre todo en la de bajos recursos, además de ser un importante ingrediente en la elaboración de alimentos concentrados para animales (Benacchio y Avilan, 1991).
Durante todo el ciclo del cultivo, el arroz está expuesto a una serie de factores bióticos y abióticos que pueden afectar en alto grado el rendimiento y calidad del grano. Dentro de las enfermedades más importantes se encuentran aquellas causadas por hongos, las cuales desde el punto de vista de su severidad e incidencia producen disminución en los rendimientos que pueden alcanzar hasta un 70% (Rodríguez y Nass, 1991). El principal problema fitopatológico de la producción del arroz en Venezuela es ocasionado por Pyricularia oryzae (teleomorfo: Magnaporthe oryzae), agente causal de la piricularia o añublo del arroz. La alta variabilidad de P. oryzae tiende a desestabilizar las medidas de control genético y químico; el éxito de los programas de mejoramiento en este cultivo está determinado por los conocimientos sobre la variabilidad de este hongo y los genes de resistencia contra el patógeno presentes en las variedades de arroz (Correa-Victoria et al., 1994).
Para el año 1969 se conocían 256 razas del hongo en cuestión (Ling y Ou, 1969), cifra que se ha incrementado a medida que avanzan los estudios en el área; esta alta variabilidad patogénica ha conllevado a agrupar a estas razas en familias genéticas o linajes según su patrón (Zeigler et al., 1995). Por lo que se han desarrollado estudios sobre la diversidad genética del hongo utilizando la técnica rep-PCR (reacción en cadena de la polimerasa basada en elementos repetitivos) con los iniciadores Pot2 (George et al., 1998). En Venezuela, se realizó la estandarización de esta técnica (Arnao et al., 2003) e igualmente se caracterizó mediante el Pot2 la estructura genética y la diversidad de linajes de P. oryzae en los estados Barinas, Cojedes, Guárico y Portuguesa para finales de la década de 1990 (Zambrano et al., 2006). El conocimiento de la resistencia de variedades mejoradas ante la variabilidad de éste patógeno existente en Venezuela, facilitaría la selección de los progenitores a ser utilizados al momento del cruce para la obtención de las nuevas variedades (Arnao et al., 2008; Lugo et al., 2008).
Dado el nivel de importancia de esta enfermedad en Venezuela, el programa de mejoramiento de arroz de la Fundación Danac (San Javier, estado Yaracuy) propone integrar los análisis de estructura genética, caracterización de la virulencia del hongo y la combinación de múltiples fuentes de resistencia para lograr un comportamiento más estable de los cultivares de arroz que reduzcan el riesgo del rompimiento de la resistencia. La combinación de estos tres aspectos, dentro de la metodología para la obtención de nuevas variedades, podría garantizar la estabilidad de la resistencia lograda en la nueva variedad. El objetivo fue determinar la diversidad de linajes y virulencia de una población venezolana del hongo P. oryzae causante de la piricularia en arroz, colectada en los años 2005 y 2006.
MATERIALES Y MÉTODOS
Se usaron seis aislados de los diferentes linajes venezolanos de P. oryzae (Ven-1, Ven-2, Ven-3, Ven-4, Ven-5, Ven-7) identificados por Zambrano et al. (2006) y 81 aislados pertenecientes a la colección 2005 y 2006 del banco de patógenos de la Fundación Danac, almacenados en papel filtro a -20 ºC.
Por su parte, se utilizaron los siguientes 26 cultivares de arroz: D-Oryza, Fedearroz 50, Fonaiap 1, Fanny, Teptep, Sha tia sao, Aichi Asahi, Nato, K3, Toride, Norin 2, Ou 244, Rico 1, Moroberekan, K59, F80-1, CT13432-107, F145-1, FD03B01, CT13432-219, CT13432-68, F128-1, F98-7, F129-1, CT13432-55, F124-1, entre los cuales se incluyen variedades nacionales, foráneas y diferenciadores (18 con genes de resistencia conocidos). La variedad Fanny (sin genes de resistencia) fue considerada como control susceptible. Las semillas de estas variedades se obtuvieron del banco de germoplasma de la Fundación Danac.
Para promover el crecimiento del hongo, se colocaron trozos de papel filtro con cultivo monoconidial de cada aislado en medio PDA (papa-dextrosa-agar) contenido en microplatos de 24 pozos, en dos réplicas por aislado. Los microplatos se incubaron a 27 °C y 58 % HR promedio, por cinco días. Posteriormente, discos de PDA con micelio fueron colocados en medio líquido de Fries (George et al., 1998) en agitación a 190 rpm, a una temperatura de 25 °C promedio por ocho días en oscuridad. La masa de micelio se filtró eliminando el resto de agar, luego fue transferida a micro tubos 1,5 mL, congelada por tres días y liofilizada (-0,75 bar, -84 ºC). Posteriormente, se realizó la extracción de ADN siguiendo la metodología de Murray y Thompson (1980) modificada para CTAB.
Se realizó una corrida en gel de agarosa 0,8 % para determinar la calidad y la cantidad de ADN extraído. La cuantificación de ADN se realizó a través de un análisis visual con Gel-Doc. Posteriormente, se realizó un ajuste en la concentración de ADN a 30-50 µg·µL-1 adecuándola para la reacción de amplificación del iniciador.
En la prueba de PCR, se utilizó el iniciador para P. oryzae (Pot2), cuya secuenciación de oligonucleótidos es la siguiente: Pot2-1 5CGGAAGCCCTAAAGCTGTTT3 y Pot2-2 5CCCTCATTCGTCACACGTT C3. El volumen final fue de 25 µL por reacción, correspondientes a 2 µL (10 ng) de ADN molde; 2,5 µL de tampón buffer; 1,2 µL de MgCl, 2 µL de dNTPs; 3 µL del iniciador (Pot2, con sus dos secuencias); 0,3 µL (5 Un/µL) TAQ polimerasa de Promega y 14 µL de agua. La amplificación del ADN se efectuó en un termociclador (AB Applied Biosystems, modelo 2720) bajo los siguientes perfiles de temperatura: un ciclo de desnaturalización inicial por 2,5 min a 95 °C; seguido por 30 ciclos de amplificación a 94 °C y 58 °C por 1 min, y 65 °C por 10 min. La extensión final fue de 7 min a 72 °C y se dejó a 4 °C por 18 min. La verificación de los productos PCR, se realizó mediante una corrida en agarosa 1,2 %, permitiendo la separación de las bandas de cada uno de los aislados; esta electroforesis se llevó a cabo en una cámara horizontal. Los aislados que mostraron igualdad de bandas se corrieron en un gel final junto al marcador molecular lambda 500 µg·mL-1 (Promega). La visualización fue registrada con Gel-Doc.
La interpretación de los datos moleculares se realizó a partir de la matriz binaria, mediante un análisis multivariado para calcular el coeficiente de Dice (el cual puede indicar que tan similares son dos grupos entre sí) empleando el software NTSYSpc versión 2.10 j. 2000. Para esto, se construyó un fenograma a partir del algoritmo de UPGMA y se efectuó un análisis de coordenadas principales por los coeficientes de similitud de Dice, lográndose agrupar los aislados, de acuerdo al patrón de bandas, dentro de linajes ya existentes o asignándolos en nuevos linajes o patrones.
Para la siembra de los aislados en el medio de crecimiento, inicialmente se mezcló jugo V8 (jugo de remolacha, apio, tomate, perejil, zanahoria, lechuga, espinaca y berro) con agua destilada (1:1). Este jugo se esterilizó a 121 ºC por 15 min, se dejó enfriar, se introdujeron en él cinco trozos de papel filtro colonizado con los aislados y se agitó a 200 rpm por 24 h a temperatura ambiente. Luego, se preparó el medio agar-salvado de arroz (20 g de agar + 20 g de salvado de arroz + 10 g de dextrosa en 1 L de agua), el cual fue esterilizado a 121 °C por 15 min, se añadieron 250 mg de oxitetraciclina, se dispensó en platos Petri y fue dejado a temperatura ambiente por 24 h. Después, se colocó una gota del jugo V8 con aislado en cada plato Petri y se esparció por todo el medio. En promedio, se sembraron 15 platos por cepa. Finalizado este proceso, se colocaron todas las cepas en bolsas plásticas por separado, se sellaron y se incubaron por 8 días a 27 °C. Posteriormente, se procedió a realizar la poda del hongo, pasando una espátula esterilizada por encima del micelio, con la finalidad de estimular la producción de conidios, colocándose en la cámara de crecimiento con luz continua por un lapso de 8 días a temperatura de 23-27 °C.
Para la preparación del inóculo, por cada aislado se procedió a desprender el micelio con una espátula esterilizada y se diluyó en agua destilada (1 mL por plato). Los conidios fueron separados filtrando con tela tul. Para el contaje de conidios, se colocó una gota de la solución filtrada en una cámara de Neubauer y se ajustó a una concentración de 5x105 conidios por mL y se añadieron dos gotas de Tween 20 a la suspensión total.
La siembra del arroz en invernadero se realizó utilizando sustrato de suelo mineral previamente esterilizado. En un matero plástico de 10 x 8 cm, se sembraron 10 semillas de cada uno de cultivares de arroz para asegurar la germinación de al menos seis semillas. El procedimiento de siembra se realizó el mismo día de la reactivación del hongo en jugo V-8. Ocho días después de la siembra se fertilizó con 5 g·L-1 de fertilizante fórmula completa 15N-15P-15K.
Quince días después de la emergencia de las plántulas se realizó la inoculación con el hongo. Para ello, se asperjó la solución de inóculo a razón de 40 mL sobre las plantas colocadas en cajas plásticas de 40 materos. Luego de la inoculación, se cerraron las cajas para propiciar un microclima óptimo para la infección, y se abrieron 24 horas más tarde.
Para la prueba de virulencia en invernadero se utilizó un diseño completamente al azar, con tres repeticiones, con arreglo factorial de 26 cultivares de arroz por 45 aislados de P. oryzae. La unidad experimental estuvo constituida por seis plantas por matero. Se evaluó el tipo de lesión (TL) mediante la siguiente escala ordinal: 1= punto de aguja; 2= manchas redondeadas menores a 3mm; 3= manchas alargadas sin centro gris; 4= manchas tipo rombo típicas con centro gris o coalescente para formar un tizón. Los resultados fueron sometidos a un análisis de la varianza mediante el programa SAS JMP 7.0. Así mismo, se realizó una prueba de medias de Tukey HSD, para determinar el espectro de virulencia de los aislados o cepas, y de esta manera se seleccionaron los de mayor virulencia y severidad. Con los promedios de la interacción cultivares diferenciales x cepas se realizó el análisis multivariado para calcular el coeficiente de Dice, como se explicó anteriormente.
Finalmente, para determinar la correspondencia entre diversidad genética y la reacción fenotípica de virulencia, se seleccionaron las cepas que contaron con resultados de virulencia y moleculares, formando una matriz completa con las medias de TL (virulencia) y la presencia o ausencia de bandas (molecular), para llevarla a un análisis con el programa InfoGen y construir un Procrustes generalizado.
RESULTADOS
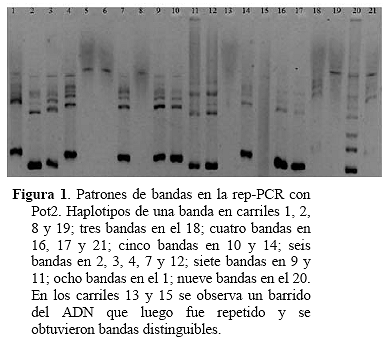
Caracterización molecular. Durante la separación de los fragmentos ampliados vía PCR se observaron 33 bandas polimórficas por el iniciador específico Pot2, de tamaño comprendido entre 564 a 4361 Pb; con un máximo de nueve y un mínimo de una banda por aislado (Figura 1).
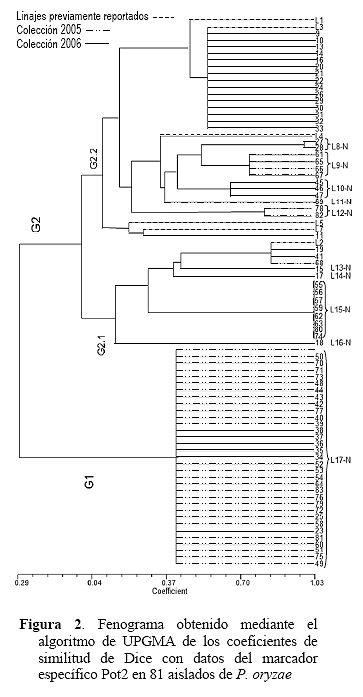
En la Figura 2 se muestra el fenograma generado con los coeficientes de similitud de Dice, que permitió caracterizar los 81 aislados, donde se pudieron observar 17 subgrupos o linajes separados en dos grandes grupos.
El primero (G1) agrupó 33 aislados, con un nuevo linaje, L17 (resaltado en la figura y es considerado un nuevo linaje, NL) con 27 aislados de la colección 2005 y 6 aislados de la colección 2006, con un índice de similitud (IS) de 0,40.
El segundo (G2) presentó dos subdivisiones G2.1 y G2.2, es más variable, con cepas iguales y totalmente diferentes, con IS entre 1 y 0. La subdivisión G2.1, tuvo cinco patrones, tres aislados fueron similares en un 0,84 y se agruparon con el linaje Ven-2, que incluyeron dos aislados 2006 y una de la colección 2005; un solo aislado constituyó el patrón del L16 (NL) con 0,14 de IS; al L15 (NL) con un IS de 1,0 se agruparon ocho aislados de la colección 2005. El L13 (NL) con 0,44 de IS tuvo un solo aislado, L14 (NL) también presentó un solo aislado con IS igual a 0,46. L13 y L14 estuvieron representados por aislados de la colección 2006.
La subdivisión G2.2 estuvo conformada por 10 patrones desde 0,36 hasta 0,99 de similitud; donde se observó un agrupamiento homogéneo conformado por 15 cepas de la colección 2006 similares al linaje Ven-3 con 0,56 de IS; también aparecen dos linajes previamente reportados Ven-1 y Ven-4 que no agruparon con aislados de ninguna colección; el aislado 11 agrupa con Ven-7 con 0,28 de IS; Ven-5 quedó sin grupo; L8 (NL) con dos aislados de la colección 2006, con IS igual a 0,92. El L9 (NL) con cuatro aislados 2005, L10 (NL) con tres aislados 2006 de 0,97, con IS de 0,72 y 0,67, respectivamente. L11 (NL) con un aislado perteneciente a la colección 2005 no agrupó y L12 (NL) con dos aislados de 2005 mostraron un IS de 0,82.
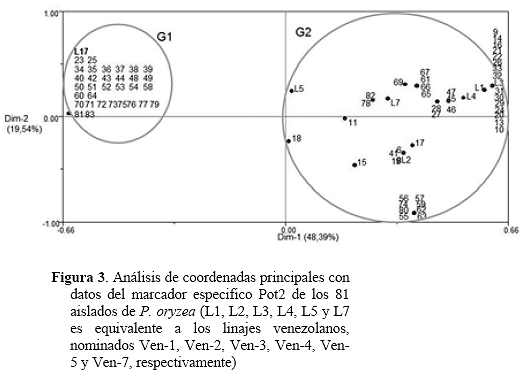
Análisis de coordenadas principales (ACP). Usando el coeficiente de similitud de Dice, el ACP permitió mediante dos dimensiones situar espacialmente los agrupamientos de cepas similares (Fig. 3). Este análisis explica el 67,93 % de la variación total, indicando alta confiabilidad del agrupamiento realizado. La primera coordenada (Dim-1) explica el 48,39 % de la variación. Separó en dos grandes grupos, situados uno en cada cuadrante (G1 y G2); se evidenció lo alejados que se encuentran estos dos grupos indicando que el índice de similitud de Dice fue muy bajo. El comportamiento de los aislados de G1 fue bastante homogéneo, no así el comportamiento del G2 que muestra mayor dispersión de los puntos de la figura.
En la frecuencia de los linajes en la población estudiada, se observó que el linaje predominante fue el L17 con un 43,24 %, seguido del linaje L3 con un 20,27 %. El linaje L2 agrupó el 10,81 %. Los linajes L1, L4, L5 y L7 no agruparon aislado alguno en la población. El linaje L17 en su mayoría está conformado por un grupo de aislados colectados en el 2005 en el Sistema de Riego Rio Guárico (SRRG).
Caracterización de la virulencia. El análisis de varianza permitió definir el comportamiento de las 45 aislados o cepas de la colección 2006 inoculados en los 26 genotipos de arroz (para un total de 1170 interacciones), el cual mostró diferencias estadísticas (P≤0,01) entre los factores evaluados (Cuadro 1).
Cuadro 1. Análisis de varianza de la virulencia de una población venezolana de Pyricularia oryzae infectiva de arroz (colección 2006)
| FV | gl | CM |
| Modelo | 1170 | 2,152 |
| Error | 1169 | 0,209 |
| Total | 2339 |
|
| R2 |
| 0,912 |
| Rep. | 1 | 0,354 |
| Cepa | 44 | 9,11** |
| Cultivar | 25 | 44,31** |
| Cultivar*Cepa | 1100 | 0,917** |
| C.V. (%) |
| 26,45 |
FV: fuente de variación; gl: grados de libertad. CM: cuadrado medio; **: P≤0,01
La prueba de Tukey HSD permitió establecer diferentes rangos para separar 21 grupos homogéneos, con comportamiento diferencial, respecto a la severidad del aislado o cepa. La cepa que presentó mayor severidad fue ISO-1005 con un valor de media de 2,825; le siguió el grupo de severidad intermedia, el cual está conformado por las cepas con valor de media que va de 2,430 a 1,095. De igual manera se observó que sólo un aislado, Irg048, mostró un promedio de severidad menor, con una media de 1,023.
En lo referente al factor cultivares de arroz (Cuadro 2), se pudieron observar las diferencias que existen en el comportamiento de los 26 cultivares inoculados con las cepas de P. oryzae permitiendo separarlos en 12 grupos diferentes.
Cuadro 2. Comparación de medias en cultivares de arroz inoculados con cepas de una población venezolana de Pyricularia oryzae (colección 2006)
| Cultivares | Gen de resistencia | Media |
| Fanny | sgr | 3,328 a |
| CT13432-55 | Pi-33 | 3,061 b |
| F98-7 | Pi-km | 3,844 bc |
| F124-1 | Pi-ta | 2,693 cd |
| F129-1 | Pi-kp | 2,574 d |
| Aichi Asahi | Pi-a | 2,283 e |
| F128-1 | Pi-t2 | 2,203 ef |
| Nato | Pi-i | 2,028 fg |
| Rico 1 | Pi-ks | 1,879 gh |
| Sha tia sao | grnc | 1,852 ghi |
| Fonaiap 1 | grnc | 1,738 hij |
| CT13432-68 | Pi-1 | 1,651 hijk |
| K59 | Pi-t | 1,623 ijk |
| CT13432-219 | Pi-2 | 1,526 jk |
| F145-1 | Pi-b | 1,455 k |
| Norin 22 | Pi-sh | 1,192 l |
| FD03B01 | grnc | 1,191 l |
| CT13432-107 | Pi-1, Pi-2 | 1,191 l |
| OU 244 | Pi-z | 1,188 l |
| Fedearroz 50 | grnc | 1,162 l |
| Te-tep | grnc | 1,091 l |
| Moroberekan | Pi-5(t), Pi-7(t) | 1,050 l |
| F80-1 | Pi-k | 1,049 l |
| D-Oryza | grnc | 1,040 l |
| Toride | Pi-zt | 1,027 l |
| K3 | Pi-kh | 1,008 l |
Letras distintas indican diferencias según la prueba de Tukey HSD (P≤0,05). sgr: sin genes de resistencia; grnc: genes de resistencia no conocidos
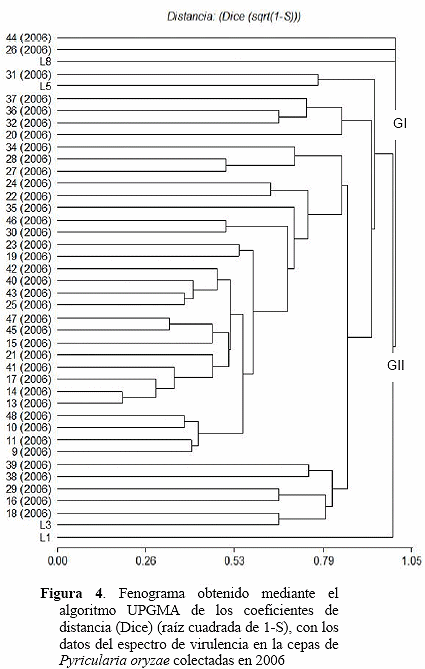
Agrupamiento por virulencia. En la Figura 4 se presenta una representación gráfica del agrupamiento de distancia de Dice, construido con los espectros de virulencia de las cepas seleccionadas para el análisis de discriminación, donde se observa la presencia de 26 grupos distintos. Éstos están distribuidos en dos grandes grupos (GI y GII) con un coeficiente de distancia alto lo que evidencia lo alejado que está uno del otro (0,97). Seguidamente, se tienen dos subgrupos que conforman el GII, donde se evidencia al L1 del otro agrupamiento mayor, a un índice de distancia de 0,95 del resto la población.
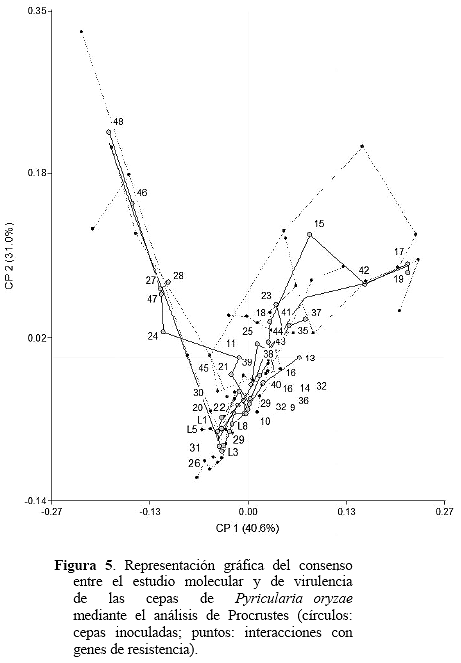
Correspondencia entre la diversidad genética y la virulencia. El análisis de Procrustes generalizado arrojó un valor de consenso de 0,68 lo cual indica que existe un alto nivel de asociación entre los resultados obtenidos en el estudio molecular y el de virulencia. Los dos métodos de caracterización de las cepas se validaron entre sí. El mismo análisis discriminó a nivel de las cepas y se pudo conocer en cuál de ellas hubo mejor concordancia entre la virulencia manifestada y el polimorfismo obtenido mediante el marcador Pot2. En general, el consenso dentro de las cepas estuvo por encima del 60 %. Entre las cepas de bajo consenso (menor de 40 %) se encuentran las 11, 18, 24, 25, 32, 36, 39, 40, 43 y 44, indicando que la discriminación de virulencia en estos casos no se corresponde con la caracterización molecular.
La Figura 5 muestra la representación gráfica del Procrustes generalizado en donde hay una ordenación de coordenadas principales para los datos moleculares y de componentes principales para el espectro de virulencia. Luego, entre éstas, se representa la ordenación de consenso en los dos ejes principales que explican más de 70 % de variación observada. Se destaca la aparición de nuevos linajes que no se agrupan con los linajes conocidos.
DISCUSIÓN
Los resultados de este trabajo difieren de los reportados por Arnao et al. (2003) y Zambrano et al. (2006), quienes indicaron que el linaje más frecuente fue el Ven-1 con 82,67 % y 76,34 %, respectivamente. También se pudo observar que los linajes Ven-1 y Ven-3 presentaron un coeficiente de similitud muy alto (0,92), resultados que coinciden con los hallados por Arnao et al. (2003), quienes indican que dichos linajes son los más cercanos.
Con respecto al porcentaje de aislados agrupados en los diferentes linajes y patrones presentes en cada localidad, se detectó que el mayor número de linajes se encontró en los estados Portuguesa (Píritu) y Guárico (Calabozo); aunque tuvieron la misma cantidad, este último presentó una mayor diversidad por tener sólo dos linajes conocidos y seis linajes nuevos, lo cual indica que es el sitio con mayor diversidad del patógeno. Esto corrobora los resultados obtenidos por Zambrano et al. (2006) quienes reportaron que el sitio con más diversidad fue Calabozo en el estado Guárico. La presión de selección sobre las cepas del patógeno es más intensa en el Sistema de Riego Rio Guárico por ser una zona de monocultivo del arroz, con efecto de los cultivares utilizados, manejo agronómico, condiciones climáticas y malezas hospedantes, lo que explica esta mayor variabilidad.
La variabilidad que revelan los resultados obtenidos coincide con lo reportado por Ling y Ou (1969) y Pantoja et al. (1997), quienes mencionan que la cantidad de razas de P. oryzae puede ser muy elevada, variando la patogenicidad y aumentando a medida que se realizan estudios debido a las diferencias entre los cultivares comerciales sembrados. Según lo reportado, Correa-Victoria y Zeigler (1994) lograron agrupar más de 100 aislados en sólo seis linajes conocidos, por lo cual, al comparar con los presentes resultados, se demuestra la alta variabilidad que existe en Venezuela donde los linajes conocidos hasta el año 2000 eran sólo siete (Zambrano et al., 2006). Por su parte Barata et al. (2009), al evaluar la diversidad fenotípica de M. oryzae, reportaron una alta variación en el comportamiento, lo que coincide con los resultados obtenidos en la presente investigación donde cada genotipo presentó un comportamiento diferente frente a los aislados inoculados.
De acuerdo a los genes de resistencia, se evidenció que el cultivar Fanny (sin genes conocidos de resistencia a P. oryzae) fue el más susceptible al presentar el mayor valor de media. También se observó que los cultivares CT13432-55, F98-7, F124-1 y F129-1 fueron los que presentaron valores de media mayores dentro del grupo de reacción intermedia con lo cual se puede inferir que el gen de resistencia que éstos poseen fue superado por alguno de los aislados inoculados durante los ensayos. Con respecto al grupo de cultivares resistentes, los resultados obtenidos indican que todos los aislados inoculados presentaron el gen de avirulencia para el respectivo gen de resistencia en cada cultivar según lo descrito por Cubero (2003), por lo que la planta logró restringir el proceso de infección del patógeno. Los cultivares cuyos genes de resistencia no han sido caracterizados revelaron reacciones intermedias y de resistencia, lo cual es un indicador de la presencia de genes de resistencia para al menos uno de los aislados inoculados.
En la interacción cepa x cultivar, los resultados coinciden con los encontrados por Arnao et al. (2008) y Lugo et al. (2008), quienes reportan que al evaluar diferentes variedades de arroz, los resultados arrojaron diferencias significativas entre cultivares, mostrando reacciones diversas entre sí respecto a la infección por P. oryzae. Igualmente, concuerdan con lo reportado por los citados autores con respecto al cultivar Fanny usado como testigo, donde indican que el mismo fue altamente susceptible con respecto a los demás genotipos evaluados, así como también que los aislados no fueron altamente virulentos en todas las variedades evaluadas.
Zeigler et al. (1995) reportaron 71 patotipos de espectro de virulencia distintos al evaluar 21 diferenciales frente a 234 aislados provenientes de dos sitios en Filipinas (4914 interacciones), lo cual representa una diversidad patotípica importante. Se presentaron múltiples espectros de virulencia en los linajes conocidos que cambiaron de acuerdo al diferencial en el que se encontraban, dándose tanto compatibilidad específica para algunos cultivares, como avirulencia para otros. Esto evidencia una variabilidad en el comportamiento de las cepas de acuerdo al cultivar o diferencial utilizado y coincide con lo reportado en el presente trabajo, en el que de 1170 interacciones se originaron 12 grupos de espectro de virulencia diferentes, que aunque en comparación con la cantidad de patotipos encontrados por los citados autores no es tan elevada, representa un comportamiento muy variable.
Las dos metodologías fueron discriminantes en un alto grado, por lo que los resultados arrojados por las pruebas moleculares y las fenotípicas (reacción fisiológica entre aislados inoculados y los cultivares con o sin genes caracterizados de resistencia) son robustas y consistentes. Existió un alto porcentaje de consenso (>70 %), lo que explica la asociación de la diversidad genética y virulencia de las cepas de la población estudiada, e indica que las cepas más apropiadas a utilizar en las investigaciones para la búsqueda de una resistencia durable en campo a P. oryzae deberían ser aquellas que se encuentran dentro de ese consenso.
En la práctica, puede seleccionarse un grupo de cepas que se encuentre dentro del consenso y producir una suspensión de inóculo con la mezcla de dos o más aislados de P. oryzae para la inoculación de líneas avanzadas o élites para resistencia a la piricularia dentro de los programas de mejoramiento de arroz. No obstante, se debe continuar con la vigilancia continua del patógeno año tras año con el fin de caracterizar su población y disponer de cepas de P. oryzae emergentes dentro del sistema arrocero, para continuar con los ciclos de mejoramiento nacional de arroz.
CONCLUSIONES
Los resultados obtenidos revelan un cambio en la conformación de la estructura poblacional de Pyricularia oryzae en el país con el paso de los años, evidenciados en la aparición de nuevos linajes y distribución de frecuencia de los linajes. Se lograron identificar diez nuevos linajes que se sumarían a los siete anteriormente reportados. Se observa que el L17 es el predominante en el Sistema de Riego Río Guárico donde se llevó a cabo la investigación, con respecto al Ven-1 anteriormente descrito. Se detectó una gran variabilidad en la virulencia de los aislados evidenciados por la reacción de los genotipos de arroz con diferentes genes de resistencia ante la inoculación de P. oryzae. A su vez, los datos de variabilidad genética y virulencia de los aislados fueron comparados y se encontró un consenso bastante alto (>70 %) entre los aislados inoculados, existiendo correspondencia entre la respuesta genética y fenotípica.
LITERATURA CITADA
1. Arnao, E., A. González, Y. Jayaro, E. Graterol y O. Borges. 2008. Evaluación de la resistencia a Pyricularia grisea en algunas variedades de arroz en Venezuela. Fitopatol. Venez. 21: 9-14. [ Links ]
2. Arnao, E., A. Vegas, Z. Gutiérrez y C. Marín. 2003. Estandarización de la técnica de PCR para la caracterización genética de una población Venezolana de Pyricularia grisea. Fitopatol. Venez. 16: 3-7. [ Links ]
3. Barata, G., A. Prabhu, M. Filippi, M. Trindade, L. Araújo y L. Zambolim. 2009. Genetic and phenotypic diversity of Magnaporthe oryzae from leaves and panicles of rice in commercial fields in the state of Goiás, Brazil. Tropical Plant Pathology 34(2):77-86. [ Links ]
4. Benacchio, S. y W. Avilan. 1991. Zonificación agroecológica del cultivo del arroz en Venezuela. Publicaciones FONAIAP. http://www.ceniap.gov.ve/pbd/monografias/benacchios/zonificacion%20arroz.pdf (consulta del 27/11/2008). [ Links ]
5. Correa-Victoria, F.J. y R.S. Zeigler. 1994. Pathogenic variability in Pyricularia grisea at a rice blast hot spot breeding site in Eastern Colombia. Plant Disease 77: 1029-1035. [ Links ]
6. Correa-Victoria, F.J., R.S. Zeigler y M. Levy. 1994. Virulence characteristics of genetic families of Pyricularia grisea in Colombia. In: R. Zeigler, S. Leong y P. Teng (eds.). Rice Blast Disease. CAB International. Wallingford, UK. pp. 211-229. [ Links ]
7. Cubero, J. 2003. Introducción a la Mejora Genética Vegetal. Edit. Mundi-prensa. Madrid. [ Links ]
8. FAO. 2013. FAO statistical yearbook. World Food and Agriculture. Food and Agriculture Organization of the United Nations. Rome. 288 p. [ Links ]
9. Fundación para la Investigación Agrícola Danac. 1996. Cultivos de arroz adaptados a las condiciones de Calabozo, estado Guárico. Boletín informativo 2(1): 1. [ Links ]
10. George, M., R. Nelson, R. Zeigler y H. Leungh. 1998. Rapid population analysis of Magnaporthe grisea by using rep-PCR and endogenous repetitive DNA sequences. Phytopathology 88: 223-229. [ Links ]
11. Ling, K. y S. Ou. 1969. Standarization of the international races numbers of Pyricularia oryzae. Phytophatology 59: 339-342. [ Links ]
12. Lugo, L., Y. Jayaro, A. González y O. Borges. 2008. Identificación de fuentes de resistencia parcial a Pyricularia grisea en cultivares y líneas experimentales de arroz. Fitopatol. Venez. 21: 51-58. [ Links ]
13. Murray, H.G. y W.F. Thompson. 1980. Rapid isolation of high molecular weight DNA. Nucleilc Acids Res. 8: 4321-4325. [ Links ]
14. Pantoja A., A. Fischer, F. Correa-Victoria, L. Sanint y A. Ramírez. 1997. MIP en arroz: manejo integrado de plagas, artrópodos, enfermedades y malezas. Centro Internacional de Agricultura Tropical (CIAT). Cali, Colombia. 141 p. [ Links ]
15. Rodríguez, H. y H. Nass. 1991. Las enfermedades del arroz y su control. Fonaiap Divulga 35(1): 1-12. [ Links ]
16. Zambrano, A., A. Vegas, R. Cardona, Z. Gutiérrez y J. Demey. 2006. Estructura genética y diversidad de linajes de Pyricularia grisea en la zona arrocera venezolana. Interciencia 31(1): 62-66. [ Links ]
17. Zeigler, R.S., L.X. Cuoc, R.P. Scott, M.A. Bernardo, D.H. Chen, B. Valent y R.J. Nelson. 1995. The relationship between lineage and virulence in Pyricularia grisea in the Philippines. Phytopathology 85: 443-451. [ Links ]