Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Salus
versión impresa ISSN 1316-7138
Salus vol.19 no.3 Valencia dic. 2015
Reacción en cadena de la polimerasa para la identificación de Salmonella spp. usando el gen invA
Teresita Luigi1,4, Legna Rojas2, Oscar Valbuena3,4
1 Laboratorio de Prácticas Profesionales de Bacteriología, Escuela de Bioanálisis Facultad de Ciencias de la Salud, Universidad de Carabobo. Venezuela.
2 Escuela de Ciencias Biomédicas y Tecnológicas. Facultad de Ciencias de la Salud, Universidad de Carabobo. Venezuela.
3 Departamento de Biología de la Facultad de Ciencia y Tecnología, Universidad de Carabobo. Venezuela.
4 Centro de Investigaciones de Microbiología Ambiental (CIMA- UC). Departamento de Microbiología, Escuela de Ciencias Biomédicas, Facultad de Ciencias de la Salud, Universidad de Carabobo. Venezuela.
Autor de correspondencia: Teresita Luigi.
E-mail: teresitaluigi@hotmail.com
RESUMEN
Se desarrolló un protocolo de PCR para identificar cepas de Salmonella de diferentes procedencias empleando un set de iniciadores PCR específico a Salmonella, para el reconocimiento de una secuencia de 119 pb del gen invA. Los resultados cumplieron con las máximas de la selectividad: inclusividad, pues todas las 14 cepas de Salmonella ensayadas presentaron señal positiva para el gen, exclusividad ya que ninguna cepa no Salmonella (Shigella, E. coli, E. coli O157:H7) mostró señal positiva, robustez porque en las 14 cepas de Salmonella se obtuvo la señal esperada, en un total de cuatro repeticiones con iguales condiciones de trabajo en cuatro días no consecutivos. Asimismo, fue reproducible, pues los resultados fueron idénticos al modificar las condiciones de reacción. Al comparar la PCR con el cultivo convencional, usando preenriquecimiento no selectivo en agua peptonada y enriquecimiento selectivo en caldo selenito-cistina, se apreció que la PCR fue 100% sensible y especifica, con 100% de inclusividad como de exclusividad.
Palabras Clave: Salmonella spp, reacción en cadena de la Polimerasa (PCR), gen invA.
Polymerase Chain Reaction (PCR) for identification of Salmonella spp. using the invA gene
ABSTRACT
A PCR protocol was developed to identify strains of Salmonella from different sources using a set of primers PCR specific to Salmonella, for the recognition of 119 bp invA gene sequence. The results met the maxims of selectivity: inclusiveness, all strains of Salmonella presented positive signal for the gene, exclusivity since no strain Salmonella (Shigella, E. coli, E. coli O157: H7) showed no signal positive robustness because in the 14 strains of Salmonella expected signal was obtained in a total of four repetitions with equal working conditions in four non-consecutive days. Also it was reproducible, results were identical by modifying the reaction conditions. Comparing PCR with conventional culture, using non-selective pre-enrichment in peptone water and selective enrichment in selenite-cystine broth, it was found that the PCR was 100% sensitive and specific, with 100% of inclusiveness, 100% exclusive and 100% of both positive and negative predictive value.
Key words: Salmonella spp - Polymerase Chain Reaction (PCR), gene invA.
Recibido: 1/8/2014 Aprobado: 6/11/2015
INTRODUCCIÓN
La reacción en cadena de la polimerasa (PCR) es una técnica sensible de amplificación in vitro de segmentos de ADN que permite detectar genes específicos de grupos taxonómicos y genes implicados en la virulencia de bacterias patógenas, y así conducir a la identificación del microorganismo de interés (1). El éxito de la identificación depende de la existencia de una secuencia de ADN específica y exclusiva del organismo bajo estudio (2). Entre los microorganismos patógenos cuya detección requiere difundir el uso de PCR se encuentran los pertenecientes al género Salmonella, ya que a pesar de las variadas medidas de salud pública desarrolladas en el siglo pasado, Salmonella sigue siendo el causante de una toxiinfección alimentaria por zoonosis más importante a nivel mundial (3). La salmonelosis es una infección de importancia tanto en salud pública como salud animal debido al impacto económico que ocasiona. Se ha calculado que en Estados Unidos de América, la salmonelosis causa más de 18 mil hospitalizaciones y 500 defunciones anuales (4). A partir de diversas muestras de alimentos se ha realizado la detección e identificación rápida de Salmonella, mediante la PCR (5-6), empleando iniciadores que copian diversas secuencias blanco obteniendo productos para el reconocimiento de genes específicos de Salmonella entre los cuales se encuentra el gen invA (7 y 8). Este gen es un factor de virulencia, relacionado con el proceso de invasión al epitelio intestinal (8). En relación a los métodos convencionales de recuperación de Salmonella sp, los mismos requieren del aislamiento en medios selectivos, posterior identificación bioquímica y caracterización serológica, lo que se traduce en tiempo prolongado para su detección (de 5 a 7 días) (9). En contraste, mediante la PCR el tiempo de detección sería de aproximadamente
24 a 48 horas, dependiendo de las modificaciones en el protocolo de trabajo (10). Cabe resaltar que desde 1999 la Comisión Europea aprobó el proyecto de investigación, FOOD-PCR, con la intención de aplicar y estandarizar el uso de PCR como diagnóstico para la detección de patógenos bacterianos en alimentos (1). No obstante, en Latinoamérica los avances han sido limitados a ciertos países de la región. Aunque a nivel mundial ya se ha empleado el gen invA para la identificación de Salmonella, en Venezuela no se conocen estudios o protocolos reportados sobre estandarización de la PCR empleando el gen invA específico, por lo que la adaptación del protocolo a las condiciones de nuestros laboratorios permitiría su implementación tanto la industria alimentaria como el sector clínico. Dada la importancia de Salmonella en la salud pública, sería beneficioso un protocolo de PCR rápido, específico y confiable. El objetivo del presente trabajo fue describir un protocolo de PCR para la detección de Salmonella spp, usando iniciadores PCR género específicos, los cuales generan un producto de 119 pb correspondiente al gen invA.
MATERIALES Y MÉTODOS
Cepas. El material de estudio estuvo constituido por 14 cepas diferentes de Salmonella, algunas provenientes de aislados clínicos del laboratorio del Departamento de Microbiología de la Facultad de Ciencias de la Salud de la Universidad de Carabobo (MFCS-UC), otras del Laboratorio de Prácticas Profesionales de Bacteriología, Departamento de Estudios Clínicos, Escuela de Bioanálisis (PPB-EB-UC), sede Carabobo, de la misma universidad y cepas de referencia del Centro Venezolano de Colecciones de Microorganismos (CVCM), Laboratorio de Biología de Plásmidos Bacterianos, Centro de Biología Celular, Instituto de Biología Experimental, Facultad de Ciencias, Universidad Central de Venezuela. Las cepas que presentaron un perfil bioquímico compatible con Salmonella spp. fueron confirmadas mediante la prueba de aglutinación en placa con Antisuero Polivalente Somático O DifcoTM (Salmonella O Antiserum Poly A-I & Vi, 222641).
Cepas controles. Como control positivo se empleó un aislado de Salmonella Enteritidis perteneciente a la colección del CVCM. Como controles negativos se usaron 2 cepas de Shigella boydii, Escherichia coli silvestre y Escherichia coli O157:H7 pertenecientes a la bacterioteca del MFCS-UC.
Método convencional para la detección de Salmonella. La detección de Salmonella fue realizada según la metodología descrita por COVENIN 1291 (11). La misma se desarrolló siguiendo tres etapas: Un pre-enriquecimiento, en 225 mL de agua peptonada al 0,1 % donde se inoculó con cada una de las 14 cepas. Posteriormente se incubó a 37 ºC por 24 horas. Transcurridas las 24 horas del pre-enriquecimiento, se inició la etapa de enriquecimiento selectivo, para la cual se tomó 1 mL de la muestra pre-enriquecida y se inoculó por separado en tubos contentivos de 10 mL de Caldo Selenito-Cistina (Merck KGaA, Darmstadt, Alemania), incubando los tubos a 37ºC por 24 horas. Al culminar el período de incubación, se procedió al enriquecimiento diferencial. Las muestras provenientes de los caldos Selenito-Cistina se sembraron por agotamiento en placas de Petri con Agar MacConkey, Agar Xilosa- Lisina-Desoxicolato (XLD) y Agar Salmonella-Shigella (SS) (BBL Becton Dickinson and Company. Sparks, MD USA) respectivamente. De esta manera, se dispusieron de seis placas por cada muestra, las cuales fueron incubadas durante 24 horas a 37°C. Posteriormente, se seleccionaron las colonias y posteriormente se seleccionaron 2 a 5 colonias para la caracterización fenotípica mediante el empleó el sistema automatizado API Lab Plus (Biomeriux).
Extracción de ADN Genómico. Las cepas bacterianas y la muestra de sangre total fueron sometidos a extracción de ADN genómico. Los inóculos experimentales de Salmonella fueron diluidos en el caldo Infusión Cerebro Corazón (BHI) (BBL Becton Dickinson and Company. Sparks, MD USA) hasta obtener recuentos desde 3 x 10 UFC/mL hasta 4 x 109 UFC/mL. Los ensayos se realizaron por triplicado mediante los siguientes métodos:
a) Extracción mediante kit comercial. El procedimiento se realizó siguiendo las especificaciones del protocolo sugerido por la casa comercial (Wizard® Genomic ADN Purification Kit Promega. Madison, WI, EE.UU) para el aislamiento de ADN genómico a partir de bacilos Gram negativo. El ADN obtenido, se almacenó a 2-8 ºC, hasta su análisis por PCR, de lo contrario fue conservado en un Biofreezer de –20°C (Cool- labTM Lab-line Instruments, Inc.).
b) Extracción mediante choque térmico. Para adecuar este procedimiento a laboratorios con poca logística instrumental el choque térmico se efectuó en un termobloque a 95°C o en baño de agua hirviente, ambos por 20 min. A tal fin 3 asadas de los cultivos bacterianos se resuspendieron en 100 μL de agua estéril y luego ambas muestras fueron centrifugadas a 13.000 rpm por 3 minutos. Cada muestra fue almacenada a 4ºC hasta su análisis por PCR.
Determinación de la concentración de ADN. Posterior a la extracción de ADN por los métodos antes descritos, se midió la absorbancia de una dilución de ADN 1:100 V/V (10 μL de solución de ADN en 990 μL de agua) a 260nm y a 280nm, y se procedió a calcular el cociente D.O.260nm /D.O.280nm. La concentración de ADN (μg/mL) fue calculado de acuerdo a Sambrook y Russell (12), empleando la siguiente formulación: [ADN]=50 (factor)* x100 (dilución) x D.O.260nm * (Factor =50 μg de ADN lee 1 unidad de Absorbancia a 260 nm/mL).
Reacción en Cadena de la Polimerasa (PCR). Los ADN genómicos obtenidos fueron sometidos a la reacción en cadena de la polimerasa utilizándose iniciadores (primers) PCR específicos para amplificar una secuencia de 119 pb del gen de invasión (invA) de Salmonella (acceso GenBank nº M90846) referido por Arnold et al (14). Los oligonucleótidos (iniciadores) empleados fueron: Styinva-JHO-2-left secuencia 5-TCGTCATTCCATTACCTACC-3 y Styinva-JHO-2-right secuencia 5-AAACGTTGAAAAACTGAGGA-3 La reacción de PCR, se llevó a cabo en microtubos Eppendorf de 1,5 mL en un volumen final de 50 μL, conteniendo: 25 μL de mezcla Master Mix 2X, 3 μL de Primer invA1 100pM/uL, 3μL de Primer invA2 100pM/uL, 14 μL de agua libre de nucleasa y 5 μL del ADN. La reacción se realizó en un termociclador (MiniCyclerTM MJResearch) bajo las siguientes condiciones: un pre-ciclo a 94ºC por 3 min, 35 ciclos a 94ºC por 1 min c/u, 1 ciclo a 55ºC por 1 min, 1 ciclo a 72ºC por 1 min y una elongación final a 72ºC por 10 min. Los amplicones se analizaron mediante electroforesis en geles de agarosa 2% teñidos con bromuro de etidio y visualizados con un transiluminador de luz UV y la fotodocumentación se realizó empleando una cámara fotográfica Polaroid.
Controles del sistema PCR. Para evaluar la selectividad de la PCR se utilizaron diferentes controles cuyos ADN moldes se especifican a continuación: Control positivo: ADN genómico Salmonella Enteritidis (CVMC). Control de mezcla: mezcla sin muestra de ADN. Controles negativos y de especificidad de iniciadores: ADN genómico de Escherichia coli silvestre, Escherichia coli O157:H7, Shigella boydii, ADN humano; mezcla de ADN de Salmonella más ADN de humano; mezcla con ADN de Salmonella más ADN de Shigella spp., siguiendo recomendaciones internacionales por expertos en el área, la extracción del ADN, la preparación de las mezclas y los reactivos, el procesamiento de las muestras y la PCR, fueron realizadas en cabinas situadas en áreas separadas y alejadas de la zona donde se analizaron los productos amplificados.
Análisis Estadístico. Para el análisis estadístico de los resultados se compararon los resultados de la PCR con los obtenidos mediante el método de cultivo convencional, considerándose positivo a todo resultado en el que se observó señal de amplificación positiva para el gen invA y negativo a todo resultado en el cual no se observó señal positiva. Por otro lado, fueron calculados los índices de sensibilidad, especificidad, inclusividad y exclusividad para la PCR y la metodología convencional para la detección de Salmonella spp. Las mismas fueron calculadas de la siguiente manera: Sensibilidad(%)=[A/(C+A)]x100, Especificidad(%)=[B/(C+B)]x100, Inclusividad(%)=[A/A+B]x100, Exclusividad%)=[D/C+D]x100. Donde B, correspondió al número de verdaderos negativos; A, al número de verdaderos positivos; D, al número de falsos negativos, y C, al número de falsos positivos (13).
RESULTADOS
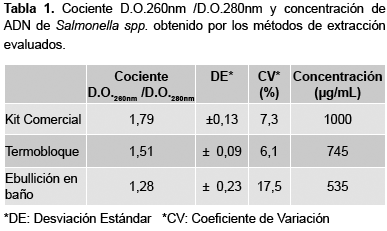
Los resultados obtenidos luego de la extracción de ADN genómico a partir de Salmonella spp, se reportan en la tabla 1. En relación a la extracción realizada mediante kit comercial (Wizard, Promega), la misma presentó un cociente D.O.260nm /D.O.280nm de 1,79 con una desviación estándar (DE) de ±0,13 y un coeficiente de variación (CV) de 7,3 %. De igual manera, los ADN genómicos obtenidos, empleando los métodos de extracción físicos, presentaron cocientes D.O.260nm /D.O.280nm de 1,28 (DE ± 0,23 y CV=17,5%) para la extracción en baño en agua hirviente, y de 1,51 (DE ± 0,09 y CV= 6,1 %) con el calentamiento en termobloque.
Respecto a la concentración de ADN obtenida con el empleo de los diferentes métodos, y partiendo de una suspensión de bacteriana con igual recuento de microorganismos (4 x 109 UFC/mL), se obtuvo mediante el kit comercial una media de 1000 μg de ADN, con el calentamiento en termobloque se obtuvieron 745 μg de ADN y mediante ebullición en baño de agua 535 μg de ADN. Los resultados evidencian una mayor concentración de ADN extraído por el kit comercial en relación a los métodos físicos. No obstante, todos los fragmentos de ADN genómicos extraídos independientemente del método usado, lograron ser amplificados por la PCR.
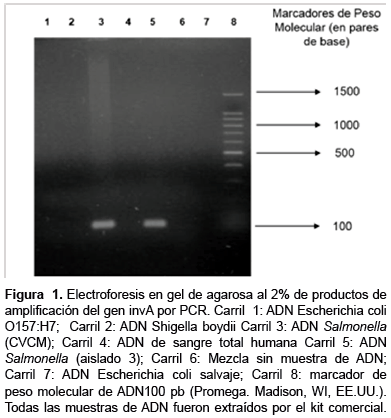
En la figura 1 se observan los productos de amplificación (amplicones) provenientes de la reacción de PCR utilizando ADN de Salmonella (CVCM y un aislado clínico), E. coli (cepas O157H7 y cepa silvestre) y de Shigella. Además se incluyó una reacción con ADN de sangre total humana y un sistema sin ADN.
Los amplicones producidos, con una masa molecular de 119 pb, sólo se detectaron en los sistemas suplementados con ADN de Salmonella (carriles 3 y 5), no detectándose en los sistemas de E. coli, Shigella, ADN humano y sin ADN.
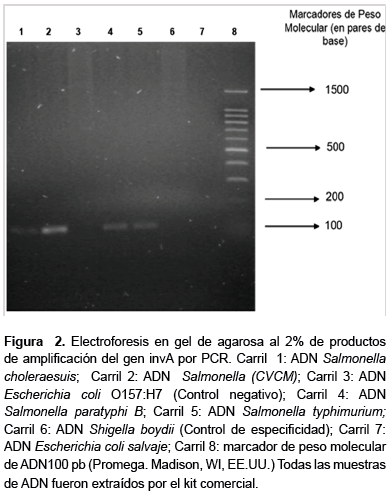
En la figura 2 se observa el amplicón de 119 pb en las reacciones de amplificación correspondientes a diferentes cepas de Salmonella (S. choleraesuis, S. Enteritidis CVCM, S. paratyphy B y S. typhimurium; carriles 1, 2, 4 y 5, respectivamente); de nuevo en las reacciones con ADN de E. coli y Shigella no se detectó producto de amplificación.
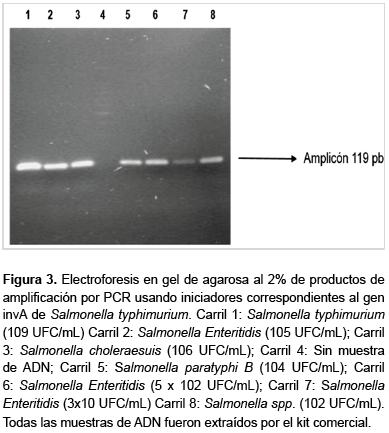
En la figura 3 se muestran los amplicones obtenidos al procesar diferentes cantidades de las cepas bacterianas (y por ende de ADN), el intervalo de concentraciones fue de 10 9 UFC/mL (S. typhimurium, carril 1) a 3x10 UFC/mL (S. Enteritidis, carril 7). En todos los casos se detectó el amplicón de 119pb, evidenciándose un límite de detección que debería ser inferior a 3x10 UFC/mL (S. Enteritidis, carril 7). Cualitativamente se observa que a mayor número de células procesadas, se obtuvo una mayor intensidad del amplicón detectado.
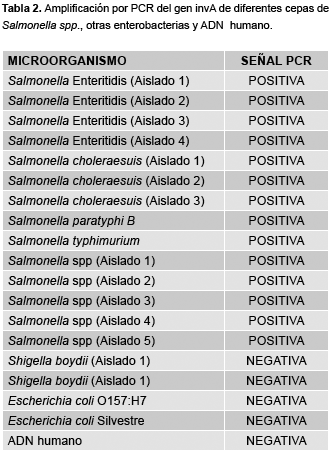
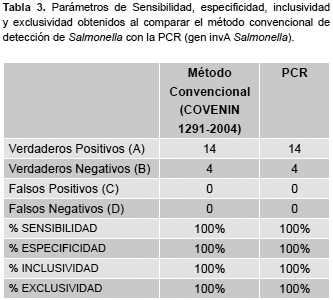
Resumiendo, en la tabla 2 se detallan los resultados obtenidos en la PCR al usar como fuente de ADN diversas cepas de Salmonella y otros microorganismos enteropatógenos de diferente procedencia. Todas las cepas de Salmonella fueron positivas para la amplificación del gen invA, mientras que fue negativa las cepas de E. coli, Shigella y ADN humano. Al analizar la robustez de la técnica, como parámetro al evaluar un método cualitativo se debe mencionar que las catorce distintas cepas de Salmonella sometidas a ensayo presentaron la señal esperada en un total de cuatro repeticiones empleando las mismas condiciones de trabajo (ensayo intralaboratorio) en cuatro días diferentes no consecutivos. El análisis estadístico de los datos obtenidos (tabla 3) indicaron los siguientes resultados: 100% de sensibilidad y especificidad, 100% de inclusividad, 100% de exclusividad, 100% de valor predictivo positivo y 100% de valor predictivo negativo. Por lo tanto, al comparar la PCR con el método de cultivo convencional, se pudo apreciar que la técnica de PCR empleando el gen invA para la identificación de Salmonella desarrollada en esta investigación, cumple con los parámetros idóneos para la óptima evaluación de un ensayo de laboratorio.
DISCUSIÓN
Entre los métodos de extracción de ADN evaluados en la presente investigación, el kit comercial permitió la obtención de una mayor concentración de ADN genómico. Sin embargo, aunque los métodos físicos de extracción arrojaron concentraciones de ADN inferiores a las obtenidas por el kit comercial, todos los fragmentos de ADN genómico extraídos lograron ser amplificados por la PCR, generándose el amplicón esperado. La diferencia en la cantidad de ADN extraídos con los diferentes protocolos es debido a que con el kit comercial se efectúa una desproteinización del ADN, mientras que en los protocolos con choque térmico no se efectúa. Aunque los cocientes D.O.260nm /D.O.280nm fueron menores en los métodos físicos y por lo tanto, la pureza del ADN extraído (evidenciado por los cocientes D.O.260nm /D.O.280nm) fue menor, ello no influyó en la amplificación correcta del gen invA. En términos de tiempo de ejecución, el método de extracción con calor por calentamiento en agua hirviente puede ser considerado como un método adecuado para ser implementado previo a la PCR, ya que resulta menos engorroso. En cuanto al procedimiento, no involucra digestión con proteinasa K, extracción con fenol/cloroformo ni los riesgos inherentes a la misma, ni de equipo calentador. Además de sencillo, económico y no dependiente de reactivos o kits importados, permite reducir el tiempo de extracción, permitiendo el procesamiento simultáneo de gran número de muestras (14), las cuales constituyen bondades que pudiesen influir en adoptar esta sencilla técnica en los laboratorios de rutina microbiológica en el país. Cabe señalar que los resultados indican que aún con bajas concentraciones de ADN, bajo estas condiciones experimentales, es posible obtener un proceso de amplificación apropiado. Por otro lado, la PCR es una técnica sensible cuyo éxito en su implementación depende, entre otros factores, de la elección correcta de la secuencia blanco, la cual debe permitir la identificación del microorganismo de interés, independientemente de la presencia de otras fuentes de ADN procedente de microorganismos concomitantes o de la muestra misma (15). La alta especificidad de la PCR empleando el gen invA para detectar Salmonella confirmó que la secuencia de 119 pb de dicho gen contiene secuencias únicas para el género, por lo que es un blanco conveniente de PCR. Es importante comentar que métodos basados en la PCR usando el gen invA han dado como resultado la detección positiva de Salmonella. Esto es s importante para que instituciones como la FDA promuevan la implementación de la técnica de forma rutinaria (16). En relación a la presente investigación, a diferencia de otros trabajos el protocolo, fue totalmente modificado, observándose resultados positivos para la detección de Salmonella, inclusive cuando se varió intencionalmente algunos pasos del procedimiento, en especial los relacionados a volúmenes de mezcla (se ensayó la PCR con volúmenes finales de 50, 25 y 20 μL), obteniéndose el amplicón esperado con el empleo de hasta 20 μL de mezcla de reacción y 2 μL de muestra de ADN. Es importante destacar que la PCR debe cumplir parámetros de confiabilidad respaldados por un adecuado empleo de controles en el sistema de PCR (7). Para obtener un resultado confiable a partir de una técnica de diagnóstico basada en PCR se sugiere utilizar un control positivo, un control negativo y un control blanco de reactivos. Además, la inclusión de un control interno de amplificación (IAC, según siglas en inglés) puede evidenciar una falla en cada tubo de reacción, ya sea por la presencia de inhibidores o por un problema particular inherente a la técnica, situación que ha sido descrita al analizar muestras de alimentos. En esta investigación se incluyeron controles positivos y un blanco. Sin embargo, no se consideró la incorporación de un IAC porque la misma fue diseñada en una primera fase de experimentación para ser utilizada como tamizaje o confirmación a partir de cultivos o suspensiones bacterianas y no directamente de muestra de alimentos, donde generalmente se pueden encontrar los inhibidores de la PCR, en especial, de la taq polimerasa. La técnica ensayada presentó buen desempeño al utilizar el ADN correspondiente 3x10 UFC/mL de S. Enteritidis. No se observaron interferencias al utilizar concentraciones elevadas de ADN (hasta 109 UFC/mL), ni al combinar distintas concentraciones de cepas bacterianas, lo cual evidencia la alta especificidad de la técnica. Se demostró que la técnica de PCR fue reproducible observándose iguales resultados inclusive cundo se varió la cantidad de ADN, obteniéndose el amplicón esperado con el empleo de hasta 20 μL de mezcla de reacción y 2 μL de muestra.
En conclusión, la PCR para el gen invA de Salmonella resultó ser altamente específica y sensible para la detección de miembros de este género en suspensiones bacterianas con recuentos desde 4 x 109 UFC/mL hasta 3 x 10 UFC/mL. Los resultados obtenidos bajo las condiciones experimentales establecidas cumplieron con los requisitos de selectividad, exclusividad, robustez y reproducibilidad, lo cual tiende a validar y estandarizar el uso de la PCR en la determinación de Salmonella spp. Asimismo, a diferencia del método convencional de detección de Salmonella, el cual resulta laborioso y requiere de medios de cultivos para su reactivación, aislamiento en medio selectivo y caracterización fenotípica mediante el empleó el sistema automatizado API Lab Plus (Biomeriux), (siendo necesario de 5 a 7 días para confirmar los resultados), con la PCR usando el gen invA, el tiempo de detección por PCR fue de 24h, lo cual se traduce en un balance positivo en la economía de las empresas, así como de las instituciones del estado encargados de velar por la salud pública al lograr un diagnóstico rápido y preciso. Se sugiere continuar desarrollando investigaciones donde se aplique este protocolo de PCR para detectar Salmonella a partir de alimentos.
Agradecimiento. Los autores desean expresar su agradecimiento al personal del Centro de Investigaciones Microbiológicas Aplicadas (CIMA) de la Universidad de Carabobo, Venezuela.
REFERENCIAS BIBLIOGRÁFICAS
1. Mandal P, Biswas A, Choi K, Pal U. Methods for rapid detection of foodborne pathogens: an overview. Am J Food Technol. 2011; 6(2): 87-102. [ Links ]
2. González J., Pereira N, Soto Z, Hernández E, Villarreal J. Microbiological Isolation of Salmonella spp. And Molecular tools for detection. Revista Salud Uninorte. 2014; 30(1): 73-94. [ Links ]
3. Arcos E, Mora L, Fandino L, Rondón I. Prevalencia de Salmonella spp. en carne porcina, plantas de beneficio y expendios del Tolima. Orinoquia. 2013; 17 (1): 59-68. [ Links ]
4. Gutiérrez A, Paasch L, Calderón N. Salmonelosis y campilobacteriosis, las zoonosis emergentes de mayor expansión en el mundo. Vet Méx. 2008; 39(1): 81-90. [ Links ]
5. Koyuncu S, Andersson M, Häggblom P. Accuracy and sensitivity of commercial PCR-based methods for detection of Salmonella enterica in feed. Appl Environ Microbiol. 2010; 76(9): 2815-2822. [ Links ]
6. Acosta L, Pinedo J, Hernández E, Villarreal J. Comparación de los métodos de inmunoensayo enzimático automatizado (VIDAS) y PCR para la detección de Salmonella spp. en expendios de la ciudad de Santa Marta (Colombia). 2013. Salud Uninorte. 2013; 29(2):174-182. [ Links ]
7. Shanmugasam M, Velayutham T, Rajeswar J. Inv A gene specific PCR for detection of Salmonella from broilers. Veterinary World. 2011; 4(12): 562-564. [ Links ]
8. Chacón L, García C, Barrantes K, Achí R. Estandarización de un método de PCR para la detección de Salmonella spp. en lechuga. Rev Soc Ven. Microbiol. 2010; 30:18-23. [ Links ]
9. Villareal J, Soto Z, Pereira N, Varela L, Jaramillo R., Villanueva D, Mendoza E. Reacción en cadena de la polimerasa para la detección de Salmonella sp. en leche en polvo: Optimización del método en 12 horas. Salud Uninorte. 2011; 24(2): 216-225. [ Links ]
10. Palomino C, González Y. Técnicas moleculares para la detección e identificación de patógenos en alimentos: ventajas y limitaciones. Rev Peru Med Exp Salud Pública. 2014; 31(3). [ Links ]
11. Comisión Venezolana de Normas Industriales (COVENIN) Norma Nº: 1291. Alimentos. Aislamiento e identificación de Salmonella. 1988 (lra. rev.). [ Links ]
12. Sambrook, J y Russell, D. Molecular Cloning: a laboratory manual. (3 a ed). CSH Laboratory Press, cold Spring Harbor. NY. 2001. [ Links ]
13. Leotta G, Chinen I, Epszteyn S, Miliwebsky E, Melamed I, Motter M., Rivas M. Validación de una técnica de PCR múltiple para la detección de Escherichia coli productor de toxina Shiga. Rev Argent Microbiol. 2005; 37: 1-10. [ Links ]
14. Osorio E, Ramírez M, López A, Mambuscay L. Estandarización de un protocolo sencillo para la extracción de ADN genómico de levaduras. Rev colomb Biotecnol; 2009; (1): 125-131. [ Links ]
15. Tamay de Dios L, Ibarra C, Velasquillo C. Fundamentos de la reacción en cadena de la polimerasa (PCR) y de la PCR en tiempo real. 2013; 2 (2): 70-78. [ Links ]
16. González N, Hammack T, Russell M, Jacobson A, De Jesús A, Brown E, Lampel K. Detection of live Salmonella sp. cells in produce by a TaqMan-based quantitative reverse transcriptase real-time PCR targeting invA mRNA. Appl Environ Microbiol. 2009; 75(11): 3714-3720. [ Links ]