Boletín de Malariología y Salud Ambiental
versión impresa ISSN 1690-4648
Bol Mal Salud Amb v.47 n.1 Maracay ene. 2007
Uso de proteínas como alternativa diagnóstica para discriminar infecciones entre Trypanosoma vivax y Trypanosoma evansi
Ana María Bolívar1, Armando Reyna-Bello2, Francisco García3, Pablo García-Lugo1, Gladys Crisante1, Agustina Rojas1 & Nestor Añez1*
1 Investigaciones Parasitológicas J. F. Torrealba, Universidad de los Andes, Facultad de Ciencias, Departamento de Biología, Mérida, 5101, Venezuela.
2 Universidad Simón Rodríguez-IDECYT, Laboratorio de Inmunología, Caracas, Venezuela.
3 Cátedra de Parasitología, Facultad de Ciencias Veterinarias, Universidad Central de Venezuela, Aragua, Venezuela
*Autor de correspondencia: nanes@ula.ve
Tripomastigotes sanguícolas de Trypanosoma vivax y Trypanosoma evansi obtenidos de infecciones experimentales en ovejas y ratones respectivamente, fueron utilizados para purificar y caracterizar proteínas citosólicas mediante el método de partición con Tritón X-114. Los resultados revelan diferencias en los patrones proteicos entre las dos especies. Asimismo, las reacciones antigénicas mediante Western blot utilizando suero de animales naturalmente infectados, permitió discriminar las infecciones entre ambos parásitos. Se sugiere la utilización de esta metodología como una prueba diagnóstica confiable.
Palabras claves: T. vivax, T. evansi, diagnostico, proteínas.
Use of proteins as diagnostic tool to discriminate between Trypanosoma vivax and Trypanosoma evansi infections
SUMMARY
Trypanosoma vivax and Trypanosoma evansi blood trypomastigotes, obtained respectively from sheep and mice experimentally infected, were used to isolate and characterize proteins using the partition Triton X-114 method. Results showed a different protein pattern between the 2 species. Additionally, results by Western blot, using sera from naturally infected animals, revealed specific discrimination between these two species of Trypanosoma. The potential use of this method as a reliable diagnostic tool is suggested.
Key words: T. vivax, T. evansi, diagnosis, proteins.
Recibido el 04/05/2006 Aceptado el 05/02/2007
INTRODUCCIÓN
Trypanosoma (Dutonella) vivax y Trypanosoma (Trypanozoon) evansi han sido incriminados como responsables de infecciones sufridas en una amplia gama de hospedadores en regiones tropicales y subtropicales de Suramérica, Asia y el norte de África. Entre los grupos mas afectados se destacan bovinos, equinos, bufalinos, caprinos, ovinos, caninos, suinos, ciervos y camellos, los cuales presentan variaciones clínicas durante la infección observándose desde una forma aguda hasta la crónica, pasando por estados completamente asintomáticos o de portador sano, situación por la cual algunos de ellos pudieran actuar como reservorios naturales (Luckins, 1988; Sandoval et al., 1996; Tamasaukas & Roa, 1996; Espinoza et al., 2002). En Venezuela, el efecto de estos parásitos sobre el contingente ganadero es poco conocido debido a los escasos estudios sobre su incidencia y su prevalencia, situación que plantea carencia de datos precisos sobre aspectos epidemiológicos para ambas infecciones (Rivera, 1996; García et al., 2000).
Las infecciones causadas por T. vivax y T. evansi se ubican entre las principales entidades nosológicas de importancia económica en América, ejerciendo un fuerte impacto sobre la salud y reproductividad en rebaños vacunos y equinos. De allí la relevancia de su estudio y el desarrollo de investigaciones que coadyuven a su control, siendo uno de los aspectos indispensables para ello, obtener un diagnóstico certero y efectivo que supere la baja sensibilidad y especificidad de los métodos convencionales empleados hasta el presente (García & Mendoza-León, 2000; Tamasaukas et al., 2000). Esto se impone ya que debido a la dificultad de aislar y caracterizar antígenos de T. vivax, es común la utilización de antígenos de T. evansi aprovechando la similitud antigénica compartida para realizar diagnósticos en infecciones por T. vivax (Uzcanga et al., 2002, 2004). Esta práctica diagnóstica pudiera arrojar falsos positivos en zonas ganaderas donde ambas tripanosomiasis coexisten (Toro et al., 1980). Justificado por lo anterior, en el presente trabajo se presenta el desarrollo de una metodología que permite discriminar infecciones causadas por ambos parásitos utilizando proteínas específicas para cada especie cuya inmunogenicidad permite un diagnóstico alternativo.
MATERIALES Y MÉTODOS
Fraccionamiento proteico de las formas sanguícolas de T. vivax y T. evansi
Las formas sanguícolas de T. vivax fueron aisladas y purificadas siguiendo la metodología descrita por González et al. (2005). Una vez obtenidos los parásitos libres de elementos sanguíneos (2x108), fueron fraccionados siguiendo la metodología descrita por Ko & Thompson (1995) y Añez-Rojas et al. (2006) utilizando el método de partición con el detergente Tritón X-114. Brevemente, el concentrado de parásitos (pellet) colectado fue resuspendido en buffer 1 (10mM Tris-HCl, 150 mM NaCl, 2% Triton X-114, pH 7.4) el cual contenía un pool de inhibidores de proteasas (Roche), en agitación durante 1h a 0ºC y luego centrifugado a 7.200x g a 0ºC por 10 min. El pellet (Pi) obtenido conteniendo las proteínas de membrana no solubilizadas por el Tritón X-114, fue lavado con buffer 2 ( 10mM Tris- HCl, 150 mM NaCl, 0.06% Tritón X-114, pH 7.4) y mantenido a -20ºC. El sobrenadante fue mantenido a -20ºC durante 24 h y luego descongelado lentamente a temperatura ambiente colocándose durante 12 min a 32ºC para obtener la primera partición (P1). P1 fue luego centrifugado a 3.000xg por 3 min obteniéndose dos fases. La fase acuosa obtenida, conteniendo las proteínas hidrofílicas (F3) fue separada y mantenida a -20ºC. La fase detergente fue resuspendida en 3V del buffer 2 arriba indicado y mantenido por 15 min a 0ºC. El proceso de partición (P1) fue repetido y la fase acuosa conteniendo el resto de proteínas hidrofílicas fue descartado. La fase detergente fue mezclada con 3V del buffer 2, mantenida a 0ºC por 10 min y centrifugada a 18.000 x g a 0ºC por 10 min. El pellet conteniendo proteínas hidrofóbicas, no ancladas por GPI (F1) fue mantenido a -20ºC. El sobrenadante fue sometido a un segundo proceso de partición. Las proteínas contenidas en la fase detergente resultante fueron precipitadas con 3V de acetona fría y centrifugadas a 3.000 x g durante 7 min a 0ºC para obtener proteínas ancladas por GPI (F2). Este procedimiento permitió la separación de las proteínas de T. vivax en cuatro fracciones: Pi conteniendo proteínas de membrana que no solubilizan con el Tritón X-114; F1 conteniendo proteínas integrales de membrana no ancladas por glicosil-fosfatidil-inositol(GPI); F2 proteínas de membrana ancladas por GPI y F3 proteínas citoplasmáticas (hidrofílicas). Las proteínas de F2 y F3 fueron precipitadas con tres volúmenes de acetona fría y cada fracción fue homogeneizada en buffer de muestra (Tris-HCl 62 mM, SDS 2%, glicerol 10%, azul de bromofenol 0,05%, pH 6,8). Para evidenciar la presencia específica de proteínas ancladas por GPI se procedió a realizar un tratamiento con fosfolipasa C como fue previamente indicado por Ko & Thompson (1995) y Añez-Rojas et al. (2006), el resto de las proteínas lograron separarse por sus características de hidrofóbicas (F1) y de hidrofílicas o citoplasmáticas (F3).
El fraccionamiento proteico de las formas sanguíneas de T. evansi se realizó siguiendo el procedimiento descrito para T. vivax. Los parásitos (1,5x108) fueron obtenidos al infectar experimentalmente ratas albinas con T. evansi aislado de Hydrochoerus hydrochoerus, procedente del estado Apure, Venezuela y purificados siguiendo la metodología descrita por Lanham & Godfrey (1970).
Electroforesis en geles de poliacrilamida con Dodecilsulfato de sodio (SDS-PAGE).
La electroforesis fue realizada en presencia de SDS, sobre geles de poliacrilamida al 12% y 0,75 m de espesor en un sistema de Mini-Protean IIIBIO-RAD, siguiendo el procedimiento descrito por Laemmli (1970). Los geles fueron corridos a voltaje constante de 100v. Las proteínas fueron coloreadas con Azul de Comassie R-250. Cada carril contenía 15% de cada fracción (15 μl) previamente calentados a 90ºC durante 3 minutos, en presencia de 50 mM de ditiotreitol (DTT).
Análisis por Western blot.
Realizada la electroforesis (SDS-PAGE) de la fracción F3 de cada parásito, las proteínas fueron electro-transferidas a membranas de PDVF (Inmobilon P), aplicando una corriente de 30V, durante 14 horas a 4ºC, según Winston et al. (1987). La inmunodetección de antígenos fue realizada previa permeabilización de la membrana con alcohol isopropilico 70% e incubadas con suero de bovino o equino (dilución 1:100). Las membranas fueron incubadas con la apropiada dilución de los anticuerpos secundarios, conjugado anti-bovino IgG peroxidasa (1:10.000) o conjugado anti-equino IgG fosfatasa alcalina (1:10.000). Finalmente, las bandas de los polipéptidos fueron visualizadas por la adición de 4-chloro-1-naphthol-H2O2 y NBT (Nitroblue tetrazolium)-BCI (5-Bromo-4-Cloro-3-indolylfosfato) respectivamente.
RESULTADOS
Aislamiento y análisis de proteínas de Trypanosoma vivax y T. evansi.
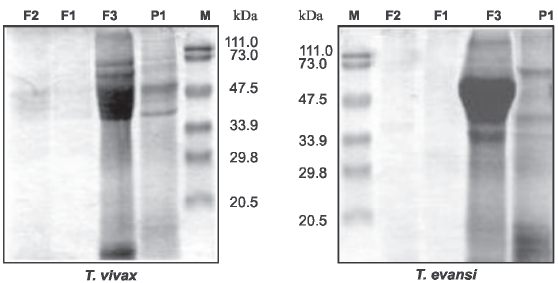
El patrón electroforético mostrado por ambos parásitos en las diferentes fracciones permitió visualizar una mayor concentración proteica en las muestras correspondientes a las proteínas del citoplasma (F3). Para T. vivax el patrón proteico detectado en la F3, reveló la presencia de bandas polipeptídicas entre los 33,9 kDa y 73 kDa de masa molecular, con dos bandas bien diferenciadas cercanas a los 47,5 kDa. El patrón detectado en las fracciones de T. evansi fue similar al observado en las fracciones de proteínas obtenidas para T. vivax. Sin embargo, fue posible detectar diferencias notables entre las fracciones. Los polipéptidos mayoritarios de la F3 de T. evansi se concentraron entre 34,0 kDa y 70,0 kDa con una banda bien diferenciada sobre los 33,9 kDa no detectada para T. vivax. Las proteínas insolubles en Tritón X-114 bajo las condiciones utilizadas (P1) presentan bandas de proteínas que pudieran ser consideradas similares a las presentes en F1 y F2 en ambas especies. Detalles sobre los patrones proteicos revelados en las dos especies de Trypanosoma (Fig.1).
Fig. 1. Fraccionamiento proteico en formas sanguicolas de Trypanosoma vivax y T. evansi. F2, F1, F3 y P1, corresponden a las fracciones obtenidas de cada parásito. Los carriles contienen 10% de las proteínas presentes en cada fracción. M: marcador de peso molecular.
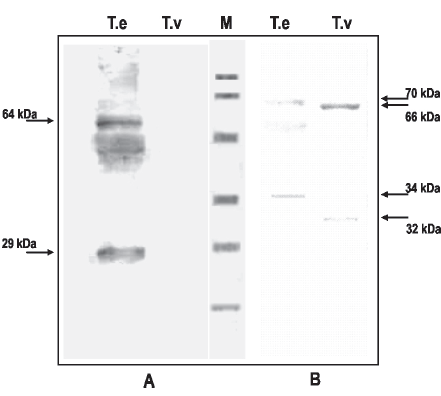
Caracterización inmunológica de proteínas citoplasmáticas de T. vivax y T. evansi. Las proteínas de la fracción F3 de T. vivax y de T. evansi fueron confrontadas con sueros de referencia de equino y bovino positivos a T. evansi (Fig. 2A) y T. vivax (Fig. 2B), respectivamente. Este ensayo permitió visualizar proteínas de T. evansi en presencia del suero de bovino positivo a T. vivax (Fig. 2B), representado por dos bandas de 34 kDa y 70 kDa y proteínas de T. evansi en presencia de suero de equino positivo a T. evansi (Fig. 2A), mostrando bandas de 29 kDa y 64 kDa. Asimismo, como era de esperarse, se revelaron proteínas de T. vivax en presencia del suero de bovino positivo a T. vivax (Fig. 2B), mostrando 2 bandas de 32 kDa y 66 kDa. Sin embargo, no se observó respuesta inmunogénica sobre las proteínas F3 de T. vivax cuando estas fueron confrontadas con suero de equino positivo a T. evansi (Fig. 2A). En las proteínas insolubles en Tritón X-114 (P1-T. vivax) se visualizó un polipéptido de 54 kDa al incubar las membranas con el suero de bovino positivo a T. vivax [no mostrado, ver Bolívar et al. (2006)].
Fig. 2. WESTERN BLOT para la detección de proteínas inmunogénicas en fracciones citosólicas (F3) de T. vivax (T.v) y T. evansi (T.e). A: fracciones reveladas con suero de equino positivo a T. evansi; B: fracciones reveladas con suero de bovino positivo a T. vivax. M: marcador de peso molecular.
Con el fin de determinar la especificidad de la reacción, fracciones proteicas de T. vivax fueron enfrentadas con sueros de bovinos positivos a Anaplasma marginale, Mycobacterium bovis, Mycobacterium paratuberculosis y suero de equino positivo a T. evansi no evidenciándose respuesta antigénica alguna.
DISCUSIÓN
En el presente estudio se registra el desarrollo y ejecución de una metodología basada en el fraccionamiento de las formas sanguícolas de T. vivax y T. evansi, la cual permitió la obtención de proteínas inmunogénicas capaces de diferenciar los dos parásitos y reconocer su capacidad antigénica. Este hallazgo preliminar pudiera ser de utilidad en la consecución de un método de valor diagnóstico potencial en áreas donde ambas parasitosis circulen considerando la especificidad detectada para cada caso.
En este respecto bandas antigénicas fueron reveladas en la fracción citoplasmática (F3) de T. vivax, evidenciándose polipéptidos de 32 kDa y 66 kDa cuando se desarrolló un Western blot en presencia de suero anti-T. vivax de bovino, las cuales no fueron detectadas cuando se utilizó el suero anti-T. evansi obtenido de un equino naturalmente infectado, indicando la gran especificidad de estas fracciones proteicas, las cuales sólo son reconocidas por anticuerpos generados por T. vivax.
Por otra parte, el Western blot de la fracción citoplasmática (F3) de T. evansi permitió la detección de dos polipéptidos de 29 kDa y 64 kDa cuando fue revelado con suero de equino anti-T. evansi, además de bandas antigénicas de 34 kDa y 70 kDa cuando se reveló con suero bovino anti-T. vivax, indicando la similitud antigénica entre ambas especies de Trypanosoma. Esto último pareciera justificar el empleo de antígeno de T. evansi en la detección de infecciones por T. vivax en bovinos, práctica diagnóstica que recientemente se ha venido llevando a cabo en Venezuela (Uzcanga et al., 2004).
En conclusión, las diferencias observadas entre ambos parásitos en estas experiencias preliminares, parecieran dar al sistema desarrollado un valor diagnóstico potencial. El hecho de que se pueda discernir específicamente entre infecciones naturales por T. evansi en suero de equinos e infecciones por T. vivax en suero de bovinos, utilizando fracciones proteicas T. evansi y T. vivax respectivamente, permite considerar estas proteínas como un marcador bioquímico candidato para identificar y diferenciar infecciones por ambos parásitos en áreas donde las dos tripanosomiasis son endémicas. Establecidas las condiciones para el desarrollo de este sistema a nivel experimental, sólo quedaría probarlo en muestras poblacionales para valorar su bondad diagnóstica en masa y concluir sobre su practicidad y costo relativo.
AGRADECIMIENTOS
A la Dra. T. Perrone por su asesoramiento y colaboración en la realización de este trabajo. Trabajo financiado por CDCHT-ULA, Proyecto C-1210-03-03-EM (NA).
REFERENCIAS
1. Añez-Rojas N., García-Lugo P., Crisante G., Rojas A. & Añez N. (2006). Isolation, purification and characterization of GPI-anchored membrane proteins from Trypanosoma rangeli and Trypanosoma cruzi. Acta Tropica. 97: 140-145. [ Links ]
2. Bolívar A. M., García-Lugo P., Crisante G., Rojas A., Teixeira M. M. G. & Añez N. (2006). Detección de infecciones subclínicas por Trypanosoma vivax en bovinos de fincas ganaderas de Mérida, Venezuela. Bol Mal San Amb. 46: 87-90. [ Links ]
3. Espinoza E., González N., Primera G., Rivero E., Hidalgo L. & González B. (2002). Efectos del Trypanosoma evansi en cabras (Capra hircus) infectadas experimentalmente. Rev. Cientf. FCVLUZ. 12: 103-107. [ Links ]
4. García H. & Mendoza-León A. (2000). Diagnóstico molecular en protozoarios Kinetoplastida. Principios y aplicaciones. Rev. Fac. Cs. Vets. UCV. 41: 109-130. [ Links ]
5. García F., Rivera M., Ortega M. & Suárez C. (2000). Trypanosomiasis equina causada por Trypanosoma evansi en tres hatos ganaderos del Estado Apure, Venezuela. Rev. Fac. Cs. Vets. UCV. 41: 91-100. [ Links ]
6. González L., García J., Núñez C., Perrone T., González-Baradat B., Gonzatti M. et al. (2005). Trypanosoma vivax: A novel method for purification from experimentally infected sheep blood. Experimental Parasitology. 111: 126-129. [ Links ]
7. Ko Y. & Thompson G. (1995). Purification of glycosyl-Phosphatidyl-Inositol-Anchored proteins by modified Triton X-114 partitioning and preparative gel electrophoresis. Annals Biochem. 224: 166-172. [ Links ]
8. Laemmli U. K. (1970). Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227: 680-685. [ Links ]
9. Lanham S. & Godfrey D. (1970). Isolation of salivarian trypanosomes from man and other mammals using DEAE-cellulose. Experimental Parasitology. 28: 521-534. [ Links ]
10. Luckins A. (1988). Trypanosoma evansi in Asia. Parasitol. Today. 4: 137-142. [ Links ]
11. Rivera M. (1996). Tripanosomiasis En: Hemoparasitosis bovinas. UCV. Consejo de Desarrollo Científico y Humanístico. Venezuela. [ Links ] 13. Sandoval E., Espinoza E. & Valle A. (1996). Leucopenia y trombocitopenia en ovejas infectadas experimentalmente con Trypanosoma vivax. Veterinaria Trop. 21: 13-33.
14. Tamasaukas R. & Roa N. (1996). Tripanosomiasis bovina (T. vivax): una revisión. Fondo Nacional de Investigaciones Agropecuarias. Maracay, Venezuela.
15. Tamasaukas R., Aguirre A., Ron J., Roa N. & Cobo M. (2000). Tetralogía hemoparasitaria en algunas fincas bovinas del Municipio Santa Rita, Estado Guárico, Venezuela. Rev. Fac. Cs. Vets. UCV. 41: 101-108.
16. Toro M., León E., López R., García J. & Ruiz A. (1980). Resultados de un muestreo sobre tripanosomiasis bovina mediante técnicas serológicas. Veterinaria Trop. 5: 43-50.
17. Uzcanga G., Mendoza M., Aso P. & Bubis J. (2002). Purification of a 64Kda antigen from Trypanosoma evansi that exhibts cross-reactivity with Trypanosoma vivax. Parasitology. 124: 287- 299.
18. Uzcanga G., Perrone T., Noda J., Pérez-Pazos J., Medina R., Hoebake J. & Bubis J. (2004). Variant surface glycoprotein from Trypanosoma evansi is partially responsible for the cross-reaction between Trypanosoma evansi and Trypanosoma vivax. Biochemistry. 43: 595-606.
19. Winston E., Fuller S. & Hurrell J. (1987). Western blotting. Chap 10.8. En: Current protocols in molecular biology. (Eds. Ausubel F., Brent R., Kingston R., Moore D., Seideman J., Smith J., Struhl K.) (g.P.A & Wiley-Interscience, NYUSA.).












 uBio
uBio 
